Discussió
L'objectiu del nostre treball és identificar les selenoproteïnes DI, SelJ i SelQ en el genoma de 14 protistes. El protocol que hem seguit el podeu trobar aquí.| SelQ | SelJ | DI | |||||||
|---|---|---|---|---|---|---|---|---|---|
| tBlastn | T_coffee | SeP? | tBlastn | T_coffee | SeP? | tBlastn | T_coffee | SeP? | |
| A. laibachii | NO | NO | NO | ||||||
| A. rara | NO | NO | NO | ||||||
| C. fasciculata | NO | NO | NO | ||||||
| D. discoideum | NO | NO | SeCys | ||||||
| D. fasciculatum | NO | NO | SeCys | ||||||
| F. cylindrus | NO | NO | NO | ||||||
| G. niphandrodes | NO | NO | NO | ||||||
| I. multifiliis | NO | NO | NO | ||||||
| L. donovani | NO | NO | NO | ||||||
| L. tarentolae | NO | NO | NO | ||||||
| P. capsici | NO | Cys | NO | ||||||
| P. polycephalum | NO | NO | SeCys | ||||||
| S. arctica | NO | NO | NO | ||||||
| T. congolense | NO | NO | NO | ||||||
DI
Tal i com hem vist a l'apartat de resultats i a la taula superior, només hem trobat hits significatius a tres organismes: D.discoideum_AX4, D.fasciculatum i P.polycephalum.D.discoideum_AX4
Hem realitzat un tblastn utilitzant com a query la DI1 humana, obtenint un hit amb un e-value de 9e-18. Per tant, donat que és un hit bo, continuarem amb el procés d'anotació. Mitjançant els programes fastasubseq i exonerate hem trobat el codó TGA, el qual ha estat assignat a l'aminoàcid unknown (X) i probablement es tracta d'una selenocisteïna. El T-coffee realitzat alinea l'aminoàcid X amb la selenocisteïna (U) de la DI1 humana i observem una alta conservació als aminoàcids de darrera. En canvi, el genewise identifica la nostra selenocisteïna amb un codó STOP i no la inclou a l'exó. Això pot ser normal, ja que ambdós programes utilitzen algorismes de cerca diferents.
Per a completar l'estudi, hem comparat la nostra selenoproteïna amb la base de dades de NCBI i hem vist que ja es trobava anotada i amb una seqüència més llarga que la predita. Hem intentat obtenir la nostra proteïna d'una forma mès llarga, però la part que ens falta i es troba a la proteïna de NCBI és molt diferent a la DI1 humana, i el T-coffee no la prediu adequadament. Aquesta nova query de NCBI la hem emprat també per a buscar homòlegs en els nostres organismes, ja que és un organisme molt proper.
Realitzant la mateixa anàlisi amb les queries DI2 i DI3 humanes, hem obtingut també uns E-values bons: 5e-19 i 9e-20, respectivament. Hem continuat el procés i també hem predit selenoproteïnes. Com hem observat que les selenoproteïnes DI1, DI2 i DI3 humanes s'alineen a la mateixa regió dels protistes i tenen E-values semblants, creiem que a D.discoideum_AX4 només hi ha una DI i en els humans s'han donat diversos events de duplicació.
Finalment, aquesta es la nostra proteïna predita amb la query més propera. La predicció del exonerate mostra que només conté 1 exó, i buscant a la regió genòmica on trobem el hit hem trobat SECIS amb el SECISearch.
Alineaments múltiples
Per tal d'analitzar les modificacions que DI ha sofert al llarg de l'evolució, hem realitzat varis alineaments múltiples:
Per comparar les tres DI de Homo sapiens:
Amb l'alineament podem veure que les tres formes que trobem a humans són diferents, tot i que també trobem regions molt conservades. És a dir, són diferents membres de la família DI.
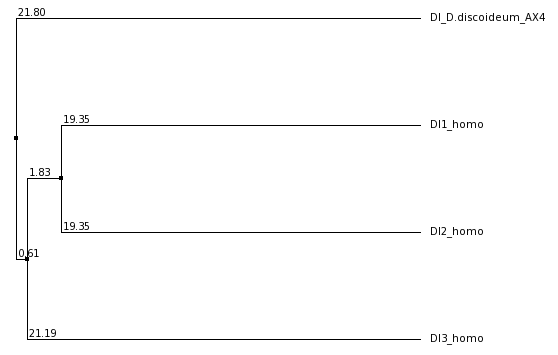
Per comparar les tres DI de D.discoideum:
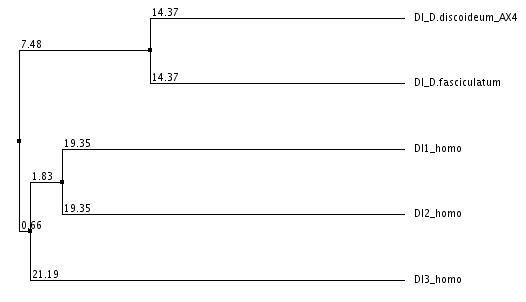
D'altra banda observem que les tres formes predites a D.discoideum_AX4 són idèntiques (tot i que la DI3 és una mica més llarga). Amb el tblastn hem vist que aquestes prediccions fan referència a la mateixa regió del genoma. Tot això ens porta a pensar que les tres formes que trobem a humans provenen d'aquesta única proteïna i que ha estat en el procés d'especiació quan s'ha produit una triplicació, adquirint així les diferents formes. Per plasmar-ho, realitzem un alineament múltiple amb les tres formes humanes (DI1, DI2 i DI3) i la de D.discoideum_AX4. Observem que la DI del protista és més diferent a les DI humanes amb regions no conservades o gaps, però tenen una llargaria similar i si que hi ha molta conservació al darrere de la selenocisteïna.
Una funció del Jalview (programa amb el que visualitzem l'alineament múltiple) permet fer un arbre de les seqüències en funció del % d'identitat. Al fer l'arbre veiem com els 3 membres de DI d'homo s'assemblen més entre sí que la DI de D.discoideum_AX4, i més la DI1 i la DI2 entre sí.
D.fasciculatum
En realitzar un tblastn amb la query DI1 humana el millor hit obtingut té un E-value de 1e-4, el que no és molt bo. Ni exonerate ni genewise han pogut resoldre l'estructura exònica de la regió. En el cas de la DI2 humana, el millor hit trobat també és fluix, ja que té un E-value de 1e-4. Genewise no pot predir aquesta estructura. En canvi, amb exonerate aconseguim alinear la U amb un aminoàcid unknown i, a més, a continuació es troba una regió conservada en el T-coffee: sembla ser una selenoproteïna, a més a més al buscar al fer blastp al NCBI hem trobat homòlegs DI en altres organismes. En el cas de la DI3 humana, el tblastn no ha trobat cap hit.Per tal d'aconseguir millors hits hem comparat amb aquest protista un organisme més proper a ell evolutivament parlant: D.discoideum_AX4. Amb el tblastn hem trobat un hit molt bo (4e-50) que es prediu correctament, tant amb genewise com amb exonerate, i amb T-coffee s'alinea l'aminoàcid Unknown amb la Selenocisteïna. A més a més mostra conservació darrera d'aquesta posició.
Les formes predites per les diferents queries a D.fasciculatum són idèntiques ja que corresponen a la mateixa regió genòmica, al igual que succeix amb el DI de D.discoideum_AX4.
Aquesta és la proteïna predita. La predicció del exonerate mostra que està formada per 3 exons, i buscant a la regió genòmica on trobem el hit hem trobat SECIS amb el SECISearch. Si comparem la DI de D.fasciculatum amb les queries humanes veiem que existeixen molts diferències ja que són espècies molt distants:
D'altre banda, si comparem la DI de D.fasciculatum amb la DI de D.discoideum_AX4, veiem que són molt més similars, tot i que la DI de D.fasciculatum predita és una mica més curta.
P.polycephalum
Hem realitzat un tblastn amb la query DI1 humana i hem obtingut un hit fluix amb un E-value de 0,003. Tot i tenir aquest E-value tan alt, amb l'exonerate hem obtingut un bon T-coffee que alinea l'aminoàcid unknown amb la U i presenta bona conservació, a la búsqueda de blastp a NCBI hem trobat homòlegs DI en altres organismes. Amb el genewise no hem obtingut cap resultat.En el cas de DI2, també hem trobat un hit fluix amb un E-value de 0,001, hem obtingut resultats amb exonerate i alineament amb T-coffee. Amb DI3 no hem trobat cap hit bo.
Al comparar la seqüència trobada en aquest protista amb la base de dades de NCBI, hem trobat un hit bo amb una DI de D.rerio, amb un E-value de e-11. Si utilitzem aquesta seq&umml;ència com a query, aconseguim un hit millor, amb un E-value de 3e-05. Amb exonerate hem predit una estructura que inclou l'aminoàcid unknown i l'alinea amb la selenocisteïna al T-coffee. Aquesta és la proteïna predita. La predicció del exonerate mostra que està formada per 2 exons, i buscant a la regió genòmica on trobem el hit hem trobat SECIS amb el SECISearch.
Les formes predites per les diferents queries a P.polycephalum són idèntiques ja que corresponen a la mateixa regió genòmica. En aquest organisme, només hem trobat una DI com en els protistes anteriors. D'altra banda la nostra proteïna predita és molt més curta que les queries, tot i que hem intentat allargar-la amb exonerate o amb altres queries sense èxit.
Aquests resultats coincideixen amb els obtinguts en l'anàlisi de la presència de la maquinària de síntesis de selenoproteïnes, ja que els tres organismes esmentats presenten SecS, eEFSec, pstk, sbp2, secp43 i sps2. Tot i això, a cap dels tres organismes hem trobat tRNAsec. Més endavant comentarem aquest punt.
SelJ
Tal i com veiem als resultats i a la taula superior només considerem els hits amb un E-value inferior a 10e-4. Tot i així són resultats dubtosos per diversos motius: en el cas de C.fasciculata, L.donovani, L.tarentolae i T.congolense obtenim hits acceptables però que no inclouen la SeCys. No obstant, decidim continuar amb l'anàlisi per veure si realment es tracta d'una selenoproteïna o no. En el cas de A.laibachii i P. capsici hem obtingut hits molt bons en els quals la SeCys s'alinea amb una Gly o una Cys (en un dels hits de P. capsici).
A.laibachii_Nc14
Inicialment vam fer un tblastn amb la SelJ de Tetraodon nigroviridis de l'arxiu TARBALL i vam obtenir tres hits significatius (e-values de 1e-15, 4e-12, 4e-12), en tots tres casos la SeCys s'alinea amb una Gly. Tot i així vam continuar l'anotació.Hit 1
Tant l'exonerate com el genewise prediuen un gen, amb un exó que inclou la SeCys substituïda per una Gly. No obstant, el t_coffee alinea la SeCys amb un gap, indicant que el gen que hem predit no es tracta d'una selenoproteïna. Com es va comentar a la introducció, i es mostra en aquest t_coffee, SelJ pertany a una família de ADP-ribosiladors, de la qual també forma part CrystallinJ1, amb la que comparteix el domini final. Per tant, possiblement el que estem predint és una proteïna d'aquesta falmília però que no és SelJ. Per a confirmar aquest resultat vam fer un blastp contra la base de dades non-reduntdant de NCBI, en la qual veiem que la proteïna predita s'assembla molt tant a SelJ d'altres organismes com a CrystallinJ1, per tant, no podem concloure amb aquesta informació de quina proteïna es tracta.Hit 2
En aquest cas l'exonerate prediu una proteïna que no inclou la SeCys i el genewise una proteïna, que igual que el tblastn, alinea la SeCys amb una Gly. Al realitzar el t_coffee ens trobem en una situació pràcticament igual a la del hit 1. El blastp contra la base de dades non-redundant del NCBI no troba cap coincidència, per tant, descartem que es tracti d'una SelJ.Hit 3
L'exonerate amb el hit 3 prediu una proteïna que no inclou la SeCys, mentre que la proteïna predita pel genewise presenta una Gly enlloc de la SeCys. El t_coffee també alinea la SeCys amb un gap i el blasp contra la base de dades non-redundant del NCBI dóna coincidències tant amb SelJ com amb CrystallinJ1 d'altres organismes, arribant a la mateixa conclusió que en el cas del hit 1.C.fasciculata
Vam començar fent un tblastn amb la SelJ de Tetraodon nigroviridis de l'arxiu TARBALL i vam obtenir un hit curt que engloba l'última part de la nostra selenoproteïna però que t&ecute; un e-value acceptable (1e-4). Tot i que no inclou la SeCys i ens fa pensar que no correspondrà a un homòleg de SelJ, decidim continuar degut al e-value acceptable. Ni la proteí predita per l'exonerate ni la predita pel genewise incluouen la SeCys. El t_coffee de l'exonerate, que és millor que el de genewise, alinea molt bé la última part de la proteïna, però no inclou la selenocisteïna ni cap fragment d'abans d'aquesta. Per aquest motiu vam decidir ampliar manualment la subseqüència a partir de la qual vam executar l'exonerate, però els resutlats del t_coffee seguien alineant la SeCys amb un gap. Aquests resultats indiquen que probablement el que estem predint sigui una proteïna amb un domini de la família dels ADP-ribosiladors a la que pertany tant la SelJ com CrystallinJ1. Els resultats del NCBI suggereixen que es tracta d'una proteïna hipotètica d'aquesta família, però descarta que es tracti de SelJ o de CrystallinJ1.Per descartar amb més seguretat que la proteïna que estem predint no és una CrystallinJ1 vam realitzar el mateix procés d'anotació amb la query de CrystallinJ1 de L.loa, però com podem veure al t_coffee la proteïna predita només compartia l'últim fragment amb la CrystallinJ1, igual que succeïa amb SelJ.
L.donovani
El tblastn amb la SelJ de Tetraodon nigroviridis de l'arxiu TARBALL i va donar un hit de e-value acceptable (9e-5) que no inclou la SeCys. Aquest resultat ens suggereix que no es tracta d'una SelJ ni d'un homòleg en cisteïna, tot i així, continuem amb l'anotació degut al e-value que s'obté. Tant l'exonerate com el genwise prediuen un gen amb un exó que no inclou la SeCys i el t_coffee només alinea el fragment final. Per estar segurs que realment la proteïna no té SeCys vam fer un t_coffee amb l'exó ampliat alinea un fragment més gran però deixa un gap en la posició de la SeCys. Els resultats del NCBI són als obtinguts amb C.fasciculata. Per tant, estem davant de la mateixa situació en la que predim un domini d'una proteïna hipotètica de la familía dels ADP-ribosiladors.Vam aprofundir més per comprobar que no es tracta d'una CrystallinJ1 pel que vam realitzar el mateix procés d'anotació amb la query de CrystallinJ1 de L.loa, però com podem veure al t_coffee la proteïna predita només compartia l'últim fragment amb la CrystallinJ1, igual que succeïa amb SelJ.
L.tarentolae
Vam començar fent un tblastn amb la SelJ de Tetraodon nigroviridis de l'arxiu TARBALL i vam obtenir un hit curt que engloba l'última part de la nostra selenoproteïna, però que t&ecute; un e-value acceptable (6e-5). Tot i que no inclou la SeCys i ens fa pensar que no correspondra a un homòleg de SelJ decidim continuar degut al e-value obtingut. L'exonerate prediu un gen amb dos exons, el primer dels quals substitueix la SeCys de SelJ per una Phe. El genewise, en canvi, prediu un gen que no inclou la SeCys. Igual que amb els organismes anteriors, el t_coffee alinea únicament l'últim fragment de la proteïna predita amb SelJ, fet que indica que estem predint un domini de la familía dels ADP-ribosiladors. Els resultats del NCBI suggereixen que es tracta d'una proteïna hipotètica d'aquesta família, però descarta que es tracti de SelJ o de CrystallinJ1.Per descartar amb seguretat que tampoc correspon a CrystallinJ1 hem realitzat el mateix procés d'anotació utilitzant com a query CrystallinJ1 de L.loa. El resutlat del t_coffee obtingut, de la mateixa manera que passava amb SelJ, alinea únicament l'últim fragment.
P.capsici
Inicialment vam fer un tblastn amb la SelJ de Tetraodon nigroviridis de l'arxiu TARBALL i vam obtenir dos hits significatius (e-values de 1e-15 i 5e-11). En el primer hit la SeCys s'alinea amb una Gly, mentre que en el segon la SeCys s'alinea amb una Cys, el que indica que podriem estar davant d'una possible SelJ. Malgrat l'alineament del primer hit amb una Gly continuem l'anotació amb els dos hits.Hit 1
Per aquest hit, tant l'exonerate com el genewise prediuen un gen amb un sol exó que inclou la SeCys substituïda per una Gly. El t_coffee de l'exonerate alinea la SeCys amb un gap, mentre que el t_coffee del genewise ho fa amb una Gly, coincidint amb el resultat del tblastn. En qualsevol dels casos, tant si s'alinea amb un gap com amb una Gly, podem considerar que la proteïna predita no es tracta de SelJ. Aquest resultat, no coincideix amb l'obtingut amb el blastp contra la base de dades non-redundant del NCBI, el qual troba similituds amb SelJ d'altres organismes amb un e-value de 4e-15, que és inferior al e-value dels hits de CrystallinJ1.Hit 2
En aquest cas, l'exonerate prediu un gen amb un únic exó que conté la SeCys substituïda per una Cys. El gen predit per genewise té les mateixes característiques. El resultat del t_coffee ens alinea la SeCys amb una Cys, concordant amb els resultats del tblastn, suggerint que la proteïna predita es tracta d'un homòleg en Cys de SelJ. Al realitzar un blastp contra la base de dades non-redundant del NCBI trobem coincidencies amb SelJ d'altres organismes amb e-values raonablement acceptables (2e-5), proporcionant una evidència més a la hipòtesi de que s'ha predit un possible homòleg de SelJ.T.congolense
Hem realitzat un tblastn utilitzant com a query la SelJ de Tetraodon nigroviridis de l'arxiu TARBALL i vam obtenir un hit curt que només engloba l'última part de la nostra selenoproteïna, però que té un e-value acceptable (8e-5), per aquest motiu continuem amb l'anotació. L'exonerate prediu un gen amb un exó que no inclou la SeCys, ja que només alinea la part final de la nostra query. El mateix passa amb genewise. El t_coffee alinea molt bé l'última part de la proteïna amb la query, però segueix sense incloure la SeCys. Al fer un blastp contra la base de dades non-redundant del NCBI obtenim un e-value de 0,001 per CrystallinJ1 i un e-value de 0,009 per SelJ. Per tant, aquests resutlats indiquen que la proteïna predita és una proteïna hipotètica que comparteix el domini d'ADP-ribosiladors amb SelJ i CrystallinJ1.Per estar-ne més segurs de que no hem predit una CrystallinJ1 hem realitzat el procés d'anotació amb la query CrystallinJ1 de L.loa. El resutlat del t_coffee alinea el fragment final de la proteïna predita amb la query, per tant també podem descartar que es tracti d'una CrystallinJ1. Un cop més hem predit el domini d'una proteïna hipotètica de la família dels ADP-ribosiladors.
SelQ
Inicialment vam fer un tblastn amb la SelQ de T.gondii de l'arxiu TARBALL, i no vam trobar cap hit significatiu (e-value < 1e-4). Vam buscar homòlegs en altres organismes mitjançant NCBI BLAST com ja hem comentat a l'apartat de materials i mètodes, per tal de contrastar les nostres observacions. Amb NCBI BLAST, vam identificar únicament una possible query SelQ a l'organisme N.caninum. Aquesta seqüència s'alineava bé amb la SelQ de T.gondii, alineant l'aminoàcid unknown amb la selenocisteïna. Tot i que, com la selenocisteïna de SelQ es troba a la penúltima posició seguida d'una cisteïna, no podem veure si existeix conservació darrera d'aquest residu, de forma que no estem 100% segurs de que veritablement sigui una selenocisteïna. Vam extreure la seqüència proteica i vam fer tblastn contra els 14 genomes. Els resultats obtinguts segueixen sent negatius.
SelQ sembla ser una família de selenoproteïnes molt restringida a T.gondii i espècies estretament relacionades, ja que hem trobat una hipotètica SelQ a N.caninum, considerant que la cerca a NCBI BLAST ha donat només un homòleg en N.caninum i que en les anotacions de genomes de protistes no s'ha observat densa de T.gondii. Consulteu els resultats a: 2010, 2011.
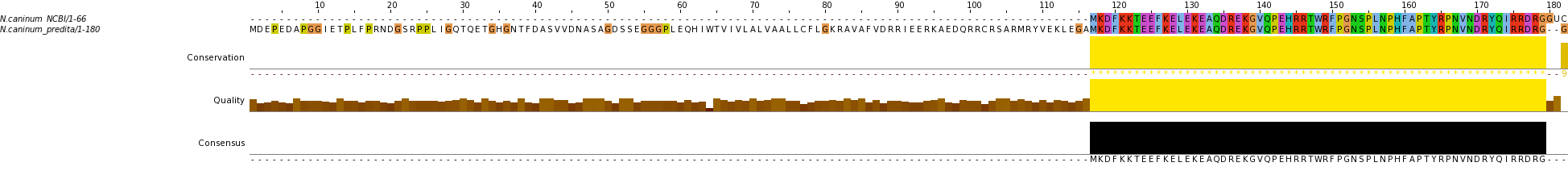
Hem buscat a NCBI la SelQ trobada a N.caninum_Liverpool amb blastp. Hem trobat una conserved hypothetical protein a N.caninum_Liverpool de 182aa. Aquesta proteïna creiem que és la que nosaltres hem predit ja que tenim una regió idèntica bastant llarga de 62aa. Amb la nostra metodologia no hem pogut predir un bon tros de l'inici de la proteïna peró d'altra banda la proteïna trobada a NCBI no conté la selenocisteïna i s'acaba justament en aquesta. Creiem que podria tractar-se d'un error d'anotació al NCBI, pensant que es tracta d'un codó STOP, de forma que faltarien afegir a l'anotació els dos aminoàcids finals UC.
Maquinària de síntesi
Per tal de corroborar si els organismes estudiats tenen o no selenoproteïnes hem cercat si presenten la maquinària de síntesi de selenoproteïnes. S'han cercat les següents proteïnes amb diferents queries: SecS, eEFSec, pstk, sbp2, secp43, sps2 i tRNASec. Podeu veure els resultats detallats aquí.| sps2 | sbp2 | pstk | secp43 | secS | eEFSec | tRNAsec | |
|---|---|---|---|---|---|---|---|
| A.laibachii_Nc14 | |||||||
| A.rara | |||||||
| C.fasciculata | |||||||
| D.discoideum_AX4 | |||||||
| D.fasciculatum | |||||||
| F.cylindrus | |||||||
| G.niphandrodes | |||||||
| I.multifiliis_strain_G5 | |||||||
| L.donovani_BPK282A1 | |||||||
| L.tarentolae | |||||||
| P.capsici | |||||||
| P.polycephalum | |||||||
| S.arctica | |||||||
| T.congolense |
A l'anàlisi control, tenim E.histolytica i T.parva com a controls negatius, ja que no presenten selenoproteïnes. D'altra banda nosaltres hem trobat en aquests organismes eEFSec i secp43 de forma que és possible que aquestes proteïnes estiguin implicades en altres processos moleculars diferents a la sintesi de selenoproteïnes. Els altres organismes control que sí que tenen selenoproteïnes i són relativament propers a: P.tetraurelia (I.multifiliis_strain_G5), D.purpureum (D.discoideum_AX4, D.fasciculatum, P.polycephalum), L.mexicana (L.donovani_BPK282A1, L.tarentolae), M.brevicollis (S.arctica), P.sojae (P.capsici, A.laibachii_Nc14, F.cylindrus), T.cruzi (C.fasciculata, T.congolense). En aquests organismes control positiu esperariem trobar tota la maquinària, però amb les nostres queries hem obtingut resultats negatius a P.tetraurelia, M.brevicollis i T.cruzi, per a pstk i sbp2. Això pot ser un artefacte ja que hem agafat un número limitat de queries diferents, i admetrem certa variabilitat al analitzar els nostres resultats.
Anteriorment, hem predit la selenoproteïna DI als organismes D.discoideum_AX4, D.fasciculatum i P.polycephalum. Els resultats obtinguts a la cerca de la maquinària coincideixen amb els esperats, ja que hem trobat totes les proteïnes de la maquinària de síntesi a D.discoideum_AX4, a D.fasciculatum (no trobem pstk però tampoc el trobem en algunes queries control positiu), i a P.polycephalum (no trobem sbp2 però tampoc el trobem en algunes queries control positiu). Ambdues espècies (D.discoideum_AX4 i D.fasciculatum) provenen del mateix ancestre i mostren resultats similars; a més l'organisme control D.purpureum, que també comparteix ancestre comú i té selenoproteïnes, presenta un patró similar. Per tant, el més probable és que l'ancestre comú també presenti selenoproteïnes. En el cas de P.polycephalum, hem trobat tota la maquinària excepte sbp2. Tal i com hem esmentat anteriorment, amb les diferents queries de sbp2 que hem emprat a vegades no el trobem en organismes que es que coneix que tenen selenoproteïnes, per tant no és un resultat fiable. D'altra banda, en tots els organismes no hem pogut predir amb seguretat el tRNAsec, com comentarem més endavant.
Altres organismes on hem trobat tota la maquinaria i segurament selenoproteïnes serien: F.cylindrus, L.tarentolae i S.arctica. D'altra banda, en el cas de P.capsici, C.fasiculata i T.congolense, hem trobat tota la maquinària excepte sbp2, igual que ens pasava amb P.polycephalum. En el cas de I.multifiliis_strain_G5 no hem pogut predir ni sbp2 ni pstk, les dues proteïnes conflictives que no trobavem en alguns controls positius. Considerem que tots aquests organismes realment tenen la maquinària i molt probablement selenoproteïnes.
D'altra banda, no hem trobat cap proteïna de la maquinària a G.niphandrodes i A.rara. Els únics membres que trobem a A.laibachii_Nc14 són secp43 i eEFSec, els quals també els hem trobat als controls negatius. A priori, tenint en compte que potser necesitariem més queries per estar segurs, pensem que aquests tres organismes no disposen de la maquinària de traducció i no esperariem trobar selenoproteïnes a no ser que aquestes es sintetitzin per altres vies.
tRNASeC
La cerca dels tRNAs s'ha basat en el programa tRNAscan-SE 1.21, un programa que escaneja el genomes i prediu els tRNAs. L'output d'aquest programa proporciona informació de tots els tRNAs presents en el genoma d'un organisme i entre altres coses, ens monstra el seu anticodó. Sabent que el codó de la selenocisteïna és TGA, buscarem el seu anticodó TCA en cadascun dels fitxers que ens ha proporcionat el programa. La presència d'aquest anticodó indica l'existència del tRNA per a selenocisteïna (SeC).
Ara bé, els nostres resultats discrepen molt dels resultats esperats. En primer lloc, no s'ha predit tRNA-Sec en els organismes control que se sap que tenen selenoproteïnes, i teòricament és necesari el tRNA-Sec per a l'existència de les selenoproteïnes. D'altra banda ens ha predit SeCys en organismes on no hem trobat maquinària de síntesi (A.laibachii_Nc14 i G.niphandrodes), i el programa ha fallat en escanejar el genoma de P.polycephalum de forma que no tenim resultat d'aquest. És segur que aquests resultats no són representatius, potser són deguts a causa d'un error del programa tRNAscan-SE, un problema al treballar amb protistes o que no sapiguem com configurar-ho correctament, en qualsevol cas, aquestes dades no són representatives i per tant, no les utilitzem per a treure conclusions.
Selenoprofiles
Per a verificar els nostres resultats hem baixat el programa Selenoprofiles del CRG. En aquest enllaç podeu trobar els resultats de forma detallada (mireu la carpeta output dins cada organisme).
Selenoprofiles no ha trobat SelQ al genoma dels protistes. De SelJ ha trobat un homòleg en cisteïna en P.capsici. A DI ha trobat la selenoproteïna als genomes de D.discoideum_AX4, D.fasciculatum i P.polycephalum, com nosaltres, i també ha predit homòlegs en cisteïna de DI a A.laibachii_Nc14, C.fasciculata, F.cylindrus, L.donovani_BPK282A1, L.tarentolae, P.capsici i S.arctica, a més a més, ha predit homòlegs en serina a P.polycephalum i F.cylindrus.
A la taula anterior trobem els hits indentificats per Selenoprofiles amb un link als arxius generats. Els colors representen: ![]() homòleg en cisteïna,
homòleg en cisteïna, ![]() homòleg en serina,
homòleg en serina, ![]() homòleg en selenocisteïna.
homòleg en selenocisteïna.
Tots aquests resultats concorden amb els que hem obtingut nosaltres, tot i que nosaltres no hem trobat els homòlegs en serina i cisteïna de DI, ja que els tblastn ens donaven hits molt dolents. Com Selenoprofiles empra altres estrategies, segurament la seva cerca és molt més acurada. De totes formes, hem volgut assegurar-nos i hem comprovat els hits de Selenoprofiles amb la nostra metodologia inicial; podeu trobar els resultats aquí. En aquesta comprovació no hem pogut trobar els homòlegs de cisteïna ni els de serina, ja que ni exonerate ni genewise ens predien l'estructura exònica. D'altra banda, la selenoproteïna DI que ha identificat selenoprofiles és diferent a la nostra, es troba a una altra regió. Al verificar aquesta regió amb la nostra metodologia, hem trobat que efectivament hi ha una selenocisteïna i es tracta d'una selenoproteïna amb 3 exons. Aquí podeu trobar els resultats del .gff i del T-coffee.