Introducció
El paper del seleni a la natura
El seleni és un element poc abundant a la natura, però és un nutrient essencial pels animals, microorganismes i altres eucariotes. El seu dèficit pot donar lloc a malalties, com ara la malatia de Keshan, molt prevalent a una regió de la Xina on hi ha nivells de seleni molt baixos. En aquests casos, s'ha demostrat que suplementant la dieta d'aquestes regions amb seleni la prevalença de la malatia es redueix, tanmateix, un excés de seleni por ser tòxic. A més, estudis recents apunten que el seleni podria prevenir el càncer de pròstata, tot i que encara no hi ha resultats concloents.
El seleni es distribueix en els tres dominis de la vida: Archea, Eubacteria i Eucariota. En els organismes, el seleni es troba a les cèl·lules en forma de selenoproteïnes, mai en forma lliure. Aquest mecanisme ha estat desenvolupat pels éssers vius per tal d'evitar la intoxicació per seleni i així poder-lo mantenir com a element essencial. Les selenoproteïnes acostumen a ser enzims redox, implicats en reaccions d'oxidació-reducció, gràcies a l'alta reactivitat de l'àtom de seleni situat al centre catalític, i per tant, tenen una possible capacitat protectora antioxidant. [1]
Les selenoproteïnes
Les selenoproteïnes
són un tipus de proteïnes que incorporen a la seva
seqüència un aminoàcid anomenat
selenocisteïna,
el qual correspon a l’aminoàcid 21, i
s’anomena
amb una lletra U.
L’aminoàcid selenocisteïna és idèntic a una cisteïna, però ha substituït el radical de sofre (SH) per un de seleni (SeH). El seleni i el sofre ocupen la mateixa columna a la taula periòdica dels elements, fet que els confereix propietats químiques similars, tot i que el seleni resulta ser un element més reactiu que el sofre. Aquest fet explica que quan es busquen els gens ortòlegs de selenoproteïnes en altres organismes, a vegades es troben Cys enlloc de SeCys, ja que en ambdós casos les proteïnes tenen funcions similars. Per exemple, la Selenoproteïna U en peixos no és una selenoproteïna en humans. Fins i tot podem arribar a trobar, dins una mateixa espècie, un gen que codifica per una Selenocisteïna i una còpia del mateix gen que conté una cisteïna.
El nombre de selenoproteïnes existents en les diferents espècies varia molt segons el tàxon. Així doncs, per exemple, es coneixen prop de 30 selenoproteïnes en peixos, 25 en humans, 3 en Drosophila melanogaster i 1 en Caenorhabditis elegans. Tanmateix, no tots els organismes tenen selenoproteïnes, per tant veiem que no són essencials per a la vida. Fins aleshores no se n'ha trobat cap en plantes. De fet, entre els eucariotes és bastant comú, mentre que en altres fílums, no tant. [2]
Síntesi de selenoproteïnes
L’aminoàcid Selenocisteïna és codificat pel codó UGA
el qual és, normalment, un codó de terminació
de la traducció. Per tant, ens trobem amb la situació
que en una mateixa espècie
hi ha un mateix codó amb dos significats diferents.
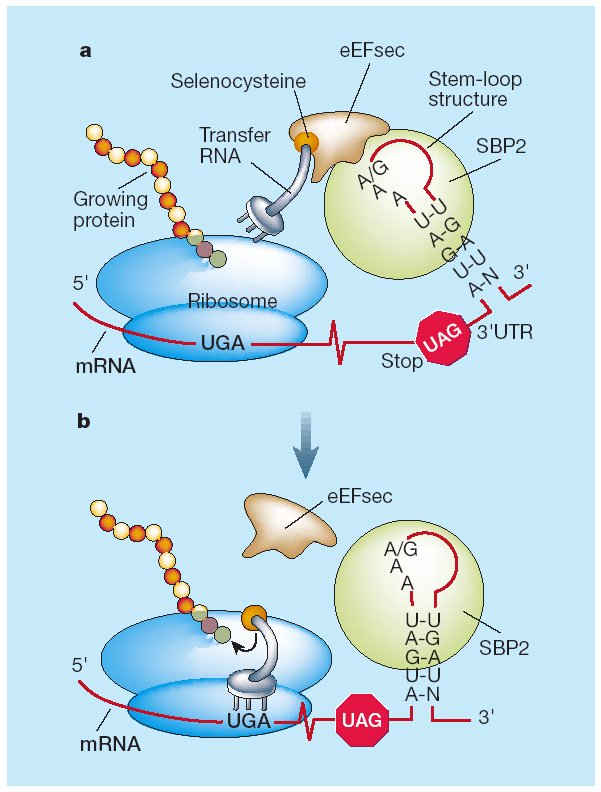
La re-codificació del codó UGA necessita d'una maquinària específica. La principal senyal que requereix el ribosoma per diferenciar correctament un codó d'stop d'un que codifica per una SeCys és un element de l’estructura secundària (tridimensional) del RNA anomenat SeCIS (SelenoCysteine Insertion Sequence). Els SeCIS consisteixen en un loop localitzat en les regions codificants dels gens de selenoproteïnes en bacteris i en l’extrem 3’-UTR dels gens de selenoproteïnes d'archae i eucariota. Aquest element recluta una proteïna anomenada SeCIS Binding Protein (SBP2), la qual s’uneix i alhora recluta un factor d’elongació específic de selenoproteïnes (EFsec). Aquest factor de transcripció selecciona específicament el tRNA unit a SeCys i l'aproxima al codó UGA del mRNA de les selenoproteïnes, determinant la incorporació de la Selenocisteïna. La traducció del mRNA continua fins que el ribosoma reconeix un nou codó de terminació. Per tant, és un procés que requereix la participació de molts gens. [3]
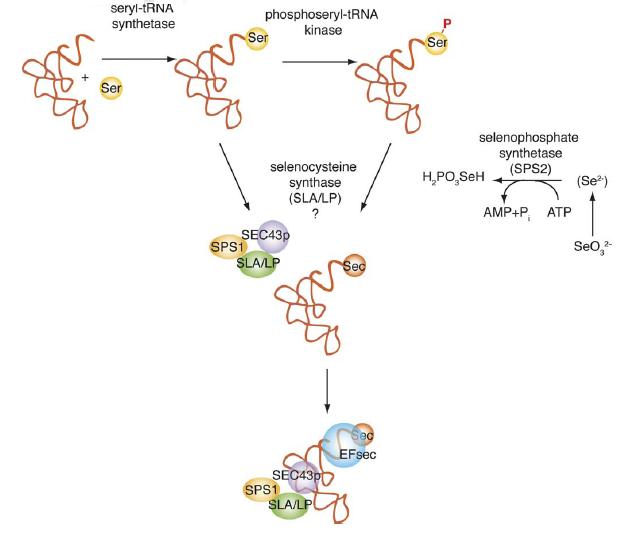
La Selenocisteïna se sintetitza directament en el seu tRNA perquè no es troba lliure a la cèl·lula. El procés que segueix és el següent:
- Una Serina s’uneix mitjançant l'acció de la seril-tRNA sintetasa al tRNA.
- La fosfoseril-tRNA kinasa (PSTK) fosforila la Serina unida al tRNA.
- L’enzim selenocisteïna sintasa (SLA/LP), amb l’ajuda de la selenocysteine associated protein (SEC43p) i les selenofosfat sintetases (SPS1 i SPS2), transformen la Serina en una Selenocisteïna i creen la seqüència EF, on s’unirà el tRNA al factor de transcripció EFsec.
Anotació i predicció
Qualsevol programa automàtic d’identificació de gens, s’equivocarà en la identificació de selenoproteïnes, ja que sempre que trobi un codó UGA el considerarà sistemàticament com un codó d'stop. És per això que les selenoproteïnes estan molt mal anotades.
Per poder-les identificar a nivell dels gens, és necessari trobar alguna manera d'identificar els SeCIS amb l'objectiu de saber quins codons UGAs no codifiquen per codons d'stop sinó per Selenocisteïnes. El problema, és que no s’ha trobat cap patró de seqüència SeCIS, és a dir, no hi ha cap conservació de seqüència que ens permeti identificar la regió SeCIS d’una Selenoproteïna.
Inicialment es creia que entre tots ells només tenen conservats un quartet de nucleòtids (AUGA), però posteriorment s'han identificat quartets diferents. Per aquest motiu, els SeCIS que tenen el quartet AUGA s'anomenen canònics i la resta no canònics.
El que sí sabem és que l’estructura tridimensional d’aquestes seqüències és igual en tots els casos, per tant, una estratègia per trobar aquests SeCIS pot ser buscar les seqüències que es puguin plegar i formar aquesta estructura 3D coneguda (loop-helix-loop). PatScan és un dels diversos programes que poden fer aquesta cerca mitjançant la predicció aproximada de l'estructura d'un RNA petit. Se li dóna el patró estructural i un conjunt de seqüències i el programa mostra quina/es d’aquestes pot adoptar aquesta estructura. Podríem aconseguir un programa per predir de forma aproximada l’estructura d’un RNA petit.
Per fer una predicció eficient de la localització de selenoproteïnes es sobreposen la informació d'elements SeCIS amb la localització d'exons del gen que estem estudiant. [4]
Història natural de les selenoproteïnes
Els selenoproteomes coneguts, ens ofereixen una
visió fins a cert punt desconcertant de
l’evolució
d’aquestes proteïnes. Cada una d’elles sembla tenir
la seva pròpia història evolutiva: poden ser presents
en una determinada espècie o grup taxonòmic,
absents en un altre grup filogenèticament proper, i en canvi, tornar a
ser presents (com a selenoproteïnes o homòlegs en Cys)
en un grup taxonòmic filogenèticament allunyat.
Cada nou genoma analitzat aporta noves sorpreses i fa que calgui replantejar fets que es donaven gairebé per establerts. Per exemple, fins fa poc es pensava que les selenoproteïnes eren essencials per la vida animal, donat que tots els animals coneguts tenien selenoproteïnes i C. elegans manté tota la maquinària molecular necessària només per sintetitzar una selenoproteïna. Tanmateix, després de l’obtenció de la seqüència del genoma de dotze espècies de Drosophila, es va descobrir, mitjançant anàlisis computacionals, que D.willistoni no té selenoproteïnes i ha perdut la capacitat de codificar-ne. L’anàlisi d’un nombre de genomes d’insectes seqüenciats fa un parell d'anys ha permès comprovar que les selenoproteïnes s’han extingit en moltes altres espècies d’insectes, tot i que n’hi ha d’altres que encara les mantenen.
Les selenoproteïnes en els genomes de protists
Entre els grups taxonòmics en el quals la distribució de selenoproteïnes és més diversa i, en conseqüència, més desconeguda, es troben els protists. Òbviament, això es deu en part, a la gran diversitat taxonòmica que caracteritza aquests organismes i a la gran distància filogenètica que els separa.
Les selenoproteïnes que hem estudiat en protists són: la família DI, SelJ i SelQ.
Protists
Els tres dominis reconeguts en la natura viva són Eucariota, Arquea i Eubacteria (els dos últims també anomenats Arqueabacteria i Bacteria, respectivament). En els últims anys, els eucariotes s’han classificat en 4 regnes: animals, plantes, fongs i protists. Mentre que els tres primers regnes són grups monofilètics ben definits, el Regne Protist és parafilètic: conté organismes que es troben més relacionats amb els membres dels altres regnes que amb altres espècies de protists.

El Regne Protist comprèn tots aquells organismes que no pertanyen a cap altre regne, per això és el més divers dels regnes. Conté més de 200.000 espècies conegudes i encara en queden moltes per descobrir, a mode d’exemple hi pertanyen algues, floridures, paràsits (Plasmodium falciparum), protozous, etc. Entre ells no hi ha gaire en comú, a part de ser organismes eucariotes unicel·lulars que existeixen com a cèl·lules independents o bé com a colònies, les quals no s’especialitzen en teixits. Una altra característica comuna entre els organismes protists és que cap es troba adaptat plenament a la vida en presència d’aire, de manera que els que no són totalment aquàtics, habiten en ambients terrestres humits o a l’interior d’altres organismes.
La classificació dels protists ha estat modificada diversos cops en els últims 20 anys. Gràcies a les noves tècniques de comparació de seqüències entre espècies, s’ha eliminat el problema de l’escassetat o ambigüitat dels caràcters morfològics dels protists a l’hora d’establir la seva classificació. Per tant, ara és quan es comença a definir grups monofilètics de protists.
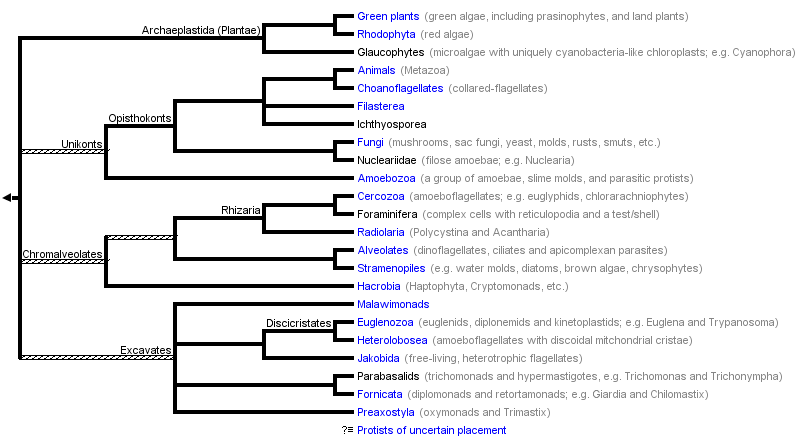
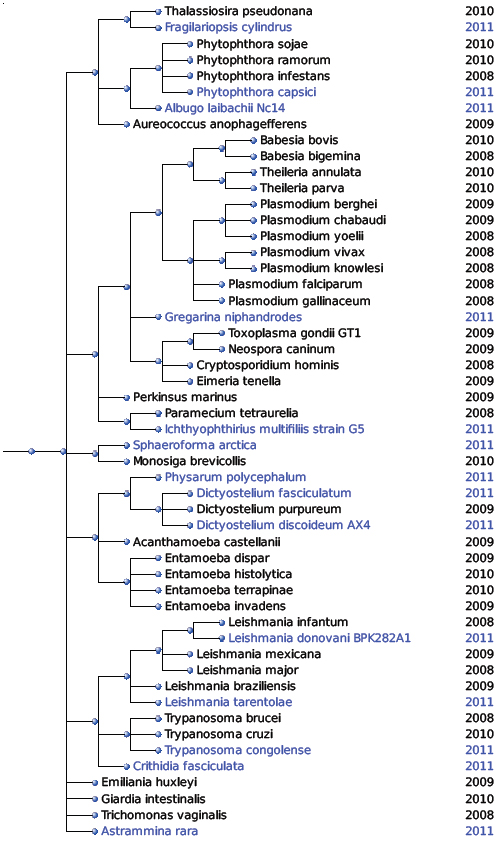
El nostre estudi consisteix en cercar els selenoproteomes dels protists dels quals s’ha seqüenciat el genoma aquest mateix any 2011. Els trobem marcats en blau a la imatge següent i descrits breument a l'apartat de genomes de protistes.