Abstract

Es denominen selenoproteïnes a aquelles proteïnes que contenen com a mínim un aminoàcid selenocisteïna (Sec/U) en la seva seqüència. Aquest és característic per la presència de l’element Seleni en la seva composició. L’aminoàcid Sec està codificat pel codó UGA, que en condicions estàndard és un codó stop. Però que gràcies a la funció dels elements SECIS, a la cadena de mRNA d’aquestes proteïnes, s’insereix aquest residu en la seva posició.
El que hem fet en aquest projecte és identificar tant les selenoproteïnes com les proteïnes encarregades de la síntesi d’estes al genoma de l’organisme Datnioides undecimradiatus, mitjançant la seva homologia amb l’espècie ja caracteritzada Danio rerio (peix zebra). Aquest organisme és un peix de la família Datnioidiae, que habita al riu Mun, a Tailàndia.
Per tal de fer-ho, hem creat un programa automatitzat per fer l’alineament entre la seqüència coneguda de cada proteïna del peix zebra i el nostre organisme. Aquest programa incorpora els programes: tBLASTn, Fastafetch, Fastasubseq, EXONERATE, FastaseqfromGFF, Fastatranslate i T-COFFEE. A més, per comprovar els nostres resultats, hem utilitzat el programa SEBLASTIAN per predir si efectivament hi havia una selenoproteïna en aquesta regió i SECISearch3 per buscar elements SECIS en esta.
De les 53 proteïnes analitzades presents als peix zebra, hem aconseguit anotar correctament 34 selenoproteïnes, 8 homòlogues a cisteïna, 6 relacionades amb la maquinària de síntesi, 2 no presents al nostre genoma i 3 que no s'han aconseguit anotar correctament.
Introducció
Selenoproteïnes
Les selenoproteïnes són proteïnes que contenen una o varies selenocisteïnes (Sec/U), l'anomenat aminoàcid 21. Aquest té una estructura similar a la cisteïna, però la diferència és que el sofre és substituït per seleni. L’element seleni es un micronutrient essencial per a tots els dominis de la vida, ja que té un paper important en moltes funcions, la majoria realitzades per selenoproteines. És per això, que el seleni té molts beneficis per la salut, entre els qual són la protecció contra el càncer i malalties cardiovasculars, així com el control de les alteracions gastrointestinals o la millora del sistema immunitari.
Les funcions de les selenoproteïnes estan implicades en molts processos fisiològics, tot i que no se’n coneixen moltes d’aquestes en mamífers. S’encarreguen de la regulació i senyalització de reaccions d’oxidació-reducció, metabolisme d’hormones tiroidees, respostes immunitàries i defensa antioxidant.
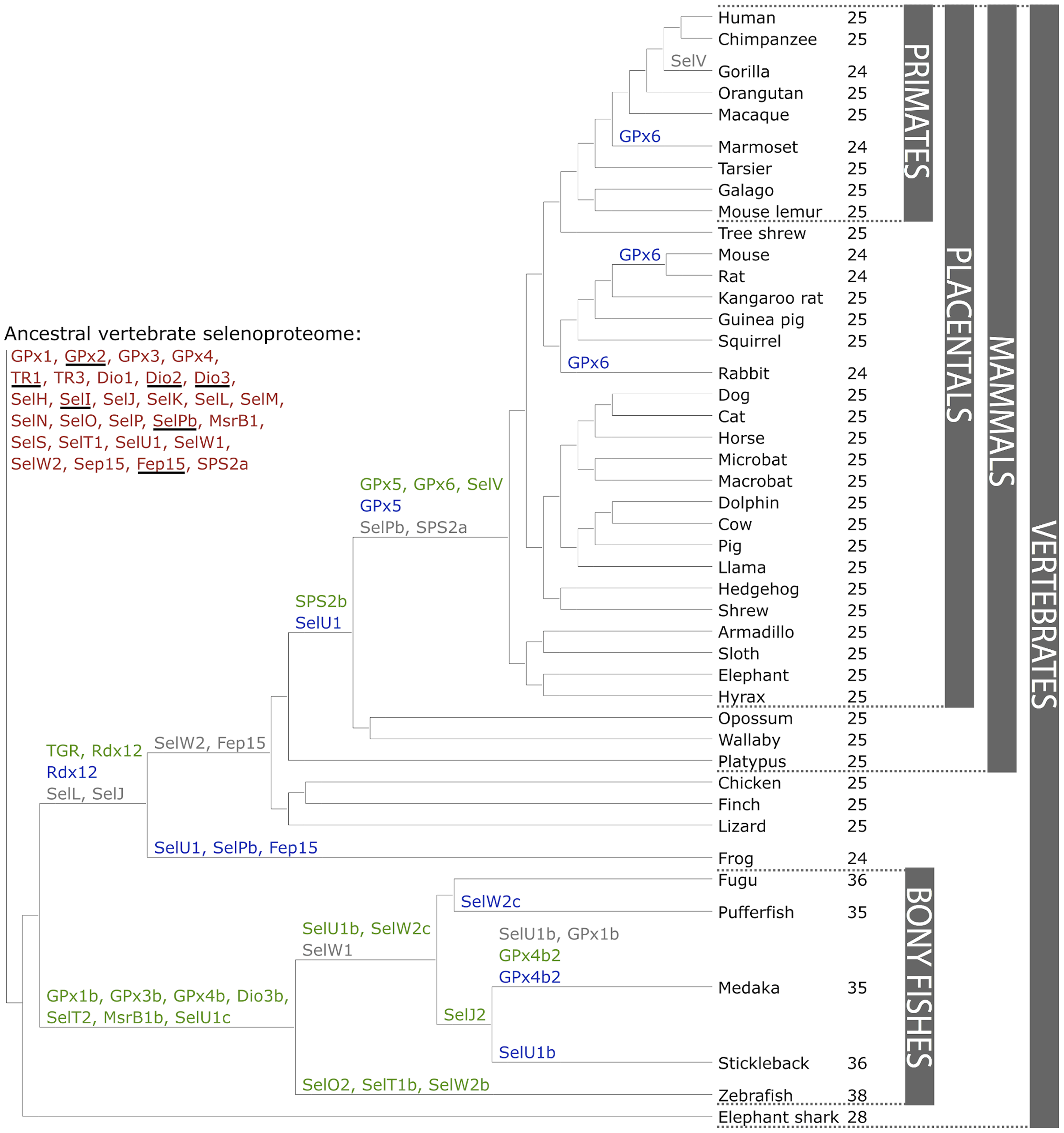
L’evolució de les selenoproteïnes no és coneguda completament. Aquestes proteïnes es troben als tres dominis de la vida i alguns dels components estan bastant conservats, és per això que és probable que hagin aparegut aviat en l’evolució. El selenoproteoma de mamífers està codificat per 25 gens, per altra banda el més llarg és el de Zebrafish que conté 38 selenoproteïnes. S’han trobat 21 selenoproteïnes comuns en tots els vertebrats: GPx1-4, TR1, TR3, DIO1, DIO2, DIO3, SelH, Sell, SelK, SelM, SelN, SelO, SelP, MsrB1, SelS, SelT1, SelW1, Sep15. Altres proteïnes només es troben en certs llinatges, la majoria de selenoproteïnes noves són generades per duplicacions i les altres han substituït la seva Sec per una cisteïna.
Biosíntesi
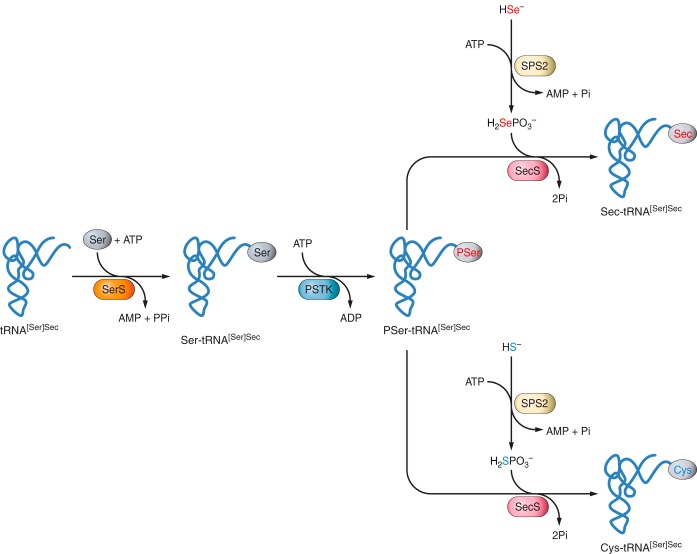
Fins fa ben poc només es coneixia el mecanisme de biosíntesi de les selenocisteïnes en eubacteria, recentment s’ha descobert com es produeix aquest procés en eucariotes. Sec és l’únic aminoàcid la biosíntesi del qual es dóna a través del seu propi tRNA, que s’anomena Sec tRNA. Això comporta un paper clau d’aquesta molècula en la biosíntesi de selenoproteïnes.
La seqüència d’aquesta mostra moltes característiques que la diferencien d’altres tRNA, com per exemple la seva llargada és de 90 nucleòtids, la molècula tRNA més gran seqüenciada. El gen que codifica per aquesta molècula és Trsp, el qual es troba en una còpia única en tots els organismes, excepte en el Zebrafish que té dues còpies.
El primer pas de la biosíntesi és l’aminoacilació del tRNA amb serina per la seril-tRNA sintetasa. El següent pas és la formació de O-fosfoseril-tRNA per la quinasa fosfoseril-tRNA (PSTK). Aquesta molècula formada servirà de substrat per la Sec sintasa, la qual insertarà la forma activa de seleni, el selenofosfat. Aquest és generat per la selenofosfat sintetasa (SPS2) a partir de selenit i ATP. L’enzim SPS2 també està implicat en la síntesi de novo de Cys, fent així que aquest s’inserti a les selenoproteïnes enlloc de Sec.
Pel que fa a la descomposició de les Sec, està involucrat l’enzim Sec liasa (SCL), el qual catalitza la degradació de Sec a L-alanina i seleni. Aquest enzim pot ser utilitzat per les cèl·lules per tal d’aconseguir una font alternativa de seleni per la biosíntesis de selenoproteïnes.
Els passos esmentats de la biosíntesi són específics d’eucariotes, encara que molts són compartits amb eubacteries i archaea.
Incorporació de la selenocisteïna
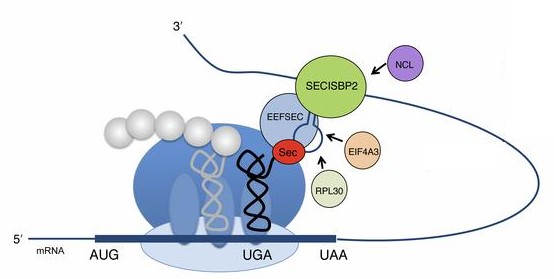
En la traducció estàndard de proteïnes, quan el ribosoma es troba amb un codó UGA es para la traducció ja que es un codó d’stop. En canvi quan s’està traduint una selenoproteïna i el ribosoma es troba amb el codó UGA, la maquinària Sec interacciona amb la maquinària de traducció per prevenir l’stop, i per tant s’introdueixi la selenocisteïna.
Aquesta maquinària consta de diferents proteïnes:
·Elements SECIS: són unes estructures del RNA en forma de loop amb acitivitat cis, que es troben en les regions 3’ del mRNA de selenoproteïnes. Tenen unes característiques que les fan distintives d’altres loops de mRNA, i de les archaea i bacteria. En el cas dels eucariotes són de tipus I, estan formats per dos loops i entre aquests es troba el quartet GA. Aquesta regió és la més important ja que és l’encarregada de la unió amb SBP2. La funció del SECIS és informar que el codó UGA s’ha de recodificar i inserir un Sec, enlloc d’un stop.
·SBP2 (secis binding protein 2): està formada per tres dominis: l’aminoterminal que sembla tenir una activitat reguladora, el domini SID que s’encarrega d’incorporar Sec i el domini COOH-terminal d’unió al RNA, aquest juntament amb SID són els responsables de la unió a SECIS. La funció de la proteïna SBP2 és la unió als elements SECIS i al eEFSec.
·eEFSec (Sec-specific eukaryotic elongation factor): és molt similar al factor d’elongació que s’encarrega d'introduir els altres 20 aminoàcids, però aquest té activitat GTPasa i una alta especificitat pel tRNA aminoacilat. La seva funció és reclutar al tRNA per introduir selenocisteïna a la cadena, perquè això es doni correctament és necessari que estigui unit a SBP2.
·Ribosomal protein L30: és un component de la subunitat gran ribosomal i té un domini que li permet la unió a SECIS. La funció exacta encara no és coneguda, però sembla que forma part de la maquinària basal d’inserció de selenocisteïna.
La regulació en la expressió de selenoproteïnes es dóna per diverses vies. Una d’elles és la quantitat de seleni disponible, però s’ha demostrat que hi ha famílies com les GPx que es troben més afectades per la deficiència de Se i en canvi, altres com les TR no estan tan regulades per la disponibilitat de Se. Per altra banda, les proteïnes eIF4a3 i Nucleolina tenen un paper important en la regulació i la jerarquia de l’expressió de les selenoproteïnes. La proteïna eIF4a3 competeix pel lloc d’unió a SECIS amb la SBP2, per tant serveix com a regulador negatiu per la incorporació de selenocisteïna. La fosfoproteïna Nucleolina és un regulador positiu per la traducció del mRNA que codifica per selenoproteïnes essencials.
Mètodes
Per tal de predir les selenoproteïnes i la maquinària implicada en la seva síntesis al genoma de Datnioides undecimradiatus, realitzarem una comparació de les selenoproteïnes homòlogues anotades en un genoma d’una espècie evolutivament propera a la estudiada. El genoma de referència utilitzat per trobar proteïnes homòlogues és el de Danio rerio (Zebrafish).
El genoma de Danio rerio conté un total de 53 proteïnes entre selenoproteïnes i maquinària implicada en la seva síntesis. Les proteïnes de Danio rerio correspondran a la query, les quals s’han extret de la base de dades SelenoDB, i són les següents:
·Selenoproteïnes:
Sel15, SELENOE, GPx (GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, GPx7, GPx8), DIO (DIO1, DIO2, DIO3a, DIO3b), SELENOH, SELENOI, SELENOJ, SELENOK, SELENOL, SELENOM, SELENON, SELENOO1, SELENOO2, SELENOP, SELENOS, SELENOT (SELENOT1, SELENOT1B, SELENOT2), SELENOU (SELENOU1a, SELENOU2, SELENOU3), SELENOW i TXNRD (TXNRD2, TXNRD3).
·Maquinària implicada en la seva síntesis:
eEFSec, MsrA, PSTK, SBP2, SecS, SEPHS, SEPHS2 , MRSB (MRSB1a, MRSB1b, MRSB2, MRSB3) i SECp43.
Tal com explicarem més endavant, també cercarem els elements SECIS i generarem un arbre filogenètic de les proteïnes de Datnioides undecimradiatus i Danio rerio.
Per tal de dur a terme la predicció de les selenoproteïnes al genoma de Datnioides undecimradiatus, hem creat un programa que, a partir de la query, ens prediu els seus homòlegs a Datnioides undecimradiatus. El programa segueix els passos del següent esquema:
ESQUEMA
1. Predicció de les Selenoproteïnes i maquinària implicada en la seva síntesis
La predicció es far doncs, a partir de les proteïnes anotades de Danio rerio i del genoma de Datnioides undecimradiatus, al qual tenim accés mitjançant el següent path:
/mnt/NFS_UPF/soft/genomes/2019/Datnioides_undecimradiatus/genome.fa
Hem creat un programa íntegrament automatitzat el qual ens genera els alineaments de les selenoproteïnes i ens permet fer la seva predicció a partir dels diferents programes exposats a l’esquema. Però abans hem creat un programa per crear els directoris de cada proteïna. El programa creat és el següent:
PROGRAMA
Seguidament, explicarem pas a pas les diferents parts del programa:
tBLASTn
tBLASTn ens permet buscar les diferents Selenoproteïnes de Danio rerio al genoma Datnioides undecimradiatus.
Abans de fer-ho, hem eliminat els caràcters que dificultaven el funcionament del programa: “%” i “#”. A més, hem substituit el caràcter de la selenocisteïna “U” per una “X”, ja que el programa utilitzat no reconeix les “U”.
L’output del tBLASTn és una taula que ens mostrarà tots els hits trobats en els diferents scaffolds, caracteritzats per diferents e-values. L’e-value és la variable que hem utilitzat com a llindar: els hits amb un e-value superior a 0,0001 han estat desestimats. La taula de valors resultants la emmagatzemarem en un arxiu (blastn$p).
Un cop triats els hits, el programa els ordena per e-value més petit a més gran. D’aquests, el nostre programa tria l’scaffold amb l’e-value més petit, de manera que la predicció la farem amb la proteïna predita en aquest scaffold. Malgrat tot, un cop el programa ha analitzat totes les proteïnes, hem tornat a estudiar els resultats del tBLASTn per anotar altres scaffolds amb e-values semblants per tal de fer la predicció de selenoproteïnes en altres scaffolds candidats. Per fer-ho, hem utilitzat aquest segon programa que ens permet l’estudi d’altres hits individualment.
Fastafetch
Mitjançant el fastafetch, el nostre programa emmagatzema la seqüència de l’scaffold del hit triat en un arxiu (scaffold$p).
FastaSubSeq
L’objectiu del fastasubseq serà guardar en un arxiu la regió d’interès on es troba la selenoproteïna en l’scaffold$p. Per fer-ho, utilitzarem les dades de les columnes de blastn$p on es troben la posició d’inici i la posició de final de la seqüència de nucleòtids de la nostra selenoproteïna.
Primer de tot, el programa agafa aquests dos valors, i interpreta si la cadena és forward o revers i ho emmagatzema en un fitxer (tipuscadena.fa).
Seguint amb la posició d’inici i final, el programa crearà un fitxer guardant la seqüència del scaffold incloent 100.000 nucleòtids anteriors a la posició d’inici que ens indica el tblastn, i amb una length de 200.000 nucleòtids. D’aquesta manera, el programa estarà cercant la selenoproteïna en el hit triat, i també en d’altres hits del mateix scaffold que es troben en posicions cercanes. La seqüència de l’scaffold la guardarà al fitxer subseq$p.
Així doncs, el nostre programa ens cercarà selenoproteïnes en l’scaffold de menor e-value 100.000 nucleòtids per davant de la posició d’inici i amb una length de 200.000 nucleòtids per no desestimar hits que es puguin trobar cercans a la posició indicada. A més, com hem dit anteriorment, aquells hits amb un e-value semblant al triat però en altres scaffolds, els correrem paral·lelament en el segon programa proposat. Hem de tenir en compte, però, que hi ha alguns hits en un mateix scaffold que no entren el el rang de nucleòtids triats, de manera que els cercarem i també els introduirem al segon programa per tal d’estudiar totes les regions susceptibles a contenir selenocisteïnes i obtenir uns resultats més acurats.
Exonerate
Mitjançant aquest pas, el nostre programa agafarà el fitxer subseq$p i ens cercarà els possibles exons que aquest contingui. Amb la funció grep ens separarà exons d’introns. El resultat d’aquest pas serà en una taula on veurem el nombre d’exons, tipus de cadena i posicions d’inici i final de cada exó. Aquestes dades les guardarem al fitxer prot$p.exonerate.gff. Aquest pas s’ha realitzat amb l'opció exhaustive, per a que la cerca d’exons sigui el més acurada possible.
FastaseqfromGFF
El fastaseqfromgff ens permet trobar la seqüència del cDNA dels exons predits en el pas anterior, dades que emmagatzemarem en el fitxer prot$p.exonerate.fa.
Fastatranslate
Seguidament, el programa transformarà la seqüència de nucleòtids a aminoàcids. A més, en aquest pas, hem transformat la selenocisteïna predita (“*”), en una X. Les dades s’emmagatzemen a l’arxiu translate$p.fa
TCOFFEE
Finalment, el TCOFFEE ens permet alinear la seqüència d’aminoàcids obtinguda amb la query.
En certes proteïnes en les que obtenim un score del TCOFFEE molt baix, hem repetit tot el procés seleccionant els exons manualment ja que l’alineament no era el correcte. La selecció d’exons es basa en escollir els que tenen el mateix tipus de cadena que la proteïna i els que es situen més pròxims a les posicions del hit seleccionat al tblastn.
2. SECIS
Els elements SECIS (Selenocystenine Insertion Sequence) son seqüències localitzades a l’extrem 3’UTR en eucariotes necessaris per la traducció del codó UGA en l’aminoàcid Selenocisteïna. La presència d’elements SECIS ens confirma la predicció de Selenoproteïnes.
Per tal de trobar els elements SECIS, hem utilitzat el programa Seblastian, el qual prediu elements SECIS a l’extrem 3’UTR de la seqüència mitjançant “SECISearch3”. A més, el mateix programari ens permetrà buscar seqüències que codifiquen per selenoproteïnes.
Per trobar els elements SECIS, l’input del Seblastian són els fitxers Subseq$p. Seblastian només permet introduir seqüències d’una longitud màxima de 120.000 caràcters, de manera que els nostres fitxers superaven aquestes longituds, i per tant els hem hagut de reduir. Per fer-ho, ens hem de fixar en la direcció de la cadena estudiada (forward o Reverse), ja que els elements SECIS es troben a l’extrem 3’ de la seqüència, de manera que haurem de tallar els fitxers pel principi o pel final depenent del tipus de cadena.
Així doncs, hem creat un programa automatitzat que ens llegeix el document ‘tipusdecadena.fa’ i ens crea un subseq nou apte per a Seblastian.
En aquelles proteïnes duplicades, com el path del subseq és diferent, hem creat un segon programa.
D’altra banda, per tal de predir selenocisteïnes mitjançant SEBLASTIAN, hem hagut de fer un programa diferent ja que els programes anteriors sovint, al agafar la regió 3’, no inclouen la regió on es troba la selenocisteïna. D’aquesta manera hem creat un programa que a partir del fitxer subseq$p que hem creat en el programa original, ens crea un altre subseq modificat el qual té una length de 120.000 nucleòtids.
En les proteïnes duplicades, com el path del subseq és diferent, hem creat un segon programa.
3. FILOGÈNIA
Un cop obtingudes les prediccions de les selenoproteïnes en Datnioides undecimradiatus, per tal d’estudiar les selenoproteïnes predites, hem realitzat diferents arbres filogenètics per cada família de selenoproteïnes utilitzant la seqüència proteica de l’espècie estudiada i de la query.
D’aquesta manera, hem utilitzat el programa Clustal omega per tal de generar els arbres filogenètics. Seguidament, hem “rerootejat” els arbres filogenètics, és a dir que hem afegit una proteïna externa a la família estudiada per tal d’obtenir arbres més acurats i obtenir distàncies a escala entre les diferents proteïnes predites. Per fer el “rerootejat” hem introduit l’arbre generat amb el Clustal al programa iTOL.
Resultats
SELENOPROTEÏNES
MAQUINÀRIA IMPLICADA EN LA SEVA SÍNTESI
Discussió
L’objectiu d’aquest treball és anotar les selenoproteïnes i maquinària necessària per la seva síntesis en el genoma de Datnioides undecimradiatus, espècie que ha estat recentment seqüenciada.
Ens hem valgut de les 49 selenoproteïnes i maquinària implicada en la seva síntesi anotades al genoma de Danio rerio, de les quals hem buscat les homòlogues al genoma de l’espècie estudiada mitjançant el programa creat. A partir dels resultats obtinguts, hem pogut realitzar la predicció de les selenoproteïnes.
Per tal de valorar les prediccions generades en cada proteïna, hem estudiat les diferents dades obtingudes a partir del nostre programa. En cada proteïna exposarem el següent:
·Filogènia de la família estudiada (en cas que la proteïna tingui més d’una variant)
·Funció de la proteïna
·Nombre d’exons, cadena on es troba (forward o reverse) i localització de la proteïna a l’scaffold
·Presència de metionina a l’inici de la proteïna
·Presència de selenocisteïna
·Alineament mitjançant el TCOFFEE: score i valoració
·SEBLASTIAN: Predicció d’elements SECIS i de la pròpia selenoproteïna mitjançant aquest programa
·Valoració final on exposarem si hem predit la selenoproteïna en la seqüència estudiada
Cal comentar, però, que en certes proteïnes de la mateixa família, hem obtingut diversos hits en els mateixos scaffolds i en les mateixes posicions dins d’aquest. Per tal d’esbrinar quin hit correspon a cada proteïna, hem generat un arbre filogenètic amb tots els hits de cada proteïna. El criteri utilitzat ha estat triar el hit que es troba més proper a la proteïna de Danio rerio, de manera que les filogènies presentades són les filogènies resultants d’haver fet la selecció.
SELENOPROTEÏNES
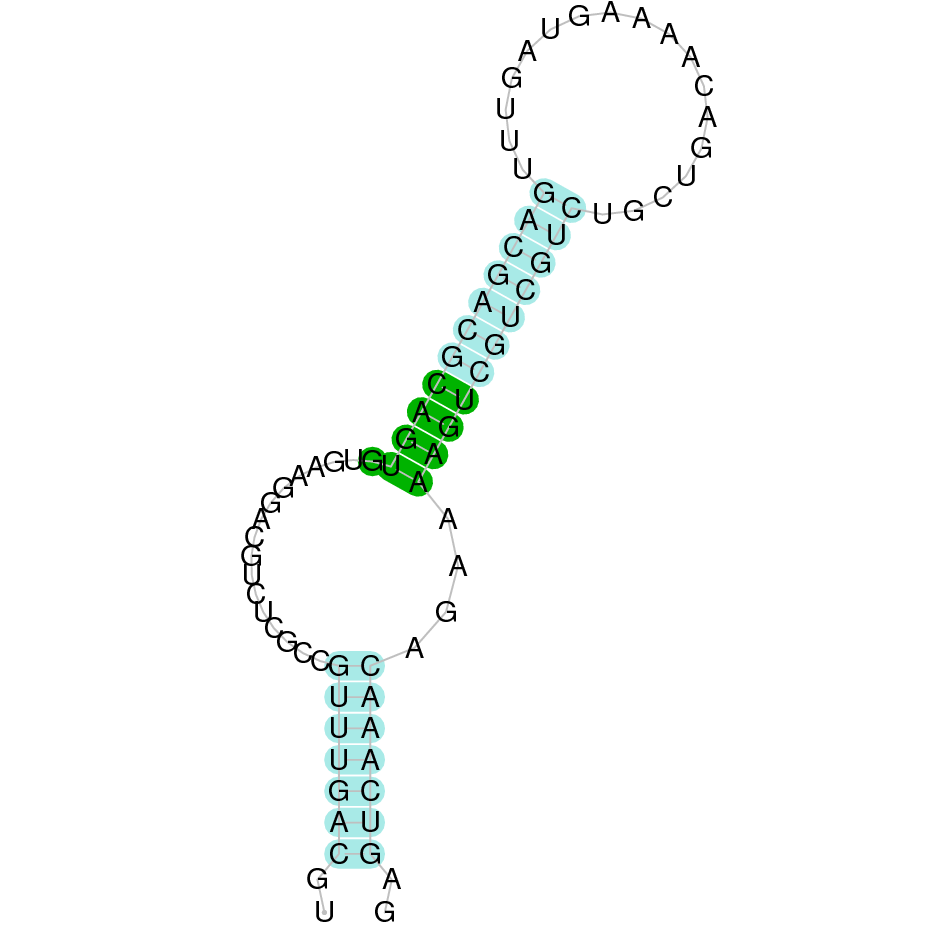
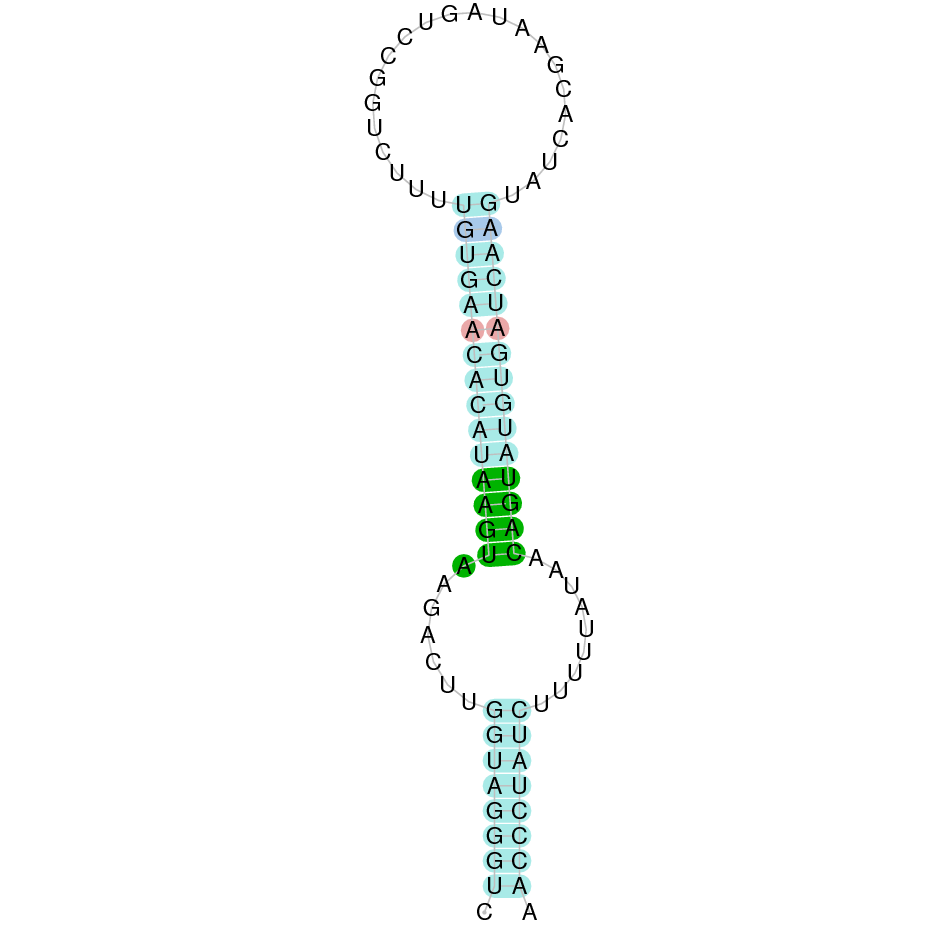
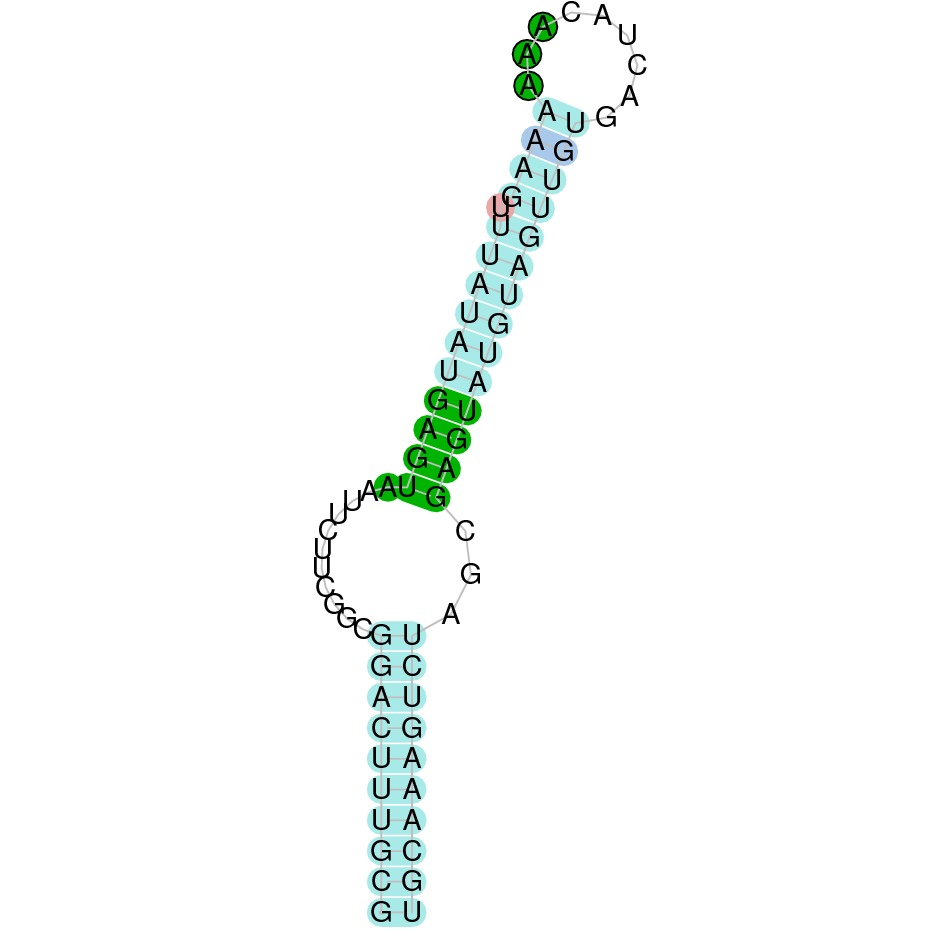
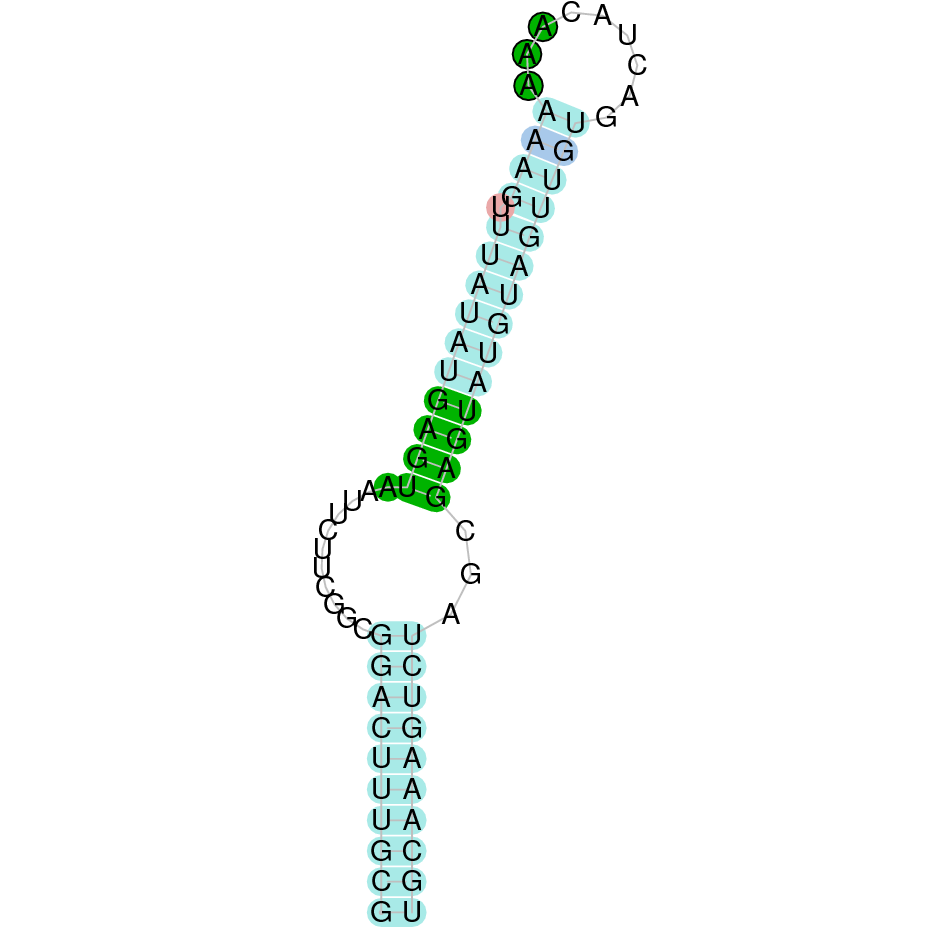
(1) 15 kDa selenoprotein (Sel15)
15 kDa selenoprotein (Sel15)
La Sel15 juntament amb la selenoproteïna M, formen part d’una família de selenoproteïnes. Aquestes es troben als plecs del reticle endoplasmàtic i són del tipus tioredoxina. La nostra espècie no té SelM per tant ens centrarem en la Sel15. La funció principal d’aquesta selenoproteïna és la regulació de l'homeòstasi redox al reticle endoplasmàtic. La seqüència conté un domini tipus tioredoxina i un pèptid senyal aminoterminal, fet que concorda amb la seva localització al reticle. També s’ha vist que pot tenir un paper en la mediació d’efectes quimiopreventius.
La selenoproteïna 15 es troba a l’scaffold WARS01000030.1 de Datnioides undercimradiatus. Aquesta proteïna està formada per sis exons i es troba a la cadena reverse, des de la posició 5957856 fins a la 6029399. No comença per metionina, sinó per leucina. Observem que conté una selenocisteïna. Segons la predicció, hem obtingut un e-value de 1.15e-19. L’alineament té un score de 1000, la proteïna s’ha predit completament. Aquesta proteïna conté un element SECIS de grau B.
SEBLASTIAN ens ha predit una selenoproteïna, per tant podem concloure que la proteïna Sel15 es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna Sel15
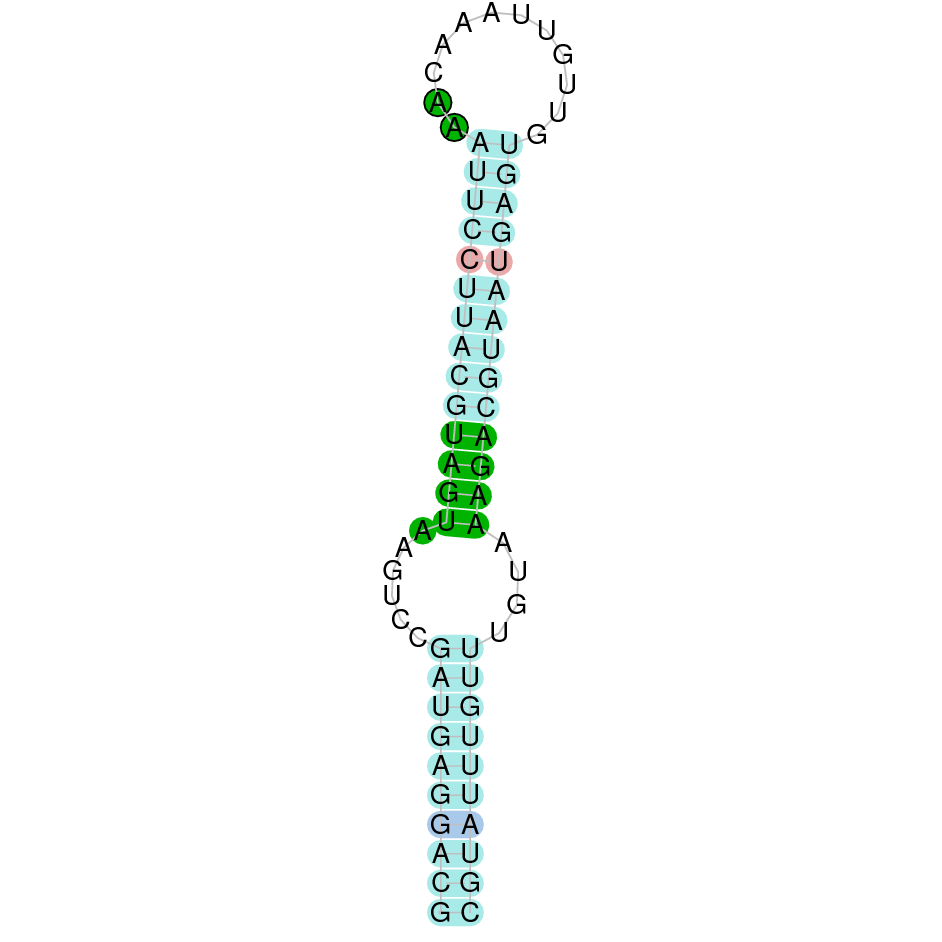
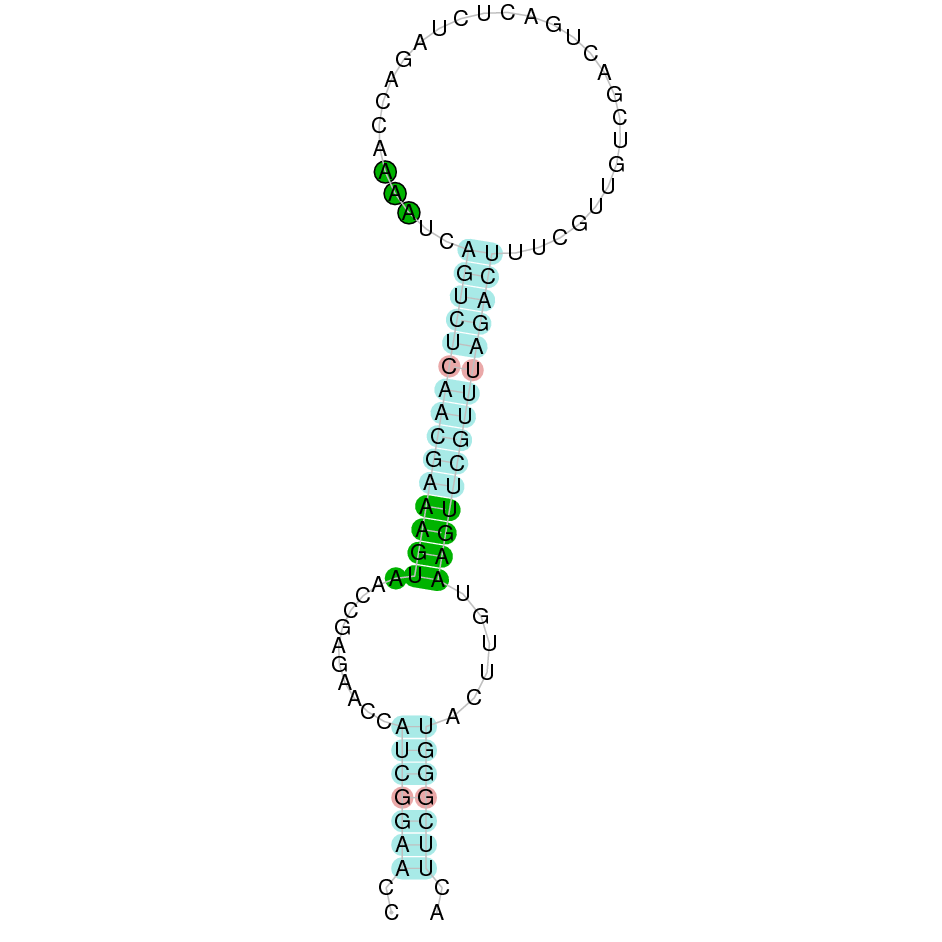
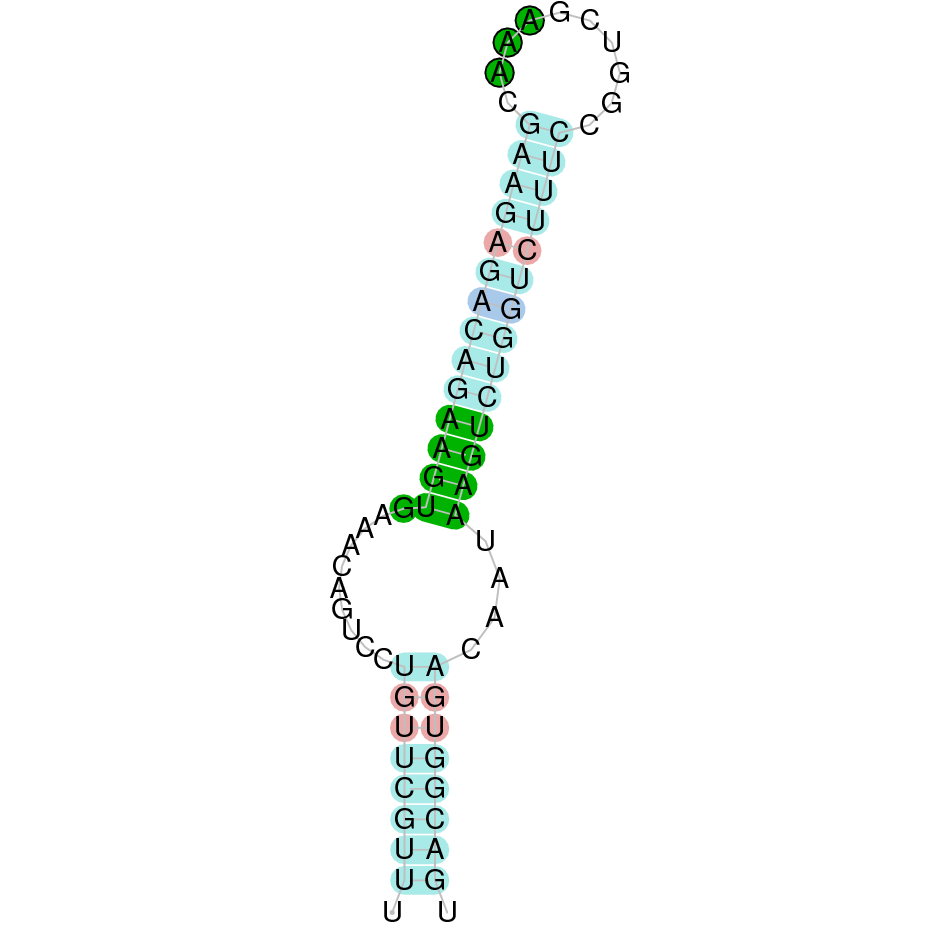
(2) Fish selenoprotein 15 (SELENOE)
Fish selenoprotein 15 (SELENOE)
La proteïna SELENOE o també anomenada Fep15, forma part de la familia de selenoproteïnes Sep15, tot i ser homòloga es troba distant. Aquesta proteïna només es troba en peixos, es absent en mamífers inclús en la forma ortóloga que conté Cys. Fep15 es troba al reticle endoplasmàtic i es creu que té una funció especialitzada que difereix de les Sep15 i SelM. Els anàlisis filogenètics suggereixen que Sep15 va evolucionar per duplicació gènica.
Aquesta proteïna es situa a l’scaffold WARS01000015.1 de Datnioides undecimradiatus. Està formada per sis exons i es troba a la cadena forward, entre les posicions 12243083 i 12329977. No comença per metionina, sinó per fenilalanina. La nostra proteïna conté una selenocisteïna. L’e-value obtingut és 4.36e-17, i té un score de 980. La proteïna està ben predita, però al principi la predicció no és gaire bona. No s’ha trobat cap element SECIS.
SEBLASTIAN ens ha predit la selenoproteïna SELENOE, per tant concloem que es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna SELENOE
(3) Glutathione peroxidase (GPx)
Les selenoproteïnes d’aquesta família es troben en els tots els tipus d’organismes, Eubacteria, Archaea i Eukarya. En el cas del Zebrafish, es troben vuit subtipus de GPx, les primers cinc contenen selenocisteïna, en canvi la GPx7 i GPx8 contenen Cys en comptes de Sec. Aquesta família de proteïnes abarquen moltes funcions fisiològiques importants i estan implicades en el manteniment l’homeòstasi redox cel·lular, la desintoxicació de hidroperòxids i la senyalització del peròxid d’hidrogen. Tot i que els residus del lloc actiu de tots els enzims GPx estan conservats i els seus mecanismes catalítics són idèntics, les funcions fisiològiques de les diferents selenoproteïnes GPx són molt diverses.
Durant l'evolució, els peixos vertebrats han patit duplicacions dels gens de les selenoproteïnes, degut a la duplicació sencera del genoma i a duplicació gènica. En el cas de les GPx, les principals duplicacions són les GPx1b, GPx3b i GPx4b. A més, les proteïnes GPx7 i GPx8 han evolucionat de la GPx4 ancestral, la qual és la proteïna més antiga d’aquesta família. Les proteïnes GPx1 i GPx2 es troben més relacionades entre elles, presenten més homologia, que respecte les proteïnes ortòlogues respectives d’humans. Aquest fet ens mostra que és probable que aquestes proteïnes hagin evolucionat per duplicació gènica.
A l’analitzar les diferents selenocisteïnes d’aquesta família, hem vist que al fer el tBLASTn, teníem hits idèntics (en scaffold i posició genòmica) en les següents selenoproteïnes: GPx1a, GPx1b, GPx2 i GPx3b. Per tal de saber quin hit correspon a cada selenoproteïna, hem realitzat un arbre filogenètic per determinar quin d’aquests hits correspon a cadascuna de les selenoproteïnes del genoma de referència.

Arbre filogenètic de la família de proteïnes GPx
Enllaç per a l'arbre filogenètic de GPx intermedi
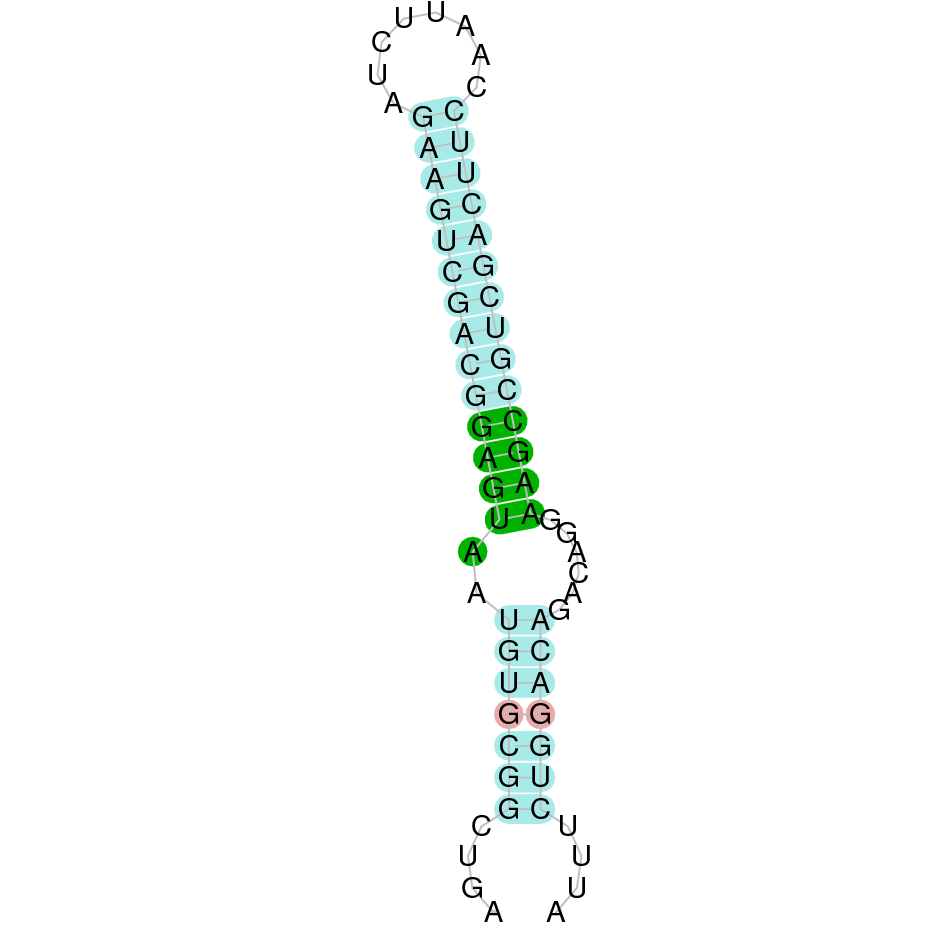
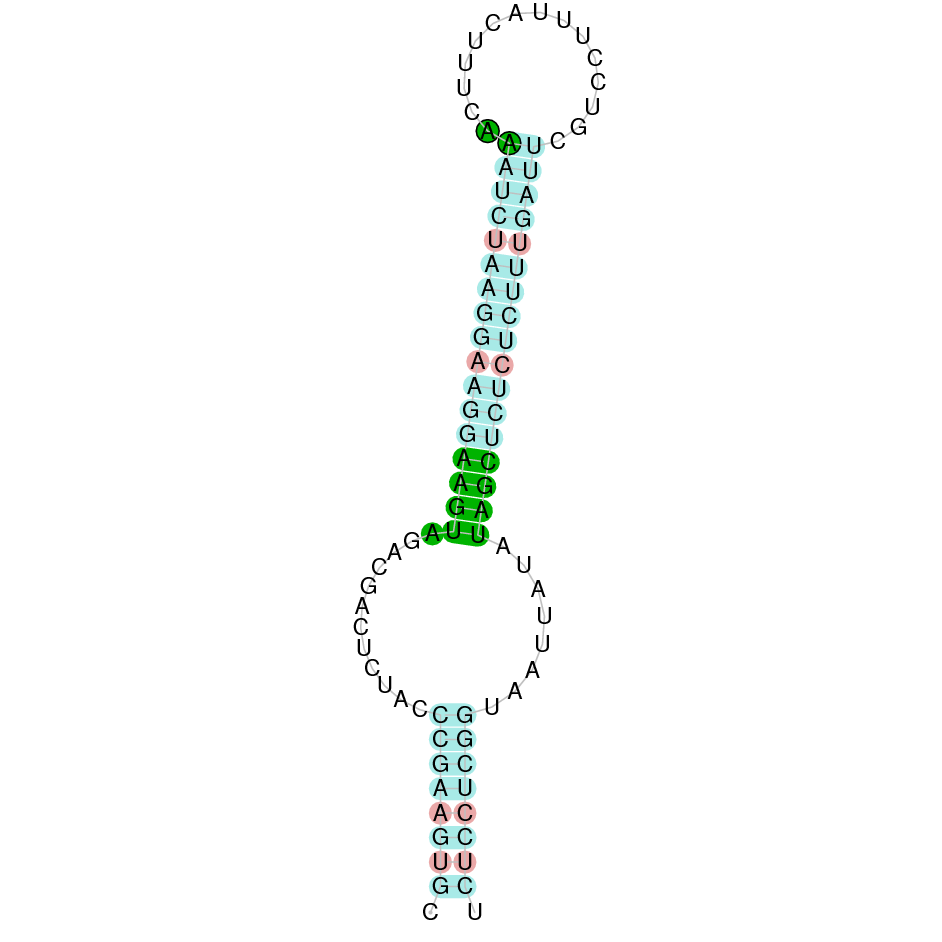
Glutathione peroxidase 1a (GPx1a)
A la GPx1a obtenim dos scaffolds (WARS01000013.1 i WARS01000017.1) on es pot trobar la proteïna de Datnioides undecimradiatus. A més, dins l’scaffold WARS01000013.1 observem dos hits amb un e-value semblant però que es troben a posicions diferents. Segons els resultats observats a la filogènia hem escollit l’scaffold WARS01000013.1.2, ja que aquest és el que es troba més a prop de la proteïna GPx1a del Zebrafish. Aquesta elecció coincideix amb que és el hit que té un e-value major (1.58e-61) i un score major (999). La proteïna està formada per dos exons, es troba a la cadena forward entre les posicions 9739075 - 9740862. Comença per treonina, tot i que ha perdut els 5 primers aminoàcids i hauria de començar per metionina. Conté una selenocisteïna a la seqüència. La proteïna s’ha predit correctament ja que l’score obtingut al TCOFFEE és 999. Té un element SECIS de grau C.
SEBLASTIAN ens ha predit la selenoproteïna, això afirma que GPx1a es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna GPX1a
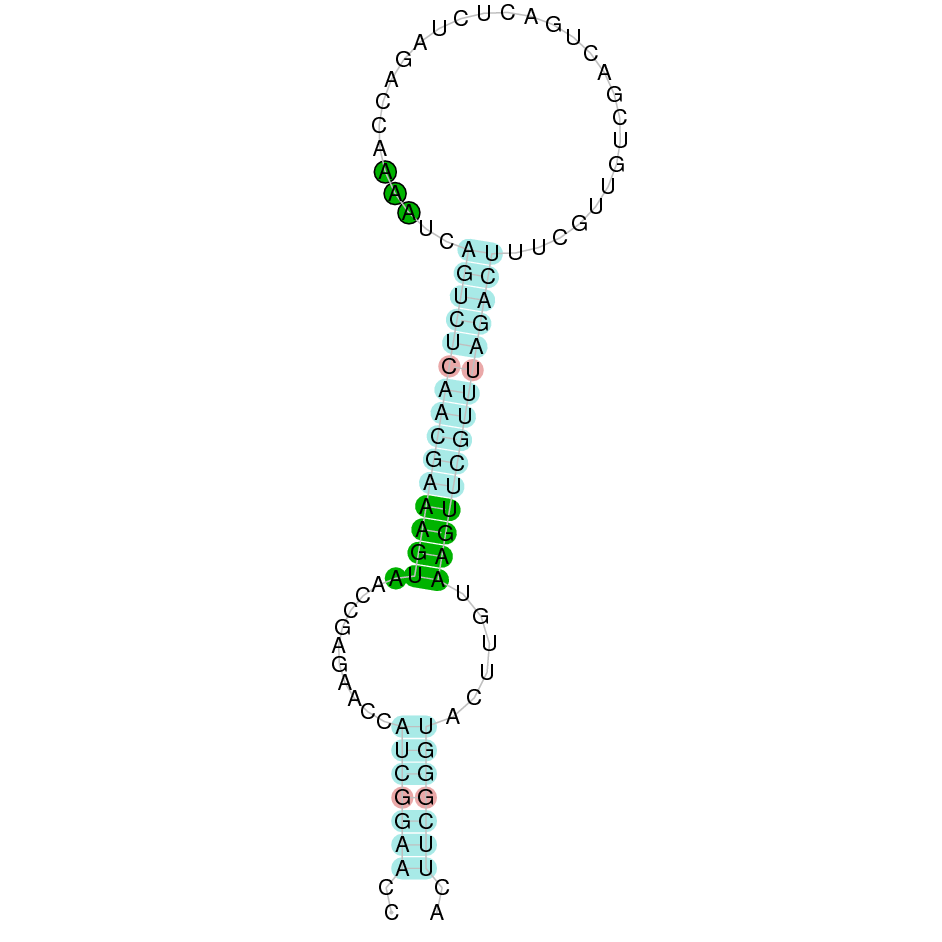
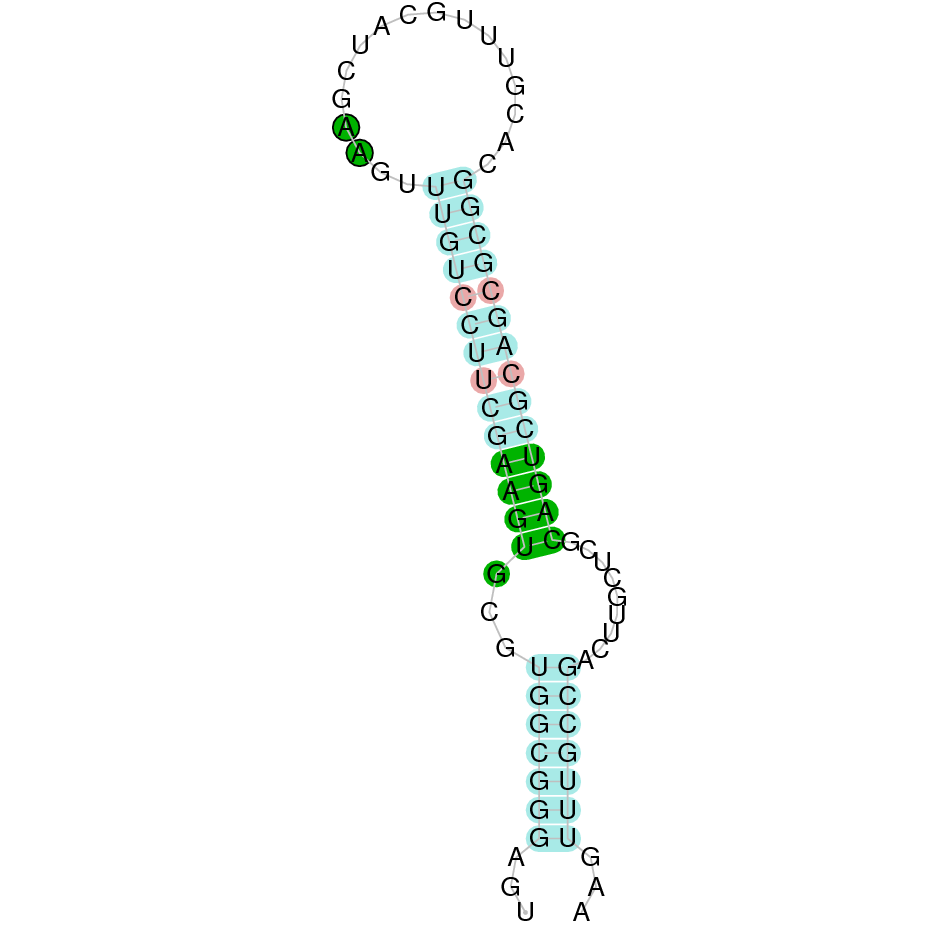
Glutathione peroxidase 1b (GPx1b)
En aquest cas també observem dos scaffolds (WARS01000013.1 i WARS01000017.1), i en el WARS01000013.1 s’obtenen dos hits diferents amb e-value semblants, però que es troben a posicions diferents. A partir de l’arbre filogenètic, observem que l’scaffold que correspon a la proteïna GPx1b de Datnioides undecimradiatus és WARS01000013.1.1 ja que és el que té més homologia amb aquesta proteïna del Zebrafish. A més coincideix que l’scaffold triat es el que té un major score (1000). GPx1b està formada per dos exons i es troba a la cadena reverse, entre les posicions 7479077 - 7480250. La proteïna comença per metionina i conté una selenocisteïna. Hem obtingut un e-value de 1.21e-59 i un score de 1000, per tant la proteïna s’ha predit completament. Té dos elements SECIS, ambos a la cadena reverse i de grau B.
SEBLASTIAN ens ha predit la selenoproteïna, per tant confirmem que GPx1b es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna GPX1b
Glutathione peroxidase 2 (GPx2)
El tBLASTn d’aquesta proteïna ens mostra dos scaffolds candidats (WARS01000013.1 i WARS01000017.1) en els que podem trobar la proteïna GPx2. A l’scaffold WARS01000013.1 trobem dos hits amb e-values similars però que es situen a posicions diferents. Segons l’arbre filogenètic l’scaffold més proper a la proteïna corresponent del Zebrafish és el WARS01000017.1. A més, com ha passat anteriorment, és l’scaffold que té un major e-value (3.50e-49) i un major score (1000). La proteïna està formada per dos exons, es troba a la cadena reverse i entre les posicions 2714071 - 2715193. Comença per metionina. Conté una selenocisteïna. L’score obtingut a l’alineament és de 1000, de tal manera que la proteïna s’ha predit completament. Té un element SECIS de grau B.
Hem obtingut predicció de la nostra proteïna per SEBLASTIAN, per tant concloem que GPx2 es troba a Datnioides undecimradiatus.
Aquestes tres proteïnes (GPx1a, GPx1b i GPx2) contenen scaffolds les unes de les altres. Com podem veure clarament a l’arbre filogenètic, tots els scaffolds WARS01000013.1.2 obtinguts de les diferents proteïnes de Datnioides undecimradiatus corresponen a la GPx1a de Zebrafish. Tots els scaffolds WARS01000013.1.1 de Datnioides undecimradiatus corresponen a la proteïna GPx1b, i el mateix passa amb els scaffolds WARS01000017.1 que corresponen tots a la proteïna GPx2.
Per tant podem concloure que cada scaffold, encara que es trobi en diferents proteïnes de Datnioides undecimradiatus, equival a una proteïna (de les tres esmentades) de Zebrafish. Això és perquè les proteïnes de Zebrafish es troben més diferenciades, en canvi a Datnioides són molt semblants, per tant el programa les alinea juntes interpretant-les com la mateixa. Com hem explicat anteriorment, aquesta homologia que presenten les diferents proteïnes es deu a que han evolucionat a través de duplicació gènica.
Exó de la proteïna GPX2
Glutathione peroxidase 3a (GPx3a)
La proteïna GPx3a es troba a l’scaffold WARS01000014.1 de Datnioides undecimradiatus. Esta composada per sis exons i es troba a la cadena reverse, se situa entre les posicions 3342798-3396768. La proteïna comença per triptòfan, ja que el principi s’ha perdut. Conté una selenocisteïna a la seqüència. Hem obtingut un e-value de 1.24e-27 i té un score de 999, per tant la proteïna s’ha predit bé. Té tres elements SECIS de grau B, però només un d’ells és el correcte, el que es troba a la cadena reverse com la nostra proteïna.
SEBLASTIAN ens ha predit la selenoproteïna, per tant confirmem que GPx3a es troba a Datnioides undecimradiatus.
Exó de la proteïna GPX3a
Glutathione peroxidase 3b (GPx3b)
En aquesta proteïna obtenim varis scaffolds (WARS01000013.1, WARS01000017.1 i WARS01000012.1). A l’hora d’escollir l’scaffold més adequat on es troba la seqüència corresponent a la proteïna GPx3b, ens hem fixat en la filogènia per determinar l’scaffold que es troba més a prop de la proteïna del Zebrafish. A més, els valors d’e-value relativament baixos i el percentatge d’identitat suficientment alt d’aquest mateix scaffold, han concordat amb les observacions de l’arbre filogenètic. Per tant, l’scaffold que correspon a la proteina GPx3b de Datnioides undecimradiatus és WARS01000012.1. Està formada per cinc exons i es troba a la cadena reverse, entre les posicions 1354517 i 1329491. Comença per metionina i conté una selenocisteïna. L’e-value obtingut és 3.39e-20 i l’score de l’alineament és de 1000, per tant la proteïna s’ha predit completament. Té un element SECIS de grau B.
SEBLASTIAN no ha predit la selenoproteïna, però tot i així, amb els resultats obtinguts podem concloure que la proteïna GPx3b es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna GPX3b
Glutathione peroxidase 4a (GPx4a)
Aquesta proteïna es troba a l’scaffold WARS01000029.1 de Datnioides undecimradiatus. Està formada per quatre exons, es troba a la cadena forward entre les posicions 18136314-18188610. El primer aminoàcid es la fenilalanina, i la proteïna conté una selenocisteïna. Hem obtingut un e-value de 3.09e-23 i un score de 1000. La proteïna s’ha predit correctament i té un element SECIS de grau B.
Hem obtingut una predicció de la selenoproteïna per SEBLASTIAN, d’aquesta manera confirmem els resultats obtinguts previament. GPx4a es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna GPX4a
Glutathione peroxidase 4b (GPx4b)
La proteïna GPx4b es troba a l’scaffold WARS01000029.1 de Datnioides undecimradiatus. Està composta per quatre exons, es situa a la cadena forward des de la posició 6281867 fins a la 6283223. Aquesta proteïna no comença per metionina, sinó per arginina, tot i que sembla que s’ha perdut el primer aminoàcid, que en la proteïna del Zebrafish observem que es fenilalanina. Conté una selenocisteïna a l’inici de la seqüència. L’e-value obtingut és 1.56e-19 i l’alineament obtingut pel TCOFFEE ens dona un score de 999, per tant la proteïna s’ha predit correctament. La proteïna conté un element SECIS de grau B.
S’ha obtingut una predicció de la selenoproteïna per SEBLASTIAN, així concloem que la proteïna GPx4b es troba a Datnioides undecimradiatus.
Exó de la proteïna GPX4b
Glutathione peroxidase 7 (GPx7)
Aquesta proteïna es troba a l’scaffold WARS01000029.1 de Datnioides undecimradiatus. Està formada per tres exons i es situa a la cadena forward entre les posicions 23882492 i 23883457. La proteïna GPx7 no comença per metionina, sinó que comença per glutamina. No té cap selenocisteïna, la té substituida per una Cys. Segons la predicció l’e-value obtingut és 3.35e-48 i l’score de l’alineament es de 1000. La proteïna s’ha predit completament. Té un element SECIS de grau B.
SEBLASTIAN no ha predit la selenoproteïna, però segons els resultats anteriors podem concloure que Datnioides undecimradiatus té la proteïna GPx7.
Exó de la proteïna GPX7
Glutathione peroxidase 8 (GPx8)
La proteïna GPx8 es situa a l’scaffold WARS01000015.1 de Datnioides undecimradiatus. Aquesta proteïna la formen tres exons, es troba a la cadena forward i entre les posicions 10288279 - 10289186. El primer aminoàcid de la seqüència es l’arginina, per tant no comença per metionina. No conté cap selenocisteïna, però a Zebrafish tampoc, aquesta està substituida per una Cys. L’e-value obtingut segons la predicció és 9.06e-43 i l’score obtingut al TCOFFEE és 1000, la proteïna s’ha predit completament. No hem trobat cap element SECIS.
No s’ha predit la selenoproteïna per SEBLASTIAN, però els resultats ens mostren que GPx8 es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna GPX8
(4) Iodothyronine deiodinase (DIO)
La família de les selenoproteïnes DIO està formada per tres proteïnes paralogues, una d’elles es troba en dos subtipus diferents (DIO3a, DIO3b). Aquestes tenen diferents localitzacions, la DIO1 i DIO3 es troben a la membrana plasmàtica, en canvi la DIO2 al reticle endoplasmàtic. Totes contenen un domini transmembrana i el residu Sec del lloc actiu es troba a la regió aminoterminal. La seva funció és regular l’activitat de l’hormona tiroide per desiodinació reductiva. Les selenoproteïnes DIO1 i DIO2 s’encarreguen de convertir la tiroxina (T4) en hormona tiroide activa (T3), a través de l’eliminació de iode de l’anell exterior. Per que es doni la inactivació de l’hormona tiroidea, DIO3 catalitzarà la desiodinació interna de l’anell, donant lloc a la formació de T2 inactiva i T3 inversa (rT3).
L’evolució de les selenoproteïnes DIO té els seus inicis en la proteïna ancestral DIO1, de la qual van derivar DIO2 i DIO3 a la ramificació dels vertebrats. L’estructura d’aquestes proteïnes es troba molt conservada. Tal i com hem mencionat prèviament, els peixos vertebrats han estat sotmesos a duplicacions gèniques. En aquesta família de proteïnes trobem una proteïna que ha evolucionat per duplicació, la DIO3b, la qual deriva de DIO3.

Arbre filogenètic DIO1, DIO2, DIO3a i DIO3b
Enllaç per a l'arbre filogenètic DIO3a i DIO3b intermedi
Iodothyronine deiodinase 1 (DIO1)
Aquesta proteïna es troba a l’scaffold WARS01000029.1 de Datnioides undecimradiatus. Està formada per quatre exons i es troba a la cadena reverse, entre les posicions 15853037 i 15917458. La proteïna no comença per metionina, sinó per leucina. Conté una selenocisteïna a la seqüència. Segons la predicció l’e-value obtingut és 2.98e-46, al TCOFFEE obtenim un score de 999, la proteïna DIO1 s’ha predit correctament. Té un element SECIS de grau B.
SEBLASTIAN ens ha predit la selenoproteïna, per tant concorda amb els resultats anteriors, així Datnioides undecimradiatus conté DIO1 al genoma.
Exó de la proteïna DOI1
Iodothyronine deiodinase 2 (DIO2)
Aquesta proteïna es troba a l’scaffold WARS01000045.1 de Datnioides undecimradiatus. Està composta per tres exons i es troba a la cadena reverse, entre les posicions 5343707 i 5349279. La proteïna comença per metionina. Conté una selenocisteïna. Hem obtingut un e-value de 8.09e-86 i l’alineament té un score de 996, la proteïna s’ha predit correctament. Té un element SECIS de grau B.
SEBLASTIAN ha predit la selenoproteïna, d’aquesta manera concloem que DIO2 es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna DOI2
Iodothyronine deiodinase 3a (DIO3a)
En aquest cas la proteïna es troba a dos scaffolds significatius diferents, ja que ambdós tenien uns valors d’e-value molt baixos i un score molt alt. Hem escollit l’scaffold WARS01000046.1 de Datnioides undecimradiatus, ja que com observem a l’arbre filogenètic és el que es troba més a prop de la proteïna DIO3a del Zebrafish, i per tant el més similar. La proteïna està formada per un exó i es troba a la cadena forward, des de la posició 10536696 fins a la 10537487. Comença per glicina, no per metionina. Conté una selenocisteïna a la seqüència. L’e-value obtingut és 8.84e-109 i l’alineament té un score de 983, la proteïna s’ha predit correctament tot i que hi ha zones on l’alineament no és del tot bo. Conté dos elements SECIS de grau B, però només és vàlid el que es troba a la cadena forward, igual que la nostra proteïna.
Hem obtingut la predicció de la selenoproteïna per SEBLASTIAN, per tant confirmem que la proteïna DIO3a es troba a Datnioides undecimradiatus.
Exó de la proteïna DOI3a
Iodothyronine deiodinase 3b (DIO3b)
Aquesta proteïna igual que l’anterior es troba a dos scaffolds diferents. En aquest cas hem escollit l’scaffold WARS01000017.1 de Datnioides undecimradiatus, ja que com observem a l’arbre filogenètic és el que es troba més a prop de la proteïna DIO3b del Zebrafish, i per tant el més similar. Està formada per un exó i es troba a la cadena forward, entre les posicions 7057159 i 7057899. No comença per metionina, sinó per lisina. Conté una selenocisteïna. Hem obtingut un e-value de 6.76e-109 i un score de 996, per tant la proteïna s’ha predit correctament. Té quatre elements SECIS de grau B, però només un d’ells és troba a la cadena forward, per tant és l’únic vàlid.
SEBLASTIAN ha predit la selenoproteïna, fet que concorda amb els nostres resultats, per tant DIO3b es troba al genoma de Datnioides undecimradiatus.
Podem observar segons les dades analitzades i l’arbre filogenètic, que els mateixos scaffolds obtinguts de diferents proteïnes corresponen a la mateixa proteïna de Zebrafish. És a dir, l’scaffold WARS01000046.1 obtingut tant de la proteïna DIO3a com DIO3b de Datnioides undecimradiatus, correspon a la proteïna DIO3a de Zebrafish. Per altra banda, l’scaffold WARS01000017.1 obtingut de les proteïnes DIO3a i DIO3b de Datnioides corresponen a la proteïna DIO3b. Per tant, un dels scaffolds correspon a una proteïna i l’altre scaffold a l’altra.
Això pot ser degut a que les proteïnes de Zebrafish es troben més diferenciades, en canvi les de Datnioides són molt semblants, per tant el programa les alinea juntes interpretant-les com la mateixa. L’homologia observada entre les proteïnes es deguda a com han evolucionat, per duplicació gènica.
Exó de la proteïna DOI3b
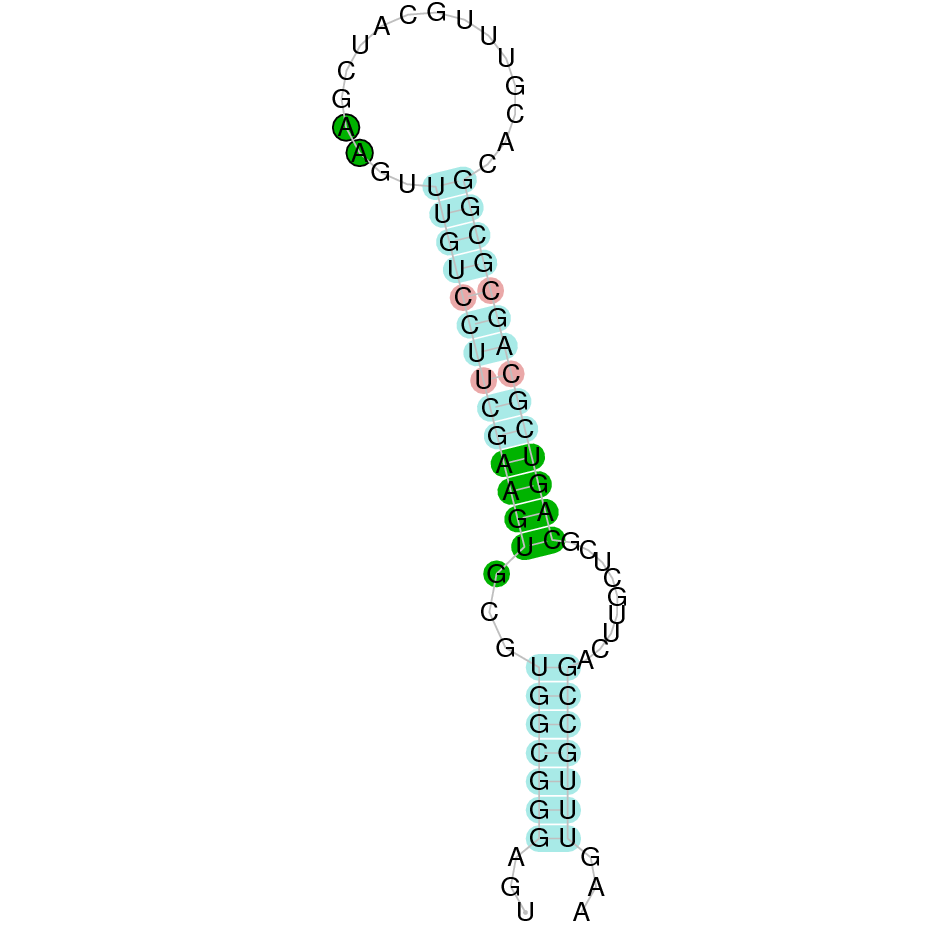
(5) Selenoprotein H (SELENOH)
Selenoprotein H (SELENOH)
És una proteïna que es localitza específicament al nuclèol, i s’expressa principalment durant el desenvolupament embrionari. Està relacionada amb la resposta front a xoc i estrès tèrmic. A més té activitat glutatió peroxidasa. S’ha vist que la seva activitat protegeix les neurones contra les radiacions UV inhibint les vies de senyalització relacionades amb l’apoptosi.
Aquesta proteïna es troba a l’scaffold WARS01000042.1 de Datnioides undecimradiatus. La proteïna conté 4 exons i se situa en la cadena forward entre les posicions 598107-609190. La proteïna predita no comença per metionina ja que ha perdut els 3 aminoàcids inicials, i sí que conté una selenocisteïna, igual que la del Zebrafish.
L’evalue de la predicció és de 2.97e-11, i l’alineament realitzat amb el T-COFFEE té un score de 959.
Hem trobat elements SECIS de categoria B en la seqüència analitzada i SEBLASTIAN ha predit que efectivament en esta regió del genoma hi ha una selenoproteïna que conté una cisteïna. Podem dir que esta proteïna es troba ben anotada al genoma del peix.
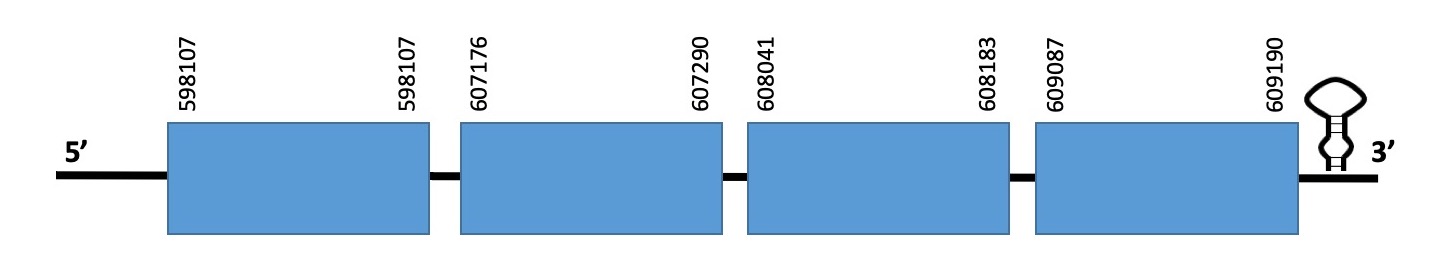
Exó de la proteïna SELENOH
(6) Selenoprotein I (SELENOI)
Selenoprotein I (SELENOI)
És una selenoproteïna específica de vertebrats. Codifica per una proteïna transmembrana la funció de la qual és catalitzar l’últim pas de la síntesis de novo de dos fosfolípids: permet el pas de fosfocolina i fosfoetanolamina a fosfatidilcolina i fosfatidiletanolamina respectivament, fosfolípids necessaris per la formació de vesícules i plegament cel·lular.
Esta proteïna es troba al scaffold WARS01000046.1 i presenta un nombre elevat d’exons entre les posicions 12287222 i 12474204 de la cadena reverse del genoma. La proteïna comença per metionina, a l’igual que la SELENOI de Zebrafish. Ambdues conserven la selenocisteïna.
Observem com al TCOFFEE, amb una score de 891, l’alineament de la proteïna no és gens bo. La major part de la proteïna de Zebrafish no s’alinea amb la proteïna predita al nostre peix.
No s’ha trobat cap element SECIS, però SEBLASTIAN sí que ha predit una selenoproteïna en el genoma de Datnioides undecimradiatus, on ambdues proteïnes comencen per metionina i conserven selenocisteïna.
En conclusió, podem dir que amb els resultats obtinguts amb el TCOFFEE no podem concloure que la proteïna SELENOI estiga ben anotada.
(7) Selenoprotein J (SELENOJ)
Selenoprotein J1 (SELENOJ1)
És una selenoproteïna que es troba únicament en peixos de clase Actinopterygii. No té funció enzimàtica: s’expressa principalment a l’ull en etapes d’embriogènesis, on la seva funció és el manteniment la transparència d’aquest i la correcta difracció de la llum a l’ull.
La proteïna es troba al scaffold WARS01000030.1 del genoma, presentant també com l’anterior un gran nombre d’exons entre les posicions 3438838 i 3617512 de la cadena forward. La SELENOJ predita al nostre genoma no comença per metionina, però conserva la selenocisteïna.
El resultat del TCOFFEE, amb una score de 827, mostra un alineament molt dolent de les proteïnes; encara que el SEBLASTIAN sí que ha predit que hi ha una selenoproteïna en el Datnioides undecimradiatus. Tampoc s’ha trobat cap element SECIS.
Així doncs, la conclusió extreta amb els resultats obtinguts és que no podríem afirmar que la SELENOJ1 estiga ben anotada al nostre genoma.
(8) Selenoprotein K (SELENOK)
Selenoprotein K (SELENOK)
És la selenoproteïna que està més difosa entre organismes eucariotes. Codifica per una proteïna transmembrana del reticle endoplasmàtic i participa en la degradació de proteïnes mal plegades al reticle endoplasmàtic: reconeixement, ubiquitinació i redirecció de les proteïnes al citosol. Aquest procés es duu a terme per diverses proteïnes com SelS.
Aquesta proteïna es troba a l’scaffold WARS01000026.1 de Datnioides undecimradiatus. La selenoproteïna està formada per 5 exons i es troba en la cadena forward entre les posicions 800477-846053. La proteïna predita comença per metionina i conté una selenocisteïna al final de la seva seqüència. L’evalue de la predicció és de 6.86e-08, i l’alineament realitzat amb el T-COFFEE té un score de 984.
No hem trobat cap element SECIS en la seqüència de la selenocisteïna. SEBLASTIAN ha predit una selenoproteïna en este alineament (començant ambdues amb metionina i conservant la selenocisteïna). Podem concloure que la proteïna SELENOK es troba ben anotada al genoma.
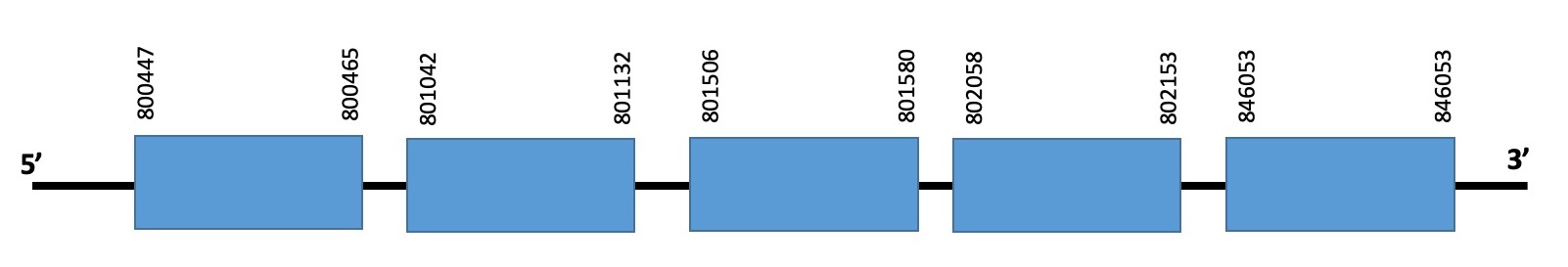
Exó de la proteïna SELENOK
(9) Selenoprotein L (SELENOL)
Selenoprotein L (SELENOL)
És una selenoproteïna que es troba únicament en peixos. Conté dues selenocisteïnes, les que li permenten formar un plegament de tipus tioredoxina (UxxU). Aquest plegament tioredoxina li confereix capacitat per intervenir en les reaccions redox.
Aquesta proteïna es troba a l’scaffold WARS01000046.1 de Datnioides undecimradiatus i està duplicada al scaffold WARS01000017.1 del mateix.
La selenoproteïna està formada per 11 exons, i es troba a la cadena forward entre les posicions 183638-347785. La proteïna predita no comença per metionina, i tant la selenoproteïna del genoma de referència com la de l’espècie estudiada contenen dues selenocisteïnes, tal i com hem comentat anteriorment.
L’evalue de la predicció és de 4.30e-08, i l’alineament realitzat amb el T-COFFEE té un score de 1000. Podem veure que la selenoproteïna de l’scaffold WARS01000046.1 està més conservada que la de l’scaffold WARS01000017.1, ja que aquesta ha perdut una gran part de l’inici de la proteïna. SEBLASTIAN sí que ha predit efectivament una selenoproteïna al scaffold WARS01000046.1. Conté un element SECIS, de categoria B.
Podem concloure que SELENOL es troba duplicada al genoma del nostre peix, però una més conservada respecte a la proteïna del Zebrafish.
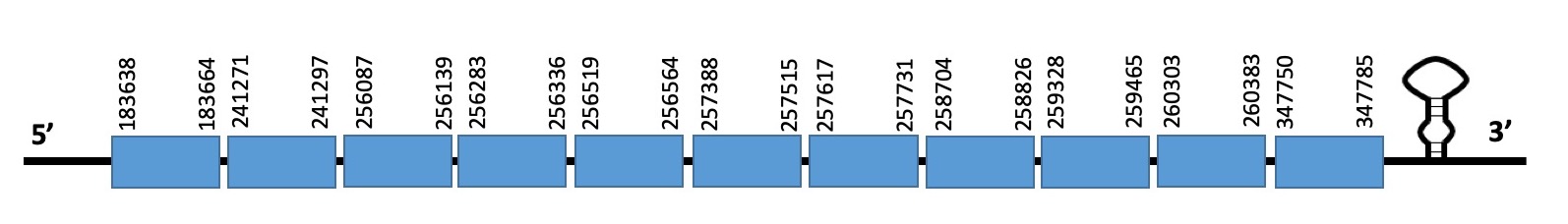
Exó de la proteïna SELENOL
(10) Selenoprotein M (SELENOM)
Selenoprotein M(SELENOM)
SELENOM és una selenoproteïna expressada principalment al cervell. És una proteïna transmembrana que participa en el plegament de proteïnes al reticle endoplasmàtic. Es desconeix la seva funció, tot i que es creu que podria tenir algun paper en malalties neurodegeneratives.
Aquesta proteïna es troba a l’scaffold WARS01000015.1 de Datnioides undecimradiatus. La selenoproteïna està formada per 6 exons i se situa a la cadena forward entre les posicions 4162416-4205559. Ni la proteïna predita ni la selenoproteïna de Zebrafish comencen per metioninacomença per metionina. La selenoproteïna conté una selenocisteïna.
L’evalue de la predicció és de 8.23e-17, i l’alineament realitzat amb el T-COFFEE té un score de 999.
No hem trobat cap element SECIS en la seqüència de la selenocisteïna. SEBLASTIAN sí que ha predit una selenoproteïna en este alineament, ambdues conservant la selenocisteïna.
L’alineament és bastant bo per tant, i podem dir que la proteïna està ben anotada.
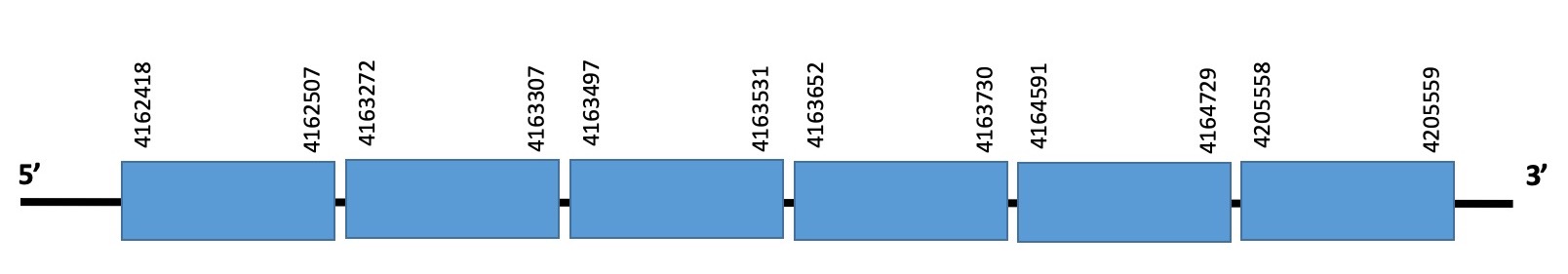
Exó de la proteïna SELENOM
(11) Selenoprotein N (SELENON)
Selenoprotein N (SELENON)
És una glicoproteïna transmembrana que es troba al reticle endoplasmàtic i que s’expressa principalment durant el desenvolupament embrionari i al múscul esquelètic. Diversos estudis han revelat que té un paper en el desenvolupament i diferenciació del teixit múscular.
Aquesta proteïna es troba a l’scaffold WARS01000017.1 de Datnioides undecimradiatus. La selenoproteïna està formada per 12 exons i es troba en la cadena reverse entre les posicions 6846247-6851566. La proteïna predita comença per metionina, i conté una selenocisteïna.
L’evalue de la predicció és de 1.36e-47, i l’alineament realitzat amb el T-COFFEE té un score de 999. SEBLASTIAN ha predit una selenoproteïna, on es conserva la selenocisteïna. Hem trobat un element SECIS de categoria B.
Podem concloure per tant, que la proteïna es troba ben anotada al genoma del peix.
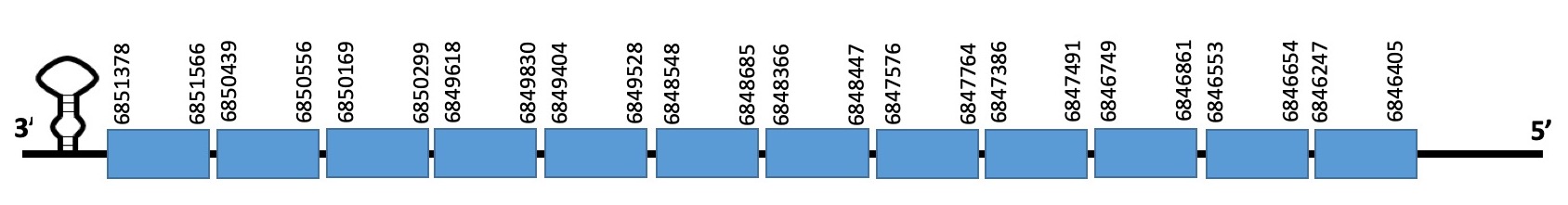
Exó de la proteïna SELENON
(12) Selenoprotein O (SELENOO)
És una proteïna que es localitza al mitocondri. Actualment, no se sap quina és la seva funció, tot i que es creu que té alguna funció en les reaccions redox.
Valorant la filogènia obtinguda, veiem que SELENOO1 de Datnioides undecimradiatus es troba proper a SELNOO1 de Zebrafish, mentre que SELENOO2 de l’espècie estudiada, veiem que es troba filogenèticament més allunyada de SELENOO2 de Zebrafish.

Arbre filogenètic de la família de proteïnes SELENOO
Selenoprotein O1 (SELENOO1)
Aquesta proteïna es troba a l’scaffold WARS01000011.1 de Datnioides undecimradiatus.
La selenoproteïna està formada per 9 exons i es troba en la cadena forward entre les posicions 3409417-3414876. La proteïna predita comença per metionina, i té una selenocisteïna al final de la seva seqüència.
L’evalue de la predicció és de 2.66e-78, i l’alineament realitzat amb el T-COFFEE té un score de 996. SEBLASTIAN ha predit una selenoproteïna, que comença amb metionina i presenta selenocisteïna. No hem trobat cap element SECIS en aquesta seqüència.
Es pot concloure que la SELENOO1 es troba ben conservada al genoma de Datnioides undecimradiatus.
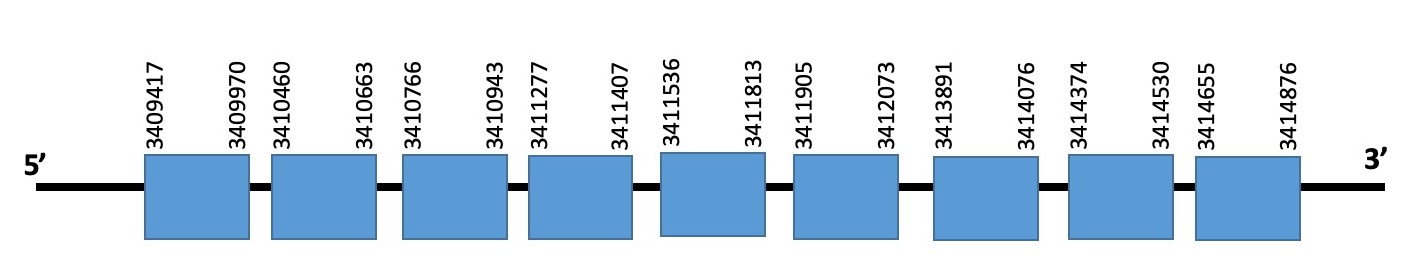
Exó de la proteïna SELENOO1
Selenoprotein O2 (SELENOO2)
El hit amb el millor e-value obtingut per SELENOO2 es troba a l’scaffold WARS01000011.1 en les mateixes posicions que la proteïna SELENOO1. D’aquesta manera, hem agafat el un hit amb un e-value molt semblant, el qual es troba a l’scaffold WARS01000311.1 de Datnioides undecimradiatus. Ja que, a més, al realitzar l’alineament amb el TCOFFEE amb el primer scaffold, la score era bastant dolenta.
A l’analitzar la proteïna veiem que hi ha hagut un fenomen d’splicing alternatiu mitjançant el qual veiem molts exons els quals es van repetint consecutivament; d’aquesta manera hem seleccionat només una d’aquestes repeticions.
La proteïna resultant està formada per 10 exons i es troba en la cadena forward entre les posicions 3375688-3414876. Ni proteïna predita ni la seqüència de referencia no comencen per metionina. A més, la proteïna resultant no conté una selenocisteïna: això és degut a que l’espècie en qüestió ha perdut alguns exons finals on es localitza la selenocisteïna al genoma de Zebrafish.
L’evalue de la predicció és de 1.10e-61, i l’alineament realitzat amb el T-COFFEE té un score de 989, però no ha alineat la majoria del final de la proteïna (es pot deure a les sèrie d’exons elegit).
No hem trobat cap element SECIS en aquesta seqüència. SEBLASTIAN ha predit una selenoproteïna.
Podem dir que la proteïna es troba bastant anotada, però que degut probablement als exons escollits, l’alineament no ha sigut del tot bo.
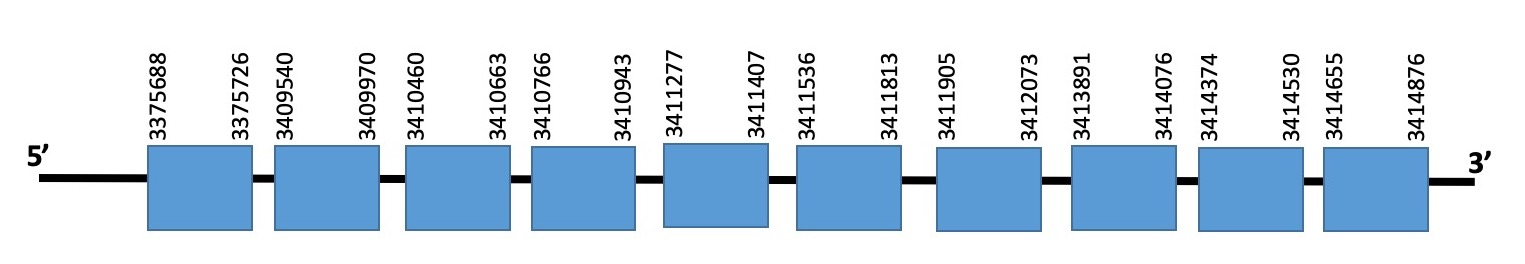
Exó de la proteïna SELENOO2
(13) Selenoprotein P (SELENOP)
És una selenoproteïna que es troba altament expressada al plasma. La proteïna resultant se secreta al plasma dels organismes, i desenvolupa un paper de donadora de seleni als teixits perifèrics, de manera que funciona com antioxidant i proteïna transportadora de seleni.
En l’arbre filogenètic obtingut porem veure que SELENOP2 de Datnioides undecimradiatus es troba pròxima a la homòloga de Zebrafish, mentre que SELENOP1 de l’espècie estudiada es troba poc conservada, i per tant es troba lluny de la seva homòloga en Zebrafish.

Arbre filogenètic de la família de proteïnes SELENOP
Selenoprotein P.1 (SELENOP.1)
AAquesta proteïna es troba a l’scaffold WARS01000030.1 de Datnioides undecimradiatus. La proteïna està formada per 12 exons i es troba en la cadena reverse entre les posicions 2525523 i 2334203. La proteïna predita no comença amb una metionina, i sí que conté una selenocisteïna.
L’evalue de la predicció és de 5.76e-26, i l’alineament realitzat amb el T-COFFEE té un score de 818. Tot i que la seqüència no comença per metionina, veiem que l’alineament és correcte. A més, SELENOBLASTIAN sí que ha trobat la selenoproteïna al nostre peix.
La seqüència conté un element SECIS de categoria B.
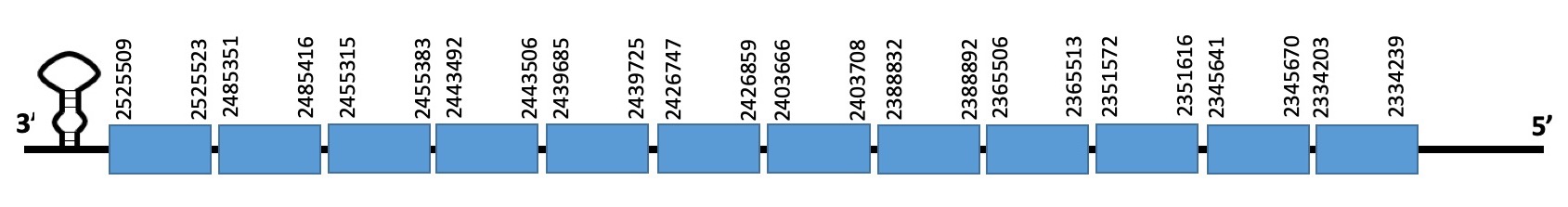
Exó de la proteïna SELENOP.1
Selenoprotein P.2 (SELENOP.2)
Amb aquesta proteïna solament es troben 2 hits al scaffold WARS01000015.1 del genoma del peix; amb un E-value de 1,04e-33. Hem pogut observar que l’alineament d’esta proteïna no és gens òptim, la qual cosa ens pot estar indicant que esta proteïna del zebrafish no es troba al genoma del Datnioides undecimradiatus.
Primerament, amb el programa exonerate predia un total de 49 exons, obtenint un resultat al T-COFFEE (score de 586) que no mostrava un alineament massa correcte. Ho vam tornar a repetir, seleccionant els exons manualment més pròxims a les posicions on s’havien trobat els hits al tblastn. Aquesta vegada, el resultat va millorar lleument, però no de forma massa significativa: un score al t-coffee de 633.
En conclusió, pels resultats obtinguts podríem dir que SELENOP.2 no es troba al genoma de Datnioides undecimradiatus, ja que l’alineament no ha sigut satisfactori.
(14) Selenoprotein S (SELENOS)
Selenoprotein S (SELENOS)
La Selenoproteïna S és una proteïna transmembrana que es troba al reticle endoplasmàtic. S’ha vist que participa en el procés de degradació de proteïnes mal plegades al reticle endoplasmàtic. Hi ha estudis que indiquen que és possible que estigui relacionada amb el control del procés inflamatori.
La selenoproteïna S es troba a l’scaffold WARS01000067.1 de Datnioides undecimradiatus. La proteïna trobada està formada per 6 exons i es troba a la cadena forward localitzant-se entre les posicions 2334345-2337323 de l’scaffold en qüestió. Veiem que la proteïna obtinguda conté una selenocisteïna al final de la seva seqüència. L’e-value de la predicció realitzada és de 5.92e-14. L’alineament realitzat ha tingut un score del T-COFFE de 993.
La proteïna SELENOS a Datnioides undecimradiatus no comença per metionina ja que a la proteïna predita li falta la primera part d’aquesta. No hem trobat cap altre hit amb un e-value estadísticament significatiu on fer la predicció, de manera que, veient l’alineament de la proteïna, hipotetitzem que Datnioides undecimradiatus ha perdut l’inici de SELENOS.
Hem trobat dos elements SECIS, essent el de categoria A el que té millor score i SEBLASTIAN ens ha predit una selenoproteïna. D’aquesta manera, confirmem la presència de SELENOS en Datnioides undecimradiatus.
Exó de la proteïna SELENOS
(15) Selenoprotein T (SELENOT)
SELENOT codifica per diverses proteïnes que es troben al reticle endoplasmàtic, on de la mateixa manera que la família SELENOR, tenen una funció important en la protecció de les neurones dopaminèrgiques front a l'estrès oxidatiu en el Parkinson, a més de controlar l’homeòstasis de la glucosa en les cèl·lules beta-pancreàtiques. Altres estudis també indiquen que aquestes proteïnes tenen un rol en la regulació de les reaccions redox als fibroblasts i la contracció del múscul llis gàstric.
És una família de selenoproteïnes que, a diferència dels mamífers, inclou 3 proteïnes: SELENOT1, SELENOT1b i SELENOT2, sent aquesta última l’única que no té proteïnes ortòlogues en altres espècies.
A l’estudiar SELENOT1 com SELENOT1b hem obtingut els mateixos dos hits significatius en els mateixos dos scaffolds i en les mateixes posicions, de manera que en un scaffold trobaríem una proteïna, i en l’altre, la segona proteïna. Per veure quina proteïna correspon a cada scaffold, hem hagut de realitzar un arbre filogenètic. Valorant-lo, hem determinat que la que es troba més propera a SELENOT1 de Zebrafish és SELENOT1 de Datnioides undecimradiatus, mentre que l’altra proteïna serà SELENOT1b.
Finalment, hem de comentar que SELENOT1b de Datnioides undecimradiatus és més propera a SELENOT2 de Zebrafish que a la pròpia SELENOT1b del mateix.

Arbre filogenètic de la família de proteïnes SELENOT
Selenoprotein T1 (SELENOT1)
Aquesta proteïna es troba al scaffold WARS01000032.1 de Datnioides undecimradiatus. SELENOT1 està formada per 5 exons i es troba a la cadena forward entre les posicions 4200995-4206282.
La predicció realitzada comença per metionina tot i que veiem que la seqüència de Datnioides undecimradiatus ha perdut 3 aminoàcids al seu inici. La proteïna conté una selenocisteïna.
L’evalue de la predicció és de 1.78e-15, i l’alineament realitzat amb el T-COFFEE té un score de 976. Veiem que la proteïna predita està sencera i molt conservada.
Hem trobat dos elements SECIS de categoria B en l’scaffold WARS01000032.1 i SEBLASTIAN ens ha predit una selenoproteïna. D’aquesta manera, afirmem que SELENOT1 es troba ben anotada en l’espècie estudiada.
Exó de la proteïna SELENOT1
Selenoprotein T1b (SELENOT1b)
Com hem comentat anteriorment, el hit amb millor e-value per SELENOT1b es troba a l’scaffold WARS01000032.1 en les mateixes posicions que SELENOT1, fet que es deu a la semblança entre ambdues proteïnes. D’aquesta manera, tal i com ha succeït amb la família de SELENOO, triarem el segon hit amb millor e-value, el qual és WARS01002953.1 de Datnioides undecimradiatus. Està formada per 6 exons i se situa a la cadena reverse entre les posicions 109393-160362. La proteïna predita no comença per metionina i conté una selenocisteïna.
L’evalue de la predicció és de 6.88e-15, i l’alineament realitzat amb el T-COFFEE té un score de 987.
Hem trobat un elements SECIS de categoria B tant en ambdues seqüències. SEBLASTIAN ens ha predit una selenoproteïna en la seqüència analitzada, de manera que amb els resultats obtinguts concloem que hem SELENOT1b es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna SELENOT1b
Selenoprotein T2 (SELENOT2)
La selenoproteïna es troba a l’scaffold WARS01002953.1 de Datnioides undecimradiatus. Aquesta està constituida per 6 exons, i es localitza a la cadena reverse entre les posicions 1327267-138893. La proteïna predita comença amb una metionina, i conté una selenocisteïna.
L’evalue de la predicció és de 6.92e-33, i l’alineament realitzat amb el T-COFFEE té un score de 993. Veiem que la seqüència de la selenoproteïna esta íntegra.
Hem trobat dos elements SECIS de categoria B i SEBLASTIAN ens ha predit una selenoproteïna. D’aquesta manera, SELENOT2 es troba ben conservada en Datnioides undecimradiatus.
Exó de la proteïna SELENOT2
(16) Selenoprotein U (SELENOU)
Es una família de proteïnes amb un paper en les reaccions redox. Encara que la seva funció no és coneguda del cert, estudis recents indiquen el seu paper en el la supervivència i funcionament de les cèl·lules de Sertoli.

Arbre filogenètic de la família de proteïnes SELENOU
Selenoprotein U1a (SELENOU1a)
La selenoproteïna es troba a l’scaffold WARS01000009.1 de Datnioides undecimradiatus. Aquesta proteïna es troba comunament duplicada en diferents espècies de peixos, doncs al valorar la proteïna predita, hem observat es troba duplicada, trobant-se una còpia consecutiva a l’altra.
La primera còpia està formada per 5 exons i la segona per 8. Es localitza a la cadena reverse entre les posicions 8949328-8951386 (1a còpia) i 8955515-9051891 (2a còpia). Cap de les dues còpies ni la proteïna a Zebrafish comença amb una metionina. La proteïna predit conté selenocisteïna.
L’evalue de la predicció és de 3.74e-31, i l’alineament realitzat amb el T-COFFEE té un score de 958. En aquest as, veiem que tenim alineada només una part de la proteïna: això és degut que la duplicació consecutiva es troba únicament en Datnioides undecimradiatus i no a l’espècie de referència.
No hem trobat cap element SECIS en la seqüència estudiada, però SEBLASTIAN si que ens ha predit una selenoproteïna en la seqüència. Tot i no haver trobat elements SECIS, afirmem que SELENOU1a es troba ben anotada al genoma de l’espècie estudiada.
Exó de la proteïna SELENOU1a
Selenoprotein U2 (SELENOU2)
La selenoproteïna es troba a l’scaffold WARS01000015.1 de Datnioides undecimradiatus. Està formada per 6 exons, i es troba a la cadena forward entre les posicions 8121923-8123910. Ni la proteïna predita ni SELENOU2 de Zebrafish comencen per metionina. Tampoc cap de les dues conté selenocisteïna, aminoàcid el qual ha estat substituït per una cisteïna.
L’evalue de la predicció és de 3.66e-33, i l’alineament realitzat amb el T-COFFEE té un score de 994. Veiem que la predicció tot i tenir un alt score, no resulta idònia especialment en les parts inicials de la selenoproteïna .
No hem trobat cap element SECIS ni SEBLASTIAN ens ha predit una selenoproteïna en la seqüència estudiada.
Veient els resultats, concloem que SELENOU2 es troba al genoma de Datnioides undecimradiatus, tot i que no es troba gaire conservada respecte la selenoproteïna de Danio rerio.
Exó de la proteïna SELENOU2
Selenoprotein U3 (SELENOU3)
La selenoproteïna es troba a l’scaffold WARS01000013.1 de Datnioides undecimradiatus. Aquesta està constituida per 6 exons, i es troba a la cadena forward entre les posicions 6362436-6368790.
Veiem que ni la proteïna predita ni la del genoma de referència comencen per metionina; ambdues començen amb una valina; a més, cap de les dues conté una selenocisteïna.
L’evalue de la predicció és de 8.81e-31, i l’alineament realitzat amb el T-COFFEE té un score de 1000. En aquest cas, l’alineament té un SCORE idòni i veiem que la proteïna es troba molt conservada.
Hem trobat un element SECIS de categoria C en la seqüència estudiada, però SEBLASTIAN no ens ha predit una selenoproteïna en la seqüència analitzada.
Analitzant els resultats, concloem que SELENOU3 es troba ben anotada al genoma de Datnioides undecimradiatus.
Exó de la proteïna SELENOU3
(17) Selenoprotein W (SELENOW)
Es una família de proteïnes que s’expressa principalment al múscul esquelètic, al cor i al cervell, on participa a les reaccions redox. Específicament, intervé al creixement i diferenciació muscular i a la protecció neuronal front l’estrès oxidatiu durant el seu desenvolupament. SELENOW al genoma de Danio rerio inclou SELENOW1, SELENOW2 i SELENOW3. Mitjançant la metodologia utilitzada, hem vist que Datnioides undecimradiatus només posseeix SELENOW1.
Selenoprotein W.1 (SELENOW)
La selenoproteïna es troba a l’scaffold WARS01000035.1 de Datnioides undecimradiatus. La proteïna està formada per 4 exons, localitzant-se a la cadena forward entre les posicions i es troba a la cadena reverse entre les posicions 8408045-8408794. La proteïna predita comença amb una metionina, i conté una selenocisteïna cap a l’inici de la seva seqüència.
L’evalue de la predicció és de 3.23e-14, i l’alineament realitzat amb el T-COFFEE té un score de 1000. Podem dir doncs, que la predicció de la proteïna ha estat correcta.
Hem trobat un element SECIS de categoria B però mitjançant SEBLASTIAN no hem predit cap selenoproteïna en la seqüència estudiada. D’aquesta manera, concloem que SELENOW està ben anotada al genoma de Datnioides undecimradiatus.
Exó de la proteïna SELENOW
(18) Thioredoxin reductase (TXNRD)
La família de selenoproteïnes Thioredoxin reductasa són proteïnes que funcionen com a homodímers, i catalitzen la reducció del grup tioredoxina. S’ha vist que, a diferència d’altres espècies, en el genoma dels peixos només hi trobem dues formes. TXNRD codifiquen per proteïnes localitzades al mitocondri, la funció de les quals és la captació de ROS (Reactive oxygen species) al mitocòndri, de manera que tenen funció antioxidant.

Arbre filogenètic de la família de proteïnes TXNRD
Thioredoxin reductase 2 (TXNRD2)
La selenoproteïna es troba a l’scaffold WARS01000015.1 de atnioides undecimradiatus. Aquesta està constituida per 16 exons, i es troba a la cadena forward entre les posicions 8121923-8123910.
Veiem que ni la proteïna predita ni la del genoma de referència comencen per metionina; ambdues començen amb àcid aspàrtic. Veiem que la protïna conté una selenocisteïna al final de la seva seqûencà.
L’evalue de la predicció és de 5.28e-33, i l’alineament realitzat amb el T-COFFEE té un score de 1000. En aquest cas, l’alineament té un SCORE idòni, però ni la nostra proteïna ni la de Zebrafish comencen per metionina.
No hem trobat cap element SECIS en la seqüència estudiada, però sí que hem predit una selenoproteïna mitjançant SEBLASTIAN. Amb tot això, afirmem que TXNRD2 està ben anotat al genoma de l’espècie estudiada.
Exó de la proteïna TXNRD2
Thioredoxin reductase 3 (TXNRD3)
La selenoproteïna es troba a l’scaffold WARS01000013.1 de Datnioides undecimradiatus. Aquesta està constituida per 17 exons, i es troba a la cadena reverse entre les posicions 19075510-19087042. TXNRD3 conté una metionina com a primer aminoàcid, i té una selenocisteïna al final de la seva seqüència. L’evalue de la predicció és de 3.87e-38, i l’alineament realitzat amb el T-COFFEE té un score de 999.
Veiem molta homologia entre les seqüències de les proteïnes TXNRD2 i TXNRD3, sent TXNRD3 la única que comença per metionina. A més, TXNRD2 té 16 exons mentre que TXNRD3 en té 17. Valorant els resultats obtinguts, tot això ens fa pensar que TXNRD2 ha perdut l’exó inicial, el qual es troba conservat en TXNRD3.
No hem trobat cap element SECIS, tot i que hem predit una selenoproteïna mitjançant SEBLASTIAN. Amb aquests resultats, afirmem que TXNRD2 està ben anotada al genoma de l’espècie estudiada.
Exó de la proteïna TXNRD3
MAQUINÀRIA IMPLICADA EN LA SEVA SÍNTESI
(1) Eukaryotic elongation factor (eEFsec)
Eukaryotic elongation factor (eEFsec)
Aquest factor d’elongació és l’encarregat de reclutar tRNA. Forma un complex amb SBP2, el qual està mediat per un element SECIS, i s’encarreguen d’insertar Sec a les cadenes de les proteïnes en resposta als codons UGA. La proteïna eEFsec té una estructura en forma de calze composta per quatre dominis. Els dominis I,II i III formen la copa del calze i tenen una alta homologia amb altres factors d’elongació. Per altra banda, el domini IV format per una extensió COOH-terminal única, està implicat en les interaccions amb SBP2. Conté cisteïna en comptes de selenocisteïna.
La proteïna eEFsec es troba a l’scaffold WARS01000013.1 de Datnioides undercimradiatus. Conté 7 exons, es troba entre les posicions 10176101 i 10184649, i la cadena es reverse. El principi de la proteïna predita s’ha perdut, i per tant la proteïna comença per treonina. No conté cap selenocisteïna, però la proteïna del Zebrafish tampoc en té, tenen una cisteïna. Hem obtingut un e-value de 9.88e-90, i un score de 988. La proteïna està ben predita però hi ha parts que no estan bé del tot. Té dos elements SECIS, escollirem el que té un grau major, en aquest cas B.
El SEBLASTIAN no ens ha predit cap selenoproteïna, tot i així podem concloure que eEFsec es troba al genoma de Datnioides undecimradiatus, basant-nos en els resultats obtinguts al TCOFFEE.
Exó de la proteïna eEFsec
(2) Methionine sulfoxide reductase A (MsrA)
La selenoproteïna MsrA és un enzim depenent de tiol que catalitza la conversió de sulfòxid de metionina a metionina. En aquest cas s’encarreguen de la reducció dels residus de metionina-S-sulfòxid en proteïnes, i a més és capaç de reduir la metionina-S-sulfòxid lliure. Contenen un residu Cys en lloc de Sec al seu lloc actiu. Aquestes proteïnes estan implicades en la protecció de les proteïnes cel·lulars front l’estrès oxidatiu, i mitjançant aquesta funció poden regular l'esperança de vida en diversos organismes model.
En aquest cas hem obtingut tres scaffolds significatius, ja que tenen tots un e-value i un score molt semblant. Hem escollit l’scaffold WARS01000017.1 de Datnioides undecimradiatus, perquè és el que es troba més a prop filogenèticament de la proteïna de Zebrafish segons l’arbre, per tant el més similar. La selenoproteïna està formada per set exons i es troba a la cadena reverse, entre les posicions 7496821 - 7602197. Comença per leucina, però ha perdut els 9 primers aminoàcids, tot i així al Zebrafish tampoc comença per metionina. No conté cap selenocisteïna i al Zebrafish tampoc, per tant és homòloga amb Cys. Hem obtingut un e-value de 8.45e-16 i un score a l’alineament de 999, per tant la proteïna s’ha predit completamet. Té un element SECIS de grau B.
SEBLASTIAN no ha predit la selenoproteïna, però tot i així considerem que a partir dels resultats del TCOFFEE la proteïna MsrA es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna MsrA
El que observem als altres scaffolds és una duplicació de la proteïna en dos scaffolds diferents. La seqüència dels scaffolds WARS01002753.1 i WARS01002804.1 és idéntica excepte en els primers 20 aminoàcids, el quals difereixen. Com veiem a la filogènia la duplicació estarà menys conservada respecte la proteïna original, degut a molts canvis d’aminoàcids.

Arbre filogenètic de MsrA
Enllaç per a l'arbre filogenètic de MsrA intermedi
(3) Phosphoseryl-tRNA kinase (PSTK)
Phosphoseryl-tRNA kinase (PSTK)
Aquesta proteïna és una quinasa que fosforila la seril-tRNA(Sec) a O-fosfoseril-tRNA(Sec), el qual és un substrat intermediari per la proteïna SecS, proteïna necessària per la biosíntesis de la selenocisteïna.
La proteïna es troba a l’scaffold WARS01000012.1 de Datnioides undecimradiatus. Està formada per 3 exons i es troba en la cadena forward entre les posicions 1671562 i 1672521. Ni la proteïna predita ni la proteïna de Zebrafish comencen per metionina ni contenen selenocisteïna.
L’evalue de la predicció és de 2.97e-11, i l’alineament realitzat amb el T-COFFEE té un score de 954. En l’alineament veiem que la proteïna estudiada ha perdut part de la seqüència a l’inici i al final d’aquesta. Veiem, també, que la part de la proteïna que sí que està alineada, tot i tenir un score correcte, no està gaire conservada.
Hem trobat un elements SECIS de categoria B en la seqüència analitzada. Però SEBLASTIAN no ens ha predit cap selenoproteïna en la seqüència estudiada. Concloent, podem dir que aquesta proteïna es troba massa ben anotada al genoma de l’organisme estudiat.
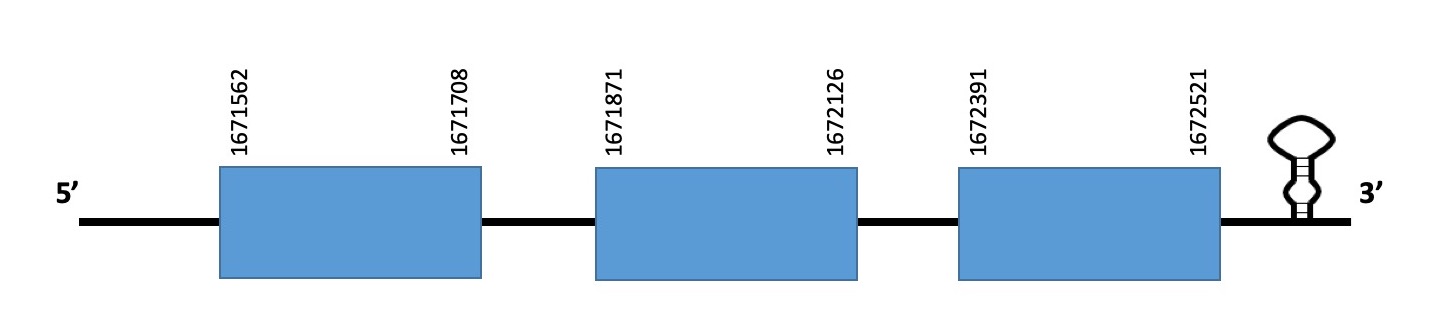
Exó de la proteïna PSTK
(4) SECIS binding protein 2 (SBP2)
Es una proteïna que conté tres dominis amb diferents funcions. Mentre la funció del domini N-terminal no ha estat dilucidat, conté un domini central d’unió específica a elements SECIS i un domini C-terminal d’unió al RNA. Aquesta proteïna és un factor limitant en la síntesis de selenoproteïnes ja que el procés requereix la presència d’elements SECIS, de manera que quan SEBP2 està absent, hi ha una disminució de l’expressió de les diferents selenoproteïnes.
Aquesta proteïna es troba a l’scaffold WARS01000015.1 de Datnioides undecimradiatus. Està formada per 10 exons i es localitza en la cadena forward entre les posicions 11386269-11425710. Ni la proteïna predita ni la selenoproteïna de Zebrafish comencen per metionina. A més, no conté una selenocisteïna.
L’evalue de la predicció és de 3.22e-29, i l’alineament realitzat amb el T-COFFEE té un score de 979. Veiem que l’alineament és molt acurat en l’inici i el final de la proteïna, mentre que certs aminoàcids de la part central de la proteïna, veiem que estan poc conservats.
No hem trobat elements SECIS en la seqüència analitzada. I SEBLASTIAN tampoc ens ha predit cap selenoproteïna en la nostra seqüència. Però, així i tot, podem dir que l’alineament de la SBP2 del Zebrafish i del nostre genoma és bastant bo.
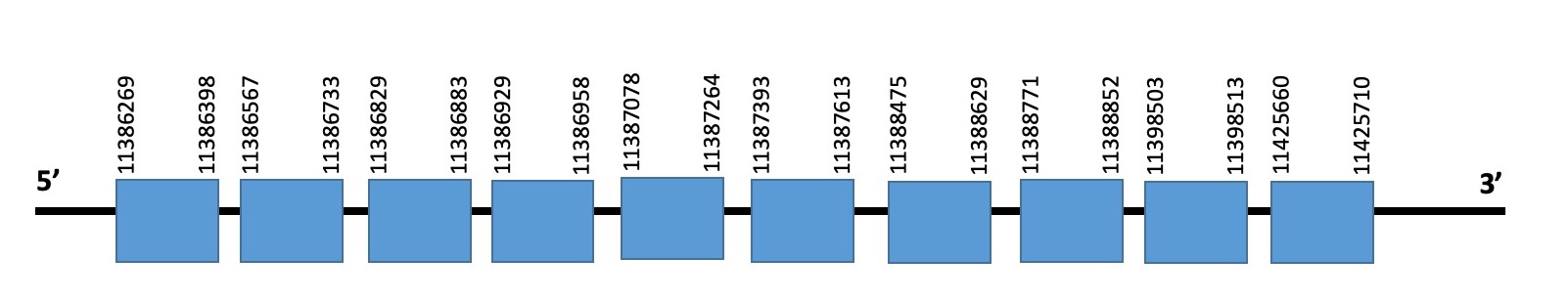
Exó de la proteïna SBP2
(5) Selenocysteine synthase (SecS)
Selenocysteine synthase (SecS)
SecS és un enzim que intervé en la biosíntesis de la selenocisteïna incorporant un selenofosfat all tRNA[ser]Sec, transformant-lo a selenocisteil-tRNA (Sec-tRNA).
Es troba a l’scaffold WARS01000065.1 de Datnioides undecimradiatus. A l’analitzar la proteïna veiem que hi ha hagut un fenomen de splicing alternatiu mitjançant el qual veiem molts exons els quals es van repetint consecutivament; d’aquesta manera hem seleccionat només una d’aquestes repeticions.
La proteïna conté 12 exons i es troba en la cadena forward entre les posicions 2714060 i 2860502. La proteïna predita comença per metionina i no conté una selenocisteïna.
L’evalue de la predicció és de 6.61e-41, i l’alineament realitzat amb el T-COFFEE té un score de 971. Degut al fenomen de splicing alternatiu veiem que l’alineament realitzat no és tant idoni com si només haguessim alineat una repetició, ja que veiem que al principi i al final de la seqüència hi ha una part de la nostra proteïna que no està alineada
No hem trobat elements SECIS en la seqüència analitzada, ni SEBLASTIAN ens ha predit cap selenoproteïna en esta seqüència. Per això podem concloure que, encara que observem que l’alineament no és complet, sí que podríem dir que la proteïna SecS es troba al genoma de Datnioides undecimradiatus.
Exó de la proteïna SecS
(6) Selenophosphate synthetase (SEPHS)
Es una proteïna encarregada de la síntesis del selenofosfat, el qual és el donador de seleni per la síntesis de la selenocisteïna. S’ha determinat que té dues isoformes: SEPHS1 i SEPHS2. És la isoforma 2 la que és capaç de sintetitzar selenofosfat de novo, mentre que la primera només ho pot fer reciclant productes generats anteriorment.
A l’analitzar els diferents resultats de SEPHS I SEPHS2, hem vist que ocupaven la mateixa posició en un mateix scaffold (WARS01000011.1), amb la diferència que que SEPHS té 9 exons, SEPHS2 en té 12. D’aquesta manera, al comparar seqüències i posicions, determinem que a Datnioides undecimradiatus només trobem una forma.
Veient l’alineament, SEPHS de Zebrafish és la proteïna que es troba més a prop d’ambdues SEPHS i SEPHS2 de Datnioides undecimradiatus. És per això, que concloem que la proteïna que es troba al genoma de l’espècie en qüestió és SEPHS.
Tot i això, les presentarem com dues proteïnes diferents (on la millor predicció està a SEPHS), encara que en la nostra espècie nomès en trobem una.

Arbre filogenètic de la família de proteïnes SEPHS
Selenophosphate synthetase (SEPHS)
Aquesta proteïna es troba a l’scaffold WARS01000011.1 de Datnioides undecimradiatus. La selenoproteïna està formada per 9 exons i es localitza en la cadena forward entre les posicions 70077014-7081119. La proteïna predita comença per metionina i ni la proteïna predita ni la de Zebrafish contenen una selenocisteïna, la qual per tant ha estat substituïda per una cisteïna.
L’evalue de la predicció és de 3.18e-50, i l’alineament realitzat amb el T-COFFEE té un score de 1000. Veiem que la selenoproteïna predita està gairebé totalment conservada respecte la selenoproteïna de Zebrafish.
No hem trobat elements SECIS en la seqüència analitzada. I tampoc SEBLASTIAN ha predit cap selenoproteïna. Malgrat això, la conclusió és bastant favorable a què SEPHS es troba ben anotada al genoma del nostre organisme.
Exó de la proteïna SEPHS
Selenophosphate synthetase 2 (SEPHS2)
La selenoproteïna es troba a l’scaffold WARS01000011.1 de Datnioides undecimradiatus. Aquesta està constituïda per 12 exons, i es troba a la cadena forward entre les posicions 7077026- 7172052.
La proteïna predita no comença per metionina ja que no conté part dels aminoàcids inicials de la proteïna de Zebrafish, fet que es pot deure a que hagi perdut algun exó respecte l’espècie de referència. La proteïna predita no conté selenocisteïna, encara que la del Zebrafish sí.
L’evalue de la predicció és de 1.33e-39, i l’alineament realitzat amb el T-COFFEE té un score de 996. Tal com hem dit, la proteïna ha perdut part de la seqüència al seu inici i veiem que també ha perdut una petita part de la seqüència cap al final d’aquesta.
Tot i que la proteïna predita no conté selenocisteïna, hem trobat dos elements SECIS de categoria B. SEBLASTIAN no ha predit cap selenoproteïna en este genoma. Però, a pesar de què haja perdut algunes parts al nostre organisme, la proteïna està significativament ben anotada.
Exó de la proteïna SEPHS2
(7) Selenoprotein R (MSRB)
Es una família de selenoproteïnes, les quals es troben al reticle endoplasmatic. Diversos estudis indiquen que tenen una funció crucial en la protecció de les neurones dopaminèrgiques de l'estrès oxidatiu en la malaltia del Parquinson. A més, s’ha vist que tenen un paper important en el control de l’homeostatsis de la glucosa a les cèl·lules beta-pancreàtiques.
Les selenoproteinR compren 4 proteïnes: MSRB1a, MSRB1b, MSRB2 i MSRB3. A Datnioides undecimradiatus trobem 5 selenoproteïnes R ja que té una duplicació a MSRB1b.
Mitjançant la filogènia hem pogut determinar quina de les duplicacions de la proteïna MRSB1b és més propera a la proteïna de Zebrafish. A més, podem observar que la selenoproteïna MSRB1a de Zebrafish és més propera a MRSB1b de Datnioides undecimradiatus que la pròpia MRSB1a de Datnioides undecimradiatus.
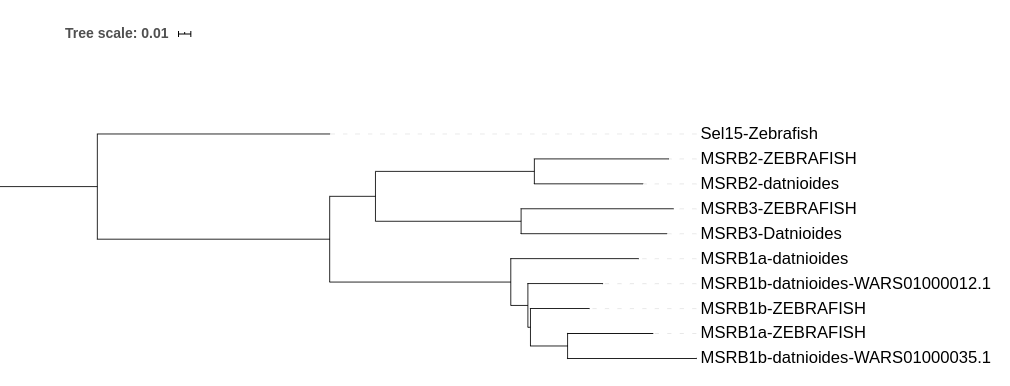
Arbre filogenètic de la família de proteïnes MRSB
Methionine-R-sufoxide reductase 1a (MSRB1a)
Aquesta proteïna es troba a l’scaffold WARS01000035.1 de Datnioides undecimradiatus. Aquesta està constituida per 8 exons, i se situa a la cadena forward entre les posicions 1623758 i 1720240. La proteïna predita comença amb una metionina, i conté una selenocisteïna.
L’evalue de la predicció és de 3.42e-38, i l’alineament realitzat amb el T-COFFEE té un score de 956. Podem veure que l’alineament és correcte però el final d’aquest tenim que la proteïna predita té una seqüència més llarga que la proteïna del genoma de referència; aquest fet es pot deure a que la nostra espècie, o té un exó més, o que el nostre programa hagi agafat erròniament un tros de seqüència quan no ho és.
Hem trobat un element SECIS de categoria B. SELENOBLASTIAN també ha trobat una selenoproteïna, que quadra amb la part alineada al TCOFFEE.
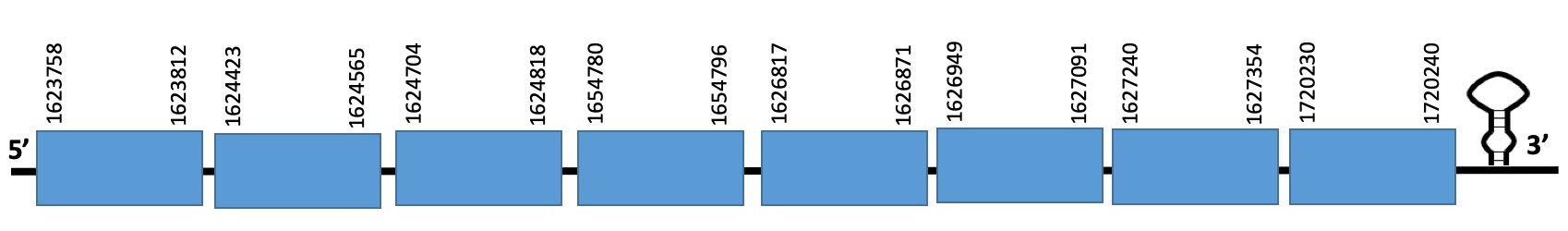
Exó de la proteïna MSRB1a
Methionine-R-sufoxide reductase 1b (MSRB1b)
Aquesta proteïna es troba duplicada a Datnioides undecimradiatus, als scaffolds WARS01000012.1 i WARS01000035.1. Després d’haver realitzar la filogènia, podem veure que la més propera a la selenoproteïna de Zebrafish és la proteïna present en l’scaffold WARS01000035.1.
La proteïna està formada per tres exons, i es troba en la cadena forward entre les posicions 9183614-9184757. La selenoproteïna predita comença per metionina i conté una selenocisteïna.
Pel que fa a la predicció realitzada, hem obtingut un e-value d’1.30e-35. L’alineament obtingut al T-COFFEE té un score de 1000, on veiem que la proteïna predita està sencera.
Hem trobat un element SECIS de categoria B en cadascuna de les dues proteïnes dels diferents scaffolds i SEBLASTIAN ens ha predit una selenoproteïna en cadascun dels dos scaffolds. Finalment, afirmem que MRSB1b està ben anotada al genoma de l’espècie estudiada.
Exó de la proteïna MSRB1b
Methionine-R-sufoxide reductase 2 (MSRB2)
Aquesta proteïna es troba a l’scaffold WARS01000010.1 de Datnioides undecimradiatus. La proteïna està formada per 5 exons, i es localitza en la cadena reverse entre les posicions 1982092-1983890. La selenoproteïna predita comença per metionina i ni la proteïna de Zebrafish ni la de Datnioides undecimradiatus conté cap selenocisteïna, la qual ha estat substituida per una cisteÏna.
Pel que fa a la predicció realitzada, hem obtingut un e-value de 2.74e-20. L’alineament obtingut al T-COFFEE té un score de 998, on veiem que la proteïna predita està sencera.
Hem trobat un element SECIS de categoria B en la seqüència estudiada i SEBLASTIAN no ens ha predit cap selenoproteïna en la seqüència estudiada. Malgrat això, determinem que MSRB2 es troba ben anotada al genoma de Datnioides undecimradiatus.
Exó de la proteïna MSRB2
Methionine-R-sufoxide reductase 3 (MSRB3)
Aquesta proteïna es troba a l’scaffold WARS01003208.1 de Datnioides undecimradiatus. La proteïna està formada per 5 exons, i es troba en la cadena forward entre les posicions 200180-204404. La selenoproteïna predita comença per metionina i ni la proteïna de Danio rerio ni la de l’espècie estudiada contenen cap selenocisteïnaM la qual ha estat substituida per una cisteïna.
Pel que fa a la predicció realitzada, hem obtingut un e-value de 3.65e-14. L’alineament obtingut al T-COFFEE té un score de 986, on veiem que la proteïna predita està sencera
Hem trobat dos elements SECIS, sent el de categoria A el que té millor score, i SEBLASTIAN no ens ha predit cap selenoproteïna en la seqüència estudiada.
Amb els resultats exposats, concloem que MRSB3 es troba ben anotada al genoma de l’espècie estudiada.
Exó de la proteïna MSRB3
(8) tRNA Sec 1 associated protein 1 (SECp43)
És família de proteïnes relacionades amb els primers passos de síntesis de la selenocisteïna: té un paper important en la incorporació de la selenocisteïna a les selenoproteïnes estabilitzant les proteïnes SECISBP2, EEFSEC i el complex tRNA. Altres estudis la relacionen amb la metilació del tRNA.

Arbre filogenètic de la família de proteïnes SECp43
tRNA Sec 1 associated protein 1.1 (SECp43.1)
LLa selenoproteïna es troba a l’scaffold WARS01000050.1 de Datnioides undecimradiatus. Aquesta està constituïda per 7 exons, i es troba a la cadena forward entre les posicions 1565141-1648812.
La proteïna obtinguda comença per metionina, i no conté cap selenocisteïna en la seva seqúència (doncs la seva funció és introduir selenocisteïnes a les selenoproteïnes).
L’evalue de la predicció és de 1.28e-15, i l’alineament realitzat amb el T-COFFEE té un score de 1000.
No hem trobat cap element SECIS ni selenoproteïna mitjançant SEBLASTIAN en la seqüència estudiada. Tot i això, veient els resultats, concloem que SECp43.1 està ben anotada al genoma de Datnioides undecimradiatus.
Exó de la proteïna SECp43.1
tRNA Sec 1 associated protein 1.2 (SECp43.2)
L’scaffold es troba aquesta proteïna és WARS01000085.1. A l’analitzar la proteïna veiem que hi ha hagut un fenomen d’splicing alternatiu mitjançant el qual veiem molts exons els quals es van repetint consecutivament; d’aquesta manera hem seleccionat només una d’aquestes repeticions. La proteïna resultant està formada per 10 exons i es localitza en la cadena forward entre les posicions 196412-341584.
La proteïna comença per metionina i en la regió alineada no hi ha selenocisteïna. L’e-value de la predicció és de 8.42e-13 i l’alineament té un score de 975. Degut al fenomen d’splicing que ha ocorregut en aquesta proteïna, veiem que l’alineament no és òptim, ja que conté altres exons que no conté la selenocisteïna de Zebrafish.
Hem trobat un element SECIS de categoria C però SEBLASTIAN no ens ha predit cap selenoproteïna.
D’aquesta manera, concloem que SECp43.2 està ben anotada al genoma de Datnioides undecimradiatus.
Exó de la proteïna SECp43.2
Conclusió

L’objectiu d’aquest treball ha estat predir, anotar i caracteritzar les selenoproteïnes, proteïnes homòlogues que contenen cisteïna i la maquinària encarregades de la síntesis d’aquestes, al genoma d’una espècie recentment seqüenciada:
Datnioides undecimradiatus. Això s’ha fet mitjançant la comparació de les proteïnes anotades al genoma de Danio rerio, mitjançant una sèrie de programes que ens han permès alinear proteïnes i generar arbres filogenètics, entre d’altres.
Un cop obtinguts els resultats, afirmem que hem pogut trobar i anotar correctament les següents proteïnes del genoma de Danio rerio a Datnioides undecimradiatus:
·Selenoproteïnes: Sel15, SELENOE, GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, DIO1, DIO2, DIO3a, DIO3b, SEPHS2, SELENOH, SELENOK, SELENOL, SELENOM, SELENON, SELENOO1, SELENOO2, SELENOP1, SELENOP2, MSRB1a, MSRB1b, SELENOS, SELENOT1, SELENOT1b, SELENOT2, SELENOU1a, SELENOW1, SEPHS2, TXNRD2 i TXNRD3.
·Proteïnes homòlogues que contenen cisteïna: GPx7, GPx8, MrsA, MRSB2, MRSB3, SELENOU2, SELENOU3 i SEPHS1.
·Maquinària encarregada de la síntesis de Sec: eEFsec, PSTK, SBP2, SecS SECp43.1 i SECp43.2.
·Selenoproteïnes no presents: SELENOW2 i SELENOW3.
·Selenoproteïnes no anotades correctament: SELENOI, SELENOJ i SELENOP2.
No hem aconseguit anotar correctament les proteïnes SELENOI i SELNOJ. En ambdues, tot i haver obtingut uns bons e-value i un bon score al TCOFFEE, quan ens fixem en l’alineament generat, veiem que té moltes espais no alineats al llarg de tota la seqüència i sovint, els aminoàcids alineats no es corresponen als Danio rerio. És per això que amb el programa utilitzat, no hem pogut predir aquestes dues selenoproteïnes en l’espècie estudiada.
Amb aquests resultats, concloem que el selenoproteoma de Datnioides undecimradiatus es troba ben conservat respecte l’espècie de referència Danio rerio.
Sovint ens hem trobat amb proteïnes que no comencen per metionina. Algunes d’aquestes tampoc comencen per metionina en proteïnes de Danio rerio, però d’altres sí; aquest fet l’atribuim en part a la pèrdua de part dels aminoàcids inicials en la proteïna.
A més, com podem veure, l’espècie estudiada, Datnioides undecimradiatus, conté un gran nombre de selenoproteïnes, les quals moltes vegades es troben duplicades. Això es deu a que el genoma dels peixos es va duplicar després de divergir evolutivament dels mamífers, de manera que tot i que moltes duplicacions s’han anat perdent, algunes s’han mantingut. Així doncs, certes selenoproteïnes s’han duplicades en aquests, fet que explica que els peixos siguin les espècies amb el major nombre de selenoproteïnes.
Una de les limitacions d’aquest treball ha estat que, sovint ens hem trobat amb que certes proteïnes de la mateixa família, tenien els mateixos hits en els mateixos scaffolds i posicions compartides, de manera que hem realitzat una aproximació basada en els arbres filogenètics generats. Així doncs, hem assignat els hits a les diferents proteïnes basant-nos en la seva proximitat a l’arbre filogenètic. Aquesta aproximació no ens assegura totalment que el hit triat correspongui a la proteïna assignada, però amb les dades amb les que comptem, creiem que és la millor manera de fer-ho.
Ens hem trobat també, amb certes proteïnes que contenen elements SECIS però no selenocisteïna, fet que hem associat a modificacions de la seqüència de nucleòtids de la proteïna, deguts, per exemple, a mutacions espontànies. D’altra banda, certes proteïnes contenen selenocisteïna però no hem trobat elements SECIS. Aquest fet pot ser degut a que el programa utilitzat per trobar aquests elements no hagi inclós en el fitxer de l’input la seqüència de l’element SECIS, o que el programa no ens l’hagi trobat perquè aquesta part de la seqüència no ha estat conservada en l’espècie estudiada.
D’altra banda, hem vist que el programa SEBLASTIAN no ens ha predit algunes selenoproteïnes que nosaltres sí que havíem predit. Això es deu a que, per anotar les proteïnes, SEBLASTIAN compara la seqüència a estudiar amb una base de dades amb informació diferent a la que nosaltres fem servir per comparar, de manera que la query que utilitza no és la mateixa que la nostra. Així doncs, el fet que haguem predit una selenoproteïna mitjançant el nostre programa, primarà respecte els resultats del SEBLASTIAN, que en tot cas ens serviran per confirmar la nostra predicció.
Finalment, exposem una limitació relacionada amb el programa realitzat: al fer el nostre programa, per tal d’obtenir una predicció el més acurada possible i escombrar tots els possibles hits a la vegada, el programa subseq ha inclòs 100.000 nucleòtids per davant de la posició d’inici de la selenoproteïna obtinguda al tBLASTn, i una longitud total de 200.000 nucleòtids. Això, en un inici, ens ha beneficiat ja que ens ha permès estudiar els diferents hits a la vegada.
Malgrat tot, a l’hora de buscar els elements SECIS i fer la predicció mitjançant el SEBLASTIAN hem vist que el fitxer, superava la mida máxima permesa, de manera que hem hagut de realitzar diferents programes que ens obtinguessin un subseq més petit, un per trobar elements SECIS i un altre per predir la selenoproteïna al SEBLASTIAN. Creiem que si en comptes d’haver agafat 100.000 nucleòtids per davant de la posició indicada al tBLASTn, n’haguéssim agafat menys i haguéssim posat una longitud del fitxer de 120.000 nucleòtids, no hauria estat necessari fer els diferents programes ja que el primer subseq hauria resultat òptim per realitzar els diferents passos.
REFERÈNCIES
Mix H, Lobanov AV, Gladyshev VN. SECIS elements in the coding regions of selenoprotein transcripts are functional in higher eukaryotes. 2007; 35 (2): 414-423.
Sattar H et al. Selenoprotein-U (SelU) knockdown triggers autophagy through PI3K-Akt-mTOR pathway inhibition in rooster Sertoli cells. Metallomics. 2018; 10 (7): 929-940.
Byung CL, Dikiy A, Kim HY, Gladyshev VN. Functions and evolution of selenoprotein Methionine Sulfoxide Reductases. Biochim Biophys Acta. 2011; 1790 (11): 1471-1477.
Sergey V, Deame H, Alexey VL, Gladyshev VN. Identification and characterization of Fep 15, a new selenocysteine-containing member of the Sep15 protein family. Biochem J. 2006; 394 (3); 575-579.
Labunskyy VM, Hatfield DL, Gladyshev VN. Selenoproteins: molecular pathways and physiological roles. Physiol Rev. 2014; 94 (3): 739-815.
Jiang L, Ni J, Liu Q. Evolution of selenoproteins in the metazoan. BMC Genomics. 2012;13:446.
Lobanov AV, Hatfield DL, Gladyshev VN. Eukaryotic selenoproteins and selenoproteomes. Biochim Biphys Acta. 2009;1790/11): 1424-1432.
Mariotti M, Ridge P, Zhang Y, Lobanov A, Pringle T, Guigo R et al. Composition and Evolution of the Vertebrate and Mammalian Selenoproteomes. PLoS ONE. 2012;7(3):e33066.
Kryukov G, Gladyshev V. Selenium metabolism in zebrafish: multiplicity of selenoprotein genes and expression of a protein containing 17 selenocysteine residues. Genes Cells. 2000;5(12):1049-1060.
Darras VM, Van Herck SLJ. Iodothyronine deiodinase structure and function: from ascidians to humans. J Endocrinol. 2012;215(2):189–206.
Castellano S, Andrés AM, Bosch E, Bayes M, Guigó R, Clark AG. Low exchangeability of selenocysteine, the 21st amino acid, in vertebrate proteins. Mol Biol Evol. 2009;26(9):2031–2040.
Sunde RA. Molecular Biology of Selenoproteins. Annu Rev Nutr. 1990;10(1):451–74.
Moghadaszadeh B, Beggs AH. Selenoproteins and their impact on human health through diverse physiological pathways. Physiology (Bethesda). 2006;21:307–315.
Lu J, Holmgren A. Selenoproteins. J Biol Chem. 2008;284(2):723–7.
Qazi IH, Angel C, Yang H, et al. Selenium, Selenoproteins, and Female Reproduction: A Review. Molecules. 2018;23(12):3053.
Selenoproteins - an overview | ScienceDirect Topics [Internet]. Sciencedirect.com. 2019 [cited 26 November 2019].
Components del grup
Equip de Treball
Grau de Biologia Humana (103), UPF
Sobre Nosaltres
·Albiñana Climent, Elisa
Estudiant a la UV
·Aranda Cuesta, Alba
Estudiant a la UPF
·Pérez Rigau, Guillem
Estudiant a la UPF