

Resultats
| tBLASTn | Exonerate | cDNA e | Proteïna e | T_coffee e | Genewise | Proteïna g | T_coffee g | SECIS | |
|---|---|---|---|---|---|---|---|---|---|
| P.tricornutum | |||||||||
| B.hominis | |||||||||
| H.arabidopsidis | |||||||||
| P.ultimum | |||||||||
| S.parasitica | |||||||||
| E.siliculosus | |||||||||
| C.muris | |||||||||
| C.parvum | |||||||||
| A.taiwanensis | |||||||||
| C.owczarzaki | |||||||||
| P.pallidum | |||||||||
| C.merolae | |||||||||
| T.trahens | |||||||||
| N.gruberi |
Hem fet un blast de les selenoproteïnes SelJ, SelQ i SelTryp contra els genomes dels 14 organismes assignats, hem predit els gens a partir dels hits resultants mitjançant l’exonerate i el genewise, i finalment hem alineat les proteïnes resultants amb les querys inicials. L’objectiu és trobar homòlegs d’aquestes selenoproteïnes en els genomes analitzats.
Després de fer el blast vàrem observar que no hi havia cap homòleg de SelQ en els genomes dels protists analitzats ja que no vàrem obtenir cap hit significatiu (e-value < 0,001).
Pel que fa a SelJ i SelTryp, vàrem obtenir quatre hits significatius per la primera, i dos per la segona en organismes diferents.
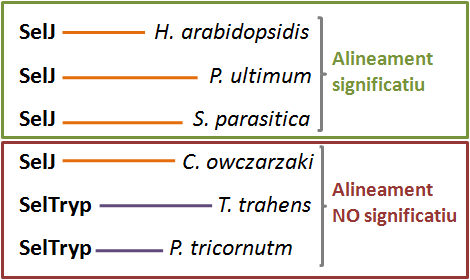
Esquema dels resultats obtinguts.
Després de predir els gens amb l’exonerate i amb el genewise vam observar que en general la predicció era més completa amb el genewise, amb alguna excepció. Per exemple, amb l’organisme H. arabidopsidis l’exonerate alineava la query SelJ de la posició 106 fins a la 177, mentre que el genewise ho feia des de la posició 10 fins a la 177.
No ha estat possible trobar elements SECIS amb el programa SECISearch.pl dintre de la regió dels gens obtinguts.
Altres resultats
Aquests resultats apareixen durant la discussió
Proteïnes
Blast
Exonerate
Genewise
T-coffee