

Discussió
Hem fet un blast de les selenoproteïnes SelJ, SelQ i SelTryp contra els genomes dels 14 organismes assignats, hem predit els gens a partir dels hits resultants mitjançant l’exonerate i el genewise, i finalment hem alineat les proteïnes resultants amb les querys inicials. L’objectiu és trobar homòlegs d’aquestes selenoproteïnes en els genomes analitzats.
Pel que fa a SelJ i SelTryp, vàrem obtenir quatre hits significatius per la primera, i dos per la segona en organismes diferents. No vam obtenir cap hit per a SelQ.
A l’alinear els gens predits amb les respectives querys mitjançant el T-Coffee vam obtenir un bon alineament en tres dels quatres hits predits per SelJ, però amb el quart, corresponent a l’organisme C. owczarzaki, ni pels dos hits predits per SelTryp (Figura 1).
ALINEAMENTS SIGNIFICATIUS
Homòlegs de SelJ en H. arabidopsidis, P. ultimum i S. parasitica
Hem trobat homòlegs de SelJ als organismes H. arabidopsidis, P. ultimum i S. parasitica. Tots tres presentaven una e-value d’entre 10-12 i 10-16 (superiors als altres hits trobats) i observàvem un bon alineament amb SelJ. Per comprovar la relació entre els tres hits vàrem fer un alineament múltiple mitjançant T-Coffee amb aquests i la query, que pots consultar aquí. Tot i observar un bon alineament múltiple, la selenocisteïna de la SelJ (query) no estava alineada amb una cisteïna, en el cas de S. parasitica, ni amb una glicina en el cas de H. arabidopsidis i P. ultimum, tal com havíem observat als resultats del blast i en les prediccions dels gens. Després de nombrosos intents, vam solucionar el problema utilitzant la ultima versió del programa T-Coffee (Figura 2). El fet de trobar homòlegs de SelJ en aquests tres organismes és consistent amb el fet que aquests són molt propers filogenèticament, tal com es pot observar en aquest arbre.

Figura 2. En aquesta figura podem observar l’alineament efectuat amb el T-Coffee de les proteïnes homòlogues (H. arabidopsidis, P. ultimum i S. parasitica) amb la seqüència query SelJ. Observem que les glicines i la cisteïna estan correctament alineades amb la X de la query SelJ.
El paper de SelJ
SelJ és una proteïna que només ha estat descrita en peixos i amb homòlegs en cisteïna en cnidaris. Així doncs aquests resultats, juntament amb els presentats pels nostres companys de l’any passat (consultar), revelen que aquesta proteïna, que es considera molt restringida en la distribució fil·logenètica, potser no ho és tant, encara que sigui en forma d’homòlegs [3] [5]. Si analitzem els resultats observem que no hi ha SelJ amb selenocisteïnes en cap organisme, sinó que només hi ha homòlegs en cisteïna i glicina. Aquests resultats suggereixen que la família de SelJ podria haver aparegut molt abans de l’aparició dels vertebrats o els cnidaris; com a mínim abans de la separació entre metazous (tàxon al qual pertanyen peixos i cnidaris) i stramenopiles (tàxon al qual pertanyen H. arabidopsidis, P. ultimum i S. parasitica). Les espècies Phytophtora sojae i Phytophtora infestans, que segons els companys de l’any passat també contenen homòlegs amb cisteïna i glicina de SelJ, també pertanyen a stramenopiles i estan estretament relacionades amb els organismes que nosaltres hem analitzat. A més a més, el fet que no hàgim trobat cap selenocisteïna sinó homòlegs amb cisteïna i glicina, juntament amb el fet que les espècies de cnidaris també tinguin una cisteïna, obre la porta a la hipòtesi que el gen ancestral tingués una cisteïna. La incorporació d’una selenocisteïna hauria estat posterior, després de la separació entre peixos i cnidaris, aprofintant la maquinària per les altres selenocisteïnes ja presents al genoma. La caracterització de genomes relacionats amb aquestes espècies i altres que tinguin una separació filogenètica anterior aportarà llum a aquesta qüestió.
ALINEAMENTS NO SIGNIFICATIUS
El motiu del mal alineament de SelJ amb C. owczarzaki: Crystallin J1
Pel que fa als l’alineaments amb el T-Coffee dels altres hits, en els tres casos (C. owczarzaki per SelJ i P. tricornutum i T. trahens per SelTryp) vam observar que només alineava una part petita de la proteïna. Observant les prediccions dels gens vàrem comprovar que això era degut a una mala predicció d'aquests, és a dir que tant l’exonerate com el genewise predeien només una part de la proteïna en comptes de predir-la sencera.
Pel que fa a SelJ en C. owczarzaki el resultat suggeria dues possibilitats: que el genewise estigués predint un alineament amb un hit que no fos una proteïna real o que el hit que alineava el BLAST fos una proteïna diferent a SelJ. Per sortir de dubtes vam agafar el hit alineat pel BLAST i vam fer un tBLASTn contra NCBI non redundant (base de dades que conté tots els genomes [12]). Dels resultats que vàrem obtenir (Figura 3), ens va sobtar que els dos únics hits amb significança pertanyessin no a SelJ, sinó a una proteïna diferent anomenada Crystallin J1. Aquesta, juntament amb SelJ, forma part d’una subfamília dins una gran familia d’enzims ADP-ribosiladors. Això ens va fer pensar que potser el hit que havia trobat amb SelJ com a query en l’organisme C. owczarzaki no corresponia realment a la família de la nostra query sinó a Crystallin J1. S’ha publicat que SelJ i Crystallin J1 són famílies molt properes i amb molta similaritat entre elles, i per tant, sembla plausible que Crystallin J1 pugui aparèixer com a hit en un blast de SelJ [5].
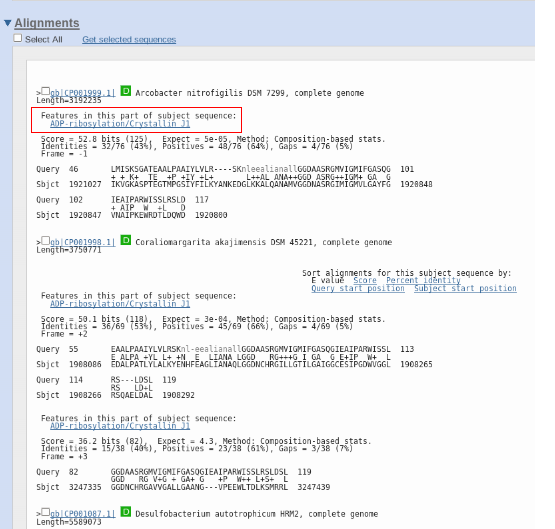
Figura 3. Resultat del BLAST contra la base de dades NCBI non redundant amb la proteïna hit per SelJ. Observem que els hits significatius corresponen a Crystallin J1. En vermell destaquem el millor hit.
Per confirmar aquesta hipòtesi vàrem obtenir la seqüència de Crystallin J1 de l'organisme Arcobacter nitrofigilis per fer un tBLASTn contra el genoma de C. owczarzaki i vam observar que apareixien dos hits en la mateixa regió en la què apareixia el hit fent servir la proteïna SelJ. Aquests dos hits alineaven amb parts diferents de la query, i per tant ens va fer sospitar que es tractava de dos exons. Vàrem fer la predicció tant amb genewise com amb exonerate. El primer només va ser capaç de predir l’ultim exó (que era el que tenia una e-value més petita) mentre que el segon va ser capaç de predir una proteïna amb dos exons. Comparant el resultat de l’exonerate fet amb SelJ i de l’exonerate fet amb Crystallin J1 observem que les regions es solapen, i que per tant estem parlant del mateix gen. Tenint en compte que amb Crystallin J1 som capaços de predir una proteïna més gran i amb un exó amb una e-value significativament més baixa que no pas amb SelJ (Taula 1), creiem que és molt probable que el gen que vam trobar correspongui a Crystallin J1.
| Llargada proteïna | nº Exons | e-value | % identitat | |
|---|---|---|---|---|
| SelJ | 129 aminoàcids | 1 | 2·10-5 | 26% |
| Crys-J1 | 326 aminoàcids | 2 | 2·10-13 | 42% |
| 2·10-4 | 23% |
Taula 1. En aquesta taula mostrem les característiques de cadascun dels hits trobats en el genoma de C. Owczarzaki per les querys SelJ i Crystallin J1 (Crys-J1): llargada de la proteïna, nº d’exons, e-value i % de identitat.
Els passos que hem anat seguint amb l’organisme C. Owczarzaki s’esquematitzen en la Figura 4:
A més a més hem alineat amb el T-Coffee el hit trobat pel BLAST amb la query SelJ (A en la Figura 4) amb el resultat de la proteïna predita mitjançant exonerate o genewise fent servir SelJ (B) i Crystallin J1 (C).
El resultat d’alinear amb el T-Coffee el hit del BLAST (amb SelJ) i Crystallin J1 (Figura 5X) és significativament millor que l’alineament del hit del BLAST de SelJ i la proteïna predita amb la pròpia SelJ (Figura 5Y). Així doncs creiem que la hipòtesi correcta és que el BLAST alineava la query SelJ amb el gen de Crystallin J1 degut a una alta similaritat.
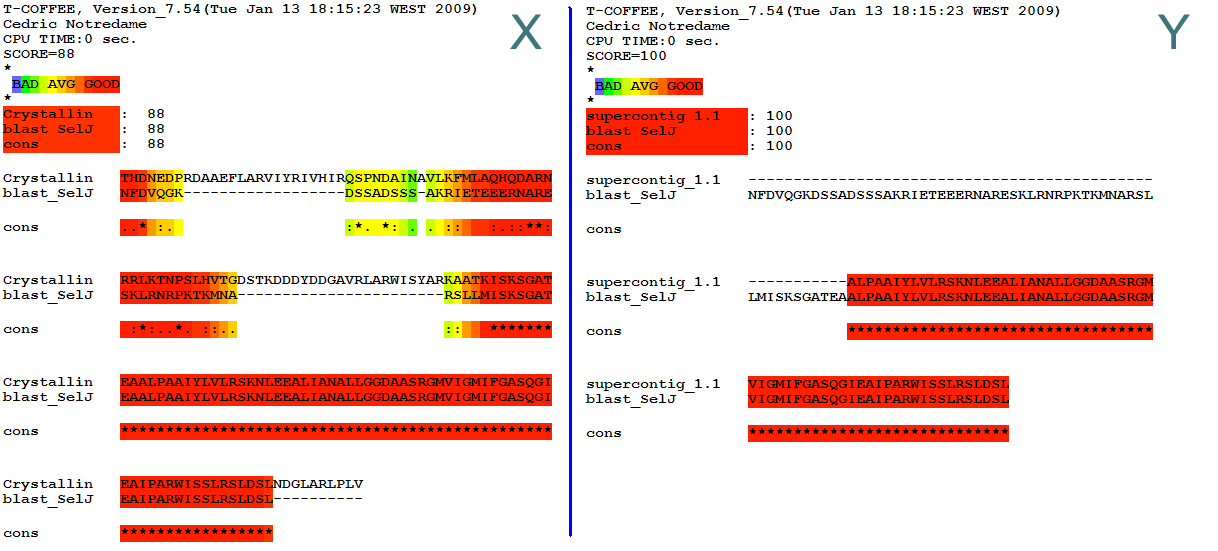
Figura 5. Podem observar els dos alineaments realitzats pel T-Coffee. X) Alineament del hit trobat pel BLAST amb SelJ i la proteïna predita amb Crystallin J1. Y) Alineament del hit trobat pel BLAST amb SelJ i la proteïna predita també amb SelJ.
Crystallin J forma part d’una família de proteïnes que tenen una funció estructural en el cristal·lí de meduses. L’estreta relació entre aquesta i SelJ suggereix una possible funció estructural al cristal·lí per part la selenoproteïna, amb alguns resultats que apunten en aquesta direcció [5]. És estrany, per tant, trobar la proteïna Crystallin J en un organisme tant allunyat com l’Arcobacter nitrofigilis, i sense la presència d’una estructura de cristal·lí. De la mateixa manera les espècies que hem analitzat no tenen aquesta estructura, ja que es tracten de fongs, i per tant no esperarem aquesta funció en els homòlegs de SelJ. Tanmateix aquestes dues proteïnes formen part d’una gran família d’enzims ADP-ribosiladors i per tant hi ha la possibilitat que ancestralment tinguessin aquesta funció enzimàtica i que posteriorment, en metazous, adquirissin la funció estructural. Una altra possibilitat és que en els primers tinguessin una funció estructural diferent i que en metazous haguessin adquirit la funció de formar el cristal·lí.
El motiu del mal alineament de SelTryp amb T. trahens i P. tricornutum: SelT
Quant a la família SelTryp, després d’executar el tBLASTn vam obtenir dos hits: un per P. tricornutum i l’altre per T. trahens tal i com ja hem comentat anteriorment. En tots dos casos la proteïna predita amb el genewise presentava una seqüència molt curta (considerablement més curta que el hit trobat amb el BLAST). Aquest fet, similar al de SelJ comentat anteriorment, suggeria les dues mateixes possibilitats. Per resoldre aquesta qüestió vam fer un tBLASTn contra NCBI non redundant amb el hit que havíem obtingut amb el tBLASTn (Figura 6) i vam observar que els resultats eren d’una altra família de selenoproteïnes: la família SelT. Tanmateix la proteïna SelTryp ha estat identificada recentement i encara no està anotada a la base de dades, tal com vam comprovar al fer un BLASTp contra NCBI non redundant amb SelTryp. Vàrem obtenir com a primers hits anotat proteïnes similars a SelT, concretament el primer era una proteïna hipotètica de Trypanozoma brucei que nosaltres sabem que és SelTryp però que no està anotada a la base de dades.
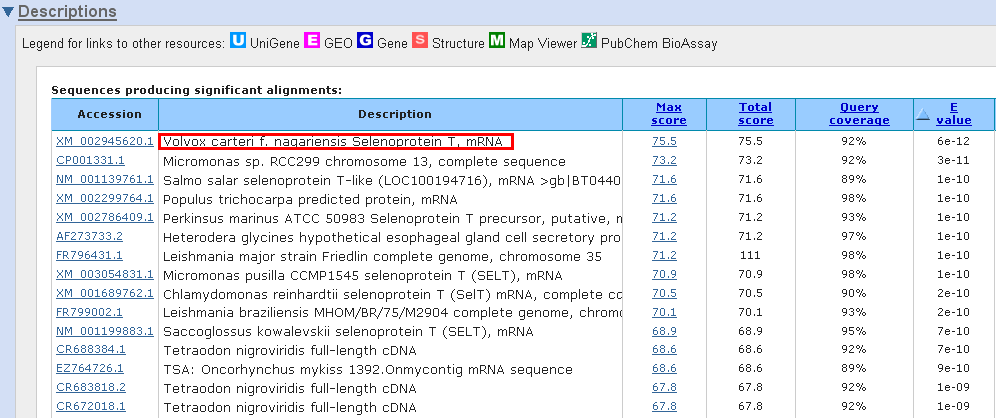
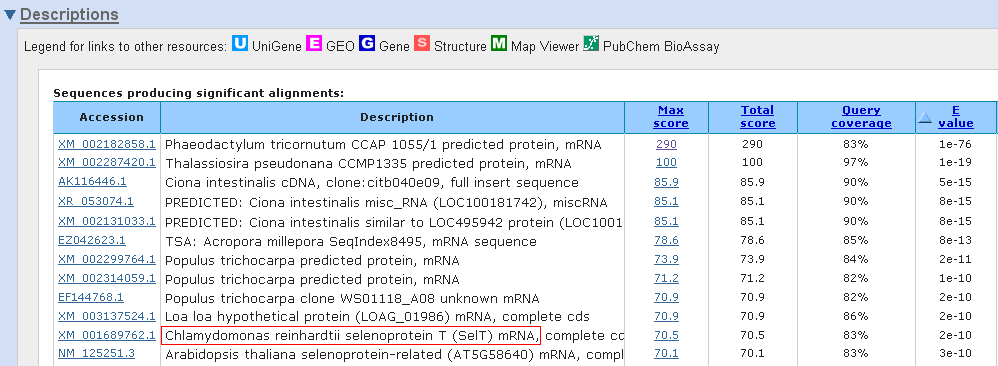
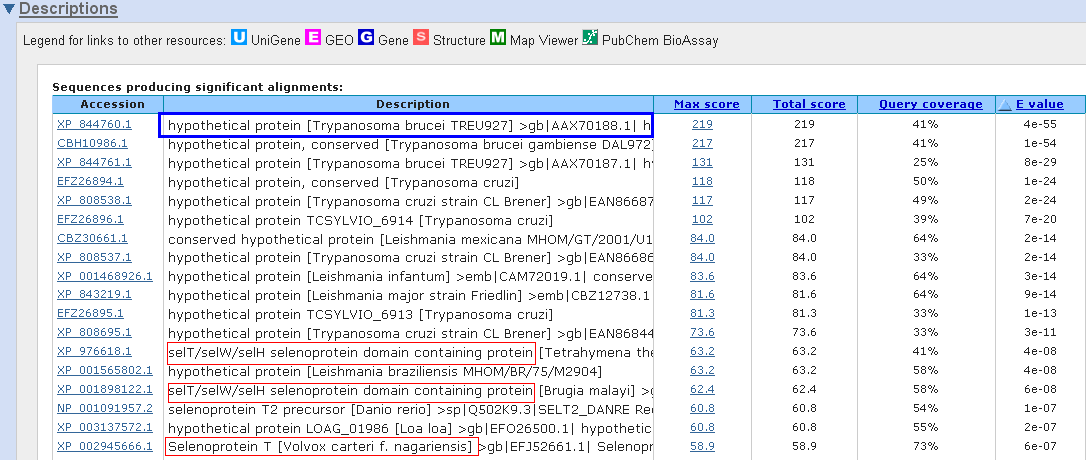
Figura 6. La figura superior mostra l’output obtingut fent un BLAST contra NCBI non redundant amb el hit alineat mitjançant el BLAST per SelTryp contra l’organisme T. trahens. La proteïna que troba és SelT de l’organisme Volvox carteri. A la figura del mig observem l’output amb els BLAST contra NCBI non redundant del hit alineat amb el BLAST per SelTryp contra P. tricornutum. La proteïna SelT que obtenim com a hit és de l’organisme Chlamydomonas reinhardtii. La figura inferior mostra el resultat de fer un BLASTp contra NCBI non redundant amb la proteïna SelTryp.
A continuació, vam proseguir a confirmar que el hit predit al principi del procés es tractava d’una regió que pertany a la proteïna SelT. Per aquest motiu vam fer servir la seqüència de SelT com a query (de l’organisme Volvox carteri, el qual pertany a la divisió Chlorophyta o algues verdes) que havíem obtingut amb el tBLASTn contra NCBI non redundant i vam realitzar un nou tBLASTn contra P.tricornutum i T.trahens, obtenint millors hits: amb una e-value més petita, una identitat més alta i un alineament més precís amb l’exonerate (per P. tricornutum i T.trahens) així com amb el genewise (per P. tricornutum i T.trahens).
Els passos que hem anat seguint amb els organismes P. tricornutum i T. trahens s’esquematitzen en la Figura 7:
En aquest punt hem alineat amb el T-Coffee el hit trobat pel BLAST amb la query SelTryp (A en la Figura 7) amb el resultat de la proteïna predita , mitjançant exonerate o genewise, fent servir SelTryp (B) i SelT (C). Aquest procés l’hem fet tant per T. Trahens (Figura 8) com per P. Tricornutum (Figura 9).
En T. trahens el resultat d’alinear amb el T-Coffee el hit del BLAST de SelTryp i la proteïna predita amb la pròpia SelTryp (Figura 8X) és molt pobre, a diferència del resultat d’alinear el hit del BLAST (amb SelTryp) i la proteïna predita amb SelT (Figura 8Y), que és quasi perfecte.

Figura 8. Aquesta figura mostra els alineaments efectuats amb el hit trobat pel tBLASTn amb la query SelTryp en l’organisme T. trahens amb X) la proteïna predita amb SelTryp i Y) la proteïna predita amb SelT.
Pel que fa al hit trobat en el genoma de P. tricornutum el que observem és que l’alineament entre el hit del BLAST (A en la Figura 7) i la proteïna predita a partir de SelTryp és bo tot i que és significativament més curt (aproximadament 100 aminoàcids, Figura 9X) que el que es produeix per l’alineament del hit del BLAST (A) amb la proteïna predita a partir de SelT, que també és molt bo (Figura 9Y).
Així doncs creiem que la hipòtesi correcta és que el BLAST alineava la query SelTryp amb els gens de SelT en els genomes de T. trahens i P. tricornutum degut a una alta similaritat.
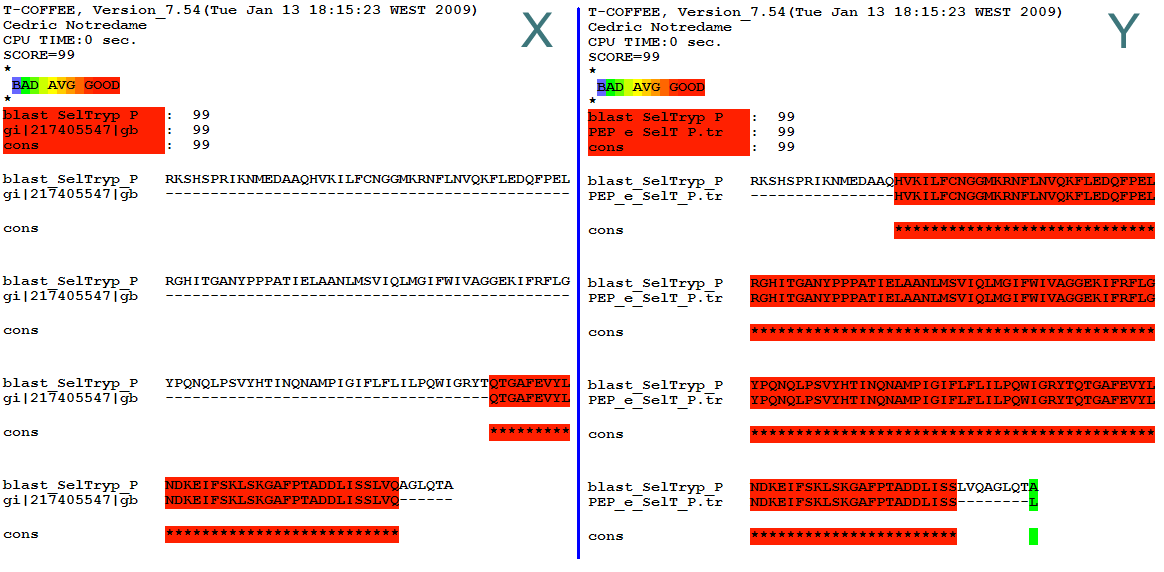
Figura 9. En aquesta figura es mostren els alineaments efectuats amb el hit trobat pel tBLASTn amb la query SelTryp en l’organisme P. tricornutum amb X) la proteïna predita amb SelTryp i Y) la proteïna predita amb SelT.
El fet que haguem obtingut SelT com a hit en un blast utilitzant SelTryp com a query indica que tenen bastanta similaritat, com a mínim en una part de la proteïna. A més a més comparteixen el centre catalític CXXU present SelT, SelW, SelH i Rdx12 [13]. Creiem que és possible que estiguin relacionades, però són necessaris més estudis per a verificar la relació entre SelTryp i SelT.
