

Introducció
El paper del seleni a la natura
El seleni és un element poc abundant a la natura. És un nutrient essencial pels animals, microorganismes i altres eucariotes; una deficiència de seleni pot portar a malaltia (com per exemple la malatia de Keshan) tot i que en excés és tòxic. En els organismes (animals, protists, procariotes...) el seleni es troba a les cèl·lules en forma de selenoproteïnes, mai de forma lliure. La majoria d’aquestes proteïnes són enzims redox i efectuen redaccions de oxidació-reducció, de manera que juguen un paper molt important en la protecció com a antioxidants. [1]
Les selenoproteïnes
Una selenoproteïna és un tipus de proteïna que es caracteritza perquè conté selenocisteïna en la seva estructura. La selenocisteïna és un anàleg de la cisteïna, que conté un àtom de seleni en lloc de sofre.
Les selenoproteïnes es troben en les espècies en tots tres dominis de la vida: arquea, eubacteria i eucariota, tot i que el nombre de selenoproteïnes existents en les diferents espècies varia molt segons el tàxon. Així dons, per exemple, es coneixen 25 selenoproteïnes en mamífers, 3 en drosophila melanogaster i 1 en caenorhabditis elegans . [2]
A més no tots els organismes tenen selenoproteïnes. De fet entre els eucariotes és bastant comú mentre que en altres fílums no tant. Entre bactèries i arquees, les selenoproteïnes es troben només en alguns de llinatges, mentre que estan totalment absents en molts altres grups filogenètics.[3]
Podem trobar homòlegs de selenoproteïnes entre organismes però que difereixen en la selenocisteïna i presenten en comptes d’aquesta, una cisteïna. Moltes vegades l’ortòleg d’una selenoproteïna conté Cys en comptes de Sec. Com per exemple la Selenoproteïna U en peixos no és una selenoproteïna en humans. Fins i tot podem arribar a trobar, dintre una mateixa espècie un gen que codifica per una selenocisteïna i una còpia del mateix gen conté cisteïna. [3]
Anotació i predicció
L’aminoàcid selenocisteïna és codificat pel codó UGA el qual és, normalment, un codó de terminació de la traducció. Per tant, ens trobem davant de que en una mateixa espècie hi ha un codó que té dos significats diferents.
És per això que les selenoproteïnes estan molt mal anotades ja que la majoria de programes assumeixen que l’UGA és un codó de stop i, per tant, les selenoproteïnes no són reconegudes. [4]
Síntesi de selenoporteïnes
La re-codificació del codó UGA necessita maquinària específica. La principal senyal per la re-codificació de l’aminoàcid és un element de l’estructura secundària (tridimensional) del RNA anomenat SECIS (SElenoCysteine Insertion Sequence) en l’extrem 3’ UTR del gen que codifica per la selenoporteïna. Aquest element recluta una proteïna anomenada SECIS Binding Protein (SBP2), la qual s’uneix i recluta un factor d’elongació específic de selenoproteïnes (Efsec). Efsec aproxima el tRNA de la selenocisteïna al codó UGA de l'mRNA de les selenoproteïnes i en determina la seva traducció a selenocisteïna; la traducció de l'mRNA continua fins que, en algun moment, el ribosoma reconeix un nou codó de terminació. Per tant és un procés que requereix la participació de molts gens. [4]
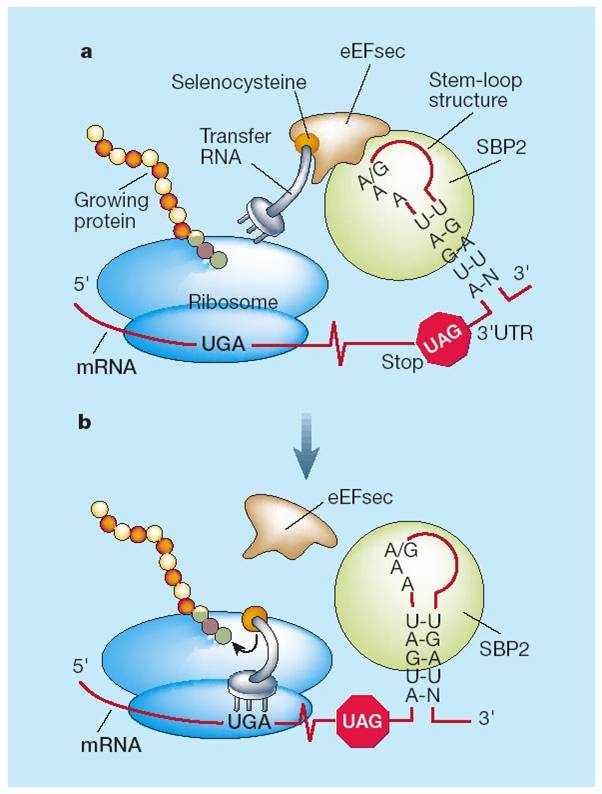
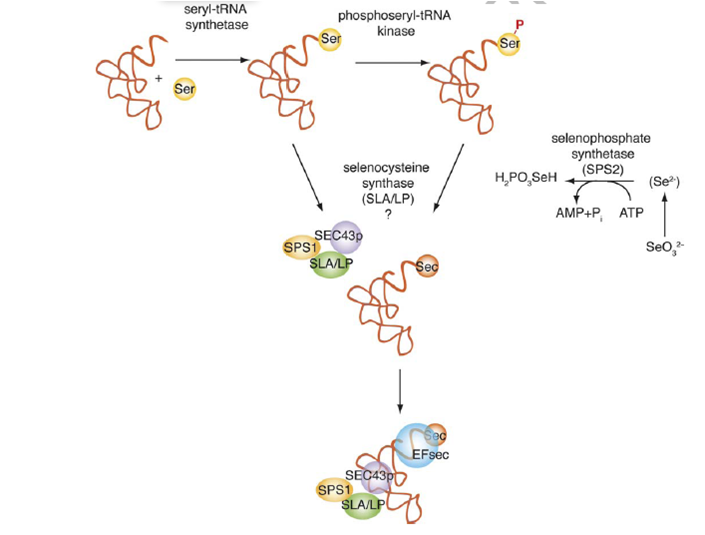
Per la biosíntesi d’una selenoproteïna, per tant, és necessària la participació de almenys 7 o 8 gens.
Els més importants són els que codifiquen per proteïnes necessàries per:
Les selenoproteïnes s’identifiquen per la presencia d’elements SECIS (a 3’ UTR) ja que tot i que a nivell de seqüència poden arribar a ser molt diferents, mantenen una estructura secundària bastant conservada que podem predir gràcies a programes informàtics.
Els programes de predicció de gens permeten UGA i segueixen analitzant el DNA. Si els nucleòtids que segueixen a aquest codó tenen potencial codificant pot ser que ens trobem davant d’una possible selenoproteïna.
Per fer una predicció eficient de la localització de selenoproteïnes es sobreposa la informació d’elements SECIS amb la localització del d’exons del gen que estem estudiant. [5]