Selenoproteïna T(SelT)
La Selenoproteïna T és una selenoproteïna que no es troba anotada a selenoDB en Myotis lucifugus. És per això, que hem usat la query de la selenoproteïna d'Homo sapiens.
El blast ens dona dos hits, tots dos en el mateix sentit a l'anotació (+). El primer, KBKB102544.1, tot i que té una alta homologia, en fer el tcoffee, veiem un alineament amb puntuació baixa. En canvi, al fer l'anàlisi amb el segon scaffold, obtenim una proteïna amb menor homologia, però un tcoffee de puntuació més alta. Per això, considerem que la proteïna SelT es troba a l'scaffold KB114215.1.
L'exonerate i el genewise han predit tots dos una proteina. En el cas de l'exonerate té 5 exons i 4 introns, i una raw score de 969. El genewise, prediu també una proteina de 5 exons i 4 introns, amb un score de 385.19 bits.
L'estructura predita tant amb exonerate com la predita amb genewise presenten un codó stop en la mateixa posició que la selenoproteina de la query (aminoàcid 49). Deduim així que la selenoproteïna està conservada. Pel que fa al tcoffee, com ja hem dit, trobem una puntuació alta que implica que la estructura està ben conservada.
Pel que fa a la predicció d'elements SECIS, el SECISearch 3 ens prediu un element SECIS a la posició 3', que ens valida la teoria que SelT és una selenoproteïna en Myotis davidii. D'altra banda, Seblastian ens confirma que en el nostre subseq trobem SelT, mostrant l'homologia del nostre scaffold amb la Selenoproteïna T de Mus muscullus. Amb tota aquesta informació, podem predir l'estructura de la selenoproteïna:
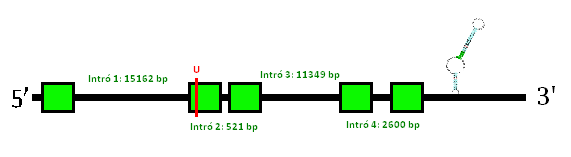
Per últim remarcar que la Selenoproteïna T no es troba anotada en Myotis lucifugus, però si que es troba en la nostra espècie d'estudi, Myotis davidii. Donat que en l'ancestre comú (l'Homo sapiens) si que la trobem, hipotetitzem que es tracta d'una estructura que Myotis lucifugus ha perdut posteriorment a la diferenciació d'aquest i la nostra espècie, o bé un error en l'anotació.
