Les selenoproteïnes són proteïnes en les quals trobem un aminoàcid selenocisteïna en la seva seqüència. La Sec consisteix en una cisteïna en la qual el radical sulfur s’ha substituit per un àtom de seleni.
L’objectiu d’aquest projecte ha estat predir totes les selenoproteïnes i proteïnes de maquinària especial de traducció del genoma de Myotis davidii, una espècie de ratpenat endèmica a la Xina. Per fer-ho, hem utilitzat els programes tBLASTn, Exonerate, Genewise i TCOFFEE mitjançant un programa automàtic creat per nosaltres mateixos.
Per obtenir les seqüències problema a buscar al genoma de Myotis davidii, hem consultat la base de dases selenoDB, on hem trobat la predicció de selenoproteïnes de l’espècies Myotis lucifugus. Aquesta espècie és del mateix gènere que la nostra, i tot i que dins del gènere Myotis no són molt properes, hem hipotetitzat que hauríem de trobar selenoproteïnes semblants entre elles. és per això que inicialment hem utilitzat les seqüències trobades a Myotis lucifugus per alinear-les amb el nostre genoma problema, però degut a algun error d’anotació en la base de dades, fer servir aquestes seqüències no sempre ha estat possible i hem hagut de recórrer a les seqüències d’humans. D’altra banda també hem comparat aquelles selenoproteïnes que només es troben en humans, així com les proteïnes de maquinària.
Els nostres resultats mostren la troballa de 21 selenoproteïnes i 7 homòlegs de cisteïna, a més de 7 proteïnes de maquinària de les quals una d’elles és una de les 21 selenoproteïnes.
El nostre estudi ha trobat un seguit de diferències entre Myotis lucifugus, Myotis davidii i Homo sapiens que no concorden amb la bibliografia que hem llegit8. S'ha observat que en Homo sapiens han aparegut noves proteïnes dintre de la família SelU que no presenten una selenocisteïna des del proteoma de l'ancestre. Aquest cas no el trobem ni en Myotis lucifugus ni en Myotis davidii, on presenten pèrdues de selenocisteïna, ja sigui per desaparició o per canvi de selenocisteïna a cisteïna. Ara bé, s'han observat unes diferències considerables en el nombre de desaparicions de selenoproteïnes entre els dos Myotis. Com que es tracta de dues espècies del mateix genère, no podem considerar les diferències com quelcom significatiu ja que no és possible que presentin menys homolgies Myotis lucifugus amb Myotis davidii que amb Homo sapiens. Per tant, podem concloure a que les proteïnes de Myotis lucifugus estan mal anotades en la base de dades SelenoDB, ja que segons aquesta no trobem algunes selenoproteïnes essencials com ara SPS2 (responsable de pasar el seleni a selenofosfat en el procés de formació del tRNASec) en Myotis lucifugus.
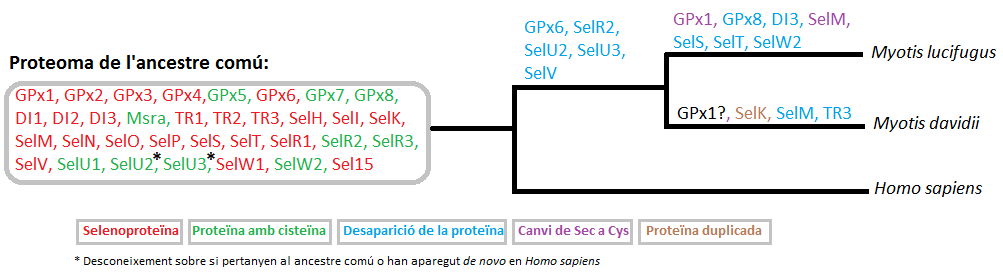
Evolució de les selenoproteïnes en Homo sapiens i el gènere Myotis
La figura pretén il·lustrar els resultats obtinguts en la nostra cerca. Cal remarcar que, com ja hem dit, no podem assegurar que les pèrdues siguin realment desaparicions de les proteïnes i no fruit d'una anotació incomplerta. D'altra banda, hem marcat algunes Selenoproteïnes (com ara SelV) com a pèrdua en el gènere Myotis, basant-nos en la literatura consultada7 , però pot tractar-se de proteïnes que han aparegut posteriorment a la separació entre espècies, i per tant, siguin adquirides en el genoma humà.
Finalment remarcar que el treball ens ha permès adquirir nous coneixements i molta pràctica en el món de la bioinformàtica. Hem validat el temari après a l'assignatura i guanyat l'habilitat per adquirir els coneixements de programació per iniciativa pròpia.
