Selenoproteïna (SelP)
La Selenoproteïna P (SelP) és molt característica pel fet de presentar més d’una selenocisteïna en la seva seqüència d’aminoà cids. Es troba conservada tant a Myotis lucifugus, en el que té 14 selenocisteïnes, com a Homo sapiens, on en pot tenir fins a 10. La SelP la podem trobar correctament anotada a SelenoDB en els dos casos, i degut a que aparentment hi havia força diferència en el nombre de selenocisteïnes entre les dues espècies, hem agafat la query de Myotis lucifugus per la seva proximitat evolutiva amb Myotis davidii.
El blast d’aquesta query només ha donat un hit amb un e-value molt bo (2e-53). Aquest hit és KB115516.1 i es troba en el sentit contrari al de l’anotació.
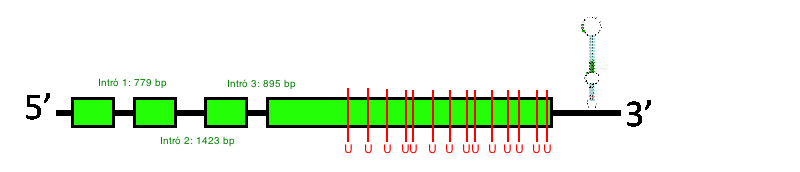
L'exonerate ha predit una proteïna que té 4 exons i 3 introns, amb un raw score de 1448. A més, en l’alineació de l’exonerate podem comprovar que hem escollit adequadament la query ja que Myotis davidii té exactament les mateixes 14 selenocisteïnes que Myotis lucifugus. En el cas de genewise, la predicció ha estat igualment bona, prediu també 4 exons i 3 introns, amb un score de 515.05 bits.
L’alineament fet pel programa t-coffee també és molt bo, amb un score de 98.
Pel que fa a la predicció d'elements SECIS, el SECISearch3 ha trobat un element SECIS de grau A i score superior a 15 a la regió 3’ de la seqüència nucleotídica. D’altra banda, el Seblastian ha predit la mateixa proteïna que l’exonerate, amb els mateixos exons i introns selenocisteïnes, alineant-la amb la SelP de Equus caballus.
Amb tota aquesta informació, podem afirmar amb força seguretat que Myotis davidii té la selenoproteïna P conservada, i amb les nostres dades en prediem aquesta estructura: