Familia Selenoproteïnes R (SelR)
La familia de Selenoproteïnes R està formada per tres proteïnes anomenades R1 (SelR1), R2 (SelR2) i R3 (SelR3). SelR1 és una selenoproteïna tant a Myotis lucifugus com a Homo sapiens, mentre que SelR3 és un homòleg de cisteïna en les dues espècies. SelR2 existeix per a humans, però no per a Myotis lucifugus. Les proteïnes es troben correctament anotades a SelenoDB. Hem utilitzat la query de Myotis lucifugus tant per SelR1 com per SelR3, mentre que per SelR2 hem utilitzat la query humana.
SelR1
El blast de la query de SelR1 ens troba un sol hit amb un e-value molt petit i una identitat molt elevada, corresponent a l’scaffold KB107323.1. Aquest scaffold es troba en la mateixa direcció d’anotació de la seqüència (+).
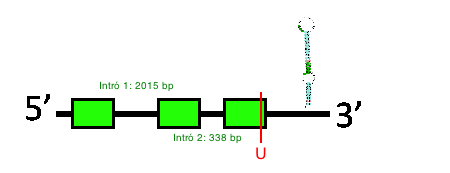
L’exonerate troba una proteïna amb tres exons i dos introns, amb una raw score de 521. En aquest alineament podem reconèixer el codó UGA on hi ha una selenocisteïna a Myotis lucifugus, per tant és força evident que la nostra espècie també la té. Tot i que l’alineament és molt bo en general, hi ha una regió de l’scaffold en que la seqüència apareix com a desconeguda i no es pot alinear. D’altra banda, el genewise també ha fet un alineament força bo, però no ha agafat tota la seqüència, de manera que només troba el segon intró, i s’ha obtingut un score de 201,93 bits.
Sorprenentment, el t-coffee no ha sortit bé, és possible que s’hagi donat algun error en el nostre programa o no s'hagi escollit bé a l'hora d'escollir entre els 6 marcs de lectura possibles.
Quant a la predicció d’elements SECIS, el SECISearch ha predit un element SECIS de grau A i score superior a 15 a la regió 3’ de la seqüència, i per tant considerem aquest resultat vàlid. D’altra banda, tal com esperàvem el Seblastian ha predit la mateixa proteïna que l’exonerate.
Així doncs, tenint en compte el correcte alineament de la proteïna en exonerate i Seblastian, la troballa del codó UGA i la presència d’un element SECIS, pensem que en Myotis davidii la SelR1 es troba en l’scaffold KB107323.1, i tindria l’estructura següent:
SelR2
El blast de la query de SelR2 ens troba quatre hits, dels quals destaca KB114609.1 amb un e-value molt petit i una homologia de t-coffee elevada. Aquest scaffold es troba en la mateixa direcció d’anotació de la seqüència (+).
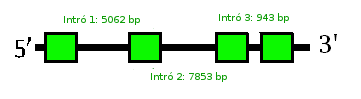
L’exonerate troba una proteïna amb quatre exons i tres introns, amb una raw score de 658. Tot i que l’alineament és molt bo, hi ha una regió de l’scaffold en que; la seqüència apareix com a desconeguda i no es pot alinear. D’altra banda, el genewise que conté quatre exons i tres introns també ha fet un alineament força bo amb un score de 261,79. Per una altra banda, el t-coffee ha donat una alta homologia.
Quant a la predicció d’elements SECIS, el SECISearch ha predit un element SECIS de grau A i score superior a 15 a la regió 3’ de la seqüència, i per tant considerem aquest resultat và lid. D’altra banda, tal com esperavem el SEBLASTIAN ha predit la mateixa proteïna que l’exonerate.
Els programes SECISearch3 i Seblastian no han pogut predir cap element SECIS ni cap selenoproteïna. Això concorda amb els resultats que hem trobat a l’exonerate, on recordem que no s’ha trobat cap selenocisteïna.
Així doncs, donats els nostres resultats seria possible afirmar que SelR2 és present en Myotis davidii en forma d’homòleg de cisteïna i no com a selenoproteïna, com en Homo sapiens.
SelR3
En fer el blast de la query de SelR3 obtenim tres scaffolds amb e-value prou bo: KB111605.1, KB116256.1 i KB114609, tots ells alineats en la mateixa direcció d’anotació (+). D’aquests, només el primer presenta una identitat notable, i com vam veure més endavant, és el que va obtenir un millor alineament al t-coffee amb diferència. Per tant l’scaffold que nosaltres hem escollit és KB111605.1.
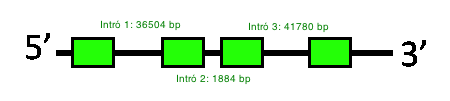
L’exonerate de la query de SelR3 troba una proteïna amb quatre exons i tres introns amb una raw score de 428, mentre que el genewise troba tres exons i dos introns amb un score de 166,61 bits. Tal com es va trobar a Myotis lucifugus, no trobem cap selenocisteïna a la seqüència, sinó algunes cisteïnes que es mantenen igual en les dues espècies de ratpenats.
En fer el t-coffee s’ha produït alguna mena d’error ja que la seqüència del nostre scaffold no es troba completa. Tot i així podem veure que l’alineament de la part que apareix és molt bo.
Els programes SECISearch3 i Seblastian no han pogut predir cap element SECIS ni cap selenoproteïna. Això concorda amb els resultats que hem trobat a l’exonerate, on recordem que no s’ha trobat cap selenocisteïna.
Així doncs, donats els nostres resultats seria possible afirmar que SelR3 és present en Myotis davidii en forma d’homòleg de cisteïna i no com a selenoproteïna, tal com ja s’havia vist en Myotis lucifugus i Homo sapiens.
Conclusió
En resum, dins la família SelR, a Myotis davidii, SelR1 és una selenoproteïna. SelR2 i SelR3 no ho són. Aquest fet es dóna també en les espècies Homo sapiens i Myotis lucifugus, amb l'excepció de que SelR2 no existeix en Myotis davidii.
