

El principal objectiu d’aquest projecte és identificar els gens que codifiquen per a selenoproteïnes al genoma del Casuarius casuarius que és un au gran no voladora que pertany a la fauna del nord d’Austràlia, essent crucial per a la conformació del bosc. Es relaciona filogenèticament amb emús i, més remotament amb els estruços, els nyandús i els kiwis.
Les selenoproteïnes són proteïnes que contenen un residu de selenocisteïna (Sec) a la seva seqüència i en conjunt formen el que es coneix com a selenoproteoma. El residu Sec es troba codificat pel codó UGA que en la majoria dels casos actua com un codó de parada. Per aquest motiu, les selenoproteïnes no estan sempre ben anotades al genoma, de manera que cal fer estudis que permetin identificar-les com és aquest cas. Per tal que es pugui incorporar el residu Sec es requereix en primer lloc la seqüència d’inserció de Sec o element SECIS. A més, es requereixen proteïnes específiques que formen part de la maquinària responsable de la síntesi de selenoproteïnes. És per això, que a més d’identificar selenoproteïnes, altre objectiu principal és identificar la maquinària necessària per la seva síntesis.
Per tal de predir el selenoproteoma del Casuarius casuarius i la maquinària relacionada amb la seva síntesis es va comparar el seu genoma amb el d’Homo sapiens el genoma del qual es troba millor anotat i amb una espècie més propera filogenèticament, Gallus gallus.
Utilitzant eines bioinformàtiques com tBLASTn, Exonerate i T-coffee en aquest treball s’han aconseguit identificar vint-i-quatre selenoproteïnes, set homòlegs que contenen Cys i sis components de la maquinària.
El seleni és un micronutrient essencial per a humans i animals. El seu excés o la seva manca poden tenir efectes rellevants en aquests organismes. A dia d'avui se sap que la importància de mantenir uns nivells constants de seleni recaual fet que el seleni és l'element que s'utilitza per a la síntesi del vint-i-unè aminoàcid natural, la selenocisteïna (Sec). La Sec és un anàleg de la cisteïna (Cys) que en comptes de tenir sulfur, conté seleni. Una de les particularitats de la selenocisteïna és que està codificada per una seqüència TGA a l'ADN, és a dir un codó stop a nivell d'ARN (UGA), i que la seva inserció a proteïna. A més a més, la Sec s'incorpora a la cadena polipeptídica de forma co-traduccional a través d'una maquinària específica i exclusiva per a aquest aminoàcid, fet que evita que hi hagi selenocisteïnes lliures. Les principals raons per les quals no ens interessa tenir Secs lliures a l'espai són dues: per un costat hi ha el risc que s'incorporin Secs en lloc de Cys, i per l'altra, la tendència de la Sec a reaccionar amb oxigen i tioredoxines, que produeix la ràpida oxidació del NADPH i la formació de radicals lliures[9].
Les proteïnes que contenen Sec són les anomenades selenoproteïnes. En el genoma humà s'han identificat vint-i-cinc gens que codifiquen per selenoproteïnes i se sap que podrien existir-ne al voltant d'unes cent en la totalitat dels mamífers. Hi ha diverses famílies de selenoproteïnes que, en part degut al fet que la Sec és un residu actiu essencial per a l'activitat catalítica, estan involucrades en diversos mecanismes com ara reaccions redox, senyalitació a través de mecanismes redox, defensa contra agents oxidants, metabolisme de l'hormona tiroïdea i respostes immunes. Per tot això i per altres raons que encara s'han d'investigar, les selenoproteïnes tenen un paper crucialals organismes i es poden relacionar amb diverses malalties en humans com ara el cancer, la malaltia de Keshan, infeccions víriques, infertilitatals homes o alteracionsal sistema immunes i en les funcions de l'hormona tiroïdea[14][16].
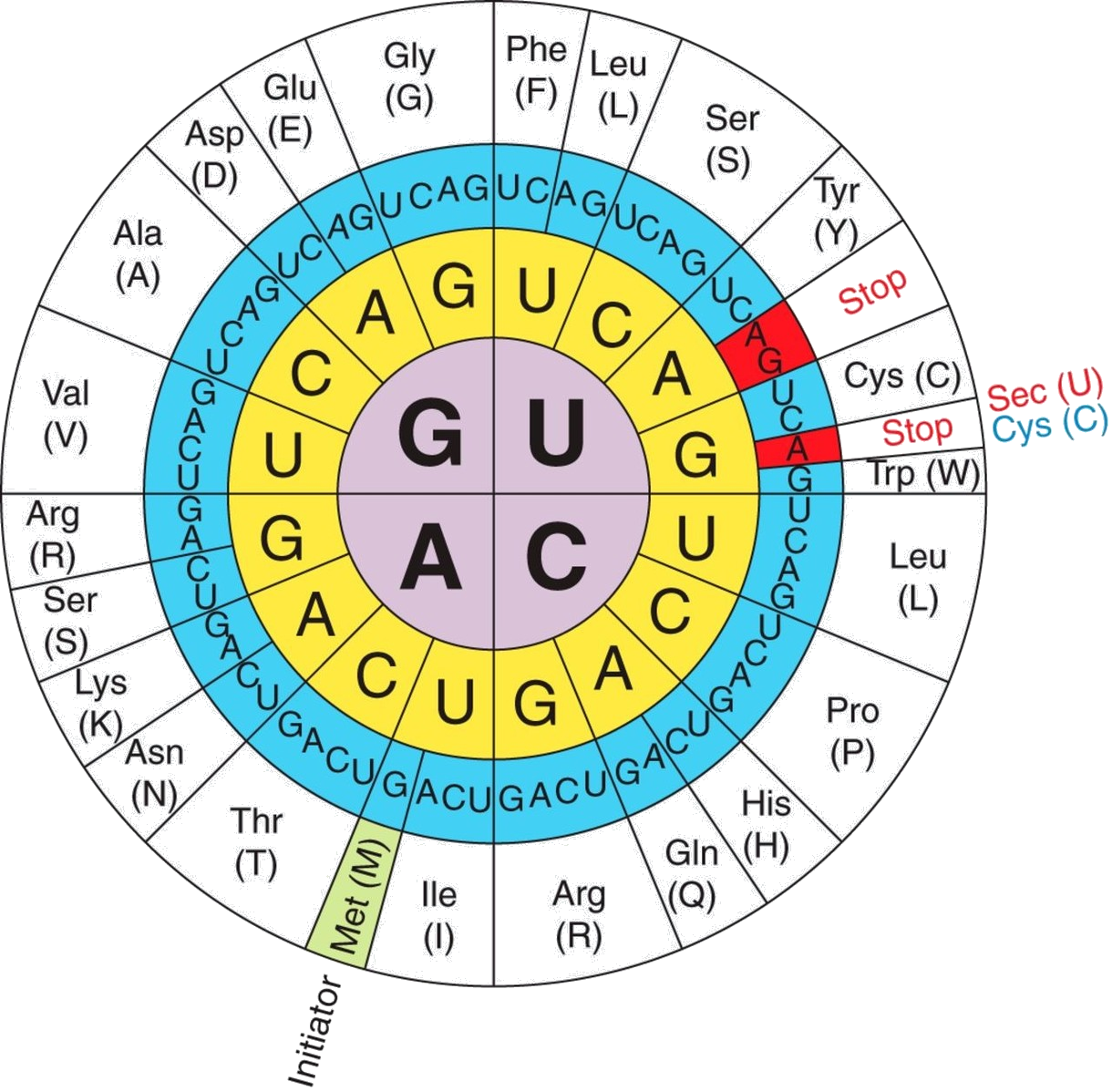
Figura 1. El codi genètic il·lustrant la funció dual del codó UGA com a codó STOP i com a codó codificant del vint-i-unè aminoàcid, la selenocisteïna (Sec). Font: [9]
Malgrat puguem trobar selenoproteïnes tantal domini bacteria, com en l'archaea, com en l'eukarya, s'ha vist que hi ha organismes que no les utilitzen o que no en tenen. Un exemple d'aquests casos seria el dels llevats, les plantes superiors i els fongs, que no tenen selenoproteïnes perquè han perdut la maquinària de síntesi d'aquestes proteïnes de manera que han quedat substituïdes per homòlegs de selenoproteïnes amb cisteïna.[15][13][12]
Tant en eucariotes com en les procariotes hi ha una gran variabilitat en la quantitat de selenoproteïnes que té cada espècie. Els selenoproteomes d'eucariotes i procariotes no coincideixen en la seva totalitat, ja que segons la distancia evolutiva que hi hagi entre les espècies, aquests selenoproteomes són més o menys similars: com més properes en termes evolutius dues espècies, més semblants són els seus selenoproteomes.[15][13][12]
Els organismes que tenen un major nombre de selenoproteïnes són els que viuen en hàbitats aquàtics, ja siguin animals o plantes. Concretament, s'ha vist que hi ha certs tipus d'algues que són molt riques en selenoproteïnes, fet que podria tenir relació amb la seva capacitat fotosintètica, de manera que la presència de selenoproteïnes seria crucial per a protegir les cèl·lules de l'alga de l'oxigen. A més a més, també s'hipotetitza que la major disponibilitat de seleni que hi ha a l'aigua, respecte altres medis, podria afavorir la introducció i l'ús d'aquest element per determinades proteïnes (selenoproteïnes) amb funcions d'oxireductases.[15][13][12]
Els hàbitats terrestres, en canvi, tenen menys quantitat de certs elements traça que els aquàtics (i entre ells, de seleni) i a més, tenen nivells d'oxigen més elevats. Aquest fet podria explicar queals organismes terrestres tinguin menys quantitat de selenoproteïnes, bé perquè les han perdudes o perquè han estat substituïdes per homòlegs amb cisteïna.[15][13][12]
Amb tot, doncs, el que els estudis suggereixen és que els organismes que viuen en medis aquàtics tenen selenoproteomes més grans que els terrestres i que, els mamífers en concret, mostren una tendència a fer un ús reduït de les selenoproteïnes. A dia d'avui, però, i malgrat l'esforç que s'ha fet i que es fa des de l'àmbit de la recerca, l'evolució del selenoproteoma en vertebrats encara no s'ha desxifrat completament.[15][13][12]
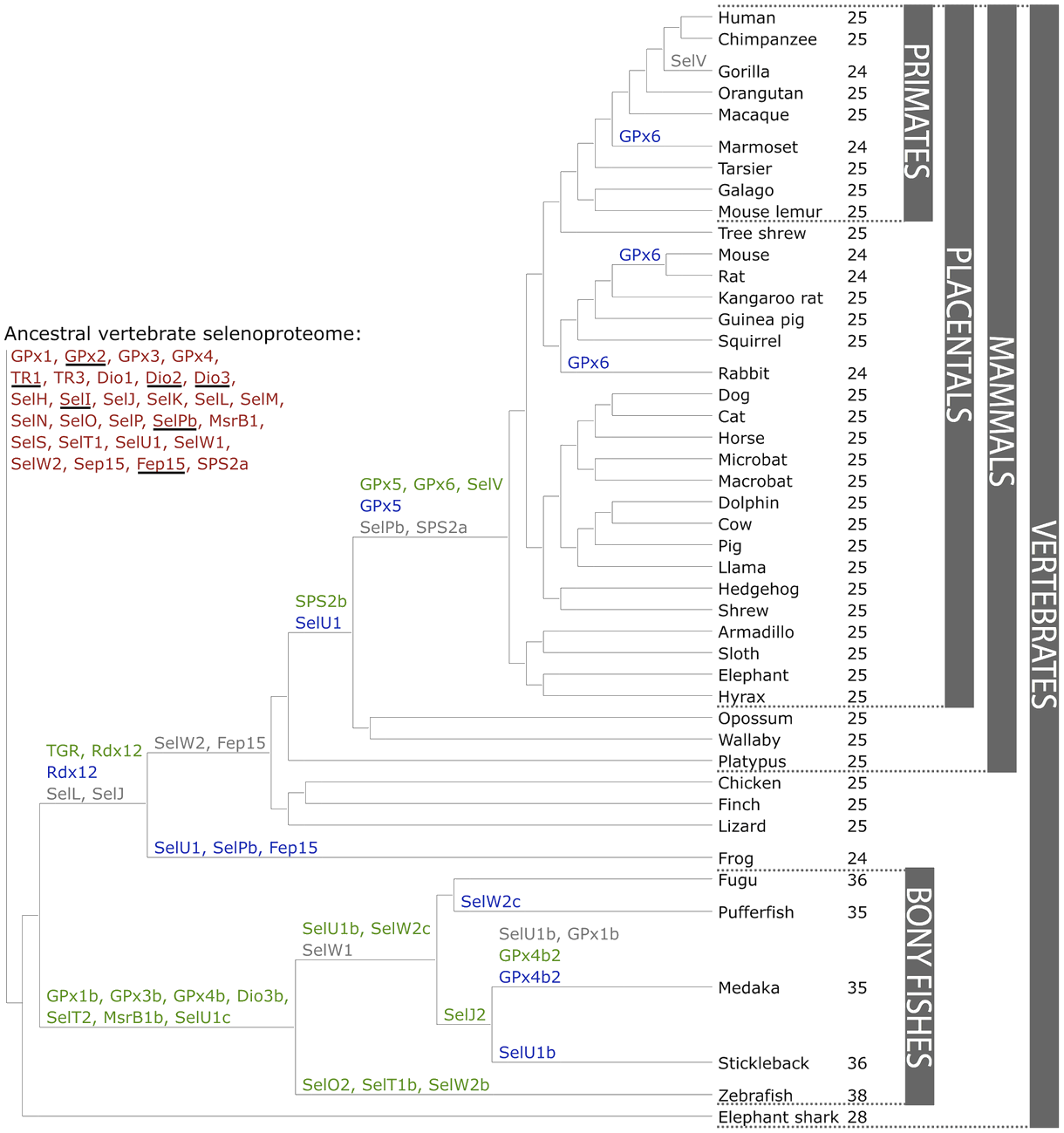
Figura 2. Evolució del selenoproteoma de vertebrats. Font: [15]
Les famílies més grans i més ben estudiades són les famílies de les glutation peroxidases, tioredoxin reductases (TR) i iodotironina deiodinases (Dio).
S'han trobat 21 selenoproteïnes en tots els vertebrats: GPx1-4, TR1, TR3, Dio1, Dio2, Dio3, SelH, SelI, SelK, SelM, SelN, SelO, SelP, MsrB1, SelS, SelT1, SelW1, Sep15. El selenoproteome més gran s'ha trobat a zebrafish amb 38 selenoproteïnes i el més petit s'ha trobat a granota i alguns mamífers amb 24 selenoproteïnes.
La funció de les selenoproteïnes no és ben coneguda, aquelles més ben descrites tenen papers importants en la regulació redox cel·lular. També s'han vist implicades en prevenció de càncer, modulació del procés d'envelliment, reproducció masculina i resposta immune.



Figura 3. Famílies de Selenoproteïnes Taula adaptada de l'article Composition and Evolution of the Vertebrate and Mammalian Selenoproteomes [15] i complementada amb les següents fonts: [9], [10], [6], [7], [20], [22].
La selenocisteïna es diferencia de la resta d'aminoàcids per ser l'únic aminoàcid en eucariotes la biosíntesi del qual necessita un tRNA específic, el tRNA[Ser]Sec. Per tal de començar el procés de síntesi de la Sec, el tRNA[Ser]Sec es sotmet a una reacció catalitzada per la seril-tRNA sintetasa (SerRS) i, d'aquesta manera, es pot formar el Ser-tRNA[Ser]Sec a partir de l'aminoacilació del tRNA[Ser]Sec amb una serina. Seguidament, la PSTK (Fosfoseril tRNA-Sec Quinasa) fosforila el Ser-tRNA[Ser]Sec, que passa a l'intermediari PSer-tRNA-Sec. Aquest intermediari és el que acaba donant lloc al Sec-tRNA després de sotmetre's a una reacció catalitzada per la SecS, que consumeix selenofosfat provinent de la reacció catalitzada per la SPS2 i ATP.[9][18]
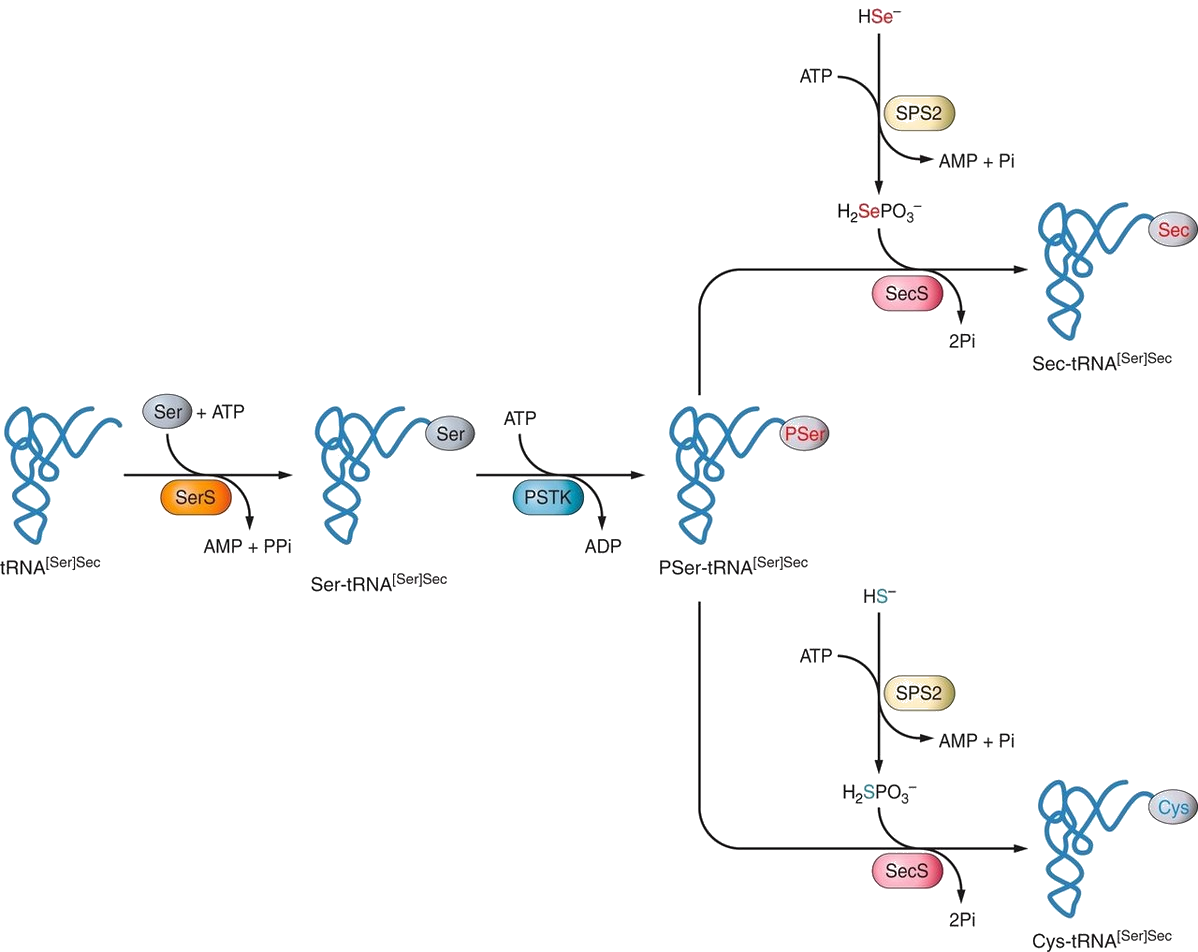
Figura 4. Procés de síntesi del tRNA-Sec i el tRNA-Cys a partir de la Ser-tRNA en eucariotes. Font: [9]
A més a més, la síntesi de selenocisteïnes també necessita que hi hagi un codó UGA, que és el que codifica per Sec, a part de factors proteics trans-activadors i els elements SECIS (Sec insertion sequence) a l'extrem 3’UTR de la cadena positiva (la mateixa on hi ha l'UGA).[9]
El moment en què un ribosoma es troba amb un codó UGA d'una seqüència d'mRNA que ha de codificar per una selenocisteïna, la maquinària de síntesi de la selenocisteïna (i en concret els elements SECIS) interacciona amb la maquinària de traducció per tal de promoure que el codó UGA sigui llegit com a codó codificant per a Sec, en comptes que sigui llegit com a codó stop i que, per tant, hi hagi una finalització prematura de la traducció. En resposta a l'acció de l'element SECIS, la Sec-tRNA[Ser]Sec pot traduir el codó UGA a una Sec. Això ho pot fer gràcies al fet que té un anticodó complementari a UGA.
En aquest procés, però, no només hi participa la maquinària mencionada, sinó que també es necessiten dos factors trans-activadors: l'SBP2 i l'eEFSEc. L'SBP2 (SECIS binding protein 2) s'associa de forma estable als ribosomes, així com també conté un domini d'unió a l'ARN (L7 Ae RNA-binding domain) que li permet unir-se als elements SECIS amb molta afinitat i especificitat. A més a més, també interacciona amb l'altre element trans-activador, l'eEFSec. L'eEFSec (Sec-specific translation elongation factor) és l'encarregat de reclutar el Sec-tRNA[Ser]Sec per a facilitar, d'aquesta manera, la incorporació de la Sec a la cadena polipeptídica que s'estigui sintetitzant.[9]
Hi ha altres SECIS-binding proteins que s'han identificat: la proteïna ribosomal L30, el factor d'iniciació eucariòtic 4a3 (eIF4a3) i la nucleolina. La L30 forma part de la maquinària d'inserció de la Sec a la cadena polipeptídica, mentre que l'eIF4a3 i la nucleolina participen de la modulació de la síntesi de les selenoproteïnes.[9]
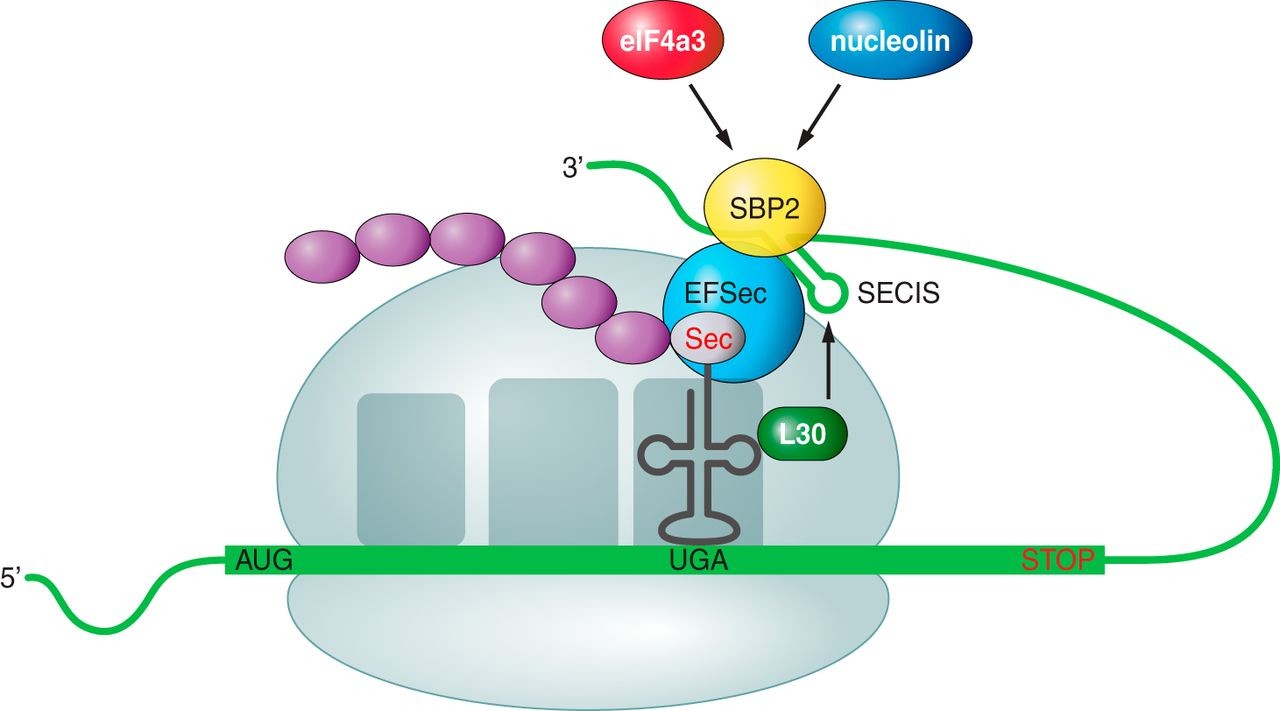
Figura 5. Mecanisme d'inserció de la Sec en eucariotes. Font: [9]


Figura 6.Taula d'el·laboració pròpia en què s'expliquen els membres de la maquinària de síntesi de les selenoproteïnes. Font:[9][4]
Cada mRNA de selenoproteïna conté una estructura de doble stem-loop formada per aproximadament 100 nucleòtids, que s'anomena element SECIS. Aquests motius de RNA juguen un paper essencialal mecanisme de la recodificació del codó UGA. Inicialment es creia que era una plataforma de reclutament de factors de recodificació, però el SECIS realment controla la traducció del mRNA de selenoproteïna mitjançant governant l'eficiència d’inserció de selenocisteïna.[9]
En els eucariotes es troba localitzat en la regió 3'UTR. En la major part dels mRNAs de selenoproteïnes UAA o UAG és utilitzat com una senyal de terminació. Es requereix una distància mínima d'uns 50 nucleòtids entre el UGA/codó selenocisteïna i el SECIS per a una recodificació eficient. Sovint trobem dos elements SECISal mRNA de selenoproteïnes amb múltiples UGAs que es recodifiquen a selenocisteïna, com la SelP en humans.[9]
Els elements SECIS en eucariotes no es troben molt conservats a nivell nucleotídic, però les estructures secundàries estan força conservades sent stem-loop-stem-loop. Dos motius conservats en l'element SECIS són essencials per a un bona recodificació del codó UGA com a selenocisteïna. Com es pot observar a la figura, el nucli del SECIS conté un quartet de parells de bases no Watson-Crick i un motiu AAA/G. La distància entre aquestes dues regions és manté per la hèlix 2. L'hèlix 1 és altament variable en grandària i composició, encara que és important per a mantenir la integritat del circuit intern. El nucli SECIS engloba dos parells G-A tàndem (denominats parells tàndem GA), que es conserven en tots els elements SECIS eucariotes.[9]
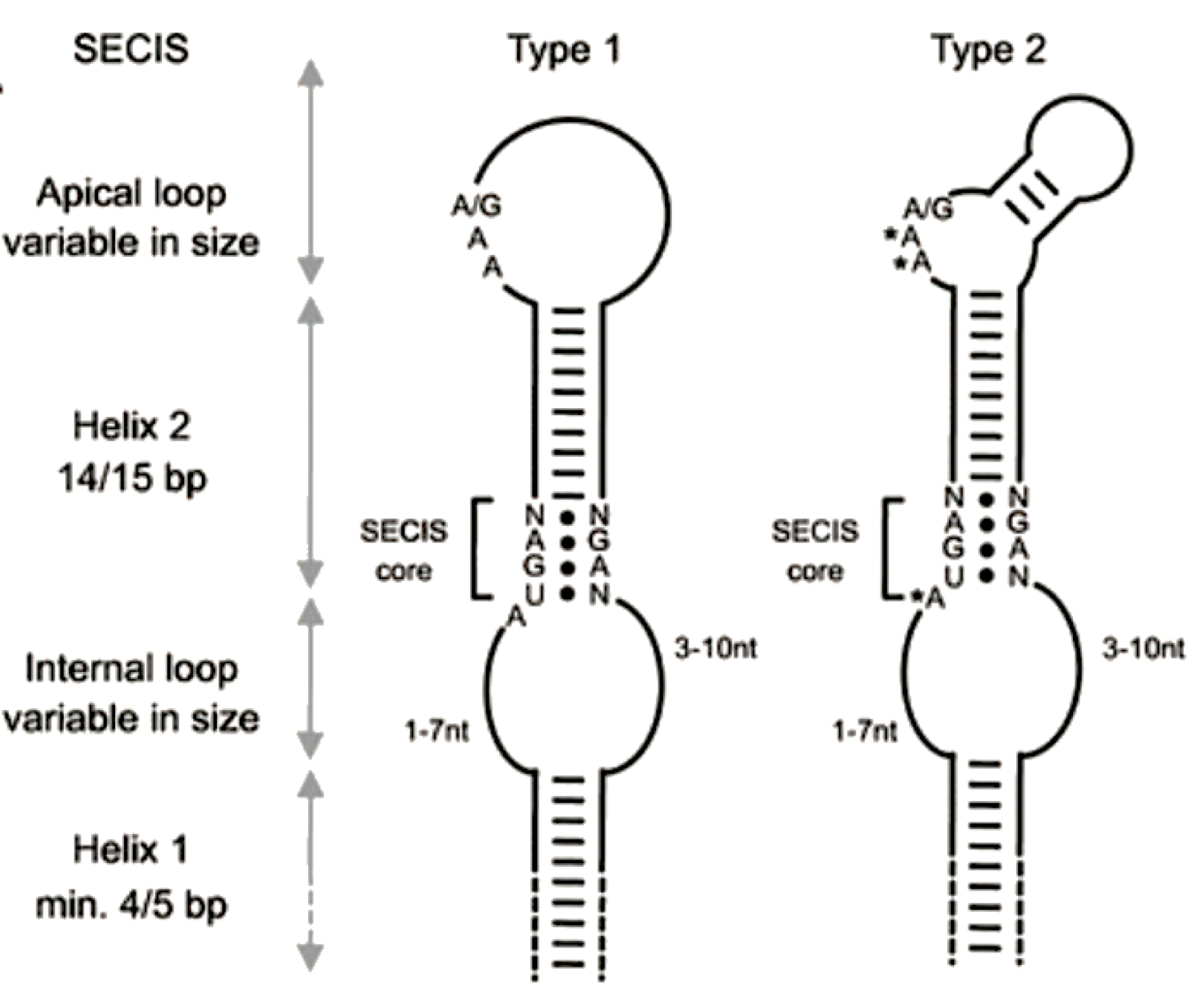
Figura 7. Estructura dels elements SECIS. Font: [4]
L'estructura secundària stem-loop-stem-loop s'utilitza per a predir els cantidadts de SECIS en genomes eucariotes acabats de seqüenciar mitjançant el programari SECISearch que és accesible en una interfície web fàcil d'utilitzar.
| SUPER REGNE | Eukaryota |
| REGNE | Animalia |
| FILUM | Chordata |
| CLASSE | Aus |
| ORDRE | Casuariiformes |
| FAMÍLIA | Casuariidae |
| GÈNERE | Casuarius |
| ESPÈCIE | Casuarius casuarius |
Figura 8. Taula taxonòmica del Casuarius casuarius adaptada de la font original. Font: [wikipedia]
El causari comú (Casuarius casuarius) és una au gran, no voladora, de la família dels Causàrids (Casuariidae). Tècnicament és l'au més gran d'Àsia (desde l'extinció de l'estruç àrab i anteriorment el moa de Nova Zelanda) i l'au astral més gran.
Un mascle adult erguit completament pot superar en alçada sense problemes una persona d'1,65 metres d'alçada i arribar a pesar més de 50 quilograms. Les femelles adultes són encara més altes i poden arribar a pesar més de 75 quilograms.
El plomatge és rígid i negre brillant. Les potes, escamoses. Són tridáctils ial dit interior de cada peu han desenvolupat una ungla modificada letal com una daga de fins a 12 cm. Les ales són minúscules, atrofiades. El coll és llarg i quasi sense pèl, cobert per una lleugera capa de plomes curtes que sembla borrissol. La pell té tons vermells, taronjes, morats i blaus. A la base del coll, per davant, pengen un parell de plecs llargs de pell vermells, denominats carúncules. Tenen els ulls grans i castanys, i el bec, llarg i corbat. Sobre el cap llueixen una protuberància anomenada casc, que sembla una banya.
Malgrat que el plomatge és sexualment monomòrfic, la femella s'identifica com a dominant, ja que té el casc més llarg, el pic més gran i les parts nues de colors més brillants.[25]

Figura 9. Casuari mascle adult amb la seva cria en una platja del sud de Cairns. Font: [25]
Busca l'alimental sòl del bosc, sobretot cerca fruites caigudes. Un jove casuari corre en busca de fruita que escolta caure a terra. Aquestes aus són ràpides. Els adults poden superar els 50 quilòmetres per hora. És capaç de digerir amb seguretat algunes fruites tóxiques per altres animals.
El casuari del sud és un au solitaria, conviu i s'aparella només en l'època de reproducció, a finals d'hivern o primavera. El mascle construeix un niu a terra, un "matalàs" de material herbaci de 5 a 10 centímetres de gruix i fins a 100 centímetres d'ample. Això és suficientment gruixut per deixar que la humitat no es quedi als ous. El mascle també incuba els ous i cria els pollets sol. Criden durant la temporada d'aparellament i fora d'aquesta època realitzen xiulen i murmuren. Els pollet criden el mascle amb freqüents xiulets de to alt.
Són hàbils donant cops de peu. Apropar-se a un mascle a càrrec d'una nidada és arriscar-se a sofrir el seu atac protector. Si se'ls intenta caçar o matar, pot defensar-se i guanyar. Les arpes com fulles són capaces de matar humans i gossos, si se'ls hi provoca. A la vegada, són tímids, pacífics i introvertits.[2][3][8][25]

Figura 10. Cria de casuari corre a cercar fruita que ha sentit que queia. La imatge mostra l'alta velocitat a la qual poden arribar els membres d'aquesta espècie. Font: [25]
El casuari sud es distribueix a Indonèsia, Nova Guinea i nord-est d'Austràlia. A Austràlia es troben als Tròpics Humits del nord de Queensland. Alguns habiten zones de bosc plujós, com el de Daintree; altres viuen en la seva perifèria i a vegades s'aventuren als jardins de les cases. Prefereixen boscos tropicals però pot fer ús dels boscos de sabana o manglars propers. Es troben a elevacions per sota d'1.100 metres a Austràlia i 500 metres a Nova Guinea.[24][25]
En el transcurs d'una jornada un adult ingereix centenars de fruites i baies. No obstant, la digestió del casuari ès delicada i, en conseqüència, les llavors no es deterioren, sino que al defecar surten intactes. Així, conforme l'au passeja pel seu territori, menjant, bevent, rentant-se i defecant, porta llavors d’un costat a un altre del bosc. Pot arribar a desplaçar-les a distàncies majors de 800 metres. També dispersa llavors pendent a dalt i riu a través. Així doncs, els casuaris són un important vehicle de disseminació i per a molts arbres, l'únic.[25]

Figura 11. La muntanya Pieter Botte (o Nulbullulul) davant del bosc plujós de Daintree, una de les àrees més extenses en què l'hàbitat del casuari es manté intacte. Font: [25]
S'alimenta d'una gran varietat de fruites del bosc que arreplega del terra. És capaç de digerir amb seguretat algunes fruites tòxiques per a altres animals. Ocasionalment menja petits animalons o els seus cadàvers i encara serps. També mengen fongs i alguns insectes.[24]

Figura 12. Casuari mascle observa com la seva cria s'alimenta de fruites del bosc colorides. Font: [25]
El casuari comú és ovípar. L'aparellament té lloc en época de cria, normalment a finals de l'hivern o primavera. El mascle fabrica un niu a terra que tapissa amb material vegetal, on la femella pon. El mascle cova els ous durant 40 dies i cura dels pollets, que són nidífugs.
La femella posa tres o quatre ous. Tenen una superfície granulada i són inicialment de color verd pebre brillant, tot i que s'esvaeixen amb l'edat. A més, varies vegades més grans que els de gallina.[23][24]

Figura 13. Ous de casuari. Són força més grans que els de les gallines i són incubats pel mascle durant quaranta dies. Font: [25]
Encara que es troba sotmès a la pèrdua de l'hàbitat en curs, l'abast limitat i la sobrecàrrega en algunes àrees, el casuari del sud a partir de 2017 va ser avaluat com a mínima preocupaci$oacute per la Llista Vermella de les Espècies Amenaçades de la UICN.[26]
La població australiana es troba a la llista de perill d'extinció segons la legislació federal i estatal de Queensland[24]. Algunes amenaces són la pèrdua d’hàbitat (explotaci´ forestal), els animals salvatges que es mengen els seus ous, la ca&ccemila i la carretera. La construcció de carreteres suposa un greu problema. A més d'una fragmentació del seu hàbitat, suposa un perill potencial per a la seva vida a causa d'atropellaments.[25]
Té un rang d'ocurrència de 396,000 km2, i entre 10,000 i 20,000 aus es van estimar en un estudi de 2002, dels quals d’entre 1.500 i 2.500 es troben a Austràlia.
Els casuaris del sud han estat criats en molts zoològics arreu del món, com a White Oak Conservation a Yulee, Florida, Estats Units.

Figura 14.Rang que determina l'estat de perill de l'espècie Casuarius casuarius com a mínim. Font: [26]
Només queden tres espècies de Causari: Casuarius casuarius, Casuarius bennetti, Casuarius unappendiculatus. Els casuarius estan emparentats amb els emús i, m´es remotament, amb els estruços, els nyandús i els kiwis.
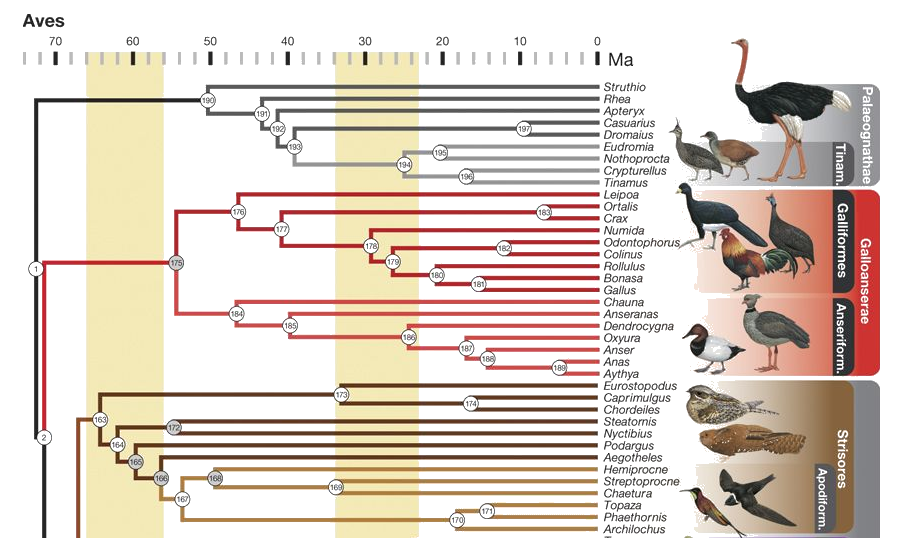
Figura 15. Filogènia del Casuarius casuarius. Figura adaptada de Prum RO, et al. 2015.
El principal objectiu d'aquest treball és identificar les selenoproteïnes del genoma del Casuarius casuarius i la maquinària necessària per la seva síntesi i funcionament. L'esquema general que es va seguir es mostra a continuació.
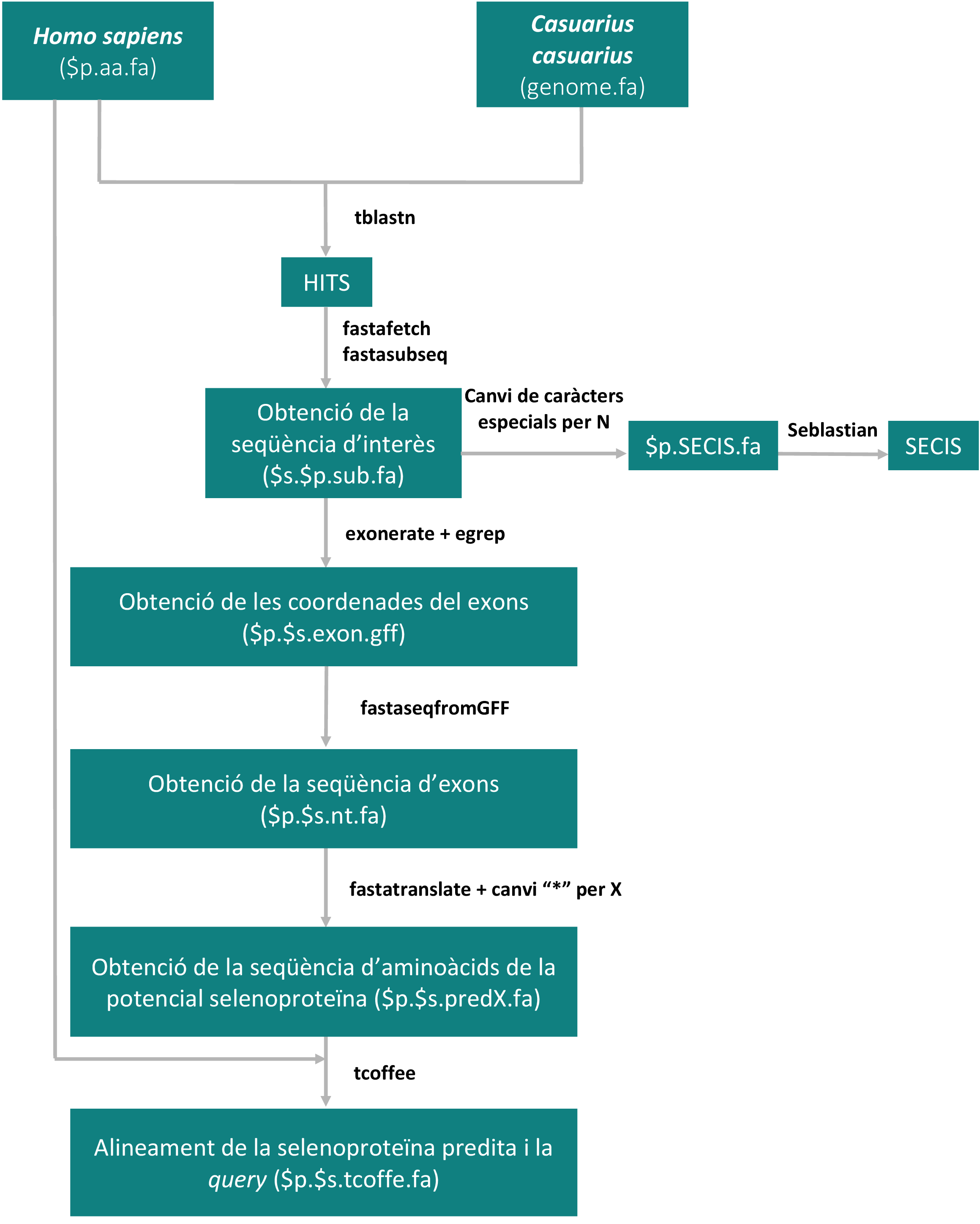
Figura 16. Esquema d'el·laboració pròpia en què s'explica el procés bioinformàtic pel qual s'han obtingut les prediccions de selenoproteïnes en la nostra espècie.
Per tal d'identificar i obtenir aquests elements, es van buscar les regions conegudes de les selenoproteïnes i la seva maquinària al genoma humà utilitzant el SelenoDB 1.0. A partir d'aquesta base de dades específica es van obtenir les seqüències proteiques humanes en format FASTA que es van utilitzar com queries per fer la comparació.
Les queries es van desar en documents de text a partir de l'editor emacs on es van eliminar els símbols del final (@, # i %) i es van nombrar com $p.fa (p.ex. DI1.fa). Un cop realitzat això, es van afegir tots els documents a la carpeta “Proteïnes humanes” dins la carpeta "public".
El genoma del Casuarius casuarius va ser facilitat pels professors de l'assignatura. Aquest es troba en l'arxiu següent:
Tot el procés es va automatitzar per tal de fer-ho més ràpid i eficaç i es va desar com a programa.pl. Després es van desar el programa i les diferents queries en una carpeta dins l'escriptori (no es pot treballar dins la carpeta "public") i es va fer la comanda que ens dóna permís per accedir i modificar el programa:
A l'hora d'utilitzar el shell hem de fer diferents comandes per tenir accés a la carpeta "public".
sudo mkdir /mnt/public
sudo mount.cifs -o username=uXXXXXX //fs-aules.ds.upf.edu/PUBLIC/20428 /mnt/public
A part, s'han de carregar els diferents programes.
exportPATH=/mnt/NFS_UPF/bioinfo/BI/bin:$PATH FastaseqfromGFF.pl
export PATH=/mnt/NFS_UPF/bioinfo/BI/soft/genewise/x86_64/bin:$PATH
export WISECONFIGDIR=/mnt/NFS_UPF/bioinfo/BI/soft/genewise/x86_64/wise2.2.0/wisecfg/ Genewise
La primera part del programa es fa servir per canviar les "U" dels documents $p.fa per "X". Les U fan referència a les selenocisteïnes, però els programes que s'executen no les reconeixen, per tant, es canvien per X que vol dir qualsevol aminoàcid. Aquestes es van desar com $p.aa.fa en la carpeta "Proteïnes amb X".
Per tal de comparar el genoma del Casuarius casuarius amb les diferents queries s'utilitza el tblastn. Aquest permet comparar una seqüència proteica ($p.aa.fa) que és la proteïna de referència humana amb el genoma de referencia (genome.fa) que és el genoma del Casuarius casuarius.
Amb aquesta comanda s'obtindrà la taula de hits significatius utilitzant l'e-value com $p.blast. L'e-value descriu el nombre de vegades que s'esperaria trobar un alineament com aquest o amb millor puntuació a la nostra base de dades (genoma del Casuarius casuarius) únicament per atzar. Per a considerar que un hit és significatiu l'e-value ha de ser superior a 0,0001. Això s'estableix amb “evalue 0.001”.
Amb aquesta comanda es podrà observar al shell la taula de hits significatius obtinguts pel blast. Aquesta taula conté el nom dels hits significatius que correspon al fragment del genoma on s'ha trobat el hit, la posició inicial (start) i final (end) del hit i l'e-value que ens dóna informació sobre la significancia estadística del hit.
Aquesta part del programa és important ja que a partir d'aquesta regió es portaran a terme els següents pasos. És per això que no ha estat automatitzada, ja que així es pot escollir el hit que es vol seguir analitzant.
Aquesta funció ens permet extreure l'scaffold d'interés, les regions potencials on es poden trobar les selenoproteïnes al genoma del Casuarius casuarius i es desa com a $s.$p.fetch.fa. En aquest pas es necessita el fastaindex el qual també va ser proporcionat pels professors de l'assignatura.
El principal problema d'aquest procediment és que els hits només representen les regions codificants del genoma. El programa ho resol expandint el hit 50.000 nucleòtids en les direccions 5' i 3' per tal que agafi tota la seqüència per la presència d'introns.
En primer lloc el programa mira si el frame és positiu, és a dir, si el nucleòtid inicial (start) és menor al final (end), o frame negatiu, és a dir, si el nucleòtid inicial (start) és major al final (end). A més, es té en compte que no les coordenades inicials i finals quedin dins l'scaffold i no es donin nombres negatius, és a dir, si al restar 50000 a la coordenada inicial dóna inferior a 0 el programa estableix que sigui la coordenada inicial serà 1. Al final s'obté la llargada total.
Aquesta funció permet obtenir la regió d'interès dins l'scaffold seleccionat amb el fastafetch generant un nou fitxer amb aquesta regió ($s.$p.sub.fa). A aquesta comanda se li ha de donar la posició inicial i la longitud de la regió d'interès que es vol seleccionar i que s'ha obtingut amb l'expansió del pas anterior.
En la primera línia es mostra el que el programa executarà en cas que el hit obtingut es trobi a la cadena +. Mentre que la segona línea s'executarà si el hit es troba en la cadena -. El document final s'anomena $s.$p.sub.fa.
Aquesta funció permet obtenir els exons del gen predit com $p.$s.exonerate.gff. Amb l'egrep s'aconsegueix unir els exons en un mateix arxiu ($p.$s.exon.gff).
Amb aquesta funció el programa obté el cDNA generat a partir del fitxer gff anterior. El nou fitxer es desa amb el nom $p.$s.nt.fa.
Aquesta funció permet l’obtenció de la seqüència d’aminoàcids a partir de la seqüència de cDNA obtinguda amb la comanda anterior. Amb això s’obté un arxiu anomenat $p.$s.pred.fa que conté la proteïna predita.
Amb aquesta funció es pot substituir els "*" on hi la les selenocisteïnes per X generant un arxiu que conté la seqüència d'aminoàcid de la selenoproteïna predita on les Sec es representen com a X i que s’anomena $p.$s.predX.fa.
A partir de l'arxiu que conté la seqüència d’aminoàcids de la seqüència predita ($p.$s.predX.fa) i la query inicial ($p.aa.fa) s'executarà aquesta funció que generarà un alineament global de les dues seqüències proteiques per tal de comparar-les.
S'utilitza el programa Seblastian per tal de trobar els gens de les selenoproteïnes mitjançant la detecció dels elements SECIS en la regió 3'-UTR. Aquests elements són estructures tridimensionals que es troben al mRNA necessaris per tal de presentar Sec a partir del codó UGA en les seves seqüències. En el cas de no trobar cap element, es fa servir SECISearch3 per corroborar aquest resultat, tot i que aquest no té en compte que ha d’estar en la regió 3'UTR. Per a poder entrar la seqüència al Seblastian, primer s’ha de fer una comanda automatitzada que canviï les lletres S, M, R, K, B, Y i W per N. El resultat d'aquesta es desa com $p.SECIS.fa i aquest serà l'input que Seblastian utilitzarà per predir les selenoproteïnes i els SECIS.
Una vegada trobades totes les proteïnes es va procedir a realitzar un arbre filogenètic de cada família de proteïnes utilitzant phylogeny.pr. Per tal de realitzar la filogènia es va generar un fitxer ($p.filo.fa) per a cada família de proteïnes. Aquest fitxer conté cadascun dels membres de la família predital Casuarius i el seu hom&ogareveleg en humans.
 TROBAT |
 NO TROBAT |
| Proteïna | Espècie | Residu | Blast | Exonerate | Scaffold | Frame | Localització | Nombre d'exons | Fastasubseq | T-coffee | Seblastian | SECIS | Imatge SECIS | Proteïna predita |
| SELENOPROTEÏNES | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DEIODINASES D'HORMONA TIROIDAL | ||||||||||||||
| DIO1 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000081.1 | Negativa | 2957511-2958002 | 1 |  |
 |
 |
 |
 |
 |
| DIO1 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000081.1 | Negativa | 2957505-2958002 | 1 |  |
 |
 |
 |
 |
|
| DIO2 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000143.1 | Positiva | 662022-674752 | 2 |  |
 |
 |
 |
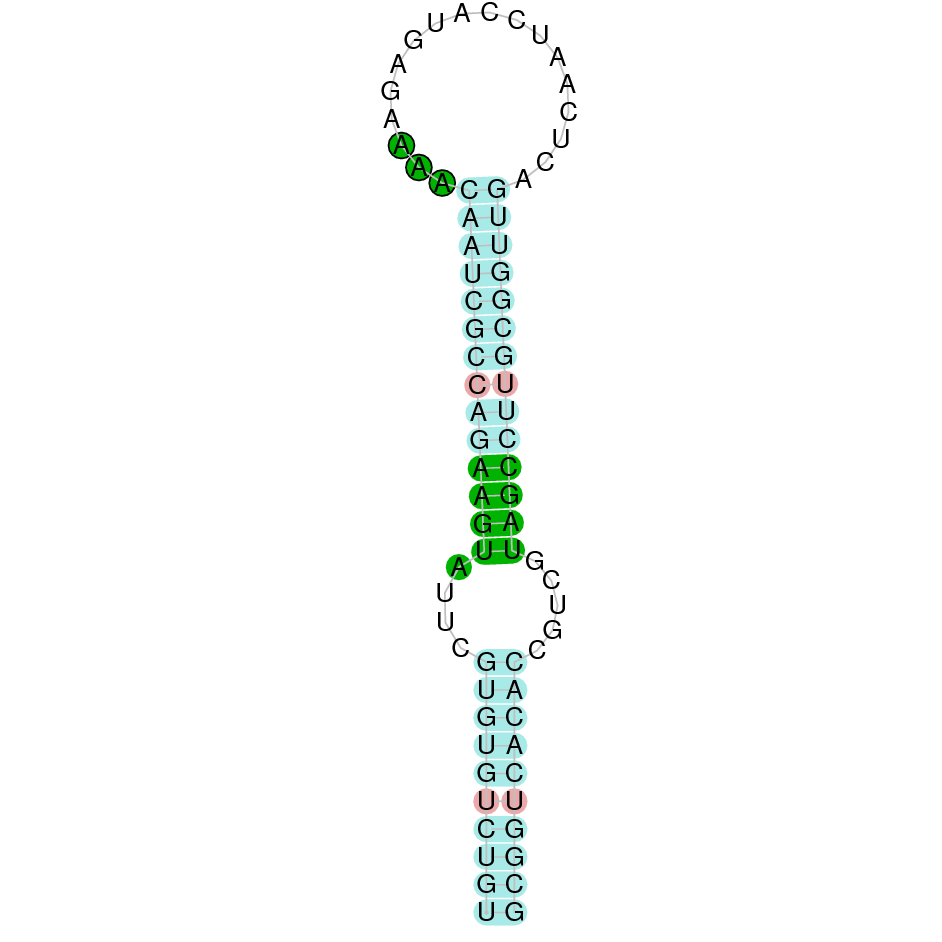 |
 |
| DIO2 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000143.1 | Positiva | 662022-674752 | 2 |  |
 |
 |
 |
 |
|
| Homo sapiens | Sec-Sec |  |
 |
PTFA01000029.1 | Negativa | 2957496-2958287 | 2 |  |
 |
 |
 |
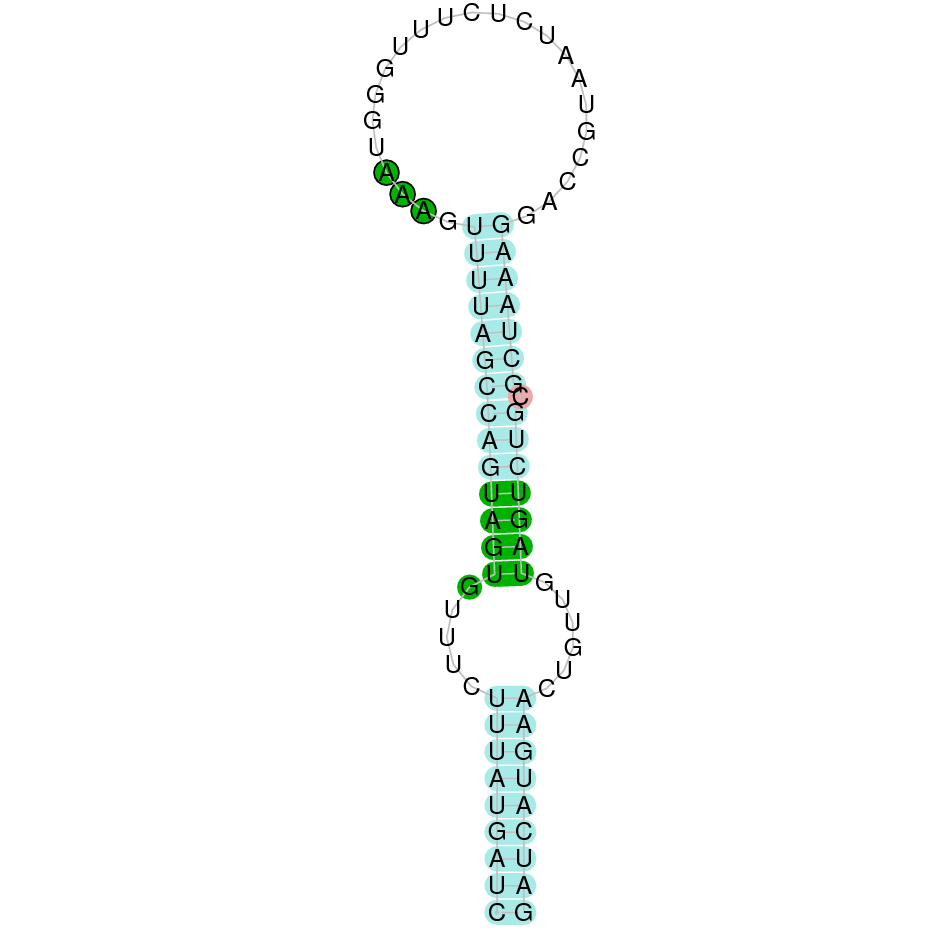 |
 |
|
| DIO3 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000029.1 | Negativa | 2957466-2958239 | 1 |  |
 |
 |
 |
 |
|
| GLUTATIÓ PEROXIDASES | ||||||||||||||
| GPx1 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000489.1 | Positiva | 151885-152826 | 2 |  |
 |
 |
 |
 |
 |
| GPx2 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000490.1 | Negativa | 149954-150712 | 2 |  |
 |
 |
 |
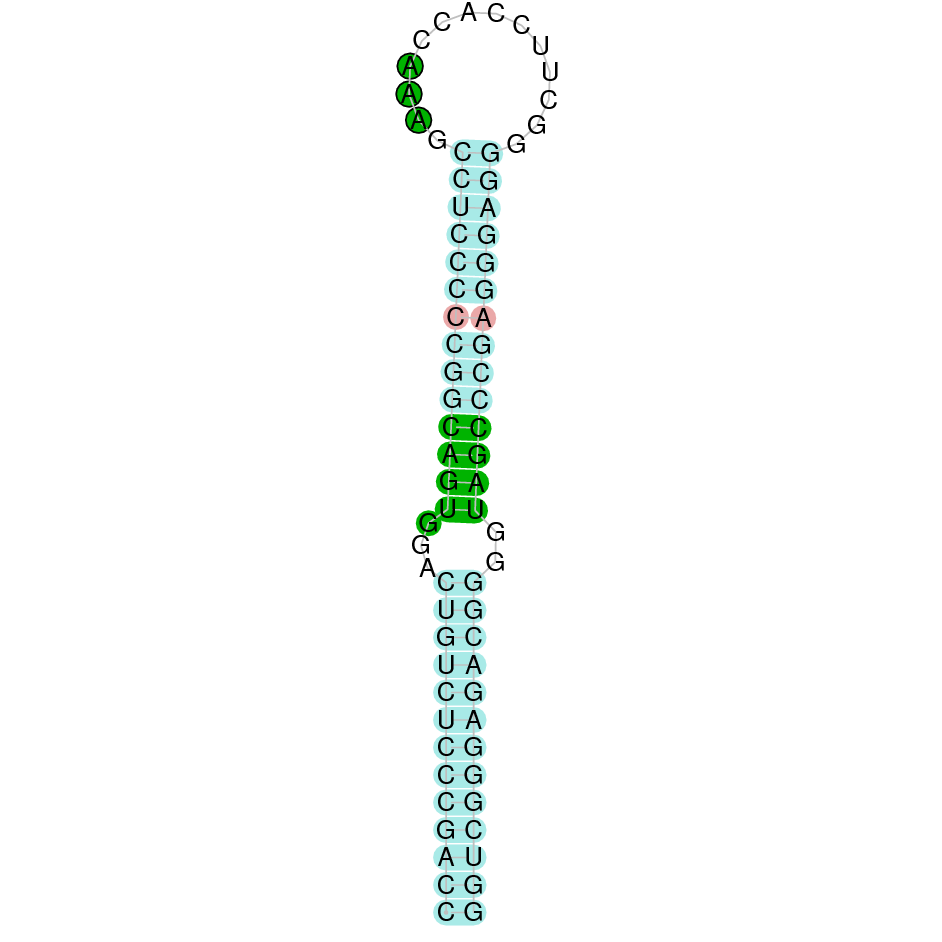 |
 |
| GPx3 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000118.1 | Positiva | 145698-146847 | 4 |  |
 |
 |
 |
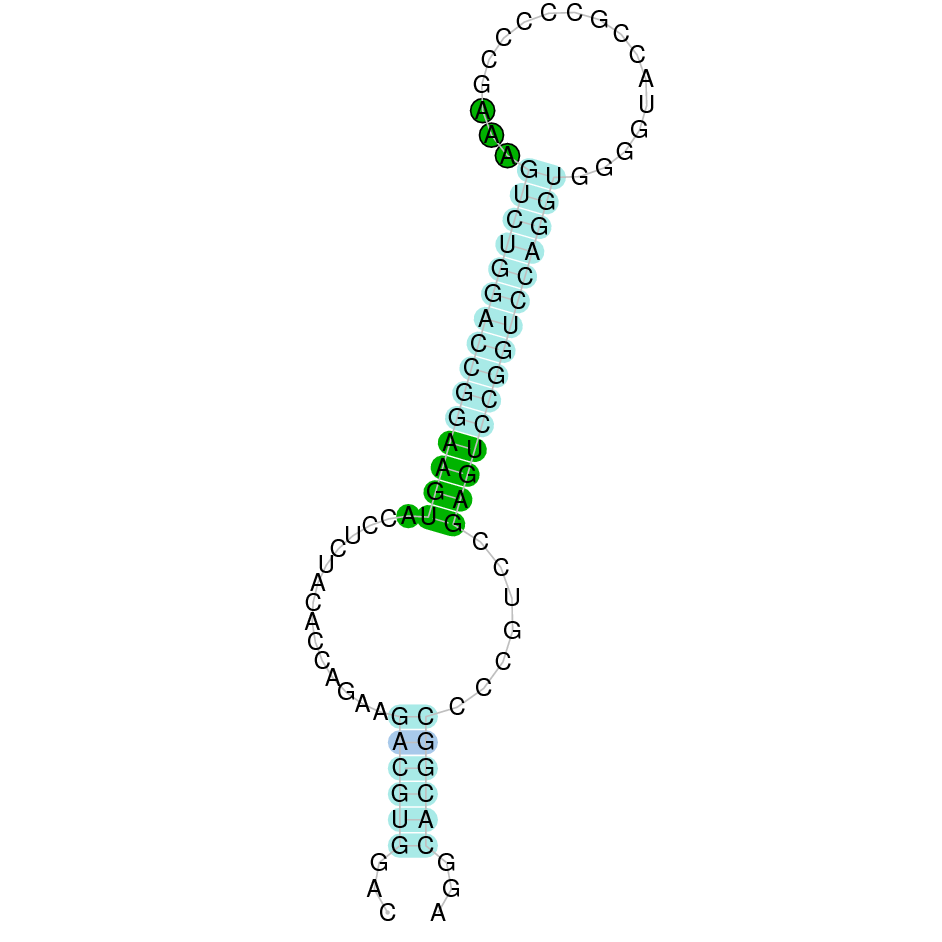 |
 |
| GPx3 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000118.1 | Positiva | 145698-146850 | 4 |  |
 |
 |
 |
 |
GPx4 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000256.1 | Positiva | 735385-736594 | 4 |  |
 |
 |
 |
 |
 |
| GPx5 | Homo sapiens | Cys-Sec |  |
 |
PTFA01000118.1 | Positiva | 144159-146838 | 5 |  |
 |
 |
 |
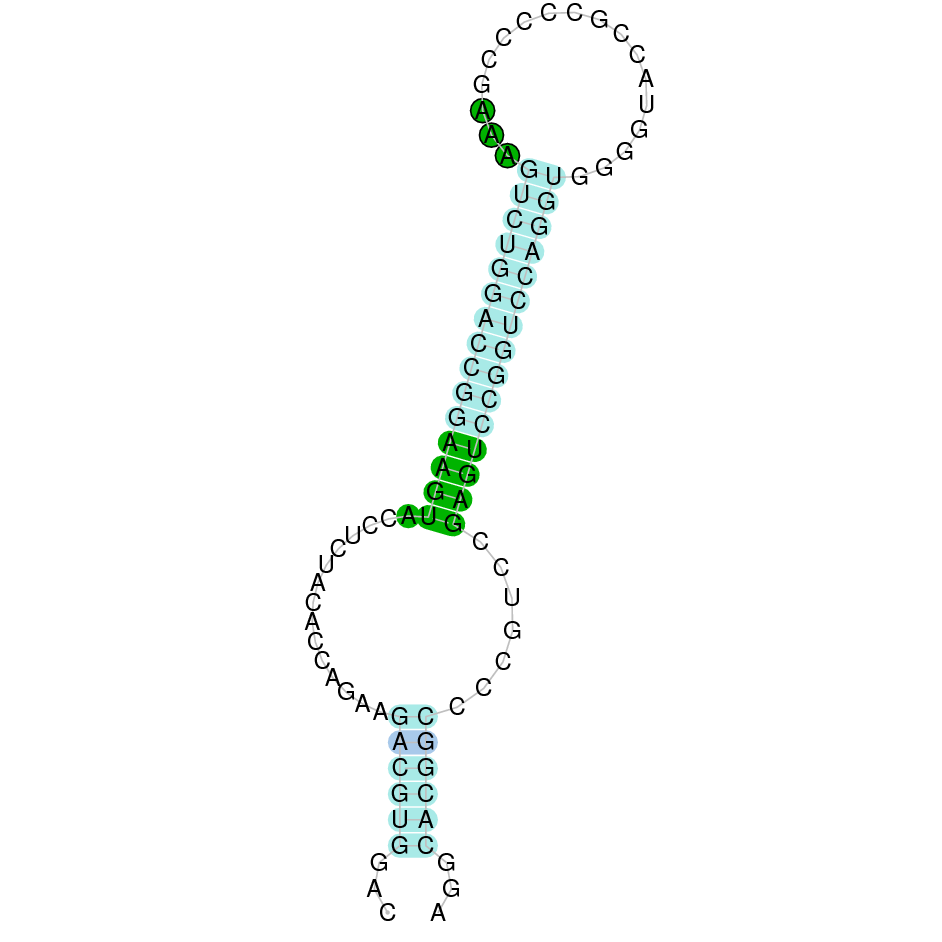 |
 |
| GPx6 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000118.1 | Positiva | 145698-146838 | 4 |  |
 |
 |
 |
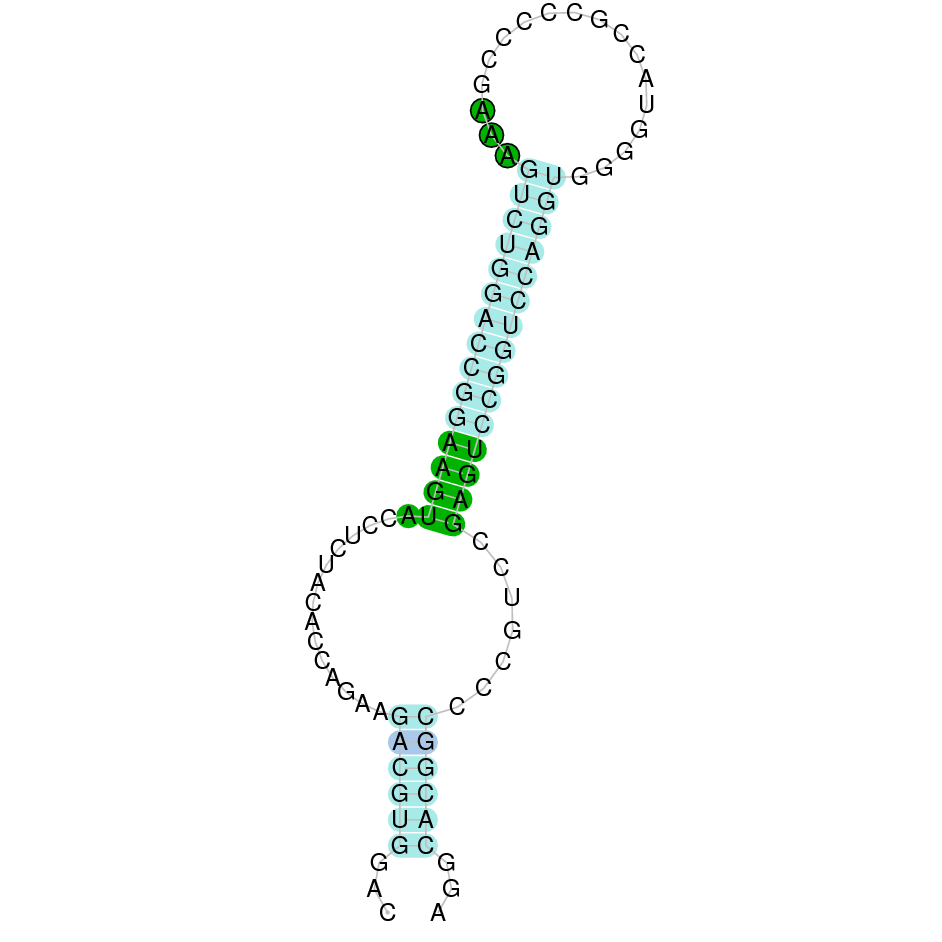 |
 |
| GPx7 | Homo sapiens | Cys-Cys |  |
 |
PTFA0100081.1 | Positiva | 1162472-1171228 | 3 |  |
 |
 |
 |
___ |  |
| GPx7 | Gallus gallus | Cys-Cys |  |
 |
PTFA01000081.1 | Positiva | 1168980-1171201 | 2 |  |
 |
 |
 |
___ |  |
| GPx8 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000055.1 | Positiva | 2037139-2041254 | 3 |  |
 |
 |
 |
___ |  |
| GPx8 | Gallus gallus | Cys-Cys |  |
 |
PTFA01000055.1 | Positiva | 2037973-2041221 | 2 |  |
 |
 |
 |
___ |  |
| SELENOPROTEÏNES O | ||||||||||||||
| SelO | Homo sapiens | Sec-Sec |  |
 |
PTFA01000225.1 | Positiva | 722058-733881 | 10 |  |
 |
 |
 |
 |
 |
| SelO | Gallus gallus | Sec-Sec |  |
 |
PTFA01000225.1 | Positiva | 722013-733881 | 9 |  |
 |
 |
 |
 |
|
| SELENOPROTEÏNES DE 15 kDa I SELENOPROTEÏNA M | ||||||||||||||
| Sel15 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000049.1 | Negativa | 380801-393167 | 2 |  |
 |
 |
 |
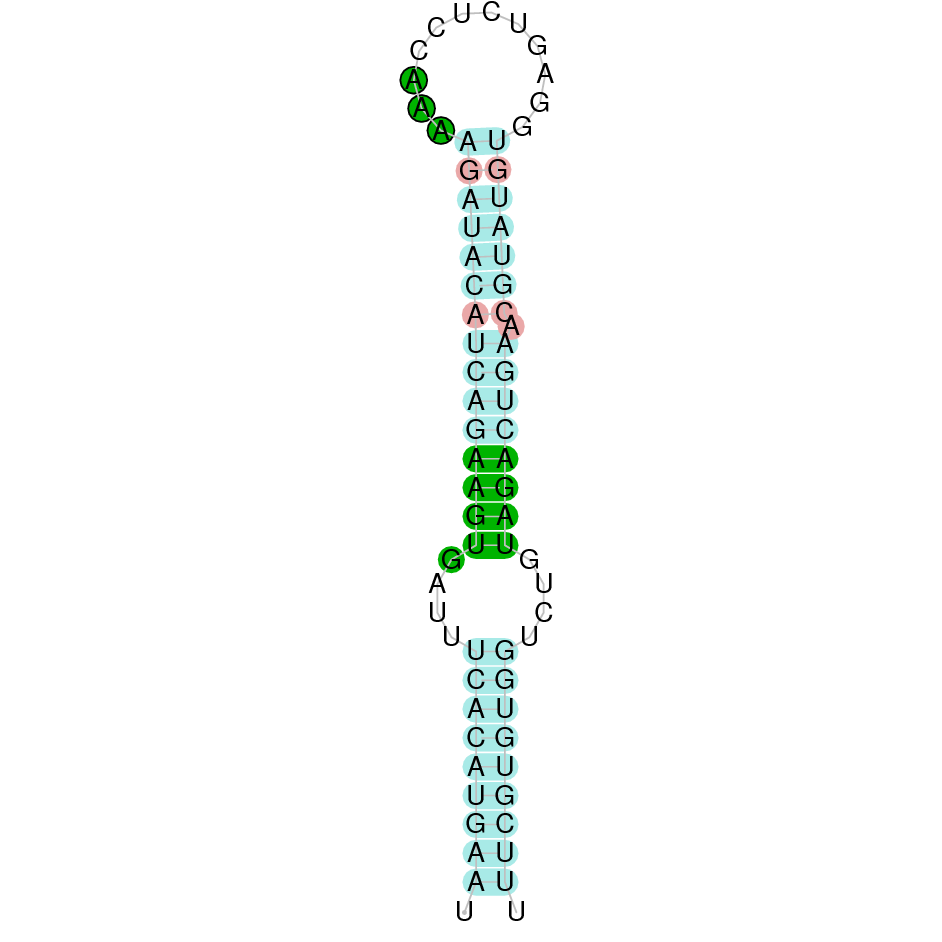 |
 |
| Sel15 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000049.1 | Negativa | 369512-393167 | 4 |  |
 |
 |
 |
 |
|
| SelM | Homo sapiens | Sec- |  |
 |
PTFA01000067.1 | Positiva | 31425-31979 | 3 |  |
 |
 |
 |
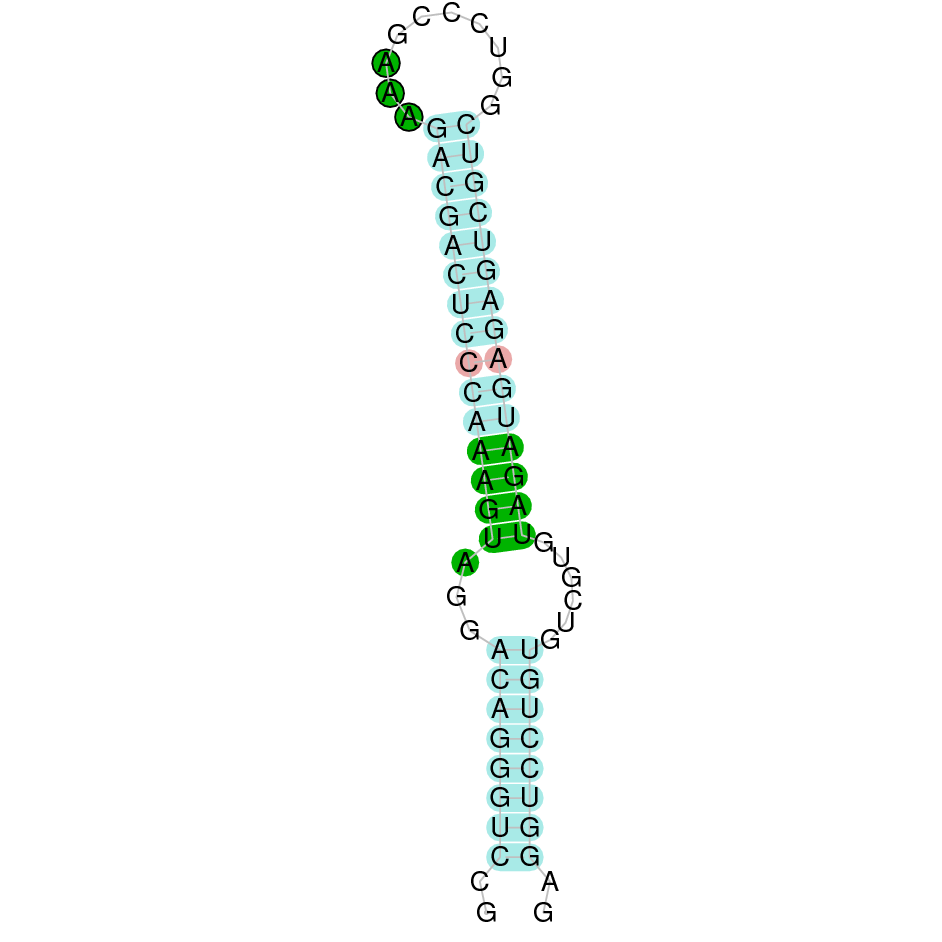 |
 |
| SELENOPROTEÏNES H | ||||||||||||||
| SelH | Homo sapiens | Sec-Sec |  |
 |
PTFA01000454.1 | Negativa | 338117-338384 | 2 |  |
 |
 |
 |
___ |  |
| SelH | Gallus gallus | _____ |  |
 |
_____ | _____ | _____ | _____ |  |
 |
 |
 |
____ | |
| SELENOPROTEÏNES I | ||||||||||||||
| SelI | Homo sapiens | Sec-Sec |  |
 |
PTFA01000351.1 | Positiva | 557690-587420 | 10 |  |
 |
 |
 |
 |
 |
| SelI | Gallus gallus | Sec-Sec |  |
 |
PTFA01000351.1 | Positiva | 557699-587438 | 10 |  |
 |
 |
 |
 |
|
| SELENOPROTEÏNES N | ||||||||||||||
| SelN | Homo sapiens | Sec-Sec |  |
 |
PTFA01000044.1 | Positiva | 4026964-4039187 | 11 |  |
 |
 |
 |
 |
 |
| SelN | Gallus gallus | Sec-Sec |  |
 |
PTFA01000044.1 | Positiva | 4022877-4039187 | 12 |  |
 |
 |
 |
 |
|
| SELENOPROTEÏNES P | ||||||||||||||
| SelP | Homo sapiens | Sec-Sec |  |
 |
PTFA01000286.1 | Positiva | 999390-1005409 | 4 |  |
 |
 |
 |
  |
 |
| SelP | Gallus gallus | Sec-Sec |  |
 |
PTFA01000286.1 | Positiva | 999390-1005643 | 4 |  |
 |
 |
 |
 |
|
| SELENOPROTEÏNES S I K | ||||||||||||||
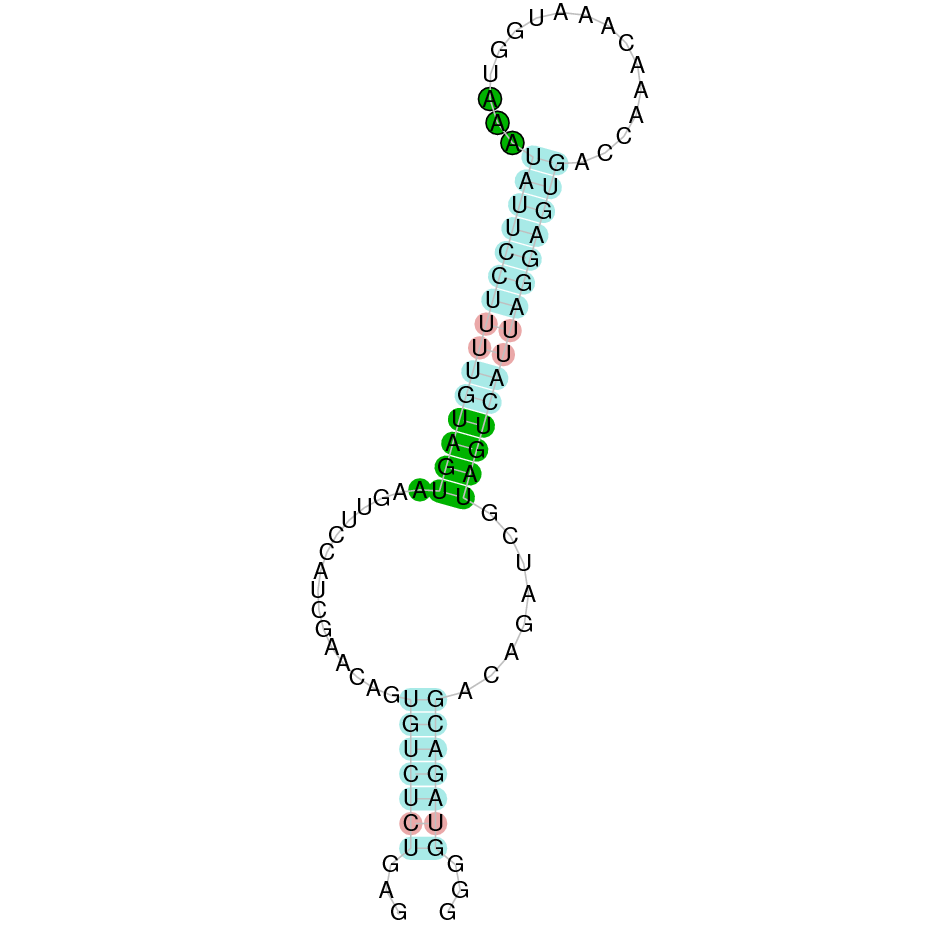
| SelS | Homo sapiens | Cys-Cys |  |
 |
PTFA01000083.1 | Negativa | 2010482-2016807 | 5 |  |
 |
 |
 |
 |
 |
| SelS | Gallus gallus | Sec-Sec |  |
 |
PTFA01000083.1 | Negativa | 2010482-2016846 | 6 |  |
 |
 |
 |
 |
|
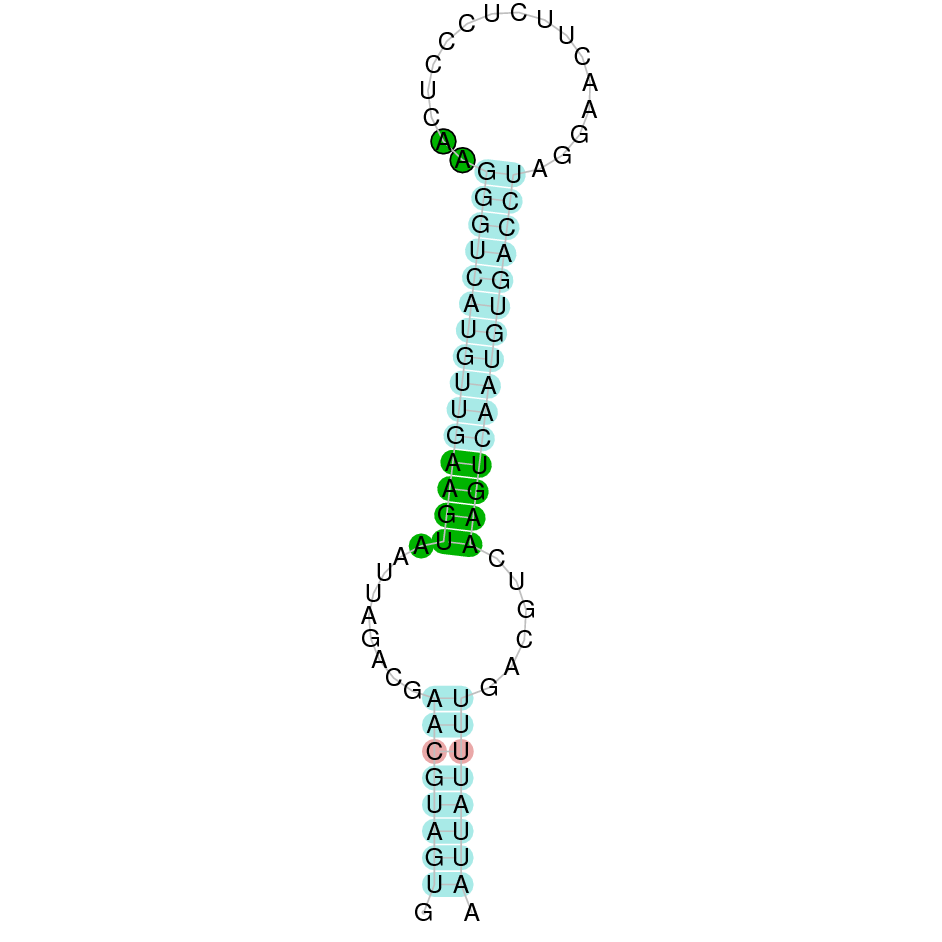
| SelK | Homo sapiens | Sec-Sec |  |
 |
PTFA01000114.1 | Negativa | 3258008-3260651 | 3 |  |
 |
 |
 |
 |
 |
| SelK | Gallus gallus | Sec-Sec |  |
 |
PTFA01000114.1 | Negativa | 3257996-3260651 | 4 |  |
 |
 |
 |
 |
|
| SELENOPROTEÏNES T | ||||||||||||||
| SelT | Homo sapiens | Sec-Sec |  |
 |
PTFA01000026.1 | Negativa | 6704276-6710238 | 5 |  |
 |
 |
 |
 |
 |
| SelT | Gallus gallus | Sec-Sec |  |
 |
PTFA01000026.1 | Negativa | 6704276-6710236 | 5 |  |
 |
 |
 |
 |
|
| SELENOPROTEÏNES U | ||||||||||||||
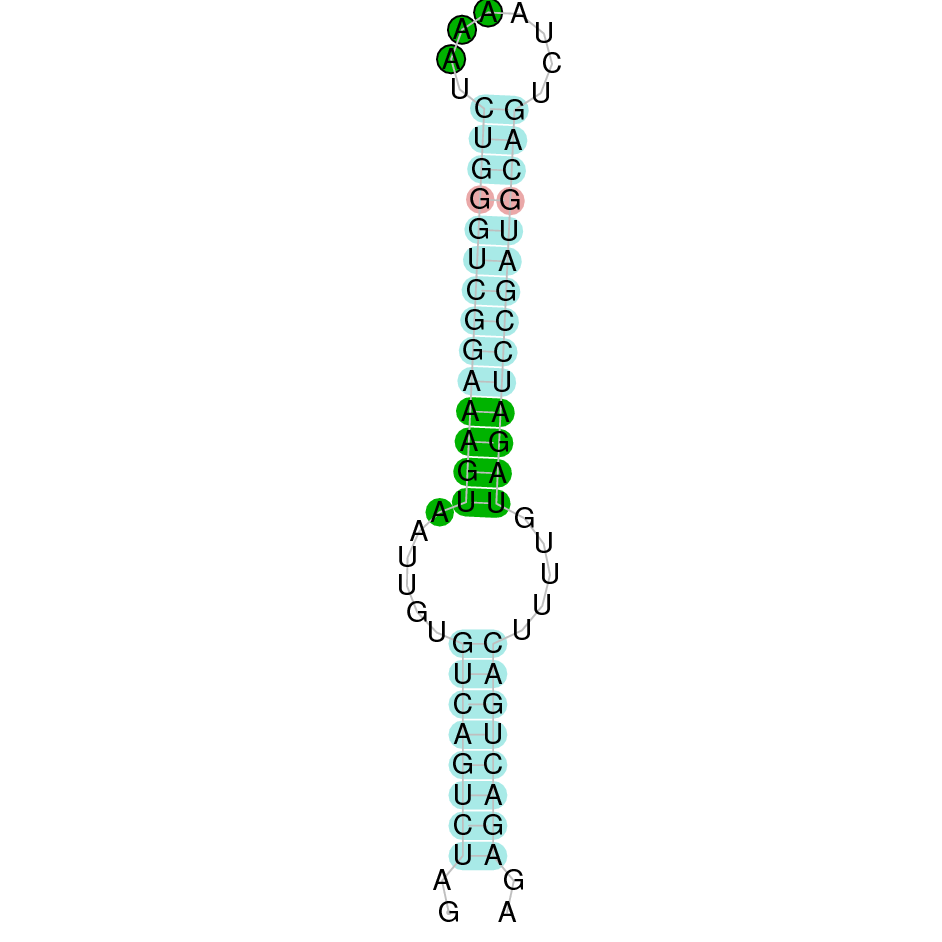
| SelU1 | Homo sapiens | Cys-Sec |  |
 |
PTFA01000016.1 | Positiva | 4732207-4737987 | 5 |  |
 |
 |
 |
 |
 |
| SelU1 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000016.1 | Positiva | 4732198-4737975 | 5 |  |
 |
 |
 |
 |
|
| SelU2 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000024.1 | Positiva | 5214638-5218545 | 6 |  |
 |
 |
 |
___ |  |
| SelU3 | Homo sapiens | ___ |  |
 |
___ | ___ | ___ | ___ |  |
 |
 |
 |
___ |  |
| SELENOPROTEÏNES W | ||||||||||||||
| SelV | Homo sapiens | ___ |  |
 |
___ | ___ | ___ | ___ |  |
 |
 |
 |
___ |  |
| SELENOPROTEÏNES W | ||||||||||||||
| SelW1 | Homo sapiens | ___ |  |
 |
___ | ___ | ___ | ___ |  |
 |
 |
 |
____ |  |
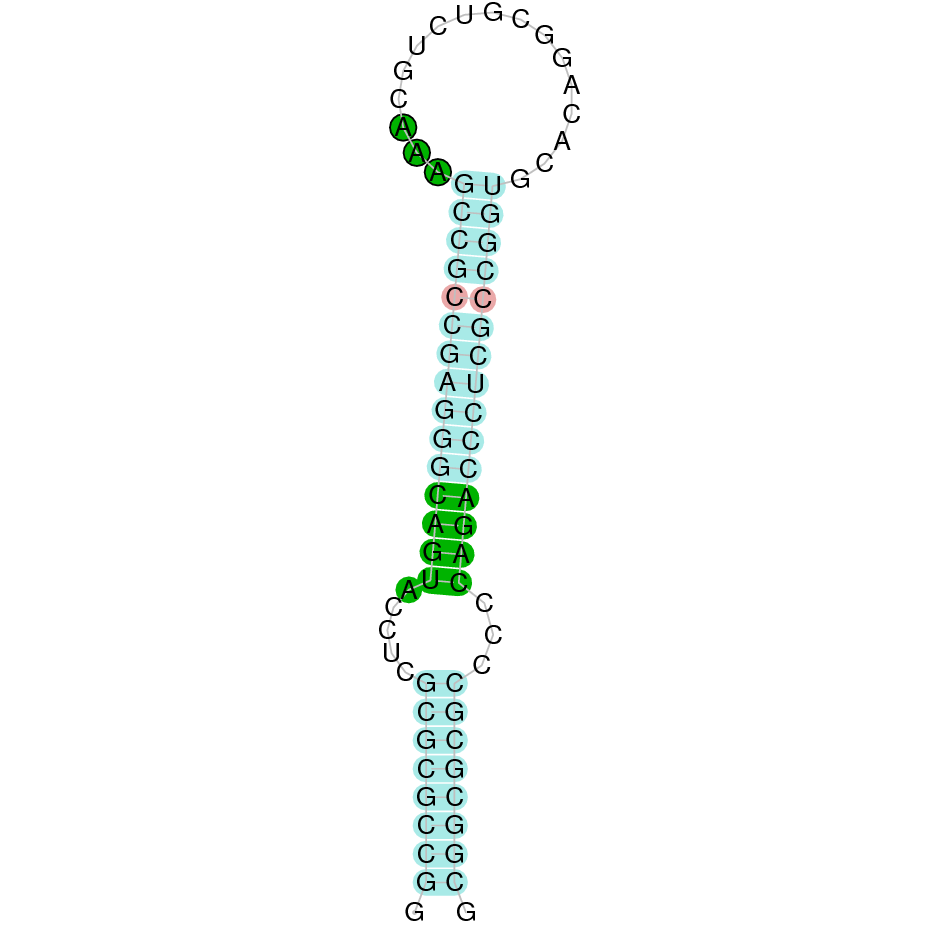
| SelW1 | Gallus gallus | ___ |  |
 |
PTFA01000548.1 | Negativa | 15984-16161 | 2 |  |
 |
 |
 |
 |
 |
| SelW2 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000190.1 | Positiva | 143433-144635 | 3 |  |
 |
 |
 |
____ |  |
| TIOREDOXINA REDUCTASES | ||||||||||||||
| TR1 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000005.1 | Positiva | 8384151-8408854 | 13 |  |
 |
 |
 |
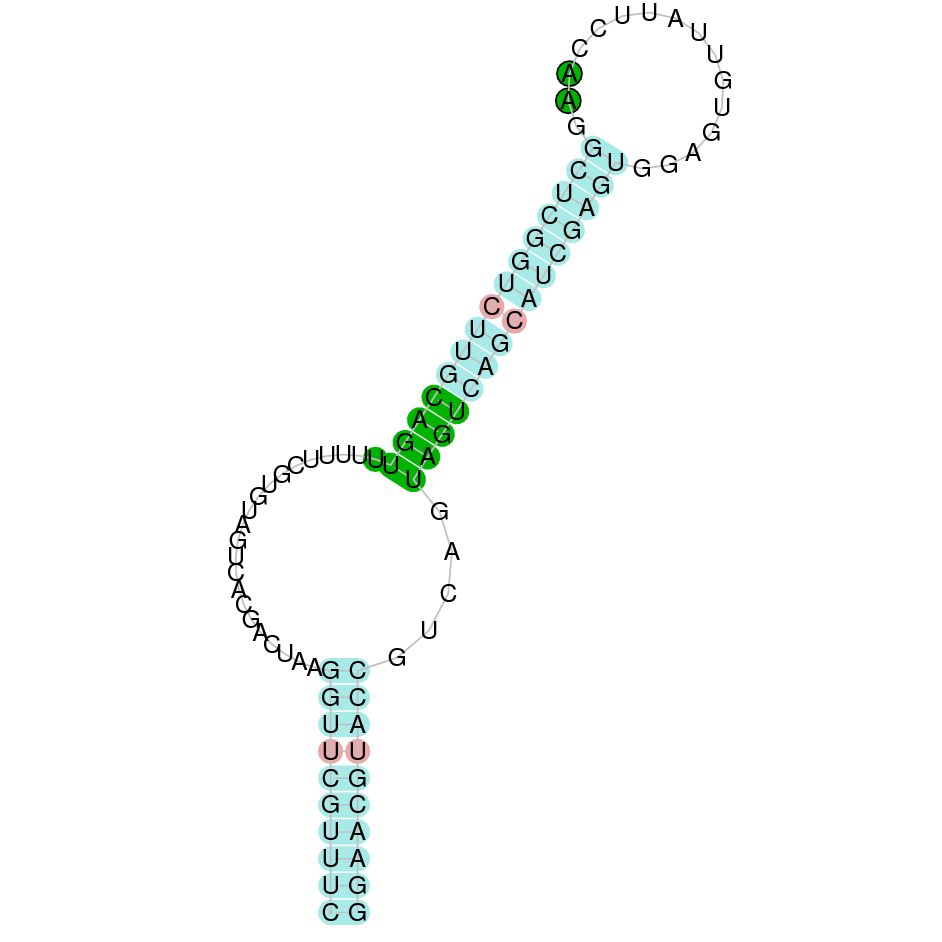 |
 |
| TXNRD | Gallus gallus | Sec-Sec |  |
 |
PTFA01000005.1 | Positiva | 8384109-8408854 | 13 |  |
 |
 |
 |
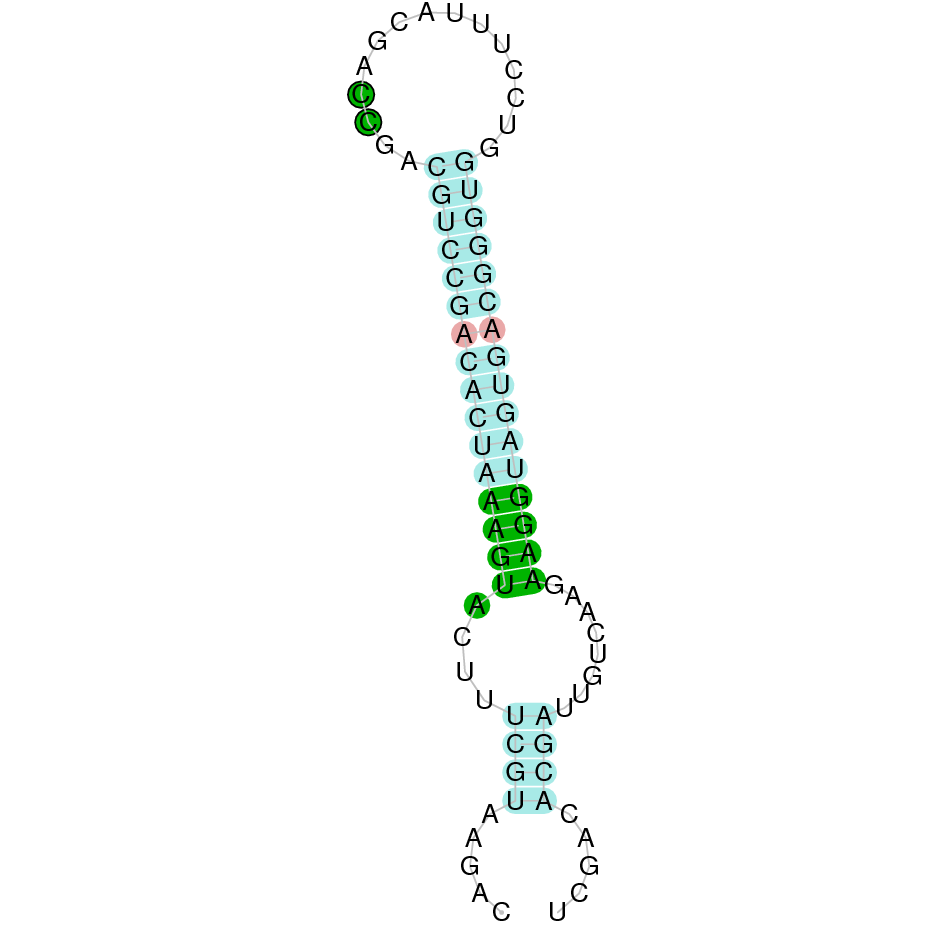 |
 |
| TR2 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000112.1 | Negativa | 1331479-1365484 | 15 |  |
 |
 |
 |
 |
 |
| TXNRD2 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000112.1 | Negativa | 1331479-1366531 | 16 |  |
 |
 |
 |
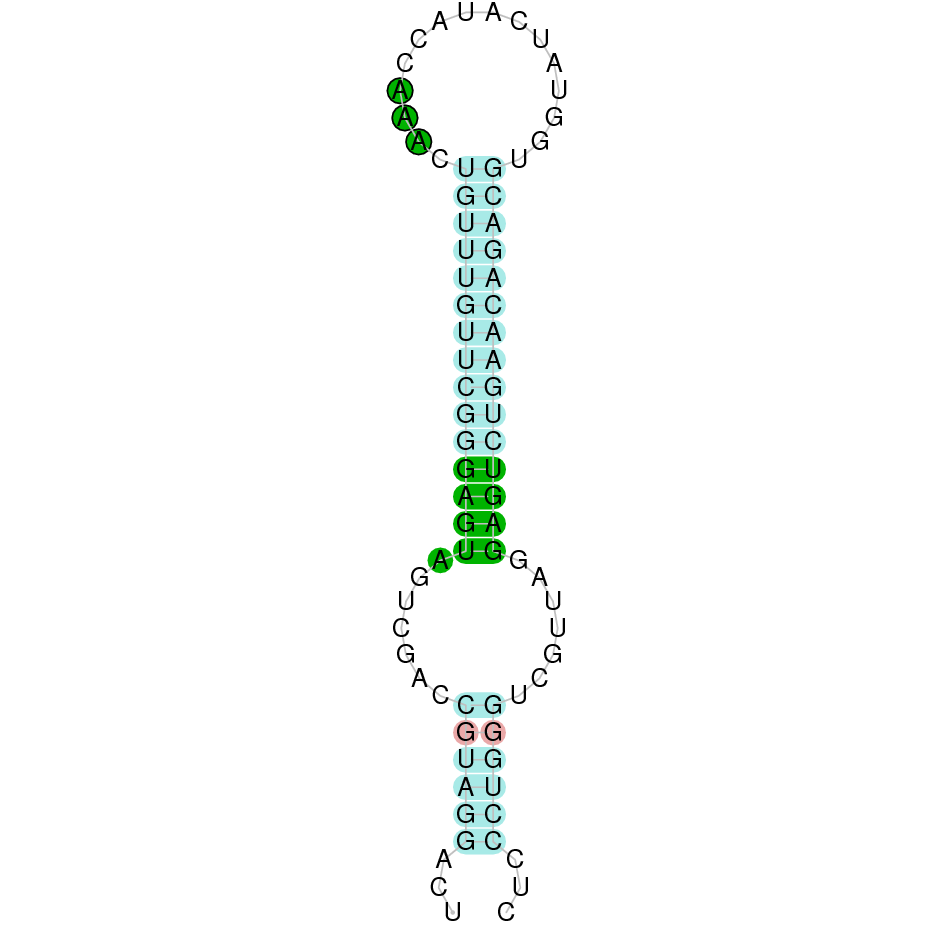 |
 |
| TR3 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000138.1 | Negativa | 1438202-1459331 | 15 |  |
 |
 |
 |
 |
 |
| TXNRD3 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000138.1 | Negativa | 1438202-1459367 | 16 |  |
 |
 |
 |
 |
|
| METIONINA SULFÒXID REDUCTASES | ||||||||||||||
| SelR1 | Homo sapiens | Sec-Sec |  |
 |
PTFA01000482.1 | Positiva | 162428-163568 | 3 |  |
 |
 |
 |
 |
 |
| MSRB1 | Gallus gallus | Sec-Sec |  |
 |
PTFA01000482.1 | Positiva | 162428-163559 | 3 |  |
 |
 |
 |
 |
|
| SelR2 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000041.1 | Positiva | 348660-355563 | 4 |  |
 |
 |
 |
___ |  |
| SelR3 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000034.1 | Positiva | 5749142-5788135 | 3 |  |
 |
 |
 |
___ |  |
| MSRB3 | Gallus gallus | Cys-Cys |  |
 |
PTFA01000034.1 | Positiva | 5749142-5788135 | 3 |  |
 |
 |
 |
___ |  |
| MrsA | Homo sapiens | Cys-Cys |  |
 |
PTFA01000233.1 | Negativa | 303531-366828 | 3 |  |
 |
 |
 |
___ |  |
| MrsA | Gallus gallus | Cys-Cys |  |
 |
PTFA01000233.1 | Negativa | 198054-198218 | 1 |  |
 |
 |
 |
___ | 
|
| MAQUINÀRIA | ||||||||||||||
| FOSFOSERIL-tRNA QUINASES | ||||||||||||||
| PSTK | Homo sapiens | Cys-Cys |  |
 |
PTFA01000061.1 | Negativa | 4935701-4938134 | 5 |  |
 |
 |
 |
____ |  |
| PSTK | Gallus gallus | Cys-Cys |  |
 |
PTFA01000061.1 | Negativa | 4935301-4934143 | 6 |  |
 |
 |
 |
____ |  |
| FACTORS D'ELONGACIÓ ESPECÍFICS DE SELENOCISTEÏNA | ||||||||||||||
| eEFSec | Homo sapiens | Cys-Cys |  |
 |
PTFA01000138.1 | Positiva | 2579842-2633193 | 5 |  |
 |
 |
 |
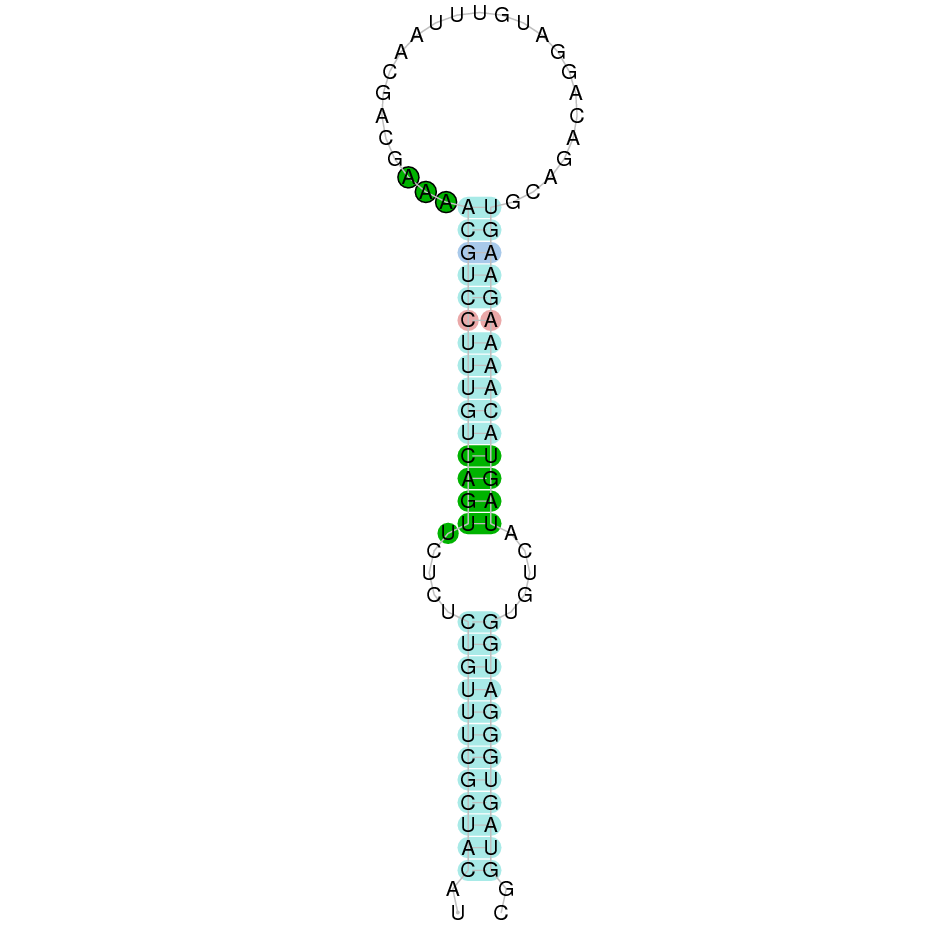 |
 |
| eEFSec | Gallus gallus | Cys-Cys |  |
 |
PTFA01000138.1 | Positiva | 257942-2633103 | 5 |  |
 |
 |
 |
 |
|
| PROTEÏNES D'UNIÓ D'ELEMENTS SECIS 2 | ||||||||||||||
| SBP2 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000007.1 | Negativa | 3076306-3101727 | 11 |  |
 |
 |
 |
____ |  |
| SBP2 | Gallus gallus | Iso-Sec |  |
 |
PTFA01000007.1 | Negativa | 5794568-5813398 | 16 |  |
 |
 |
 |
___ |  |
| SELENOFOSFAT SINTASES | ||||||||||||||
| SPS1 | Homo sapiens | ___ |  |
 |
PTFA01001172.1 | Negativa | 13137-13610 | 2 |  |
 |
 |
 |
___ |  |
| SEPHS | Gallus gallus | Cys-Cys |  |
 |
PTFA01001172.1 | Negativa | 13131-13631 | 2 |  |
 |
 |
 |
___ |  |
| SPS2 | Homo sapiens | ___ |  |
 |
PTFA01001172.1 | Negativa | 13131-13631 | ___ |  |
 |
 |
 |
___ |  |
| SECp43 | ||||||||||||||
| SECp43 | Homo sapiens | Cys-Cys |  |
 |
PTFA01000044.1 | Negativa | 5033303-5046025 | 8 |  |
 |
 |
 |
___ |  |
| SECp43 | Gallus gallus | Cys-Cys |  |
 |
PTFA01000044.1 | Negativa | 5033327-5046025 | 7 |  |
 |
 |
 |
____ |  |
| SecS | ||||||||||||||
| SecS | Homo sapiens | Cys-Cys |  |
 |
PTFA01000163.1 | Positiva | 584857-608935 | 11 |  |
 |
 |
 |
____ |  |
| SecS | Gallus gallus | Cys-Cys |  |
 |
PTFA01000163.1 | Positiva | 584857-608872 | 11 |  |
 |
 |
 |
____ |  |
Figura 17. Taula d'el·laboració pròpia on es mostren els resultats obtinguts després de córrer els programes creats per tal de fer l'anàlisi de les selenoproteïnes en Casuarius casuarius.
A la següent taula es mostren les selenoproteïnes i proteïnes de maquinària anotadesal genoma d’Homo sapiens i les que s’esperen trobar al genoma de Casuarius casuarius en funció del que se sap sobre el selenogenoma aviar. Les dades d’aquesta taula han sigut extretes de la base de dades SelenoDB i de l’estudi de Mariotti M, et al. 2012 per a Homo sapiens, mentre que per a Casuarius casuarius s’han utilitzat els estudis de Mariotti M, et al. 2012 i Shuping Li, et al. 2018 i, per a la maquinària, la base de dades SelenoDB.


Figura 18. Taula d'el·laboració pròpia basada en diverses fonts en què es mostren les selenoproteïnes esperades en Casuarius casuarius respecte Homo sapiens. Fonts: [11], [20]
Per tal d’assolir l’objectiu plantejat en aquest projecte, en primer lloc es van utilitzar com a referència les proteïnes de la base de dades SelenoDB1 perquè aquestes estan anotades manualment i, per tant, les prediccions fetes a partir d’aquestes seran més fiables. Del SelenoDB1 l’especie més propera és l’humà. No obstant, com algunes proteïnes no estaven anotades en Homo sapiens o no s predien tal i com sesperava segons l'estudiat del selenogenoma aviar [11], es va comparar amb Gallus gallus, una espècie més propera filogenèticament.Les seves proteïnes es van obtenir del SelenoDB2. A més, també es trobava Meleagris gallopavo (gall d’indi), però faltaven bastantes proteïnes per anotar i les que hi eren, estaven incompletes.
Els criteris que s’han fet servir per a considerar una proteïna, selenoproteïna són que tingui com a mínim un element SECIS [16] i, idealment, com a mínim un residu Sec.
Per a fer la selecció dels subalineaments de l’scaffold escollit s’han tingut en compte diversos criteris, entre els quals es troben que sigui significatiu, que l’alineament tingui pocs gaps i que s’aliniï amb les posicions més inicials de la proteïna de referència.
Per altra banda, també es va realitzar la filogènia de les grans famílies de selenoproteïnes per tal de comprovar si els resultats obtinguts són semblants al que s’esperava segons el que ja s’havia descrit en estudis previs[15].
A continuació, es mostra l’anàlisi de les selenoproteïnes trobades i no trobades així com dels elements de la maquinària que s’han pogut trobaral genoma del Casuarius casuarius.

Símbols utilitzats per a la realització de les estructures genòmiques de les selenoproteïnes.
Les DIO són una família formada per enzims de deionidasa importants perquè catalitzen la pèrdua d'àtoms de iode de les hormones tiroidees de manera que regulen tant l'activació com la innactivació d'aquestes. En Casuarius casuarius s'han trobat tres proteïnes DIO (DIO1, DIO2 i DIO3) que mostren homologia intra-familiar. És una de les tres famílies de selenoproteïnes més estudiades al llarg del temps (juntament amb GPx i TR) i es troben en tots els vertebrats.
Al Blast s’han obtingut 3 hits significatius corresponents a diferents scaffolds. En primer lloc es va seleccionar l’scaffold PTFA01000029.1, que era el que presentava l’e-value més baix. No obstant això, si es feia d’aquesta manera, la DIO1 es trobavaal mateix scaffold i la mateixa posició que la DIO3, suggerint això que eren la mateixa proteïna. Segons la literatura consultada, se sap que, en les aus, DIO1 i DIO3 són dues proteïnes diferents [11][14]. De la resta d’scaffolds significatius, es va seleccionar el PTFA01000081.1, ja que és el que donava una millor predicció tenint en compte la selenoproteïna esperada segons el selenogenoma aviar [11][20]. Dins d’aquest es va seleccionar el segon subalineament en funció dels criteris inicials.
Segons el T-coffee és molt probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació molt alta, té molt pocs gaps i pocs canvis d’aminoàcids.
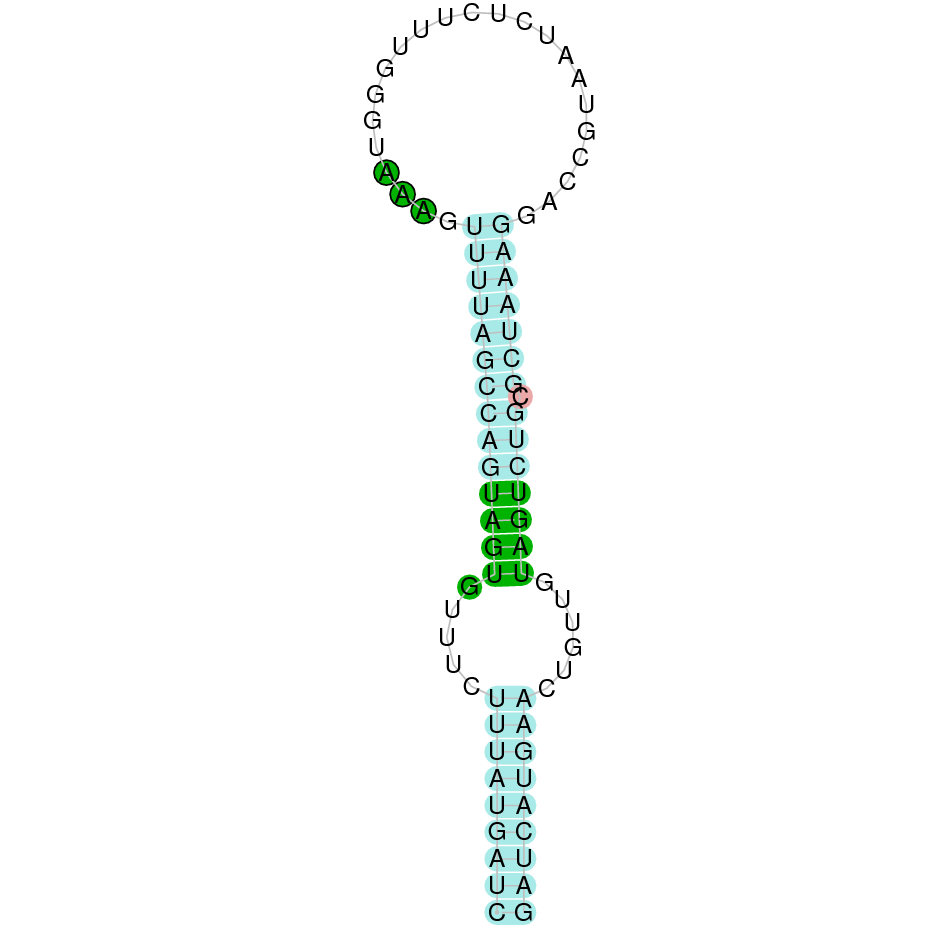
El gen predit es troba localitzat entre les posicions 2022456 i 2026567 en la cadena directa del genoma del Casuarius casuarius i conté 4111 nucleòtids.

A la figura s’observa que està formada per quatre exons i el residu Sec es troba al segon exó, més a prop de l’extrem N-terminal. Té un element SECIS a l’extrem 3’ entre les posicions 2027100 i 2027167.
A Blast s’han obtingut 3 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000143.1 que era el que presentava un e-value més baix. Dins d’aquest hi havia dos subalineaments, però només el primer s’alineava amb el residu U de la proteïna de referència i tenia un e-value menor, per tant, es va escollir aquest.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació molt alta, té pocs gaps i pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 662022 i 674752 en la cadena directa del genoma del Casuarius casuarius i conté 12730 nucleòtids.

A la figura s’observa que està format per dos exons i el residu Sec es troba a prop de l’extrem C-terminal al segon i últim exó que conté. Té un element SECIS a l’extrem 3’ entre les posicions 679284 i 679358.
Al Blast s’han obtingut 3 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000029.1 que era el que presentava un e-value més baix. Aquest només tenia un subalineament que és el que es va escollir.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació molt alta, té 1 gap gran enmig i un altre gap al final i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 2957496 i 2958287 en la cadena reversa del genoma del Casuarius casuarius i conté 791 nucleòtids.
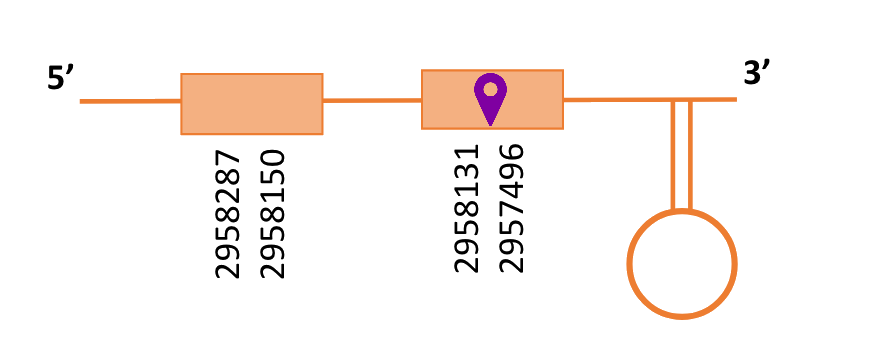
A la figura s’observa que està formada per dos exons i el residu Sec es troba al segon exó que conté. Té un element SECIS a l’extrem 3’ entre les posicions 2957084 i 2957004.

A la filogenia es pot observar que els tres tipus de selenoproteïnes DIO predites al genoma del Casuarius casuarius presenten una relació més estreta amb les selenoproteïnes del Gallus gallus que amb les de l’Homo sapiens. Això és el que s’esperava ja que ambdues espècies són aus i, per tant, són evolutivament més properes.
Aquesta família és una de les tres més estudiades al llarg del temps. A més, és la més granals vertebrats i està implicada en molts processos fisiològics. Té activitat peroxidasa amb la qual protegeix a l’organisme del dany oxidatiu.
En mamífers es troben altament conservades i hi ha 8 proteïnes homòlogues, de les quals cinc són selenoproteïnes (GPx1-4 i GPx6) i les altres han evolucionat a partir d’aquestes, amb la possibilitat que durant aquesta divergència hagin perdut la selenocisteïna. En el genoma del Casuarius casuarius s’han pogut predir les proteïnes GPx1, GPx2, GPx3, GPx4 i els homòlegs de cisteïna GPx7 i GPx8
Al Blast s’han obtingut 6 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000489.1 que era el que presentava un e-value més baix. Dins d’aquest hi havia 2 subalineaments, dels quals es va seleccionar el primer en funció dels criteris inicials.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta i té pocs gaps i pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 151885 i 152826 en la cadena directa del genoma del Casuarius casuarius i conté 941 nucleòtids.
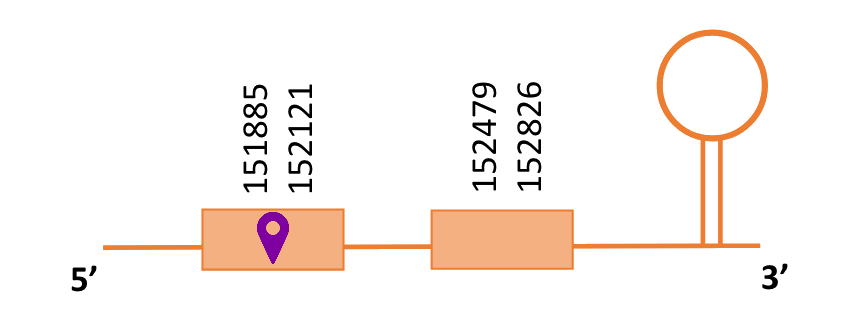
A la figura s’observa que està formada per dos exons i el residu Sec es troba a prop de l’extrem N-terminal,al primer exó. Té un element SECIS a l’extrem 3’ entre les posicions 153010 i 153081.
Al Blast s’han obtingut 6 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000490.1, que era el que presentava un e-value més baix. Dins d’aquest hi havia 2 subalineaments, dels quals es va agafar el primer segons els criteris inicials.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta, només té 2 gaps i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 150712 i 149954 en la cadena reversa del genoma del Casuarius casuarius i conté 758 nucleòtids.

A la figura s’observa que està formada per un exó i el residu Sec es troba a prop de l’extrem N-terminal, a l’únic exó que conté. Té un element SECIS a l’extrem 3’ entre les posicions 149891 i 149819.
Al blast s’han obtingut 5 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000118.1 que era el que presentava un e-value més baix. Dins d’aquest hi havia 3 subalineaments, dels quals es fa seleccionar el segon en funció dels criteris inicials.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta, té pocs gaps i pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 145698 i 146847 en la cadena directa del genoma del Casuarius casuarius i conté 1149 nucleòtids.
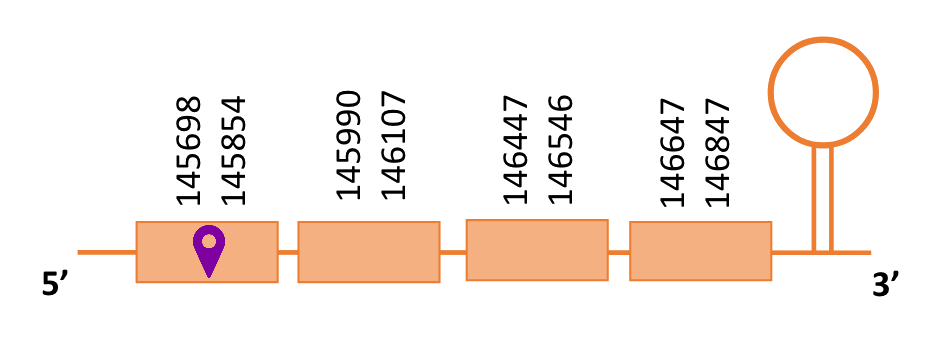
A la figura s’observa que està format per quatre exons i el residu Sec es troba a prop de l’extrem N-terminal,al primer exó. Té un element SECIS a l’extrem 3’ entre les posicions 147325 i 147412.
Al blast s’han obtingut 5 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000256.1, que era el que presentava un e-value més baix. Dins d’aquest hi havia 4 subalineaments dels quals es va escollir el primer segons els criteris inicials.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta, té pocs gaps i pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 735385 i 736594 en la cadena directa del genoma del Casuarius casuarius i conté 1209 nucleòtids.
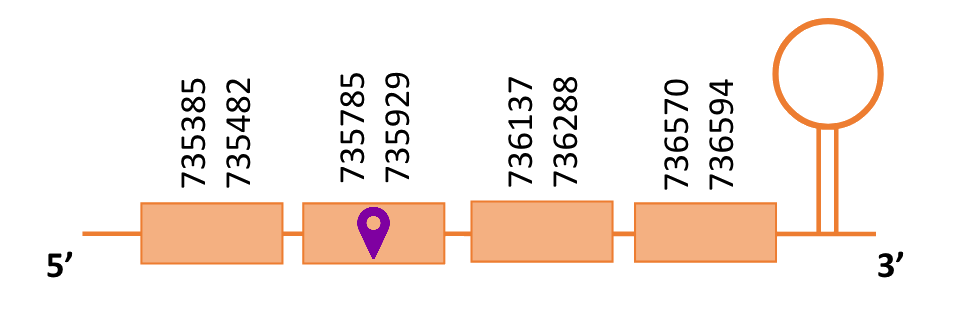
A la figura s’observa que està format per quatre exons i el residu Sec es troba a prop de l’extrem N-terminal, al segon exó. Té un element SECIS a l’extrem 3’ entre les posicions 737566 i 737637.
També s’han estudiat les proteïnes GPx5 i GPx6. S’ha vist que la localització del GPx5 i la del GPx6 es solapa amb la de GPx3 (mateix scaffold i mateixa posició). A la literatura s’ha vist que la GPx5 i la 6 es van generar per duplicació de la GPx3 en l’origen dels placentaris[15]. Per tant, al casuari, que es troba més enrere en l’escala evolutiva, no s’ha produït encara la duplicació i, per això, només presenta la GPx3.
Aquests dos membres de la família de les glutatió peroxidases també s’han estudiat. S’ha trobat que ambdós són homòlegs de Cisteïna que, a més, no tenen element SECIS. Per una banda, GPx7 s’ha trobat a l’scaffold PTFA01000081.1 entre les posicions 1162472 i 1171228 en la cadena directa i conté 3 exons. D’altra banda, GPx8 s’ha trobat a l’scaffold PTFA01000055.1 entre les posicions 2037139 i 2041254 i està formada per 3 exons.
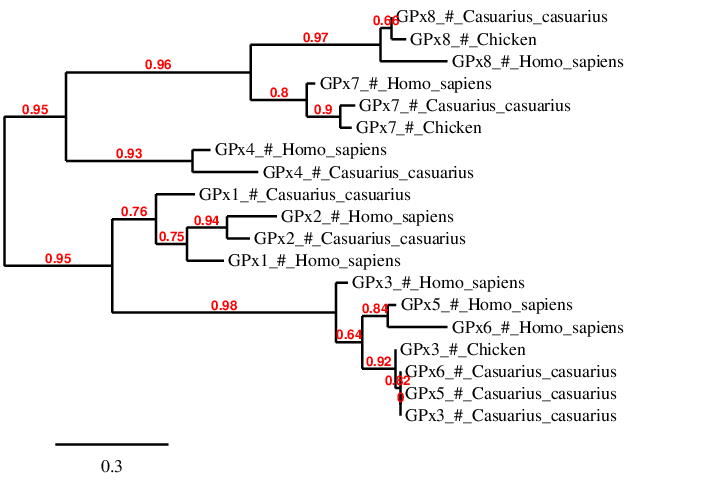
El que més destaca de la filogenia és que les proteïnes del Casuarius casuariuses troben més relacionades amb les del Gallus gallus que amb le de l’Homo sapiens.
Cal esmentar que la selenoproteïna GPx3, GPX5 i GPx6 del casuari es troben agrupades al GPx3 del pollastre. Això, juntament amb el que s’ha comentat anteriorment indica que són la mateixa proteïna.
Per altra banda, es pot observar també que GPX4 es troba més relacionada amb GPx7 i GPx8. Això concorda amb el fet que les dues últimes proteïnes van evolucionar d’un ancestre semblant a GPx4 [1].
Al Blast s’han obtingut dos hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000225.1 que era el que presentava un e-value més baix. Dins d’aquest hi havia 9 subalineaments, dels quals es va escollir el primer perquè era el més significatiu.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest scaffold degut a que, malgrat tingui dos grans gaps a l’inici, presenta una puntuació alta i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 722058 i 733881 en la cadena reversa del genoma del Casuarius casuarius i conté 11823 nucleòtids.
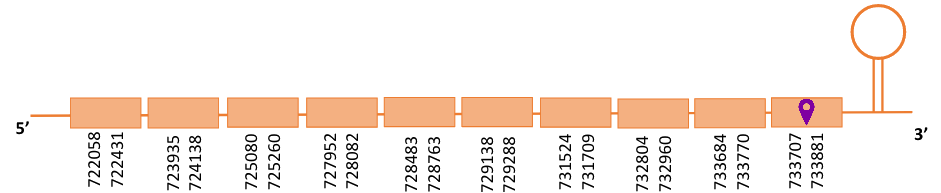
A la figura s’observa que està formada per deu exons i el residu Sec es troba a prop de l’extrem C-terminal, a l’últim exó que conté. Té un element SECIS a l’extrem 3’ entre les posicions 734204 i 734281.
Aquestes proteïnes, que són semblants a la tiorredoxina i són residents del reticle endoplasmàtic, formen una família distinctiva de selenoproteïnes. SelM i Sel15 són homòlegs distants. La seva funció encara no està clara avui dia.
En el Blast s’ha obtingut un hit significatiu corresponent al scaffold PTFA01000049.1, que presentava un e-value baix. Dins d’aquest hi havia tres subalineaments, dels quals es va escollir el primer perquè era el més significatiu i s’alineava amb les primeres posicions de la proteïna de referència.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació molt alta, només té dos gaps i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 380801 i 393167 en la cadena reversa del genoma del Casuarius casuarius i conté 12366 nucleòtids.
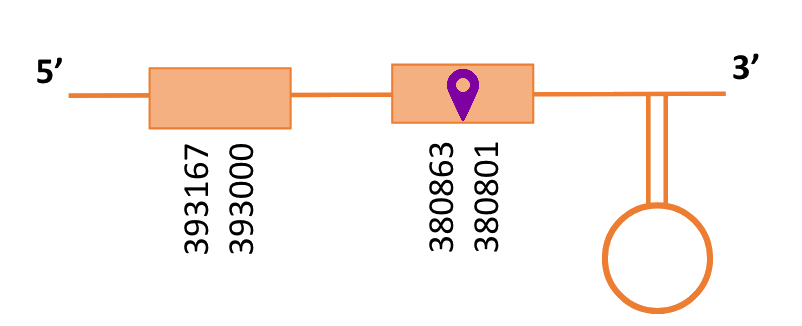
A la figura s’observa que està format per dos exons i el residu Sec es troba a prop de l’extrem N-terminal, a l’últim exó que conté. Té un element SECIS a l’extrem 3’ entre les posicions 368976 i 368906.
Al Blast s’ha obtingut un hit significatiu corresponent a l’scaffold PTFA01000067.1. Dins d’aquest, hi havia dos subalineaments i es va seleccionar el primer en funció dels criteris inicials.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta i hi ha pocs canvis d’aminoàcids. Tanmateix, hi ha un gap extens a l’inici de la seqüència que fa que la Sec de la proteïna de referència (Homo sapiens) no s’aliniï amb cap aminoàcid de la seqüència del casuari. Això podria ser per la pèrdua d’una part de la proteïna del casuari on es trobava la Sec, o bé perquè la proteïna podria estar dividida en dos scaffolds diferents que no podem determinar. En el cas de Gallus gallus, el residu Sec d’aquest s’alineava amb Valina. Tot això fa pensar que és possible que el casuari hagi perdut la Selenocisteïna.
El gen predit es troba localitzat entre les posicions 31425 i 31979 en la cadena directa del genoma del Casuarius casuarius i conté 554 nucleòtids.
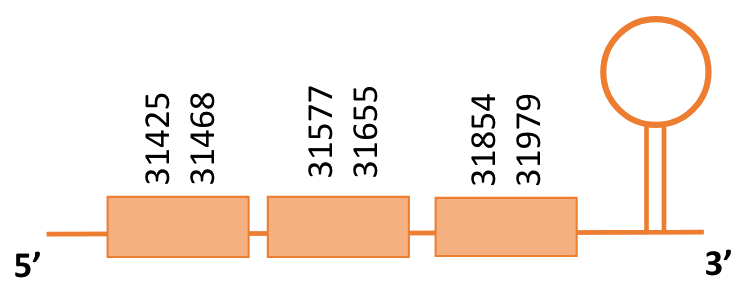
A la figura s’observa que està format per tres exons i no té residu Sec. El residu Sec de la proteïna de referència s’alinea amb un gap en la proteïna de Casuarius casuarius. Té un element SECIS a l’extrem 3’ entre les posicions 32479 i la posició 32547.
No s’ha pogut comparar amb la proteïna SelM de referència de Gallus gallus ja que no es trobava anotada al SelenoDB2.
En el blast s’han obtingut quatre hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000351.1 que era el que presentava un e-value més baix. Dins d’aquest hi havia 8 subalineaments, dels quals es va seleccionar el primer en funció dels criteris inicials.
Segons el T-coffee és molt probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació molt alta, no té cap gap i té molt pocs canvis d’aminoàcids.
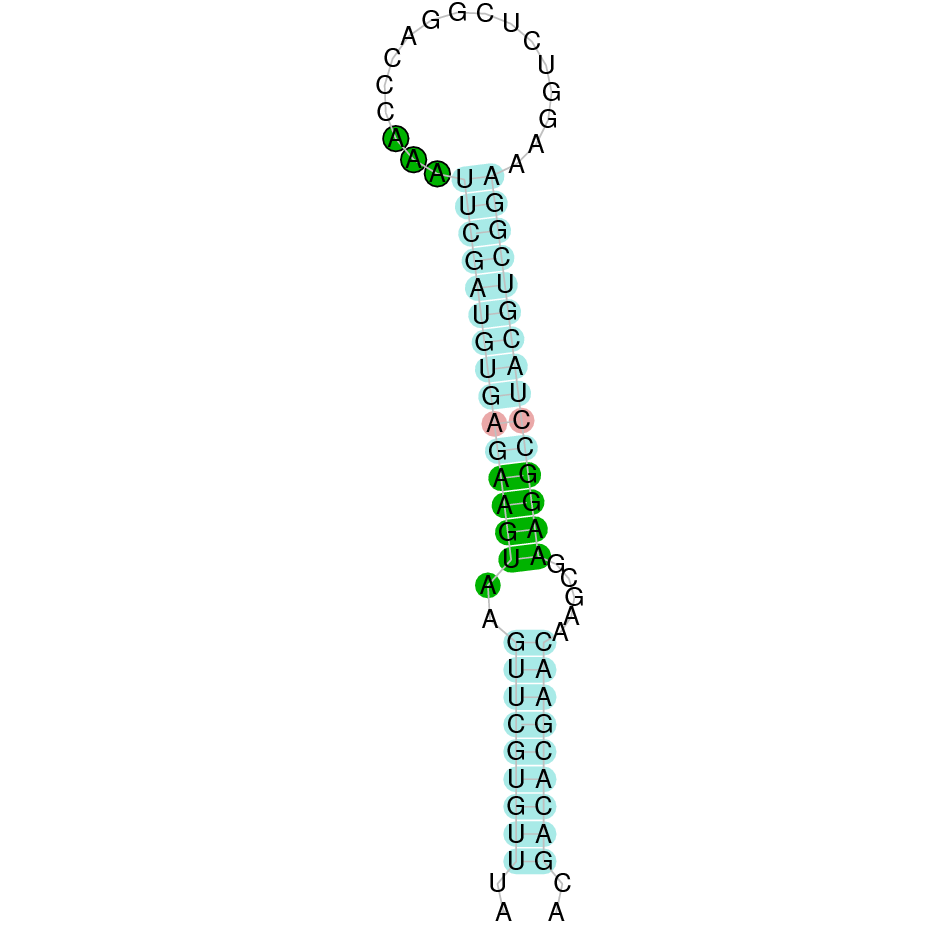
El gen predit es troba localitzat entre les posicions 557690 i 587420 en la cadena directa del genoma del Casuarius casuarius i conté 30000 nucleòtids.
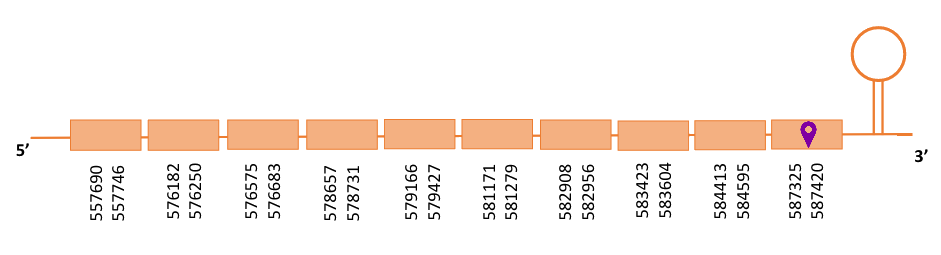
A la figura s’observa que està format per deu exons i el residu Sec es troba a prop de l’extrem C-terminal, a l’últim exó. Té un element SECIS a l’extrem 3’ entre les posicions 588619 i 588694.
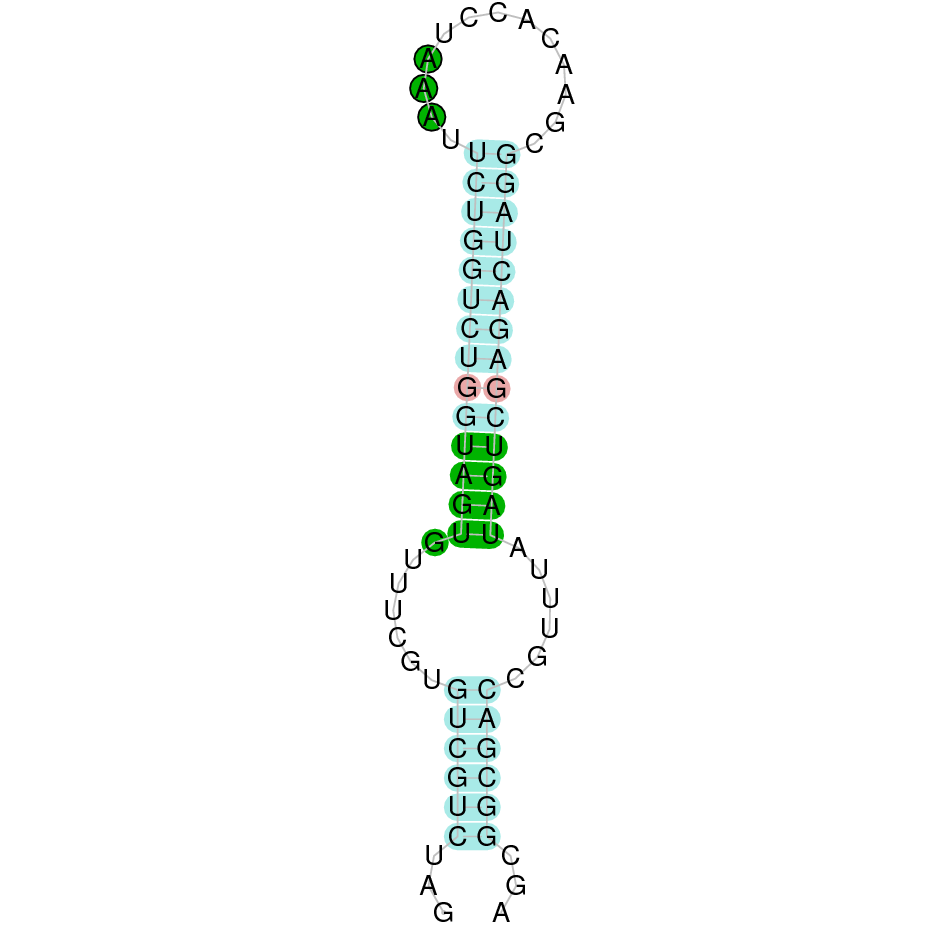
En el Blast s’han obtingut un hit significatiu corresponent a l’scaffold PTFA01000044.1 que era el que presentava un e-value més baix. Dins d’aquest només hi havia onze sub-alineaments i s’ha seleccionat el segon més significatiu i que es solapa amb posicions més inicials de la proteïna de referència d’Homo sapiens.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest contig degut a que presenta una puntuació alta i té pocs canvis d’aminoàcids. Tot i així s’ha de dir que presenta 2 gaps grans.
El gen predit es troba localitzat entre les posicions 4026964 i 4039187 en la cadena directa del genoma del Casuarius casuarius i conté 12223 nucleòtids.
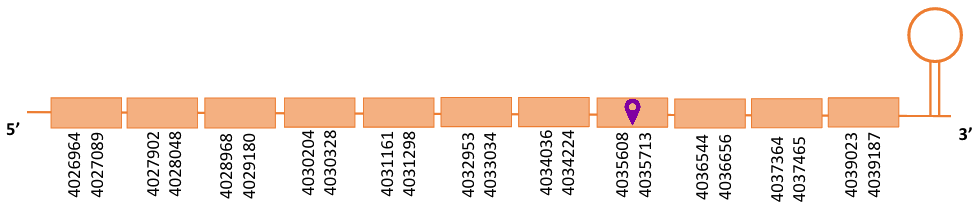
A la figura s’observa que està format per onze exons i el residu Sec es troba a prop de l’extrem C-terminal, concretament a l’exó 8. Té un element SECIS a l’extrem 3’ entre la posició 4041899 i la posició 4041970.
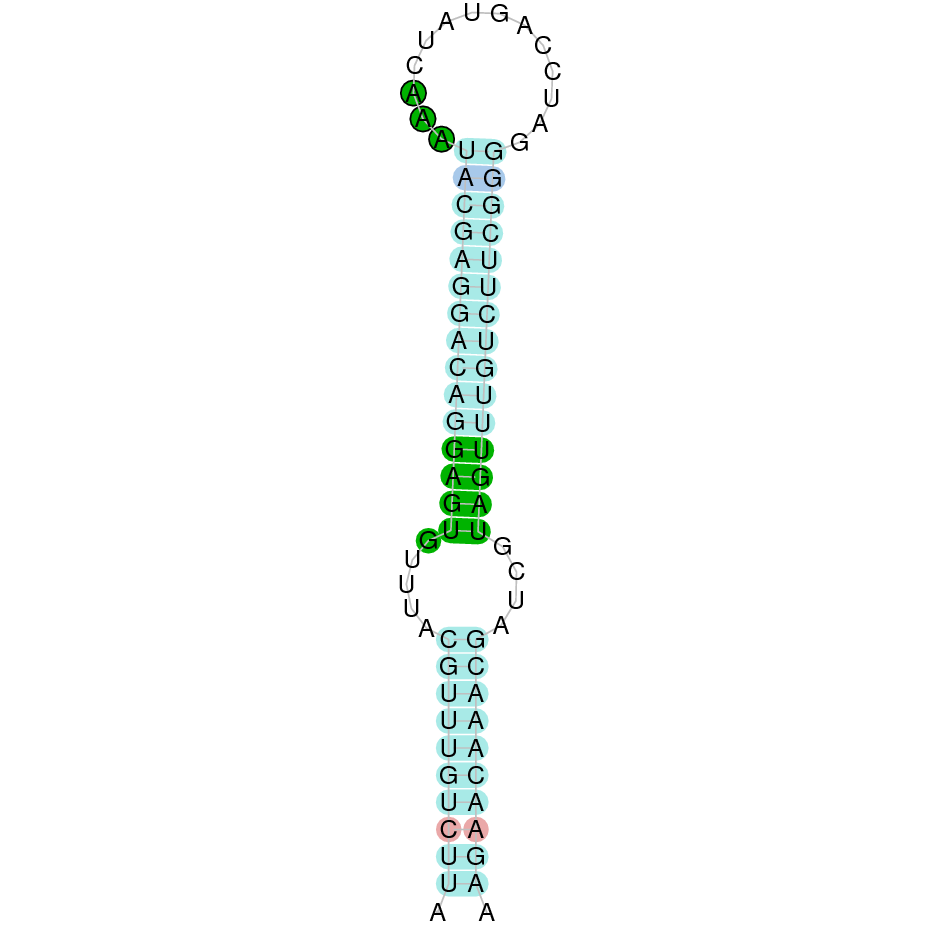
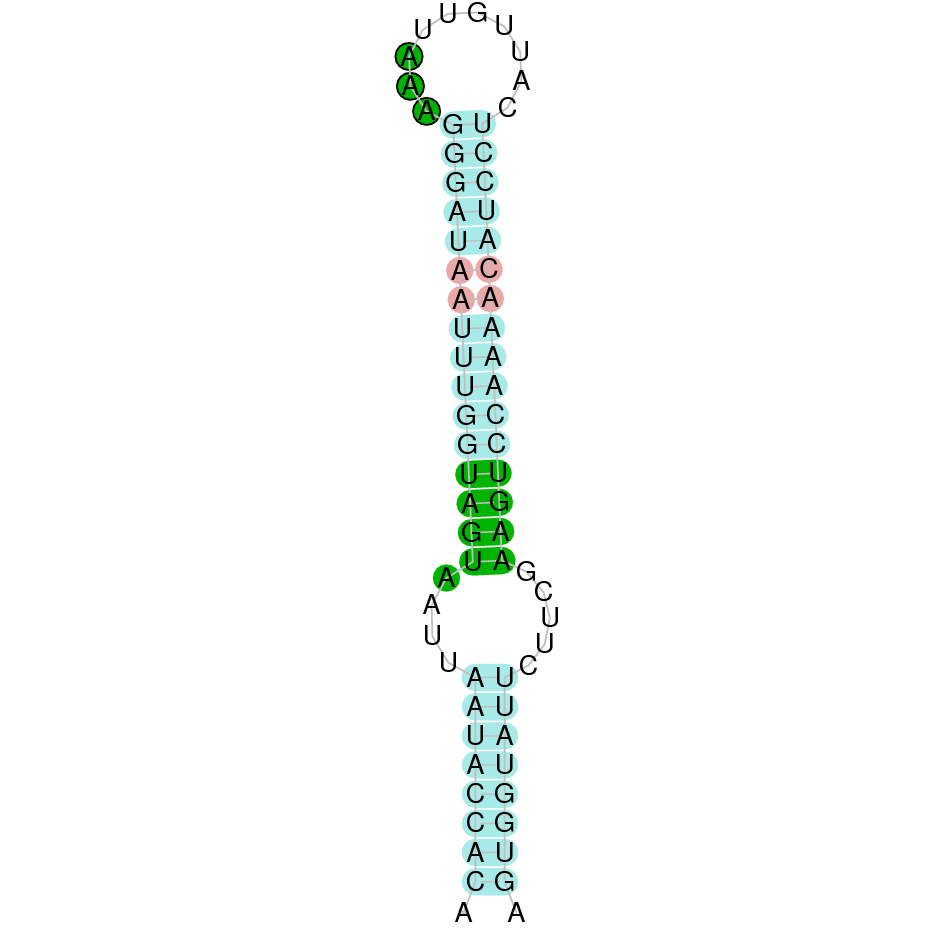
En el blast s’han obtingut dos hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000286.1 que era el que presentava un e-value més baix. Dins d’aquest hi havia 4 subalineaments, i s’ha escollit el més significatiu.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta, presenta pocs gaps i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 999390 i 1005409 en la cadena directa del genoma del Casuarius casuarius i conté 6019 nucleòtids.
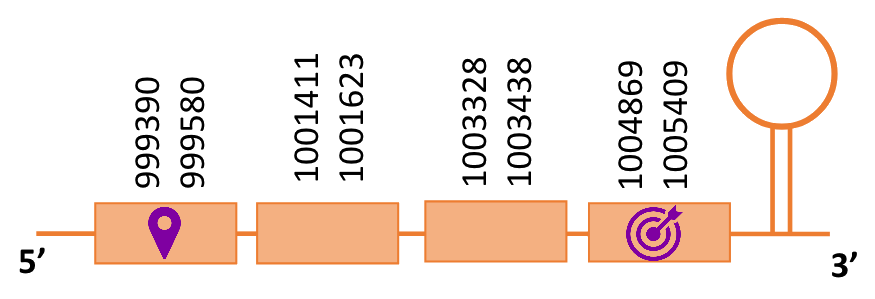
A la figura s’observa que està format per quatre exons i presenta caracteristicament una Sec al primer exò, a prop de l’N-terminal i dotze Sec a l’últim exò, a prop del C-terminal. Té un element SECIS a l’extrem 3’ entre les posicions 1005800 i 1005874.
Ara bé, s’ha de dir que utilitzant com a proteïna de referència la SelP de Gallus gallus s’han obtingut 2 SECIS que és el que s’esperava[15]. Aquest es troben entre la posició 1005800 i 1005875 i entre la posició 1006462 i 1006530, ambdós separats per 587 nucleòtids.
Família composta per tres enzims diferents els MsrB1, MsrB2 i el MsrB3 (també conegudes com SelR). Aquestes contenen zinc i es troben en tots els vertebrats i reparen els residus de metionina oxidats de les proteïnes. Redueixen la forma R de la sulfoximetionina a metionina.
En el Blast s’ha obtingut només 1 hit significatiu corresponent a l’scaffold PTFA01000482.1. Dins d’aquest hi havia tres sub-alineaments i s’ha escollit el primer, ja que és el que s’alineava amb les posicions més inicials de la proteïna de referència.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest contig degut a que presenta una puntuació molt alta, té pocs gaps i pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 162428 i 163568 en la cadena positiva del genoma del Casuarius casuarius i conté 1140 nucleòtids.

A la figura s’observa que està formada per tres exons i el residu Sec es trobaal tercer exó a prop de l’extrem C-terminal. Té un element SECIS a l’extrem 3’ entre les posicions 165331 i 165402.
MsrB2 i MsrB3 també es van estudiar però es va trobar que no tenien residu Sec sinó Cys i, a més, no tenien element SECIS. és a dir, aquests són homòlegs que contenen Cys i mantenen la seva eficiencia catalítica[15][22]. MsrB2 es troba a l’scaffold PTFA01000041.1 del Casuarius casuarius a la cadena directa entre les posicions 348660 i 355563. Segons l’Exonerate s’ha observat que aquesta proteïna presenta 4 exons. Per altra banda, MsrB3 es troba a l’scaffold PTFA01000034.1 a la cadena positiva entre les posicions 5749142 i 5788135. Segons l’Exonerate s’ha observat que aquesta proteïna presenta 3 exons.
La MsrA pot catalitzar la reducció dels residus de metionina lliures o de metionines presents en seqüències de proteïnes. En alguns organismes aquesta té un residu Sec, però en vertebrats té un residu Cys. En el cas del Casuarius casuarius s’ha trobat com a homòleg de cisteïna que no té element SECIS. En concret, s’ha trobat en l’scaffold PTFA01000233.1 entre les posicions 303531 i 366828 en la cadena reversa i conté 3 exons.

Tal i com s’esperava les proteïnes que són del mateix tipus s’agrupen entre elles. També destacar que MSRB1 i 3 es troben més relacionades entre elles que amb MSRB2. A més s’observa que la proteïna MsrA es troba menys relacionada amb la resta de MSRBs.
Les selenoproteïnes W (SelW), T (SelT), H (SelH) i V (SelV) pertanyen a la família Rdx de selenoproteïnes. S'assemblen entre elles per la seva estructura, posseixen un domini semblant a tiorredoxina i un fragment d'aminoàcids conservats tGxFEI(V). La seva funció encara no està clara.
En el Blast s’ha obtingut 1 hits significatiu corresponent a l’scaffold PTFA01000026.1. Dins d’aquest hi havia cinc contigs, dels quals s’ha escollit el primer, ja que era el que tenia l’e-value més significatiu.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest contig degut a que presenta una puntuació alta, té pocs gaps i té molt pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 6704276 i 6710238 en la cadena reversa del genoma del Casuarius casuarius i conté 5.692 nucleòtids.
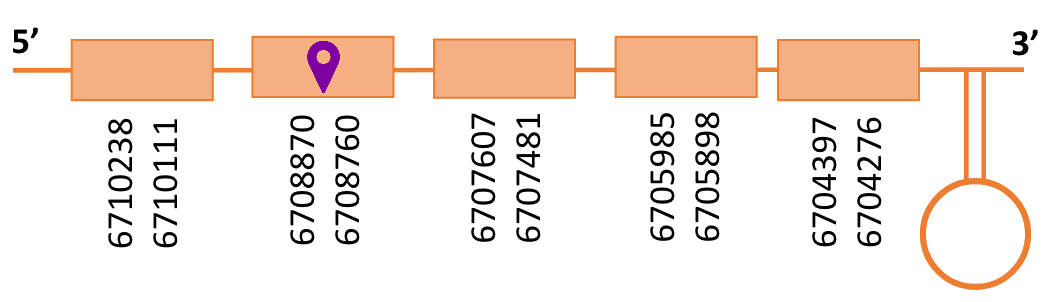
Segons la figura, s’observa que està format per cinc exons i el residu Sec es trobaal segon exó a prop de l’extrem N-terminal. Té un element SECIS a l’extrem 3’ entre les posicions 6702341 i 6702260.
Per a aquesta proteïna no s’han observat hits al blast. A més, tampoc es troba anotadaal genoma de Gallus gallus. Per tant, aquesta selenoproteïna no es troba a Casuarius casuarius. Això concorda amb el que s’espera ya que SelV es va generar per duplicació a partir de la SelW a l’origen dels placentaris [15].
Al Blast s’ha obtingut 1 hit significatiu corresponent a l’scaffold PTFA01000454.1. Dins d’aquest hi havia 2 subalineaments, dels quals es va escollir el segon en funció dels criteris inicials.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que, malgrat tingui un gran gap a l’inici, presenta una puntuació alta i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 338117 i 338384 en la cadena reversa del genoma del Casuarius casuarius i conté 267 nucleòtids.
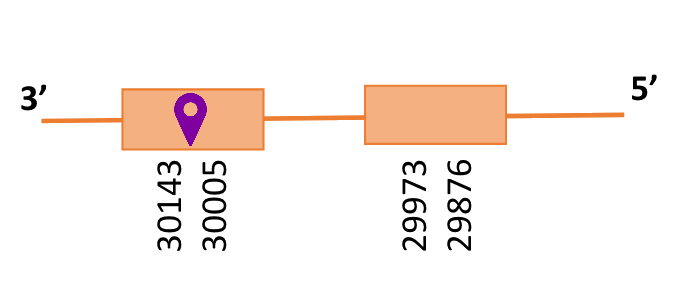
Segons la figura, s’observa que està format per dos exons i el residu Sec es troba a prop de l’extrem C-terminal,al primer exó que conté. No s’ha predit cap element SECIS.
En estudis previs s’ha determinat que en alguns gens que codifiquen per a selenoproteïnes com SelH la regió 3’UTR s’ha divit en dos exons i l’element SECIS es troba sencer a l’últim exó [15]. És per aquest motiu que es creu que no s’ha pogut predir l’element SECIS a partir de la proteïna de referència (Homo sapiens), ja que per errors del programa no s’ha seleccionat l’exó que conté l’element SECIS. Altra possibilitat és que es que el gen es trobi dividit en dos scaffolds diferents i no s’ha seleccionat el que conté l’element SECIS.
SelW s’expressa a molts teixits en funció de la disponibilitat de seleni, tot i que no es coneix molt bé la seva funció. En humans hi ha dues proteïnes d'aquesta subfamília: SelW1, que és una selenoproteïna i SelW2, que no ho és.
Utilitzant com a referència la proteïna SelW1 d’Homo sapiens es va obtenir un hit no significatiu al blast. En canvi, quan es va utilitzar la SelW1 de Gallus gallus es va obtenir un hit significatiu corresponent a l’scaffold PTFA01000548.1. Dins d’aquest scaffold només hi havia un sub-alineament que és el que es va escollir.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta un puntuació alta i pocs canvis d’aminoàcids. S’ha de dir però, que presenta un gap molt extens a l’inici.
El gen predit es troba entre les posicions 15984 i 16161 a la cadena reversa del genoma del Casuarius casuarius i conté 177 nucleòtids.
Segons l’Exonerate s’observa que SelW1 està formada per 2 exons i no s’ha trobat un residu Sec, ja que el residu Sec de la proteïna de referència s’alinea amb un gap. A més conté un SECIS que es troba entre les posicions 15839 i 15768.
A partir d’estudis previs se sap que Gallus gallus presenta aquesta proteïna i està formada per 85 aminoàcids i té 6 exons [11][15]. En canvi, la nostra predicció és d’una proteïna amb 2 exons i 48 aminoàcids. Això podria ser degut a que Casuarius casuarius no presenta aquesta part de la proteïna o bé que aquesta part es troba en un altre scaffold que no s’ha tingut en compte, la qual cosa concorda també, amb el resultat del T-coffee.
Al blast s’ha obtingut 1 hit significatiu corresponent a l’scaffold PTFA01000190.1. Dins d’aquest hi havia tres subalineaments i es va escollir el primer que era el que presentava l’e-value més petit.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest contig degut a que presenta una puntuació molt alta, té pocs gaps i té molt pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 143433 i 144635 a la cadena directa del genoma del Casuarius casuarius i conté 1.202 nucleòtids.
Segons l’Exonerate, s’observa que està formada per tres exons i no té residu Sec, en té un de Cys. S’ha predit un element SECIS, però degut a què es trobava a l’extrem 5’ i, s’ha decidit descartar-lo.
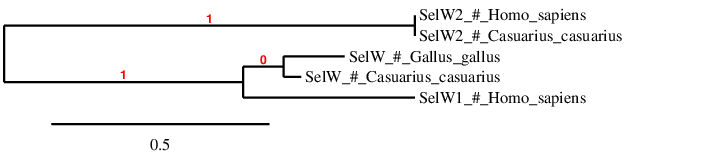
Aquesta filogenia mostra que SelW1 i SelW2 es troben allunyades entre elles, per tant, són homòlegs distants. Això concorda amb estudis previs en que es va descriure un grup de selenoproteïnes (SelW2) distintiu a la selenoproteïna W1. S’hipotetitza que SelW2 va ser duplicada abans de l’origen dels amfibis i que va ser convertida en una forma amb Cisteïna anomenada Rdx12, reportada en tots els tetràpodes. D’altra banda, SelW2 es perdria abans de la divisió dels rèptils. En el cas de Casuarius casuarius, la SelW2 conté Cisteïna i no s’ha predit cap SECIS. Per aquests motius, s’hipotetitza que el casuari pugui tenir la proteïna Rdx12[15].
SelW1 no es trobava anotada en Gallus gallus a la base de dades de referència utilitzada en aquest projecte, SelenoDB 2.0. Per tal de confirmar la seva absència al genoma del casuari, ja que amb l’Homo sapiens no es va identificar cap hit significatiu, es va recórrer a la proteïna de Gallus gallus que es trobava al NCBI (NP_001338303.1). Per aquest motiu, es creu que aquesta predicció podria ser dubtosa i es requereix un estudi més exhaustiu.
Proteïnes que redueixen tioredoxines entre d’altres substrats alhora que tenen un paper important pel que fa al metabolisme del seleni i a la protecció davant l’estrès oxidatiu. Actua com a homodímer que utilitza FAD com a cofactor.
Inicialment, es va escollir el hit més significatiu que corresponia a l’scaffold PTFA01000138.1. Al fer la predicció amb Gallus gallus el hit més significatiu corresponia a l’scaffold PTFA01000005.1. Se sap que aquesta proteïna en Gallus gallus conté 499 aminoàcids i que el residu Sec es troba a l’aminoàcid 498[11]. Això concorda perfectament amb la proteïna predita al casuari a partir de Gallus gallus. En base a això, es va tornar a mirar el BLAST d’Homo sapiens i es va veure que el segon hit més significatiu corresponia a l’scaffold PTFA01000005.1. Per això es va decidir agafar aquest scaffold per a fer la predicció.
Al Blast s’han obtingut 4 hits significatius corresponents a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000005.1 pel motiu explicatal paràgraf anterior. Dins d’aquest hi havia 11 subalineaments dels quals es va seleccionar el primer seguint els criteris esmentats inicialment.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest contig degut a que presenta un score molt alt, no té gaps i té molt pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 8384151 i 8408854 en la cadena directa del genoma del Casuarius casuarius i conté 24703 nucleòtids.
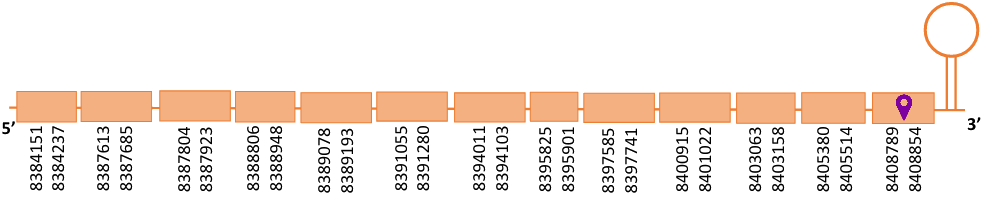
A la figura s’observa que està formada per tretze exons i el residu Sec es troba a l’últim exó, a prop de l’extrem C-terminal. Té un element SECIS a l’extrem 3’ entre la posició 8409106 i la posició 8409182.
Respecte a l’Homo sapiens, al Blast s’han obtingut 4 hits significatius corresponents a diferents scaffolds. Inicialment es va seleccionar l’scaffold més significatiu que, igual que en el cas anterior era el PTFA01000138.1. Però al utilitzar la proteïna TXNRD2 de Gallus gallus com a referència es va observar que l’scaffold més significatiu era el PTFA01000112.1. La proteïna predita a partir de Gallus gallus donava un resultat més semblant al que s’esperava segons el selenogenoma aviar [11]. Així doncs es va decidir seleccionar l’scaffold PTFA01000112.1. Dins d’aquest hi havia 14 subalineaments dels quals es va seleccionar el primer seguint els criteris esmentats inicialment.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta un score molt alt, té pocs gaps situats només a l’inici de l’alineament i té molt pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 1331479 i 1365484 a la cadena reversa del genoma del Casuarius casuarius i conté 34005 nucleòtids.
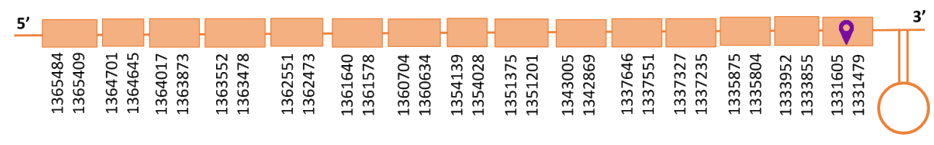
A la figura s’observa que està formada per quinze exons i el residu Sec es troba a l’últim exó, a prop de l’extrem C-terminal. Té un element SECIS a l’extrem 3’ entre les posicions 1328501 i 1328578.
Al Blast s’han obtingut 4 hits significatius corresponent a diferents scaffolds. Es va seleccionar l’scaffold PTFA01000138.1 ja que era el més clarament significatiu. Dins d’aquest hi havia tretze sub-alineaments, dels quals s’ha escollit el primer, ja que seguia els criteris esmentats a l’inici de la discussió.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta i té pocs canvis d’aminoàcids. Tot i així té un gap extens a l’inici de l’alineament.
El gen predit es troba localitzat entre les posicions 1438202 i 1459331 en la cadena reversa del genoma del Casuarius casuarius i conté 21129 nucleòtids.
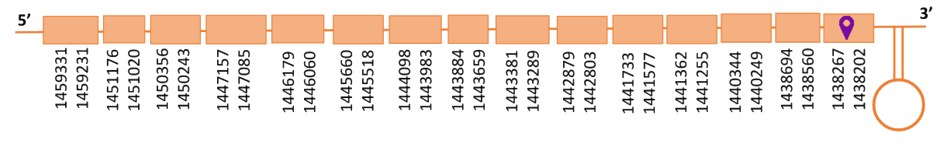
Segons la figura, s’observa que està formada per quinze exons i el residu Sec es troba a l’últim exó, a prop de l’extrem C-terminal. Té un element SECIS a l’extrem 3’ entre la posició 1437989 i la posició 1437897.
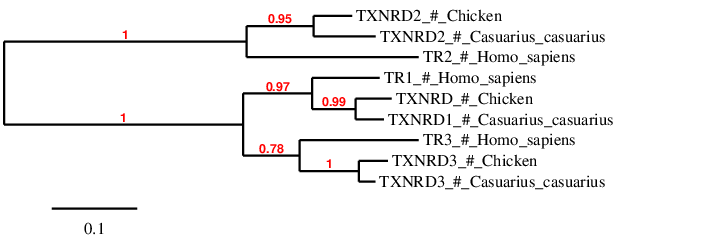
A la filogènia s’observa que les proteïnes dels mateix tipus de les diferents espècies es troben relacionades entre elles. Ara bé les proteïnes del Casuarius casuarius es troben més relacionades amb les del Gallus gallus. Això és el que s’esperava ja que ambdues espècies són aus i, per tant, són evolutivament més properes. També es pot observar que TXNRD1 i TXNRD3 s’agrupen indicant que es troben més relacionats que aquest amb TXNRD2[11].
En totes es van obtenir hits significatius corresponents als mateixos scaffolds. Es van provar els diferents scaffolds i es va seleccionar el que donava una millor predicció de la proteïna. Això últim es va determinar en funció del que s’esperava segons estudis previs on s’analitza el selenoproteoma aviar [11].
Encara que aquestes proteïnes no tenen una similaritat significativa de seqüència, però es poden agrupar en una família per la seva topologia.
En el Blast s’ha obtingut un hit significatiu corresponent a l’scaffold PTFA01000083.1. Dins d’aquest només hi havia quatre subalineaments, dels quals s’ha seleccionat el primer en funció dels criteris inicials.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest contig degut a que presenta una puntuació alta, no té molts gaps i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 2010482-2016807 en la cadena reversa del genoma del Casuarius casuarius i conté 6325 nucleòtids.

A la figura s’observa que està format per cinc exons i el residu Sec es troba a prop de l’extrem N-terminal, a l’exó 5. Té un element SECIS a l’extrem 3’ entre les posicions 2009922 i 2009832.
Al Blast s’ha obtingut un hit significatiu corresponent a l’scaffold PTFA01000114.1. Dins d’aquest només hi havia un subalimeament que és el que s’ha seleccionat.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació alta i té poc canvis d’aminoàcids. Tot i això, s’ha de comentar que al T-coffee amb Homo sapiens, apareixen bastants gaps adjacents. Aquest fet podria explicar-se per falta d’un fragment de la proteïna. Per altra banda, utilitzant com a proteïna de referència la SelK de Gallus gallus, el T-coffee presenta millor score i no hi ha cap gap. A més, a la predicció a partir de Gallus gallus, la proteïna predita apareix amb un exó més respecte la predicció feta a partir d’Homo sapiens.
El gen predit es troba localitzat entre les posicions 3258008 i 3260651 en la cadena reversa del genoma del Casuarius casuarius i conté 2643 nucleòtids.

A la figura s’observa que està format per tres exons i el residu Sec es troba a prop de l’extrem N-terminal, concretament a l’exó tres. Té un element SECIS a l’extrem 3’ entre la posició 3256828 i 3256749.
Aquesta família de selenoproteïnes és un membre important de les selenoproteïnes en les aus. Tenen activitat redox. En humans hi ha tres proteïnes anotades, però en Gallus gallus només n’hi ha una, però és una selenoproteïna. La funció d’aquestes és desconeguda.
En el blast s’ha obtingut un hit significatiu corresponent a l’scaffold PTFA01000016 que és el que s’ha seleccionat. Dins d’aquest hi havia quatre contigs i s’ha escollit el que presentava l’e-value més baix.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest contig degut a que presenta una puntuació alta, té un gap i té pocs canvis d’aminoàcids.
El gen predit es troba localitzat entre les posicions 4732207 i 4737987 en la cadena forward del genoma del Casuarius casuarius i conté 5.780 nucleòtids.
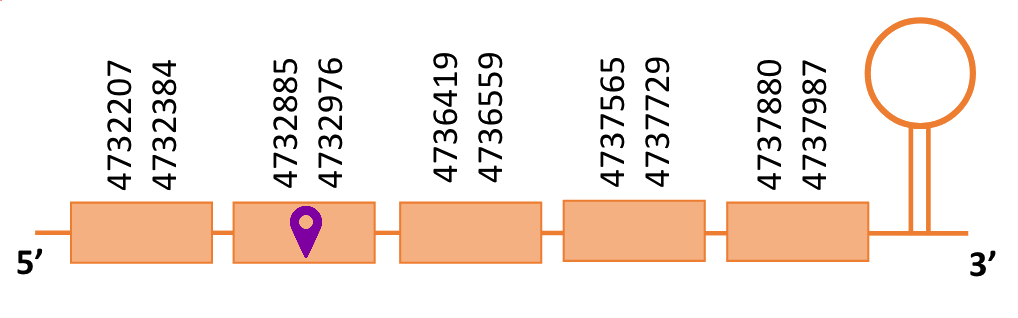
Segons la figura, s’observa que està format per cinc exons i el residu Sec es troba al segon exó, a prop de l’extrem N-terminal. Té un element SECIS a l’extrem 3’ entre les posicions 4738186 i 4738252.
S’ha observat que el residu Sec present en aquesta selenoproteïnaal Casuarius casuarius s’alineava amb un residu Cys en la mateixa posició de la proteïna d’humà, per això, s’ha decidit comparar amb altres aus per tal de contrastar si es trobava també el residu Sec i no que sigui una ganància de la nostra espècie únicament. Es va observar en Gallus gallus també la presència del residu Sec. Per tant, és possible que s’hagi perdut en humans.
Se sap que en Gallus gallus la SelU1 té 224 aminoàcids i que, igual que en la proteïna predita, la Selenocisteïna es troba a la posició 85[11]. A més, la proteïna del casuari presenta una segona U la qual cosa es veu a l'Exonerate, que no es troba en Gallus gallus i que correspon a l’aminoàcid 224. Això suggereix que en aquesta posició hi ha un codó STOP.
S’han estudiat altres membres de la família de les SelU, SelU2 i SelU3. En quant a SelU2 és un homòleg de cisteïna i es troba a l’scaffold PTFA01000024.1 del casuari entre les posicions 5214638 i 5218545 a la cadena directa. Amb l’Exonerate es va observar que aquesta proteïna estaba formada per sis exons. Per altra banda, SelU3 no es troba present al genoma del Casuarius casuarius ja que no s’ha obtingut cap hit significatiu al BLAST.
Aquests resultats concorden amb el fet que l’anàlisi filogenètic realitzat en estudis previs [15] indica que les formes del SelU que contenen Sec pertanyen al grup de selenoproteïnes SelU1.
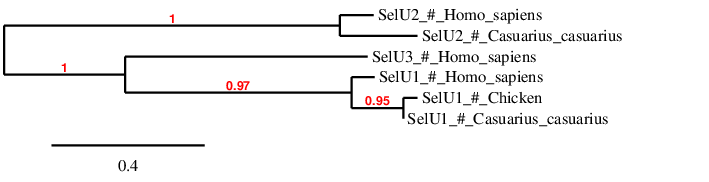
Observant la filogènia, cal destacar que SelU1 i SelU3 es troben més relacionades entre elles, mentre que SelU2 no és tan semblant amb les SelU1 i SelU3.
Les selenoproteïnes es caracteritzen per la incorporació de selenocisteïnes a la seva seqüència. Per tal d’incorporar seleni a les selenocisteïnes i incorporar aquest residu a la seqüència es requereixen diverses proteïnes sense les quals no podria haver selenoproteïnes en un organisme. En aquest treball s’han pogut identificar 6 elements que formen part d’aquest conjunt de proteïnes i s’exposen a continuació.
Al Blast s’ha obtingut 1 hit significatiu corresponent a l’scaffold PTFA01000061.1. Dins d’aquest hi havia quatre subalineaments i es va escollir el primer ja que seguia els criteris comentats a l’inici.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold degut a que presenta una puntuació molt alta, i té molt pocs canvis d’aminoàcids. A més presenta únicament dos gaps a l’inici i final de la proteïna.
El gen predit es troba localitzat entre les posicions 4935701-4938134 a la cadena reversa del genoma del Casuarius casuarius i conté 2433 nucleòtids.
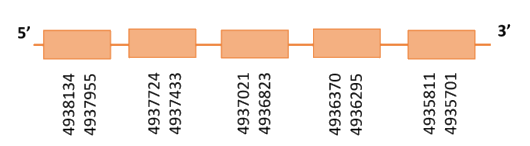
A la figura s’observa que aquesta proteïna està formada per cinc exons i no té residu Sec. S’ha predit un element SECIS a l’extrem 5’ entre la posició 4944951 i la posició 4944871. Com se sap que els elements SECIS es troben situats a l’extrem 3’, el SECIS predit va ser descartar.
Al Blast s’ha obtingut 1 hit significatiu corresponent a l’scaffold PTFA01000138.1. Dins d’aquest hi havia sis subalineaments i es va escollir el primer ja que tot i no alinear-se amb posicions tan inicials de la proteïna de referència com els altres subalineaments, era el més clarament significatiu.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold donat que presenta una puntuació molt alta, i té molt pocs canvis d’aminoàcids. A més presenta únicament dos gaps a l’inici i final de la proteïna.
El gen predit es troba localitzat entre les posicions 2579842 i 2633193 a la cadena directa del genoma del Casuarius casuarius i conté 53351 nucleòtids.

A la figura s’observa que aquesta proteïna està formada per cinc exons i no té residu Sec. Ara bé, tot i no presentar cap residu Sec s’ha pogut predir un element SECIS a l’extrem 3’ entre la posició 2659363 i 2659455.
Al Blast obtingut a partir d’Homo sapiens s’obtenen dos hits significatius que corresponen a l’scaffold PTFA01000024.1 i l’scaffold PTFA01000007.1 amb un e-value molt semblant. Per altra banda, al BLAST obtingut a partir de Gallus gallus s’han trobat 2 hits significatius corresponents als scaffolds esmentats, però l’scaffold PTFA01000007.1 és clarament més significatiu. Per la relació evolutiva entre ambdues espècies es va establir que el gen que codifica per a SBP2 es troba a l’scaffold PTFA01000007.1 de Casuarius casuarius.
Dins d’aquest scaffold, obtingut al comparar amb el genoma de Gallus gallus, hi havia 15 subalineaments i es va escollir el primer ja que seguia els criteris esmentats inicialment.
Segons el T-coffee és probable que la proteïna predita es trobi a aquest scaffold donat que presenta una puntuació molt alta, i té molt pocs canvis d’aminoàcids. Al T-coffee s’observa una Sec (X) al genoma del casuari que s’alinea amb una Isoleucina de la SBP2 de Gallus gallus. Ara bé, si analitzem el codó que codifica aquest aminòacid a l’Exonerate, s’observa que és AYG on Y podria ser una C o una T de manera que podria codificar per el codó ATG que és un codó stop, per tant, no és una selenocisteïna.
El gen predit es troba localitzat entre les posicions 5794568 i 5813398 a la cadena reversa del genoma del Casuarius casuarius i conté 18830 nucleòtids.
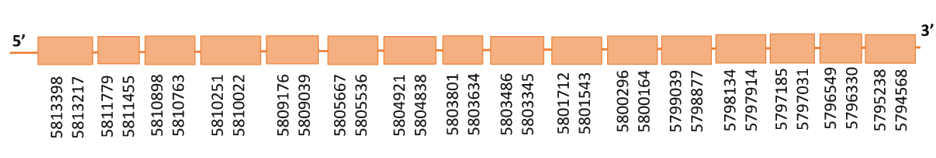
A la figura s’observa que aquesta proteïna està formada per setze exons i no té residu Sec ni cap element SECIS.
Al Blast s’han obtingut 2 hits significatius corresponents a scaffolds diferents. Es va seleccionar l’scaffold PTFA01001172.1 que era el que presentava l’e-value més baix. Dins d’aquest hi havia dos subalineaments i es va escollir el segon ja que seguia els criteris esmentats inicialment.
Segons el T-coffee, és probable que la proteïna predita es trobi en aquest scaffold donat que presenta una puntuació molt alta, i té molt pocs canvis d’aminoàcids. S’ha de comentar que presenta un gap inicial molt extens tot i que la part final de la proteïna presenta un bon alineament. Això podria donar-se perquè la proteïna es troba dividida en scaffolds diferents o que aquesta part inicial s'ha perdut en aquesta espècie.
El gen predit es troba localitzat entre les posicions 13137 i 13610 a la cadena reversa del genoma del Casuarius casuarius i conté 473 nucleòtids.

Segons la figura s’observa que aquesta proteïna està formada per 2 exons i no té residu Sec ni tampoc s’ha predit cap element SECIS. Això concorda amb el que s’ha vist en estudis anteriors en que en vertebrats SEPHS resulta no ser una selenoproteïna i conté treonina en comptes de selenocisteïna [16] tot i que en aquest estudi això no es pot contrastar.
En quant a la proteïna SPS2 predita s’ha vist que s’obtenia la mateixa proteïna que SPS1 utilitzant qualsevol dels dos scaffolds de tal manera que es va concloure que al genoma del Casuarius casuarius només hi ha una SEPHS.
Al Blast s’han obtingut 4 hits significatius corresponents a scaffolds diferents. Es va seleccionar l’scaffold PTFA01000044.1 que era el que presentava l’e-value més baix. Dins d’aquest hi havia nou subalineaments i es va escollir el primer ja que seguia els criteris esmentats inicialment.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold donat que presenta una puntuació molt alta, i té molt pocs canvis d’aminoàcids. S’ha de comentar que presenta un gap inicial molt extens tot i que la part final de la proteïna presenta un bon alineament.
El gen predit es troba localitzat entre les posicions 5033303-5046025 a la cadena reversa del genoma del Casuarius casuarius i conté 12722 nucleòtids.
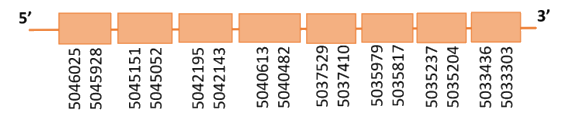
Segons la figura s’observa que aquesta proteïna està formada per vuit exons i no té residu Sec. Un element a destacar és que s’ha predit un element SECIS a l’extrem 5’ entre la posició 5089256 i 5089174, és a dir, allunyat de la proteïna predita. Aquests factors porten a descartar l’element SECIS predit.
Al Blast s’ha obtingut 1 hit significatiu corresponent a l’scaffold PTFA01000163.1. Dins d’aquest hi havia 10 subalineaments i es va escollir el primer ja que tenia un e-value més baix.
Segons el T-coffee és probable que la proteïna predita es trobi en aquest scaffold donat que presenta una puntuació molt alta, i té pocs canvis d’aminoàcids i només té un gap al final de la proteïna.
El gen predit es troba localitzat entre les posicions 584857-608935 a la cadena directa del genoma del Casuarius casuarius i conté 2478 nucleòtids.

A la figura s’observa que aquesta proteïna està formada per onze exons i no té residu Sec ni s’ha pogut predir cap element SECIS.
Utilitzant com a referència el selenoproteoma de Gallus gallus i d’Homo sapiens obtinguts ambdós a la base de dades SelenoDB s’han pogut identificar vint-i-quatre selenoproteïnes que són: DIO1, DIO2, DIO3, GPx1, GPx2, GPx3, GPx4, SelO, Sel15, SelM, SelH, SelI, SelK, SelS, SelN, SelP, MSRB1, SelT, SelU1, SelW1, TXNRD1, TXNRD2, TXNRD3 i eEFsec.
D’altra banda, s’han trobat set proteïnes homòlogues que contenen cisteïna: GPx7, GPx8, MSRB2, MSRB3, MsrA, SelU2 i SelW2.
A més, s’han identificat sis components de la maquinària que permeten la formació de selenoproteïnes: PSTK, eEFsec, SBPS2, SEPHS, SECp43 i SecS.
Algunes no comencen per Metionina i això és principalment per dos motius: o bé perquè la proteïna de referència no començava per Metionina, o perquè només s’ha pogut predir un fragment de la proteïna.
També cal esmentar que les selenoproteïnes predites utilitzant com a referència les selenoproteïnes anotades al genoma de Gallus gallus són més semblants al que s’esperava segons l’estudi de Shuping Li, et al. 2018 al que s’ha estudiat el selenogenoma aviar. Això s’ha pogut donar perquè el Gallus gallus i el Casuarius casuarius són aus i, per tant, són més propers a l’escala evolutiva. En alguns casos, s’han obtingut més exons amb Gallus gallus que amb Homo sapiens, suggerint això que podrien pertànyer a exons menys conservats o que l’hagi perdut Homo sapiens. Un cas en particular és el de SelW1 ja que no es trobava anotada en Gallus gallus a la base de dades de referència utilitzada en aquest projecte, SelenoDB 2.0. És per aquest motiu que es creu que la predicció obtinguda podria ser dubtosa i es requereix un estudi més exhaustiu.
Els SECIS són estructures de mRNA secundari que es poden predir a partir del programa informàtic Seblastian i són un dels elements necessaris per a que una proteïna es pugui considerar selenoproteïna. En altres casos s’ha predit un residu Sec, però no element SECIS. En altres casos, sobretot amb les proteïnes de maquinària, s’han predit SECIS en posicions diferents al 3’ què és el lloc on s’espera que es trobin. Per aquest motiu han estat descartats. Això pot ser degut a possibles errorsal programa, degut a que trobarà qualsevol estructura de mRNA que potencialment podria formar un SECIS.
No es pot afirmar o refutar que la presència o absència de les selenoproteïnes sigui degut a problemes de predicció o bé per events que s’han donat al llarg de l’evolució. Per això, és necessari dur a terme més estudis sobre el selenogenoma del Casuarius casuarius.
[1]Bradford, M. G., Dennis, A. J. and Westcott, D. A. (2008), Diet and Dietary Preferences of the Southern Cassowary (Casuarius casuarius) in North Queensland, Australia. Biotropica, 40: 338-343.
[2]Crome FHJ. Some observations on the biology of the Cassowary in Northern Queensland. Emu. 1976 Mar 1;76(1):8–14.
[3]Beruldsen G. Australian birds. Kenmore Hills, Qld: Beruldsen; 2004.
[4]Bulteau A-L, Chavatte L. Update on Selenoprotein Biosynthesis. Antioxid Redo Signal. 2015;23(10):775–94.
[5]Dobosz-Bartoszek M, Pinkerton MH, Otwinowski Z, Chakravarthy S, Söll D, Copeland PR, et al. Crystal structures of the human elongation factor eEFSec suggest a non-canonical mechanism for selenocysteine incorporation. Nat Commun. 2016;7(May).
[6]Jiang L, Ni J, Liu Q. Evolution of selenoproteins in the metazoan. BMC Genomics. BioMed Central. 2012 Sep 3;13:446.
[7]Jiang YY, Huang JQ, Lin GC, et al. Biol Trace Elem Res. Characterization and Expression of Chicken Selenoprotein U. 2015;166:216.
[8]Kofron CP. Attacks to humans and domestic animals by the southern cassowary (Casuarius casuarius johnsonii) in Queensland, Australia. Journal of Zoology. Cambridge University Press; 1999;249(4):375–81.
[9]Labunskyy VM, Hatfield DL, Gladyshev VN. Selenoproteins: Molecular Pathways and Physiological Roles. Physiol Rev [Internet]. 2014;94(3):739–77. Available at: http://physrev.physiology.org/cgi/doi/10.1152/physrev.00039.2013.
[10]Lee B, Dikiy A, Kim H, Gladyshev V. Functions and Evolution of Selenoprotein Methionine Sulfoxide Reductases. Biophs Acta. 2009 Nov;1790(11):1471-1477.
[11]Li S, Gao F, Huang J, Wu Y, Wu S, Lei XG. Regulation and function of avian selenogenome. Biochim Biophys Acta-Gen Subj. 2018 Nov 1;1862(11):2473–9.
[12]Lobanov A V., Fomenko DE, Zhang Y, Sengupta A, Hatfield DL, Gladyshev VN. Evolutionary dynamics of eukaryotic selenoproteomes: Large selenoproteomes may associate with aquatic life and small with terrestrial life. Genome Biol. 2007;8(9).
[13]Lobanov A V., Hatfield DL, Gladyshev VN. Eukaryotic selenoproteins and selenoproteomes. Biochim Biophys Acta - Gen Subj. 2009;1790(11):1424–8.
[14]Lobanov A V., Fomenko DE, Zhang Y, Sengupta A, Hatfield DL, Gladyshev VN. Evolutionary dynamics of eukaryotic selenoproteomes: Large selenoproteomes may associate with aquatic life and small with terrestrial life. Genome Biol. 2007;8(9).
[15]Lu J, Holmgren A. Selenoproteins. J Biol Chem. 2009;284(2):723–7.
[16]Mariotti M, Ridge PG, Zhang Y, Lobanov A V., Pringle TH, Guigo R, et al. Composition and evolution of the vertebrate and mammalian selenoproteomes. PLoS One. 2012;7(3).
[17]Mariotti M, Santesmasses D, Capella-Gutierrez S, Mateo A, Arnan C, Johnson R et al. Evolution of selenophosphate synthetases: emergence and relocation of function through independent duplications and recurrent subfunctionalization. Genome Research. 2015;25(9):1256-1267.
[18]Oudouhou F, Casu B, Dopgwa Puemi AS, Sygusch J, Baron C. Analysis of Novel Interactions between Components of the Selenocysteine Biosynthesis Pathway, SEPHS1, SEPHS2, SEPSECS, and SECp43. Biochemistry. 2017;56(17):2261–70.
[19]Santesmasses D, Mariotti M, Guigó R. Computational identification of the selenocysteine tRNA (tRNASec) in genomes. PLoS Comput Biol. 2017;13(2).
[20]Sun LH, Huang JQ, Deng J, Lei XG. Avian selenogenome: response to dietary Se and vitamin E deficiency and supplementation. Poultry Science. 2018;0:1-8.
[21]Whanger PD. Selenoprotein expression and function-selenoprotein W. Biochim Biophys Acta. 2009 Nov;1790(11):1448-52.
[22]Westcott DA, Bentrupperbäumer J, Bradford MG, McKeown A. Incorporating patterns of disperser behaviour into models of seed dispersal and its effects on estimated dispersal curves. Oecologia. 2005;146(1):57–67.
[23]Kim HY, Gladyshev VN. Methionine Sulfoxide Reduction in Mammals: Characterization of Methionine-R-Sulfoxide Reductases. Mol Biol Cell. 2004 Mar; 15(3): 1055–1064.
[24]Animals.jrank.org. (2018). Cassowaries: Casuaridae - Southern Cassowary (Casuarius casuarius): Species Account. [online] Available at: http://animals.jrank.org/pages/364/Cassowaries-Casuaridae-SOUTHERN-CASSOWARY-Casuarius-casuarius-SPECIES-ACCOUNT.html
[25]Environment.des.qld.gov.au. (2018). Southern cassowary (Department of Environment and Science). [online] Available at: https://environment.des.qld.gov.au/wildlife/threatened-species/endangered/endangered-animals/cassowary.html.
[26]National Geographic (2013). Casuarios: extrañas aves de Oceanía. [online] Available at: https://www.nationalgeographic.com.es/naturaleza/grandes-reportajes/extranas-aves-de-oceania-2_7635.
[27]Oldredlist.iucnredlist.org. (2015). Casuarius casuarius (Southern Cassowary). [online] Available at: http://oldredlist.iucnredlist.org/details/22678108/0-
Ens agradaria agraïr la col·laboració i ajuda a les diferents persones que han fet possible que aquest treball s'hagi realitzat. A la nostra tutora, Marta Badia, que ens ha ajudat en tot el que ha pogut. Als professors que coordinen i formen part de l'assignatura de Bioinformàtica: Roderic Guigó, Robert Castelo, Cedric Notredame i Toni Gabaldón. I l'Aida Ripoll com a coordinadora dels projectes.
Som un grup d'estudiants emprenedores, amb moltes ganes de treballar. El nostre objectiu és trobar totes les selenoproteïnes del nostre amic Casuarius casuarius, una au salvatge que viu en paratges exòtics i es troba en perill d'extinció. Ens ha encantat dur a terme aquest projecte. Espero que la vostra visita a aquesta web sigui útil i apreneu molt d'aquest au tan extranya i simpàtica.
