En mamífers, la família de les glutathione peroxidases (GPx) consta de 8 membres. D’aquests, són selenoproteïnes la GPx1, la GPx2, la GPx3, la GPx4 i, en algunes espècies, la GPx6. La resta de membres de la familia són homòlegs en cisteïna, ja que han substituït el residu de selenocisteïna per un residu de cisteïna. La funció general de les GPx és la de disminuir l’estrés oxidatiu mitjançant la reducció del peròxid d’hidrogen (H2O2) mitjançant el seu residu de Sec/Cys. Per tal de retornar al seu estat original, cal que l’enzim oxidat sigui reduït. Aquesta funció la pot dur a terme la GSH, en el cas de les Sec-GPx, o la tioredoxina, en el cas de les Cys-GPx. S’ha vist que el domini d’unió de les Sec-GPx per la GSH es troba totalment conservat en la GPx1, parcialment conservat en la GPx2 i la GPx3 i gens conservat en la GPx4. La pèrdua de conservació del domini d’unió a GSH està relacionat amb una pèrdua d’especificitat per aquest. Pel que fa a l’eficiència en la reducció del H2O2, les Sec-GPx són més eficients que les Cys-GPx i, dins de les Sec-GPx, l’eficiència és major en la GPx1 i va decreixent fins a la GPx4.
En la família de les GPx, tots els seus membres presenten una gran homologia entre ells. Per aquest motiu, a l’hora de còrrer el BLAST per les diferents GPx d’Homo Sapiens, els scaffolds que contenien els diferents hits eren comuns per totes les GPx. Tot i ser comuns, la distribució dels hits segons l’e-value era diferent. Per aquest motiu, a l’hora de determinar si un scaffold era una possible GPx o una altra ens hem fixat en els e-values, sent l’scaffold amb un valor menor d’e-value el candidat més probable. També ens hem fixat en l’alineament amb la query, de manera que si l’alineament era molt dolent passàvem a analitzar el següent scaffold amb la següent millor e-value. En última instància, hem comprovat que els scaffolds que hem seleccionat com a possibles candidats per les diferents GPx realment tenien la identitat de la GPx que nosaltres li havíem atribuït mitjançant l’elaboració d’un arbre filogenètic:
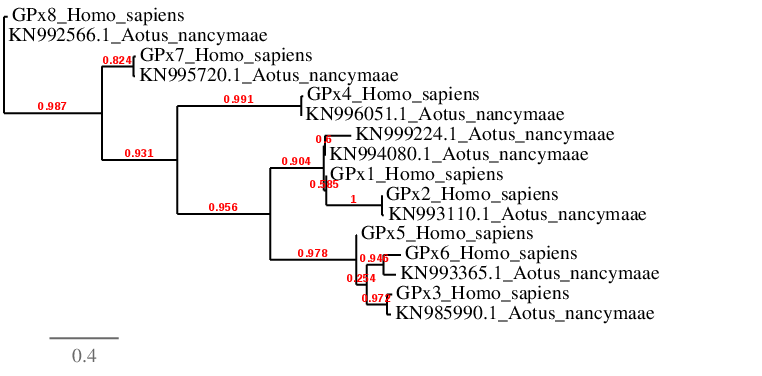
Figura 5. Arbre filogenètic de la família de selenoproteïnes GPx
GPx1
En el genoma d’Aotus nancymaae trobem una selenoproteïna homòloga a la GPx1 d’Homo Sapiens a l’scaffold KN994080.1 que presenta un alineament perfecte amb aquesta i està totalment conservada. Com a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx1 d’Homo Sapiens, podem dir que es tracta de la GPx1 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 861 nucleòtids (6813-7674) i es troba en sentit positiu. El gen consta de 2 exons i 1 intró. Nomes conté una selenocisteïna a l’exó 1 que està alineada en la mateixa posició que la selenocisteïna de la GPx1 d’Homo Sapiens.
A més a més, per aquesta proteïna s'ha pogut predir un element SECIS de grau A en sentit positiu, que es troba entre les posicions 7727 i 7796 del mateix scaffold i, per tant, en la regió 3’ UTR del gen. En aquest cas, el Seblastian ha predit que en aquest scaffold es troba la GPx1 d’Aotus nancymaae. Aquesta GPx1 predita pel Seblastian té la mateixa longitud que la GPx1 que hem trobat nosaltres, de manera que la GPx1 sí que estava ben anotada en el genoma d’Aotus nancymaae. Si tenim en compte totes aquestes evidències, podem concloure que la GPx1 en el genoma del nostre mico és una selenoproteïna que té una estructura molt ben conservada.
GPx2
En el genoma d’Aotus nancymaae trobem una selenoproteïna homòloga a la GPx2 d’Homo Sapiens a l’scaffold KN993110.1 que presenta un alineament perfecte amb aquesta i està totalment conservada. Com a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx2 d’Homo Sapiens, podem dir que es tracta de la GPx2 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 4216 nucleòtids (190876-195092) i es troba en sentit positiu. El gen consta de 2 exons i 1 intró. Nomes conté una selenocisteïna a l’exó 1 que està alineada en la mateixa posició que la selenocisteïna de la GPx2 d’Homo Sapiens.
A més a més, per aquesta proteïna s'ha pogut predir un element SECIS de grau A en sentit positiu, que es troba entre les posicions 195314 i 195378 del mateix scaffold i, per tant, en la regió 3’ UTR del gen. En aquest cas, el Seblastian ha predit que en aquest scaffold es troba la GPx2 d’Aotus nancymaae. Aquesta GPx2 predita pel Seblastian té la mateixa longitud que la GPx2 que hem trobat nosaltres, de manera que la GPx2 sí que estava ben anotada en el genoma d’Aotus nancymaae. Si tenim en compte totes aquestes evidències, podem concloure que la GPx2 d’Aotus nancymaae segueix sent una selenoproteïna i, a més a més, té una estructura molt ben conservada.
GPx3
En el genoma d’Aotus nancymaae trobem una selenoproteïna homòloga a la GPx3 d’Homo Sapiens a l’scaffold KN985990.1 que presenta un alineament molt bo amb la query. Aquesta, a diferència de les GPx anteriors, no està del tot conservada ja que li falta el fragment inicial de la proteïna. Tot i això, com a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx3 d’Homo Sapiens, podem dir que es tracta de la GPx3 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 2820 nucleòtids (4225440-4222620) i es troba en sentit negatiu. El gen consta de 4 exons i 3 introns. Nomes conté una selenocisteïna a l’exó 1 que està alineada en la mateixa posició que la selenocisteïna de la GPx3 d’Homo Sapiens.
A més a més, per aquesta proteïna s'ha pogut predir un element SECIS de grau A en sentit negatiu, que es troba entre les posicions 4221981 i 4221908 del mateix scaffold i, per tant, en la regió 3’ UTR del gen. En aquest cas, el Seblastian ha predit que en aquest scaffold es troba la GPx3 d’Aotus nancymaae. Aquesta GPx3 predita pel Seblastian té una longitud més petita que la GPx3 que hem trobat nosaltres, ja que encara li manca més tros del primer exó que a la nostra. Tot i això, el fet que en ambdós casos manqui el fragment inicial de la GPx3 sembla indicar que aquesta regió no està conservada en Aotus nancymaae. Si tenim en compte totes aquestes evidències podem concloure que, tot i que la GPx3 també és una selenoproteïna en Aotus nancymaae, és més curta que en humans. Això pot ser degut a que la regió inicial de la proteïna o bé s’hagi perdut en Aotus nancymaae, o bé que s’hagi ampliat en humans. Per determinar quina de les dues és la interpretació correcta caldria comparar la GPx3 d’Aotus nancymaae amb la GPx3 de l’ancestre comú més proper amb Homo Sapiens. Independentment d’això, aquesta variabilitat en la regió inicial denota que no és important per la funció de la proteïna. De fet, és probable que aquesta regió inicial estigui implicada en la especificitat de la GPx per diferents compostos donadors.
GPx4
En el genoma d’Aotus nancymaae trobem una selenoproteïna homòloga a la GPx4 d’Homo Sapiens a l’scaffold KN996051.1 que presenta un alineament molt bo amb la query. Amb aquesta GPx ens trobem amb el mateix escenari que en el cas anterior, ja que també li falta el fragment inicial de la proteïna. Tot i això, com a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx4 d’Homo Sapiens, podem dir que es tracta de la GPx4 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 1442 nucleòtids (12521461-12520019) i es troba en sentit negatiu. El gen consta de 7 exons i 6 introns. Nomes conté una selenocisteïna a l’exó 3 que està alineada en la mateixa posició que la selenocisteïna de la GPx4 d’Homo Sapiens.
A més a més, per aquesta proteïna s'ha pogut predir un element SECIS de grau A en sentit negatiu, que es troba entre les posicions 12519966 i 12519894 del mateix scaffold i, per tant, en la regió 3’ UTR del gen. En aquest cas, el Seblastian no ha fet cap predicció de selenoproteïna ja que, probablement la GPx4 no estava ben anotada en el genoma d’Aotus nancymaae. Tot i això, tenint en compte que la GPx4 té selenocisteïna i un element SECIS en la regió 3’ UTR, podem concloure que també és una selenoproteïna en Aotus nancymaae.
GPx5
Pel que fa a la GPx5, de tots els scaffolds pels quals s’han trobat hits en el BLAST, el que ens donava un millor alineament i tenia un millor e-value era l’scaffold KN999224.1. Els següents scaffolds amb millor e-value o bé presentaven un pitjor alineament o bé ja havien estat assignats a les altres GPx, ja que en l’arbre filogenètic s’agrupaven amb les seves GPx homològues d’Homo Sapiens. Així, hem analitzat el gen comprès en l’scaffold KN999224.1. Aquest consta de 488 nucleòtids (6242982-6243470), es troba en sentit positiu i està format per 1 sol exó. A diferència de la GPx5 d’Homo Sapiens, que és un homòleg en cisteïna, en aquest gen hi ha una selenocisteïna al principi de l’exó que s’alinea amb una cisteïna de de la query.
A més, per aquesta proteïna s'ha pogut predir un element SECIS de grau A en sentit positiu, que es troba entre les posicions 6243539 i 6243606 del mateix scaffold i, per tant, en la regió 3’ UTR del gen. En aquest cas, el Seblastian no ha fet cap predicció de selenoproteïna.Tot i això, tenint en compte que la proteïna té selenocisteïna i un element SECIS en la regió 3’ UTR, podem concloure que es tracta d’una selenoproteïna en Aotus nancymaae. El fet curiòs és que en realitzar l’arbre filogenètic, la selenoproteïna de l’scaffold KN999224.1 no s’agrupa amb la GPx5 d’Homo Sapiens sinó que apareix a un lloc proper a GPx1. Això ens indica que no és que la GPx5 d’Aotus nancymaae sigui una selenoproteïna, sinó que no hem trobat la GPx5 d’Aotus nancymaae. Això pot ser degut a que potser la GPx5 d’Aotus nancymaae i la GPx5 d’humans tenen una estructura molt diferent. Si això és cert, caldria fer servir el genoma d’una espècie més propera filogenèticament a Aotus nancymaae per tal de trobar la seva GPx5. Pel que fa a la selenoproteïna que hem trobat, es podria tractar d’una duplicació del gen GPx1 que ha mutat fins el punt en què en el T-COFFEE genera un mal alineament amb aquesta.
GPx6
En el genoma d’Aotus nancymaae trobem una proteïna homòloga a la GPx6 d’Homo Sapiens a l’scaffold KN993365.1 que presenta un alineament gairebé perfecte amb la query i que és 5 aminoàcids més curta pel final de la seqüència. Com a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx6 d’Homo Sapiens, podem dir que es tracta de la GPx6 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 10334 nucleòtids (1345541-1355875) i es troba en sentit positiu. El gen consta de 5 exons i 4 introns. Podem veure que en la posició de l’exó 2 en la què a la query hi ha una selenocisteïna, a la GPx6 d’Aotus nancymaae hi ha una cisteïna.
Tot i això, per aquesta proteïna s'ha pogut predir un element SECIS de grau A en sentit positiu, que es troba entre les posicions 1356499 i 1356572 del mateix scaffold i, per tant, en la regió 3’ UTR del gen. El fet que l’element SECIS es conservi tot i que a la seqüencia de la proteïna no hi hagi cap selenocisteïna es deu a què aquesta proteïna es va generar a partir d’una selenoproteïna en la què la selenocisteïna es va canviar per una cisteïna. En aquest cas, el Seblastian no ha fet cap predicció de selenoproteïna. Si tenim en compte totes aquestes evidències podem concloure que la GPx6 en Aotus nancymaae és un homòleg en cisteïna. Aquest fet no sorprèn, ja que tal i com s’ha explicat anteriorment, la GPx6 pot ser una selenoprorteïna o un homòleg en cisteïna depenent de l’espècie. De fet, en un altre estudi (Mariotti et al, 2012) ja van observar que en els titís, uns altres membres de la família dels primats, la GPx6 també era un homòleg en cisteina.
GPx7
En el genoma d’Aotus nancymaae trobem una proteïna homòloga a la GPx7 d’Homo Sapiens a l’scaffold KN995720.1, el qual presenta un alineament perfecte amb la query i té la mateixa llargada que aquesta. Degut a què a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx7 d’Homo Sapiens, podem dir que es tracta de la GPx7 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 6035 nucleòtids (11910713-11916748) i es troba en sentit positiu. El gen consta de 3 exons i 2 introns. Podem veure que no hi ha cap selenocisteïna ni en la query ni en la GPx7 d’Aotus nancymaae. El que sí que observem és que hi ha dues cisteïnes a l’exó 2 que estan totalment conservades en les dues GPx7. Per tant, és molt probable que aquestes siguin les cisteïnes encarregades de catalitzar la reducció de l’H2O2.
Per aquesta proteïna tampoc s'ha pogut predir cap element SECIS ni cap selenoproteïna amb el Seblastian. Si tenim en compte tot això, podem concloure que la GPx7 en Aotus nancymaae és un homòleg en cisteïna amb una estructura molt ben conservada.
GPx8
En el genoma d’Aotus nancymaae trobem una proteïna homòloga a la GPx8 d’Homo Sapiens a l’scaffold KN992566.1 que presenta un alineament perfecte amb la query i que té la mateixa llargada que aquesta. Com que a l’arbre filogenètic veiem que la proteïna d’aquest scaffold s’agrupa amb la GPx8 d’Homo Sapiens, podem dir que es tracta de la GPx8 d’Aotus nancymaae. La seqüència que codifica per aquesta proteïna és de 3750 nucleòtids (6082913-6079163) i es troba en sentit negatiu. El gen consta de 3 exons i 2 introns. Podem veure que no hi ha cap selenocisteïna ni en la GPx8 d’Homo Sapiens ni en la GPx8 d’Aotus nancymaae. El que sí que podem observar és que hi ha quatre cisteïnes, dues a l’exó 1 i dues a l’exó 2, que estan totalment conservades en les dues GPx8. Per tant, és molt probable que aquestes siguin les cisteïnes involucrades en catalitzar la reducció de l’H2O2.
Per aquesta proteïna tampoc s'ha pogut predir cap element SECIS ni cap selenoproteïna amb el Seblastian. Si tenim en compte tot això, podem concloure que la GPx8 en Aotus nancymaae és un homòleg en cisteïna amb una estructura molt ben conservada.
