SelM
En l’estudi de la Selenoproteïna SelM, primer de tot, es van emprar les query de la base de dades selenoDB (query 1 (Homo sapiens) i query 2 (Mus musculus), on els resultats obtinguts amb tblastn tingueren uns valors d’E-value massa elevats com per a poder acceptar-los i no ens permeteren continuar amb la cerca (Veure taules querySelM).
Per tal de continuar l’estudi de la presència de Selenoproteïnes, vam decidir buscar les hipotétiques SelM que s’havien obtingut en treballs d’anys anteriors [query 3 (A. Anophagefferens (2010)), query 4 (T. Pseudonana (2010)) i query 5 (E. Siliculosus (2011))]. Amb aquestes query els resultats no varen millorar, a excepció del resultat amb E. Siliculosus. Malgrat això la baixa qualitat de l’alineament no ens va permetre fer una predicció prou acurada amb exonerate (de l’ordre de -04). Aquest fet ens fa pensar que potser sí que alguns protistes presenten selenoproteïnes, tot i que només amb aquestes query no som capaços d’obtenir-les.
Amb l’objectiu d’emprar noves query en la nostra cerca vam buscar altres SelM a les bases de dades dbTEU i NCBI, i vam iniciar una nova cerca amb 13 query més (query 6 (Bos taurus), query 7 (Chlamydomonasreinhardtii 1), query 8 (Chlamydomonasreinhardtii 2), query 9 (Daniorenio), query 10 (Gallusgallus), query 11 (Ostreococcus tauri), query 12 (Pan troglodytes), query 13 (Rattusnovergicus), query 14 (Takifugurubripes), query 15 (T. Pseudonana 2), query 16 (Xenopustropicalis), query 17 (Ostreococcuslucimarinus), query 18 (Xenopustropicalisprecursos) i query 19 (Micromonaspusilla)). Tot i així, els resultats obtinguts amb aquestes query tampoc són concloents, de manera que no ens permeten arribar a una conclusió vàlida.
Després de portar a terme tot l’estudi amb les 19 query, no havíem obtingut cap resultat que ens permetés obtenir una conclusió fiable. Com que no contàvem amb més query, vam passar a emprar com a query seqüències seed. Aquestes seqüències són alineaments representatius de la superfamilia sep15 la qual està composta per Sep15 i SelM, però nomès representa el domini compartit d’ambdues famílies. Així, el que obtindrem serà aquest domini i, una vegada tinguem la proteïna predita, haurem de discernir si aquest organisme té Sep15 o, pel contrari, SelM.
Emprant els seed com a query, els resultats del tblastn són significatius en els genomes de D.fasciculatum i D.discoideum, i per tant, tenim un bon indici de que aquest dos poden contenir una selenoproteïna d’aquesta superfamília. Tot i així, fent servir aquestes seqüències el que obtindrem serà només el domini compartit d’ambdues famílies (SelM i Sel15).
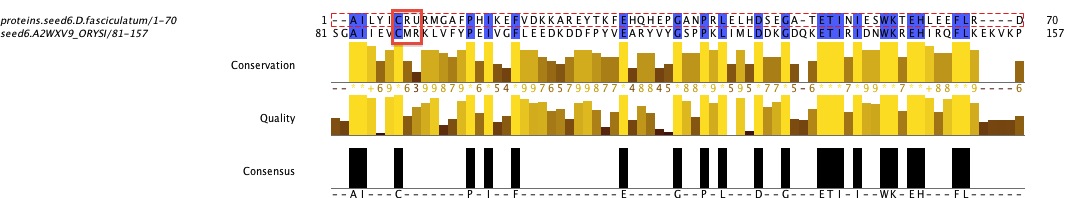
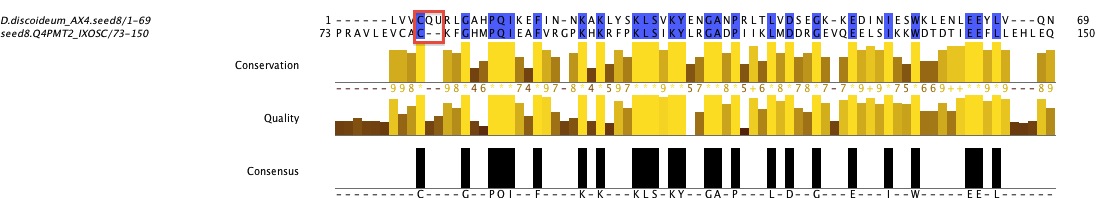
Abans de treure cap conclusió, varem buscar les proteïnes completes corresponents als seeds que donaven els resultats més significatius, de manera que repetim tot el procès però amb la proteïna sencera (seed5complete, seed6complete, seed7complete, seed8complete, seed9complete i seed10complete). Cal comentar que aquestes proteïnes són extretes d’NCBI i per tant, corresponen indistintament a SelM o Sep15. En aquest cas, el seed5complete és una SelM, en canvi, el seed7complete és una Sep15 i el seed8complete, el seed9complete i el seed10complete són homolegs en Cisteïna de Sep15.
Com veiem a la imatge superior, aquestes dades avalen la informació enunciada a la introducció (Introducció de superfamília SEP15). En aquest cas, totes les seqüències segueixen el patró descrit i veiem com les proteïnes SelM o homòlegs tenen un patróC-XX-U/C i les proteïnes Sel15 o homòlegs el tenen C-X-U/C.
Amb aquestes proteïnes completes fem tot el procés de nou, i els resultats obtinguts amb tblastn ja ens fan veure que D.fasciculatum i D.discoideum_AX4 són els que tenen uns E-values més significatius. D’aquesta manera, es confirma la nostra hipòtesi de la possibilitat de presència d’aquesta superfamilia en aquests dos organismes. Continuant el procés, podem extreure 5 proteïnes (mitjançant els seeds completes del 6 al 10) de D.fasciculatum i 4 proteïnes (mitjançant els seeds completes del 6 al 9) en el cas de D.discoideum_AX4.
A l’hora d’analitzar els resultats, ara el que volem saber és si aquestes proteïnes s’han extret del mateix contig i de la mateixa regió i, per tant, corresponen a la mateixa proteïna.
En el cas de les proteïnes de D.fasciculatum:
En el cas de les proteïnes de D.discoideum_AX4:
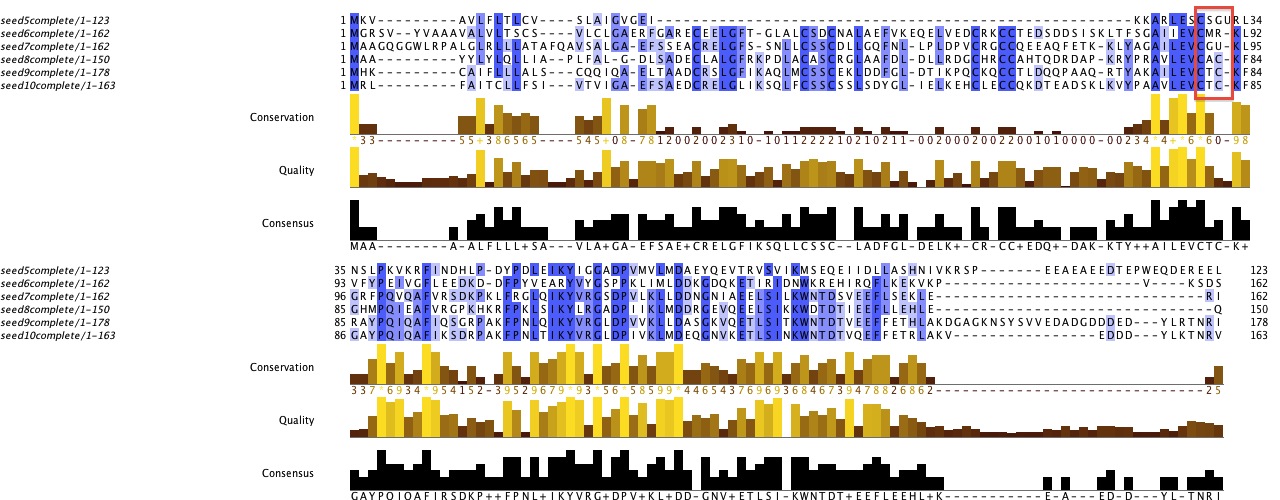
Una vegada sabem que corresponen a la mateixa proteïna, ara volem saber si el que tenim és una SelM o una Sep15. Per tal d’esbrinar-ho, primer de tot buscarem la proteïna més representativa de les que hem obtingut amb tot el procés i això ho farem analitzant paral·lelament el multialineamententre les 5 proteïnes en el cas de D.fasciculatumi les 4 en el cas de D.discoideum_AX4. Els resultats de l’alineament s’analitzen mitjançant t_coffee:


L’alineament entre les 4 proteïnes en el cas de D.discoideum ens permet veure que, tot i que hem emprat querys diferents per a la seva obtenció, al final del procés tenim la mateixa proteïna, cosa que ens confirma la validesa de l’existència de la seqüència proteica. Ara bé, de les 4 seqüències, nosaltres continuarem analitzant només la proteïna extreta amb la query seed6complete (la primera del multialineament), ja que ens proporciona un fragment més a la part upstream i no conté cap GAP dins de la seqüència. En el cas de les proteïnes de D.fasciculatum, basant-nos en les mateixes premisses, també escollim la primera seqüència per continuar amb l’anàlisi.
Un cop tenim les seqüències correctes, el que farem serà analitzar la seqüència mitjançant un alineament amb la query seed6complete, tot i que podíem haver escollit un altre, ja que el que ens interessa és estudiar el patró que segueixen les nostres proteïnes.
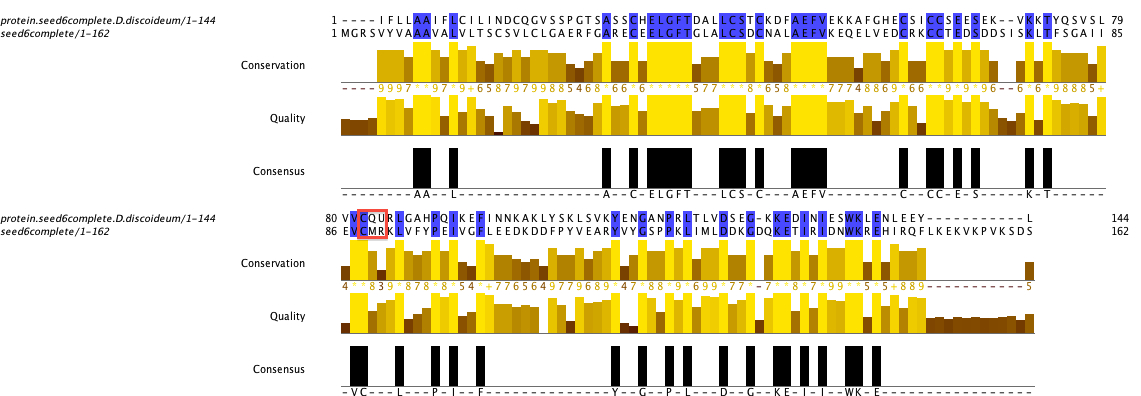
Si ens fixem en la imatge de l’alineament de la proteïna extreta de D.discoideum_AX4, veiem com conté una selenocisteïna i, per tant, serà una selenoproteïna homòloga a les nostres query. A més, si ens fixem en el patró que segueix el domini redox, veiem que té el motiuC-X-U, per tant, podem dir que el nostre homòleg es tracta d’una Sep15 i no d’una SelM. Per tal de confirmar això, si mirem la resta de proteïnes extretes amb les altres queryen un multialineament (t_coffee superior),veiem com el motiu segueix el mateix patró tot i haver sigut obtingudes amb 4 querys diferents. Podrem afirmar que la nostra hipòtesi és correcta amb un mínim marge d’error.
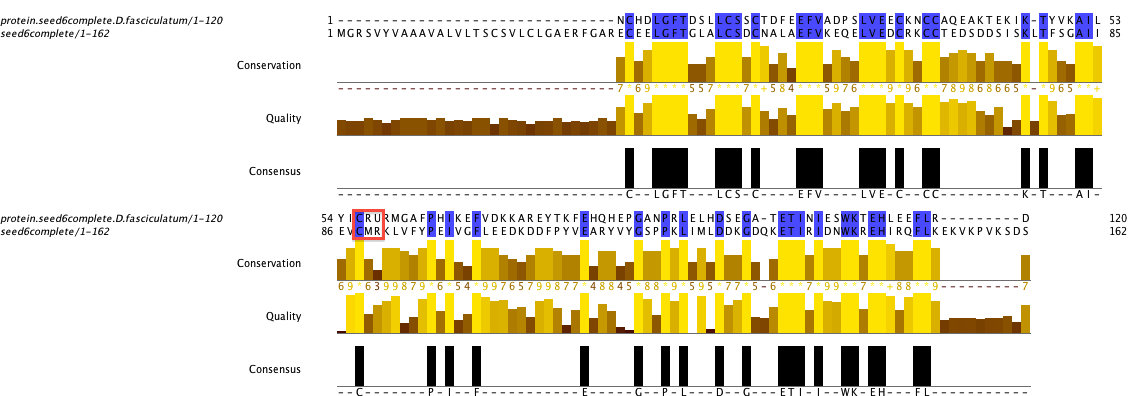
Parlant ara de la proteïna de D.fasciculatum, tenim el mateix alineament (amb seed6complete) i veiem com també conté una selenocisteïna i, per tant, també serà una selenoproteïna. A l’hora d’estudiar el patró, veiem que segueix exactament el mateix motiu C-X-U, pel que direm que es tracta també d’una Sep15. Tornant al multialineament t_coffee dels 5 resultats previs, podem confirmar el resultat ja que, tal i com passava amb l’anterior proteïna de D. discoideum_AX4, segueix el mateix patró i per tant, recolza la nostra hipòtesi.
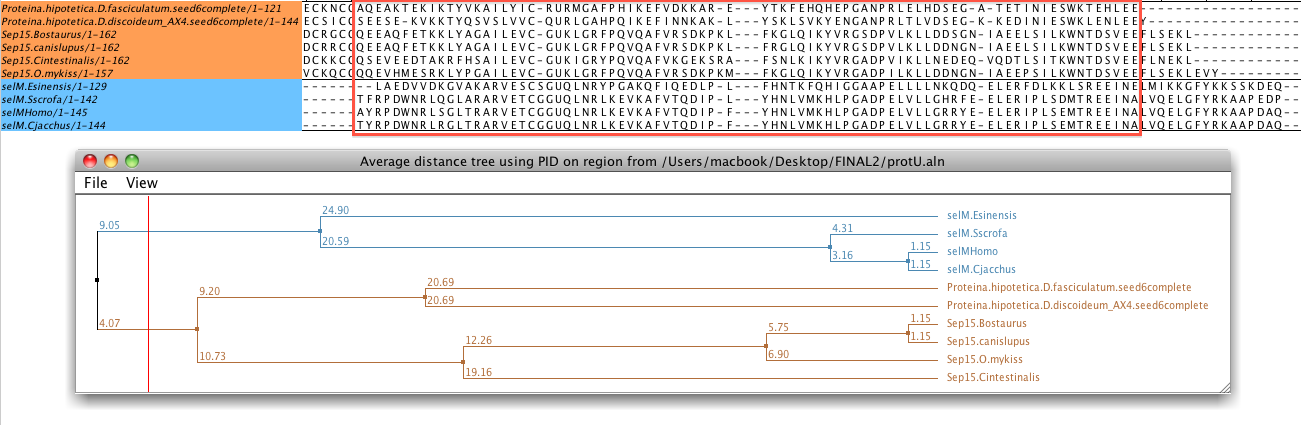
Finalment, i només per poder obtenir més dades que ens ajudessin a poder donar més pes als nostres resultats, vam realitzar una cerca blastp amb les proteïnes extretes i a més, amb una de les opcions del programa Jalview, vam analitzar la relació filogenètica per identitat (de manera aproximada), convertint els alineaments t_coffee en arbres filogenètics. D’aquesta manera, en el cas del blastpel que fem inicialment és llençar un blast amb NetBLAST. No obstant, el problema que ens trobem amb això és que tots els resultats corresponen a proteïnes “hipotètiques” i només amb el seu nom no podem saber si tenen alguna possibilitat de pertànyer a alguna família de Selenoproteïnes (clica aquí per accedir als resultats del blastp de D.discoideum_AX4 o D.fasciculatum). Llavors, el que fem és llençar un blastp via internet per poder consultar les proteïnes resultants. El que observem és que totes les que presenten hits significatius són proteïnes de 15kDa (com la nostra superfamília SEP15) i a més, comptem amb alguns hits que contenen una selenocisteïna tot seguint el mateix patró C-X-U descrit anteriorment.
En segon lloc, tal i com hem comentat anteriorment, fem un multialineament amb diferents selenoproteïnes conegudes SelM i Sep15 i les nostres proteïnes hipotètiques. Amb l’alineament es fa un càlcul de la distància mitjana emprant el % de la identitat per obtenir un arbre de relacions filogenètiques. Tal i com esperàvem, i per confirmar la nostra interpretació, les dues proteïnes hipotètiques s’engloben dins de la branca de les sep15.
Per tal d'acabar de donar pes als nostres resultats no significatius del blast, el que fem es llençar un genewise amb les regions genòmiques dels organismes en concret. Tot i així, cap dels resultats ens permet continuar ja que no proporciona cap informarció nova.
| Nom organisme | D.fasci | D.discoi | L.donov | T.congo | C.fasci |
| Seed5 | - | - | - | - | |
| Seed6 | - | - | - | - | |
| Seed7 | - | - | - | - | |
| Seed8 | - | - | - | - | |
| Seed9 | - | - | - | - | |
| Seed10 | - | - | - | - | |
| Seed8Complete | - | - | - | ||
| Seed9Complete | - | - | - |