L'anàlisi proteic es va realitzar només per als gens escollits com vàlids en l'apartat anterior.
Utilitzant el programa BLASTp, es va intentar aliniar les seqüències peptídiques codificades pels gens escollits (tretes de l'arxiu de resultats dels programes de predicció de gens), amb una base de dades de seqüències proteiques conegudes. Es van marcar els paràmetres: swissprot, Homo Sapiens, low complexity, expect 10, word size 3, matriu BLOSSUM 62, existence 11: extension 1 (per penalitzar els gaps), HTML, at the bottom i pairwise.
Un com es va córrer un BLASTp per a les 7 proteïnes a analitzar, als arxius es van guardar en format txt i html.
En realitzar el BLASTp amb la seqüència de la proteïna 1, es va veure que en aquest cas no hi havia hagut aliniaments gaire bons amb les proteïnes humanes de la base de dades. Tot i així, es va escollir el millor aliniament (BLASTPg1fgenesh.html ) per poder analitzar-ne la identitat, l'E-value, el score i, posteriorment, fer un estudi dels possibles dominis de la proteïna.
En aquest cas, el millor aliniament és el de la proteïna COL22A1 (d'Homo sapiens) de la base de dades. L'entrada de la proteïna i les característiques de l'aliniament són:
Length = 854
Score = 37.7 bits (86), Expect = 0.084
Identities = 51/192 (26%), Positives = 67/192 (34%), Gaps = 48/192 (25%)
Com es pot observar, no hi ha una elevada similaritat ni un E-value gaire baix, cosa que fa més probable l'efecte de l'atzar en aquesta predicció. No obstant, podria ser que tant baixa identitat fos degut a que la nostra proteïna en realitat comparteix algun domini amb altres proteïnes localitzades en una altre regió cromosòmica.
Es pot dir, doncs, que la regió predita no és gaire similar a res del que hi ha en la base de dades de proteïnes humanes.
Per obtenir més informació sobre aquesta proteïna problema, es va probar de fer un BLASTP swissprot però, en aquesta ocasió, contra la base de dades de rossegadors per veure si es podria trobar una identitat més elevada que podria indicar que el gen predit té algun gen ortòleg en aquests animals.
En observar les dades, es va veure que els resultats d'aliniament no milloraven gaire en rossegadors respecte als obtinguts anteriorment en la base de dades humana, ja que la identitat major que es va trobar era d'un 37% i l'E-value era de 0.12. Aquesta proteïna que va oferir un millor aliniament va ser la precursora de la cadena alfa 1 del col.lagen (gi|1168725|sp|Q05306|CA1A_MOUSE ).
Les característiques de l'aliniament van ser les següents:
Score = 34.7 bits (78), Expect = 0.12
Identities = 32/86 (37%), Positives = 38/86 (44%), Gaps = 7/86 (8%)
El que es pot concloure d'aquesta regió, que ja de per si no va ser gaire predita similarment entre els tres programes, és que no hi ha cap gen ben conegut d'aquesta regió, potser perquè encara no ha estat seqüenciada. L'únic que es pot dir és que el poc tant per cent de similaritat que troba amb humans potser es deu a que la proteïna predita de la nostra regió conté un domini similar al que conté la proteïna donada per l'aliniament.
Els arxius on hi ha els resultats del BLASTp per a la proteïna 1 són els següents:
En obtenir els resultats del BLASTp per a la proteïna 2, es va veure que el millor aliniament el tenia la proteïna precursora de la sialofosfoproteïna dentina (gi|17865470|sp|Q9NZW4|DSPP_HUMAN).
Score = 47.4 bits (111), Expect = 1e-05
Identities = 58/260 (22%), Positives = 86/260 (33%), Gaps = 11/260 (4%)
En aquesta ocasió, succeix bàsicament el mateix que en la proteïna 1, és a dir, el millor aliniament que s'ha trobat té un tant per cent d' identitat força baix (22%) que no permet acceptar la predicció com a bona. Ara bé, en aquest cas l'E value obtingut es força més petit que en la proteïna anterior, la qual cosa permetria deduir que, tot i que ha trobat poc per aliniar, el que ha aliniat ho ha fet amb un baixa probabilitat de que sigui per atzar.
S'observa que la proteïna aliniada no es localitza en la regió que estem analitzant, sinó en el cromosoma 4.
Els resultats del BLASTp s'han guardat amb els següents noms:
El millor aliniament corresponia a la proteïna ZPR1 Zinc Finger (Zinc finger protein 259), amb entrada gi|6137318|sp|O75312|ZPR1_HUMAN.
Les caracterítiques de l'aliniament van ser:
Score = 776 bits (2004), Expect = 0.0
Identities = 390/401 (97%), Positives = 390/401 (97%)
Per a la proteïna 3, hem obtingut un primer aliniament molt bo, ja que existeix un tant per cent d'identitat molt elevat (d'un 97%), molts bits (776) i un E-value de 0,0. Això dóna una fiabilitat molt alta de que la proteïna trobada té una relació amb la nostra proteïna 3 problema.
Per altra banda, s'ha comprobat que la proteïna obtinguda com a millor aliniament es troba en el cromosoma 11, igual que la seqüència a analitzar. Però mentre que la proteïna problema té 401 aminoàcids, la que dóna l'aliniament en té 459. Això vol dir que la predicció ha obviat uns 57 aminoàcids.
ZNF259
Official Symbol: ZNF259 and Name: zinc finger protein 259 [Homo sapiens]
Other Aliases: HGNC:13051, ZPR1
Other Designations: zinc finger protein ZPR1
Chromosome: 11; Location: 11q23.3
GeneID: 8882
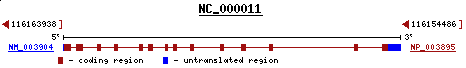
A continuació es mostra un esquema els gen de la proteïna aliniada ZPR1.
gi|27469566|gb|AAH42075.1|
Length = 680
Length = 1253
PROTEÏNA 3
Length = 459
Length = 459

Posteriorment, es va analitzar més detingudament la proteïna ZPR1 per tal d'intentar trobar dominis característics. Concretament, es va trobar que la proteïna amb la que s'havia aliniat tenia un tipus de domini tipus Zinc finger (ZPR1), per duplicat, com es veu a la imatge:
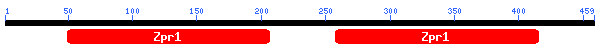
La proteïna ZNF259, també anomenada ZPR1, es caracteritza per ser una proteïna que s'uneix mitjançant els seus dominis Zinc finger als dominis citoplasmàtics tirosin kinasa dels receptors de EFG.
Aquesta proteïna es troba en el citoplasma de cèl.lules quiescents, i es translocada al nucli en presència de mitògens.
Com s'ha vist anteriorment, aquesta proteïna presenta dos dominis de tipus zinc fingers que es troben altament conservats.
Els resultats del BLASTp es van guardar amb els noms:
El millor aliniament el troba en el precursor de la proteïna Apolipoproteina A-V (ApoAV) amb la qual troba un 90 % d'identitat i un E-value de 0, la qual cosa dóna una bona confirmació de que la proteïna predita és exactament aquesta o s'hi assembla molt. Per tal d'acabar de corroborar-ho es va mirar en quin cromosoma es trobava per veure si realment es localitza en la regió que s'està analitzant.
La proteïna amb la que ha aliniat es troba el cromosoma 11, igual que la seqüència que estem analitzant.
ZNF259
Official Symbol: APOA5 and Name: apolipoprotein A-V [Homo sapiens]
Other Aliases: HGNC:17288, APOA-V, APOAV, RAP3x
Other Designations: apolipoprotein A5; apolipoprotein AV; regeneration-associated protein 3
Chromosome: 11; Location: 11q23.
GeneID: 116519
APOA5 Links
La proteïna proporcionada per l'aliniament té una longitut de 366 aminoàcids i les següents característiques:
Identities = 356/394 (90%), Positives = 356/394 (90%), Gaps = 28/394 (7%)
Score = 690 bits (1780), Expect = 0.0
L' Apolipoproteïna A-V és un important determinant dels nivells de triglicèrids en plasma, el factor de risc més important en patologies de l'artèria coronà ria. És un component de les lipoproteïnes d'alta densitat i és molt similar la proteïna de rata, que es troba up-regulada en resposta a dany hepàtic. El gen que codifica per a aquesta proteïna utilitza llocs de poliadenilació alternatius i es localitza proximal en el cluster de gens d' apolipoproteïnes, en el cromosoma 11q23.
A continuació, consta un esquema del gen de la Lipoproteïna A-V:

Pel que fa als dominis trobats per a aquesta proteïna, es veuen representats en el següent dibuix:

Per a aquesta proteïna, s'ha guardat l'esquema del gen com a PROT4rev.png i els dominis obtinguts, com a DOMINI.prot4.png.
Els resultats del BLASTp van se guardats d'aquesta manera:
El millor aliniament obtingut va ser el de la proteïna precursora de l'Apolipoproteïna C-III (Apo-CIII), amb entrada gi|114026|sp|P02656|APC3 _HUMAN. Aquesta proteïna té una longitut de 99 nucleòtids i les següents característiques en l'aliniament amb la proteïna problema:
Identities = 60/99 (60%), Positives = 60/99 (60%)
APOC3 MGC cDNA clone, Links
Official Symbol: APOC3 and Name: apolipoprotein C-III [Homo sapiens]
Other Aliases: HGNC:610, APOCIII
Chromosome: 11; Location: 11q23.1-q23.2
GeneID: 345
Score = 121 bits (303), Expect = 5e-29
La proteïna de la regió a analitzar s'assembla en un 60% a la ApoC-III,i això ens podria fer pensar que es tracta de proteïnes homòlogues. Per altra banda, es dues proteïnes es troben situades a la banda q23 del cromosoma 11.
Pel que fa a l'E-value, és molt bo, ja que és de 5e-29, la qual cosa indica que l'efecte de l'atzar en l'aliniament d'aquestes dues proteïnes és mínim. El score és de 121 bits, nombre no gaire elevat però, com s'ha comentat, els altres paràmetres si que ho són.
Tots aquests resultats indiquen que la proteïna problema podria ser pròpiament la Apolipoproteïna C-III o alguna altra proteïna, però amb una funció similar.
L'Apolipoproteïna C-III (ApoC3) és una lipoproteïna de baixa densitat (VLDL) que inhibeix la lipoproteïna lipasa i la lipasa hepàtica. Es creu que això endarrereix el catabolisme de les partícules riques en triglicèrids. Els gens APOA1, APOC3 i APOA4 estan estretament relacionats en els genomes humà i de rata. Els gens A-I i A-IV es transcriuen de la mateixa cadena, mentre que els gens A-1 i C-III es transcriuen de manera convergent. Un augment dels nivells d'apoC-III indueix el desenvolupament d'hipertrigliceridèmia.
El gen que codifica per a la proteï ApoC3 té la següent estructura:

No s'ha trobat cap informació sobre els dominis de la proteïna predita.
Els resultats del BLASTp es van guardar com a:
El millor aliniament el va tenir el precursor de l'Apolipoproteïna A-I (ApoA-I), que té l'entrada gi|113992|sp|P02647|APOA1_HUMAN.
Es tractava d'una proteïna de 267 aminoàcids i les següents característiques d'aliniament:
Identities = 240/267 (89%), Positives = 240/267 (89%)
Score = 475 bits (1223), Expect = e-134
Com es pot observar, el tant per cent d'identitat és molt elevat (89%) i, al seu torn, l' E-value és molt petit.
Podem concloure que la proteïna 6 problema és molt similar a la proteïna que ens proposa Blast.
El valor del score és força bo (475 bits) i l'E- value ho és també, ja que té un valor de e-134.
Aquesta proteïna es troba situada en el cromosoma 11, com la regió genòmica a analitzar, concretament es troba en 11q23.
Official Symbol: APOA1 and Name: apolipoprotein A-I [Homo sapiens]
Other Aliases: HGNC:600
Other Designations: amyloidosis; apolipoprotein A1
Chromosome: 11; Location: 11q23-q24
GeneID: 335
APOA1 MGC cDNA clone, Links
L'estructura del gen de la proteïna que s'ha aliniat amb la predita és la que es mostra tot seguit.

Com mostra el esquema, es pot apreciar que el gen es troba en orientació reverse al igual que el gen de la protïna predita.
Al fer un anàlisi més exhaustiu de la proteïna es pot observar que presenta un tipus de domini com el que es presenta a continuació:
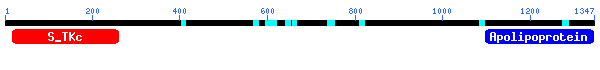
La proteïna predita és APOA1 i la seva funció és la de dirigir l'eflux de colesterol des de diferents teixits cap al fetge, per a la posterior excreció.
L' Apolipoprotein A-I és el component majoritari de les lipoproteïnes d'alta densitat plasmàtiques (HDL).La seva síntesi té lloc al fetge i a l'intestí prim i està formada per dues cadenes idèntiques de 77 aminoàcids, un pèptid senyal de 18 aminoàcids i un propèptid de 6.
L' APOA1 és un cofactor de la lecitin colesterolaciltransferasa (LCAT), la qual és responsable de la formació d'èsters de colesterol en plasma.
Els resultats del BLASTp obtinguts es poden observar tot seguit:
L'aliniament més bo que el Blastp ha trobat per la proteïna problema 7 és amb la proteïna GRC5 (PHD finger protein 2) que es caracteritza, principalment, per tenir un domini PHD. La seva entrada és gi|34098387|sp|O75151|PHF2_HUMAN.
Tant el score obtingut (73) com l'E-value (0.085) no són massa indicatius de que realment hi hagi una similitut entre les dues proteïnes. La identitat entre aquestes tampoc és gaire bona, sent només d'un 34%.
Les característiques de l'aliniament mencionades són aquestes:
Score = 32.7 bits (73), Expect = 0.085
Identities = 20/58 (34%), Positives = 26/58 (44%), Gaps = 2/58 (3%)
Length = 1101
La següent imatge mostra l'estructura del gen de la proteïna GRC5:
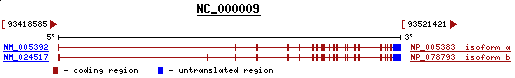
A més, aquesta proteïna que ha trobat Blast en primer lloc pertany a una regió del braç llarg del cromosoma 9, mentre que la nostra seqüncia proteica problema és del cromosoma 11.
Official Symbol: PHF2 and Name: PHD finger protein 2 [Homo sapiens]
Other Aliases: HGNC:8920, GRC5, KIAA0662
Chromosome: 9; Location: 9q22.31
GeneID: 5253
PHF2 Links
El fet que hagi aliniat la nostra proteïna del cromosoma 11 amb una proteïna del cromosoma 9, fa pensar que poder el tant per cent d'identitat trobat es deu a que ambdues proteïnes comparteixen certes similituts. No obstant, caldria comprobar si la nostra proteïna presenta similaritats amb la d'altres espècies com per exemple el ratolí, ja que aquest fet podria indicar que aquells gens són ortòlegs i tenen un origen comú.
S'ha realitzat un BLASTP amb bases de dades de rossegadors per veure si existia certa homologia amb alguna proteïna d'aquestes espècies i hem pogut comprobar que el tant per cent d'identitat no era millor que en l'aliniament amb la base de dades humana. Per tant, es dedueix que la proteïna predita comparteix regions d'homologia amb la proteïna que es troba en el cromosoma 9.
Per a aquesta proteïna GCR5, s'ha trobat un domini PHD, com es pot observar en la imatge adjunta:
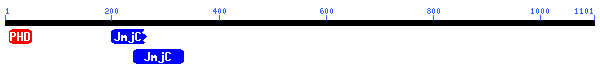
Els resultats del BLASTp es troben en els arxius: