E.dispar
Las selenoproteínas en Entamoeba dispar
SelI
Tanto para la selI (etanolaminafosfotransferasa) en Homo sapiens como para su homóloga en Drosophila melanogaster (que no se trata de selenoproteína, ya que en lugar de selenocisteína contiene cisteína) se obtuvieron varios hit significativos de fragmentos pequeños dentro del genoma de la especie E. dispar, lo que en un primer momento hizo sospechar que se trataba de la conservación de un dominio de proteína en diferentes especies.
D.melanogaster:(tblastn.out) (tblastn.hsp.out)
H.sapiens:(tblastn.out) (tblastn.hsp.out)
Una búsqueda en la base de datos de Pfam del Instituto Sanger confirmó que el dominio conservado era el de colina/etanolaminafosfotransferasa. No obstante, pese a que el resultado parecía concluir que la homología de secuencia solo se trataba de la conservación de este dominio, se prosiguió en el estudio de la secuencia para descartar la posibilidad de que se tratase de una selenoproteína presente en E.dispar.
Con la secuencia peptídica que se obtuvo una vez aplicado el programa genewise al alineamiento de SelI de Homo sapiens o la de Drosophila melanogaster(genewise.out) frente al genoma de Entamoeba dispar, se realizaron dos protein-BLAST en línea en el servidor del NCBI, frente a los genomas de H.sapiens y D.melanogaster, para ver si el mejor hit recíproco de la secuencia aminoacídica obtenida se trataba de la proteína de la cual se había partido.
Al contrario de lo esperado, el mejor resultado no fue ni la selI en H.sapiens ni su homóloga en D.melanogaster, sino una colina/etanolaminafosfotransferasa en ambas.
Para confirmar que las secuencias de la base de datos SelenoDB estaban bien anotadas, y eran realmente homólogas, se realizó un protein-BLAST en línea partiendo de la secuencia peptídica de SelI de humano frente al genoma de la mosca y viceversa, partiendo de la homóloga de SelI en la mosca frente a humano. Se trataban del mejor hit recíproco.
A continuación, se buscaron proteínas homólogas partiendo de la secuencia aminoacídica de la SelI putativa de E.dispar en la base de datos PhylomeDB, una colección de árboles filogenéticos. Así se pudo comprobar que la selI (etanolaminafosfotransferasa) y la colina/etanolaminafosfotransferasa son parálogas en H.sapiens y en D.melanogaster, y que la que es selI putativa en E.dispar, es co-ortóloga de ambas, en realidad más parecida a la colina/etanolaminafosfotransferasa. Todo parece indicar que se produjo una duplicación en la rama compartida entre Drosophila y humano dando lugar a selI y colina/etanolaminafosfotransferasa, y que la adquisición de la selenocisteína en H.sapiens se produjo posteriormente tras la separación de ambas especies, dado que Drosophila melanogaster no posee selenocisteína sino cisteína (no se trata de una selenoproteína).
Al mismo tiempo, se decidió hacer un alineamiento múltiple mediante la aplicación tcoffee, partiendo de la secuencia aminoacídica de la selI putativa en E.dispar (más parecida a colina/etanolaminafosfotransferasa) frente a las cuatro secuencias co-ortólogas siguientes:
- selI Homo sapiens
- selI Drosophila melanogaster
- colina/etanolaminafosfotransferasa Homo sapiens
- colina/etanolaminafosfotransferasa Drosophila melanogaster
El resultado puede verse aquí (tcoffee.html). Se aprecia que el alineamiento de la secuencia putativa (la primera) parece truncarse, lo que, junto con el reducido tamaño del contig en el que se encontraba, sugiere que la proteína se encuentra en dos contigs y no ha sido posible obtener la posible secuencia completa.
La estrategia que se decidió seguir en adelante fue realizar otro protein-BLAST en línea, esta vez sin filtro de especies, para encontrar una proteína homóloga más parecida a la encontrada en E.dispar (de una especie más cercana), e intentar alinearla en su genoma, esperando encontrar dos buenos hits, en diferentes contigs, que confirmaran la hipótesis de que la proteína se encontraba truncada en dos contigs.
Sin embargo, se obtuvo un resultado inesperado: el mejor hit era la colina/etanolaminafosfotransferasa de E.dispar, ya descrita en NCBI.
El siguiente paso fue alinear esta proteína encontrada con el genoma de E.dispar. Como se esperaba, se obtuvo un muy buen alineamiento, pero, sorprendentemente, al final de éste, se había introducido un gap (en la secuencia query, la obtenida desde el NCBI), con un codón stop en medio de la secuencia genómica con la que se había alineado (la secuencia subject, el genoma de E.dispar). (tblastn.out) (tblastn.hsp.out) Lo más intuitivo era pensar que podría tratarse de un pequeño intrón.
Pero… ¿podría ese codón stop tratarse de una selenocisteína, en ausencia de maquinaria?
Entonces, se decidió realizar un alineamiento múltiple partiendo de la colina/etanolaminafosfotransferasa de E.dispar (la secuencia obtenida desde el NCBI) frente a las cuatro secuencias co-ortólogas anteriormente mencionadas, para ver cómo se alineaba este gap. (tcoffee.html)
En el alineamiento múltiple obtenido mediante el tcoffee se aprecia que se añade un gran gap en todas las secuencias co-ortólogas, respecto a la colina/etanolaminafosfotransferasa de E.dispar: parece que la colina/etanolaminafosfotransferasa de E.dispar posee un fragmento (exón) más que las demás.
Se decidió pues, seguir con el alineamiento de ésta frente al genoma de E.dispar, para intentar dilucidar de qué se trataba el gap que se introducía en el análisis mediante BLAST en línea de comandas. El análisis del exonerate muestra en detalle cómo el fragmento alineado con el gap corresponde a un intrón (exonerate.out). También se muestran los resultados del análisis mediante genewise (genewise.out).
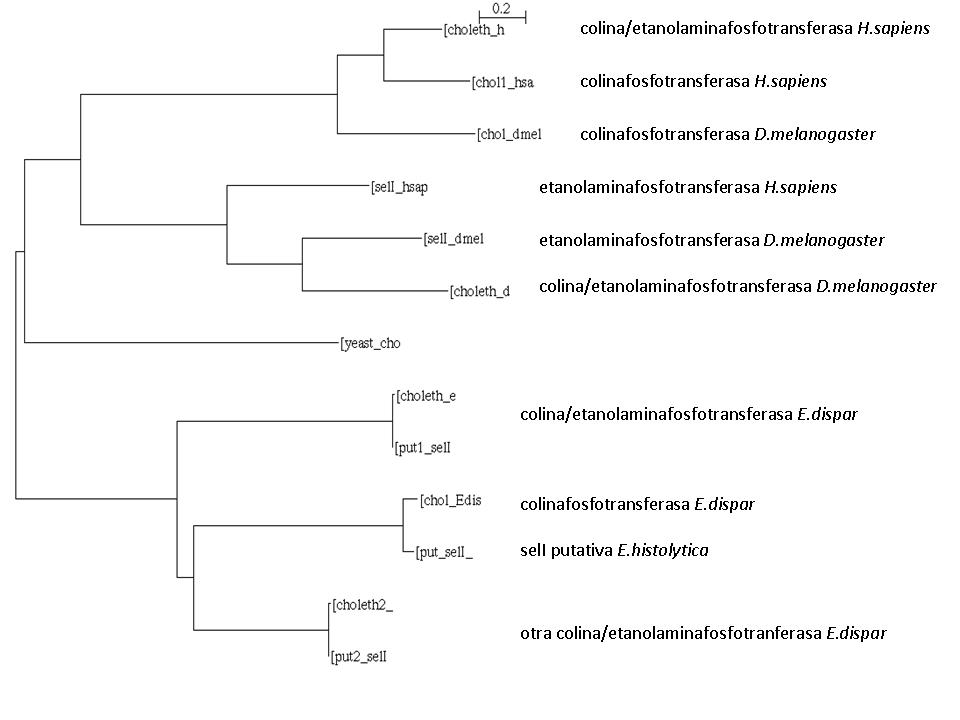
Finalmente, se decidió hacer un árbol con todas las posibles secuencias co-ortólogas encontradas, así como con la colina/etanolaminafosfotransferasa de levadura, que tras analizar la filogenia del gen en PhylomeDB se observó que únicamente poseía un homólogo. En este alineamiento múltiple (tcoffee.html) se incluyeron las secuencias de:
- las selI de H.sapiens, D.melanogaster
- las colina/etanolaminafosfotransferasas de H.sapiens, D.melanogaster, levadura y dos de E.dispar que hay descritas en NCBI
- las colinafosfotransferasas de H.sapiens, D.melanogaster (no está antada como tal pero es el mejor hit recíproco de la de H.sapiens) y E.dispar.
- las dos co-ortólogas putativas encontradas en E.dispar* y la encontrada en E.histolytica
*Partiendo de la secuencia de la SelI de H.sapiens, se obtuvieron dos buenos alineamientos en el genoma de E.dispar, en contigs diferentes (tblastn.out) (tblastn.hsp.out). Se obtuvieron las secuencias peptídicas de las proteínas putativas y se incluyeron en el árbol (genewise.out 1) (genewise.out 2).
¿Por qué se decidió incluir una nueva homóloga, la colinafosfotransferasa?
Un análisis más exhaustivo de la SelI de Homo sapiens en Ensembl, proporcionó información sobre un tercer parálogo a la SelI (etanolaminafosfotransferasa) y a la colina/etanolaminafosfotransferasa: la colinafosfotransferasa. Partiendo de esta información se encontraron secuencias homólogas tanto en D.melanogaster como en E.dispar, por lo que decidieron incluirse en el alineamiento múltiple. (árbol) (árbol).
Para poder analizar el árbol filogenético construido hay que recalcar que se incluyeron tanto las secuencias putativas encontradas en el genoma sobre el cual se realizó el trabajo, así como las secuencias ya definidas en el NCBI para el organismo en cuestión. Es por ello que en la parte inferior del árbol tanto la primera pareja como la tercera corresponden entre sí a la misma proteína: cada una de las dos colina/etanolaminafosfotranferasas definidas para E.dispar en el NCBI (alineadas idénticamente con sus putativas encontradas en el genoma de E.dispar partiendo de la selI de Homo sapiens). La segunda pareja corresponde a la colinafosfotransferasa de E.dispar alineada con la homóloga putativa encontrada en E.histolytica. E.dispar
Según este árbol filogenético puede apreciarse que ocurrió una duplicación antes de la divergencia de humano y mosca, pero que además, después, en cada una de las especies una de las copias sufrió otra duplicación: así se acabó con tres parálogas en H.sapiens y otras tres parálogas en D.melanogaster, co-ortólogas entre sí. Se aprecia también que la levadura sólo posee una de las copias, mientras que el organismo analizado posee tres (según lo anotado en el NCBI), de las cuales se lograron definir dos en E.dispar y una en E.histolytica.
EhSEP2 de Emiliania huxleyi
Para la EhSEP2 de Emiliania huxleyi se obtuvo un buen alineamiento (tblastn.out) (tblastn.hsp.out), donde la selenocisteína se alineaba con una cisteína. Para confirmar que se trataba de una proteína homóloga, con la secuencia peptídica de la EhSEP2 putativa en E.dispar, obtenida a partir del genewise (genewise.out), se realizó un protein-BLAST en línea en NCBI en con el filtro de especies de E.huxleyi.
El mejor hit obtenido fue EhSEP2. También se realizó un protein-BLAST de la secuencia de EhSEP2 proporcionada por los profesores y se confirmaron los resultados esperados. Esta vez se trababa del mejor hit recíproco, encontrándose la proteína homóloga en E.dispar, pero no una selenoproteína, ya que en lugar de selenocisteína posee una cisteína. Los resultados del tcoffee muestran el alineamiento (tcoffee.html).
Búsqueda de selenoproteínas en E. histolytica:
Además de la búsqueda de selenoproteínas designada en el genoma de E. dispar, se ha realizado una segunda búsqueda de este tipo de proteínas en una especie muy próxima: E. histolytica. A través de estudios genéticos se ha comprobado que ambas especies presentan una alta homología de genoma, a excepción de un conjunto de genes que codifican para cisteína proteasas en E. histolytica y que parecen ser los responsables de que esta especie sea patogénica, al contrario que E. dispar. Lo interesante de estos genes es que, aunque en E. dispar se conserva su loci, la secuencia está altamente degenerada por inserciones, deleciones y múltiples codones stop, hecho que plantea la posibilidad de que se trate de genes que codifican para selenoproteínas.
Sin embargo, todos los resultados obtenidos tras la búsqueda parecen indicar la ausencia de selenoproteoma en E. dispar, por lo que se decidió estudiar el genoma de E. histolytica para descartar si la diferencia entre estas dos especies se debe a la presencia de selenoproteínas en E. histolytica. Una segunda razón por la que se ha estudiado también este genoma es describir mejor el árbol evolutivo de las selenoproteínas dentro de la familia de las amebas; saber si la ausencia de selenoproteínas es solo un caso aislado en E. dispar o si se produjo antes de la separación entre especies.
La búsqueda de selenoproteínas en E. histolytica comenzó por realizar un tblastn entre el genoma de la especie y las secuencias peptídicas de selenoproteínas obtenidas de la base de datos en internet Selenodb y otras descritas en artículos de revisión y en la base de datos NCBI.
De esta manera se trataba de obtener una idea aproximada de cómo de óptimo es el alineamiento y si podría existir o no una homología de secuencia. Se obtuvieron varios hits de secuencia de los cuales solo 6 presentaban un e value significativo (con un score menor de -10) y un % de alineamiento óptimo. Sin embargo, en ninguno de los casos la selenocisteína (u) presente en las selenoproteínas se mostraba alineada frente al genoma de E. histolytica. Esto podría haber sido consecuencia de la presencia de un codón TGA en el genoma de E. histolytica reconocido como un codón de stop que interrumpiera la pauta de lectura. Resultados tBLASTN
Los hits que resultaron ser significativos correspondieron con la selenoproteína selI y eEEFsec en las especies D. melanogaster y Homo sapiens, y eEFsec en Mus musculus.
El paso siguiente fue realizar un estudio más detallado de estos hits por alargamiento de secuencia. Se realizó un exonerate y un genewise en el que en el alineamiento de la subsecuencia del genoma de la especie frente a la secuencia peptídica de los hits significativos tampoco aparecía la “u ” por lo que se barajaba la posibilidad de que esta no estuviera presente en el genoma. (para ver los resultados acceder a travé de los enlaces siguientes:
- Resultado del genewise de Homo sapiens para eFSec
- Resultado del genewise de Homo sapiens para selI
- Exonarate de Homo sapiens para selI
- Resultado del genewise de Drosophila para eFSec
- Resultado del genewise de Drosophila para selI
Para estar seguros de la homología de secuencia, se realizó un blastp de la secuencia putativa en E. histolytica frente a las proteínas de la base de datos NCBI, para comprobar si el mejor hit descrito para la supuesta selenoproteína en E. histolytica también correspondía con eEFsec/SelI en H. sapiens y D. melanogaster. Los resultados para la proteína selI mostraron como en el caso de E. dispar que el mejor hit no era SelI en H. sapiens o D. melanogaster sino la proteína colina/etanolaminafosfotransferasa. En cambio, para la proteína putativa eEFsec en E. histolytica el mejor hit tampoco resultó ser el esperado eEFsec sino otro factor de elongación: EF1α.
Tras realizar un alineamiento múltiple de proteínas con el programa tcoffee, entre la secuencia putativa de eEFsec en E. histolytica, y eEFsec y Ef1α en H. sapiens y en D.melanogaster, se comprobó que las secuencias putativas descritas en E. histolytica para eEFsec son un fragmento del gen para EF1α resultados tCoffelo que acabó por descartar la presencia del gen eEFsec en E. histolytica.
Analizando los resultados obtenidos mediante el recurso PFAM de la página web del Instituto Sanger, que se trata de una colección de familias de proteínas, se observó que la homología existente entre las dos secuencias de factores de elongación se debe a que ambos comparten dominios estructurales que pertenecen a la familia de factores de elongación TAU.
Resultados de búsqueda de tRNAs de Selenocisteína
Mediante la ejecución del programa tRNAscan no se han encontrado posibles tRNAs de selenocisteína. Pulsar aquí para ver output del programa tRNAscan.

 volver SelI
volver SelI
