


 |
 |
|
 |
Núria Martínez (nuria.martinez09@campus.upf.edu)
Mònica Mercadé (monica.mercade01@campus.upf.edu)
Gisela Miró (gisela.miro01@campus.upf.edu)
Clara Ruiz (clara.ruiz02@campus.upf.edu)
Mireia Sans (mireia.sans01@campus.upf.edu)
Meritxell Vinyoles (meritxell.vinyoles01@campus.upf.edu)
| Introducció | Selenoproteines | P.infestans | Materials i mètodes | Resultats i Discussió | Conclusions | Bibliografia |
INTRODUCCIÓ
Les Selenoproteïnes són proteïnes que incorporen en la seva estructura la selenocisteïna, un aminoàcid codificat pel codó UGA en presència d'uns elements característics, els SECIS. Aquest codó característicament codifica pel codó STOP de finalització; aquesta doble funció en la codificació del codó UGA suposa un repte en l'anotació de selenoproteïnes en nous genomes, ja que amb els programes estàndard aquests codons són anotats automàticament com a codons STOP.
Recentment s'han identificat elements que caracteritzen les seqüències codificants de les selenoproteïnes i que permeten la seva identificació i caracterització en un nou genoma. Actualment el gran repte és poder identificar selenoproteïnes en nous genomes recentment seqüenciats, però també tornar estudiar genomes ja analitzats per cercar possibles selenocisteïnes obviades, i anotades com a finalització de la proteïna.
El nostre projecte consisteix en la recerca i anotació de gens de selenoproteïnes en el genoma d'un organisme recentment seqüenciat, Phytophthora infestans. La nostra cerca l'hem realitzat tant a partir de selenoproteïnes ja anotades en el genoma d'altres organismes, com el descobriment de noves selenoproteïnes característiques del nostre organisme. La nostra investigació, però, no es centrarà només en l'estudi de selenoproteïnes en si, sinó també en la maquinària relacionada, és a dir proteïnes que participen en la síntesi de selenoproteïnes.
Torna a l'inici
SELENOPROTEINES
Les Selenoproteïnes són proteïnes que incorporen selenocisteïnes (aa 21), una cisteïna anàloga a la qual un àtom de seleni substitueix un de sofre. La incorporació de l'aminoàcid selenocisteïna és especificat pel codó UGA, normalment un codó stop. En un mRNA el codó UGA pot tenir dues lectures, la de codó stop ,molt més freqüent, o la de selenocisteïna.
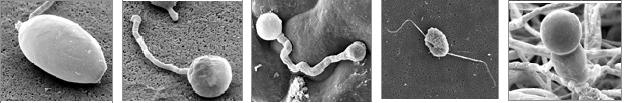
L'element SECIS: és el que determina que el codó UGA sigui llegit com a selenocisteïna. L'estructura secundària/terciària del mRNA forma aquest element SECIS, que es troba a la regió 3'-untranslated.
Tot i que s'ha vist que buscar elements SECIS potencials és útil per identificar noves proteïnes, pot donar molts falsos positius. El mètode que s'aplica per reduir el nombre de falsos positius es basa en correlacionar els elements SECIS predits amb aquells gens que s'hi trobi un fort biaix de codons característic de les regions codificants per proteïnes, extès darrere del codó UGA.
Per a la biosíntesi de selenoproteïnes hi intervenen diferents elements:
Diversos components de la maquinària de síntesi de les selenoproteïnes estan conservats entre diferents espècies suggerint un rol important de les selenoproteïnes en la funció cel·lular.
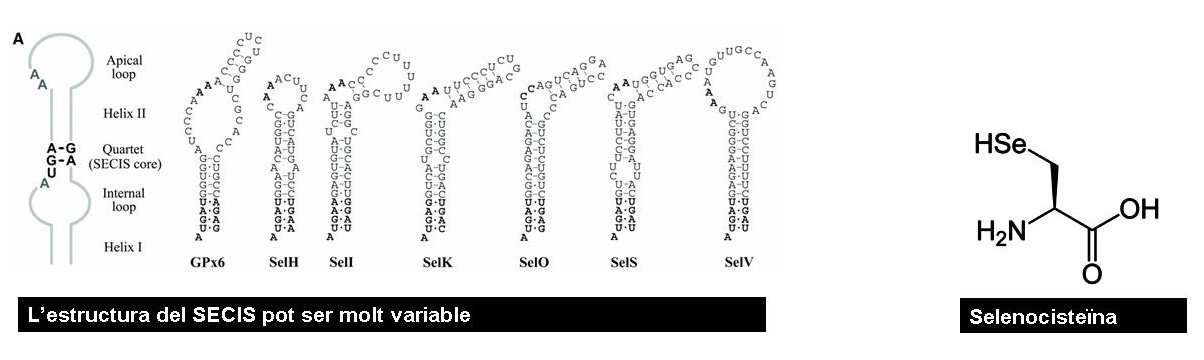
La Selenocisteïna: El seleni és un nutrient essencial pels animals, microorganismes i alguns eucariotes, per això la deficiència en seleni pot causar malalties. Aquest Seleni es troba sobretot en les selenoproteïnes, i per això aquestes són tant importants.
La majoria de selenoproteïnes són enzims redox, i se'ls hi atribueix una hipotètica capacitat antioxidant gràcies a la seva participació en la regulació de l'estat redox de la cèl·lula.
Aquestes selenoproteïnes han passat desapercebudes durant molt de temps, ja que la majoria de programes no tenen en compte que el codó UGA pot codificar, a part pel codó stop, per a una SeC. En trobar un UGA el programa consideraria que s'atura la pauta de lectura, cometent per tant l'error d'obviar la presència d'una selenocisteïna.
Recentment s'han identificat elements que caracteritzen les seqüències codificants de les selenoproteïnes i que permeten la seva identificació i caracterització en un nou genoma.
Alguns d'aquests elements són:
Actualment en la cerca de selenoproteïnes a més a més de tenir en compte la codificació del codó UGA per una SeC, per tenir la certesa que ens trobem davant d'una, el fet de seguir trobant homologia després d'un potencial codó stop (UGA), o el fet de trobar un element SECIS a prop, ens pot fer pensar que en tenim una davant.
Torna a l'inici
Phytophthora infestans
Phytophthora infestans és un protist fungoide de la classe Oomicets. Es tracta d'una paràsit de plantes que infecta sobretot patates tomàquets i altres Solanàcies, causant una malaltia que rep el nom de "tizón tardío" o "mildiu de la patata". Pot arribar a provocar grans plagues i de fet va ser la principal causa de la "The Great Famine", una passa de fam que va haver-hi a Irlanda i a Escòcia a mitjans del segle XIX.
Les espores de P.infestans hivernen als tubercles infectats i es propaguen ràpidament en condicions càlides i humides. Les espores es desenvolupen a les fulles, i es poden dispersar mitjançant la pluja, que pot arrossegar les espores al terra on infectarien tubercles joves; o el vent, que les pot dispersar.
Els tubercles infectats desenvolupen taques grises o negres que són de color marró o vermellós per sota la pell. Una infecció bacteriana, secundària a la produïda per P.infestans, farà que els tubercles es podreixin ràpidament i que desprenguin una olor desagradable. Alhora, les fulles presentaran grans taques i les tijes s'ennegriran.
Per una banda, intentar limitar la font d'infecció sembla ser la millor forma d'evitar una plaga: les llavors que es planten han de ser de bona qualitat i s'han de descartar totes aquelles patates de la temporada anterior que han patit la malaltia, ja que poden actuar com a fonts d'infecció. Per altra banda, hi ha certes característiques metereològiques que propicien l'extensió de la plaga.
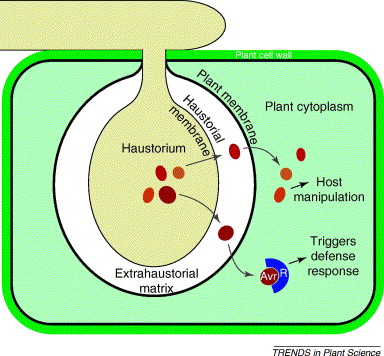
Aquesta figura mostra la interfase entre el patogen i la planta hoste. L'oomicet penetra en la paret de la planta, però no la membrana de la planta. Les proteïnes efectores (cercles vermells i taronges) són secretats pel patogen i es postula que entren al citoplasma de l'hoste per alterar el metabolisme i les vies de defensa. Quan les proteïnes de resistència de l'hoste (R) reconeixen les proteïnes efectores del patogen aquestes són reconegudes com a Avr.
S'estima que la mida del genoma de Phytophthora infestans és de 237 Mb, i un anàlisi microscopi indica que el genoma consta d'entre 8 i 10 cromosomes.
Aquest genoma, per tant, es troba ensemblat fins a les unitats de supercontigs, i cal dir que no és complet, ja que hi resten zones per acabar de seqüenciar.
Torna a l'inici
MATERIALS I MÈTODES
Cliqueu aquí per
veure el protocol d'ABP
Esquema Materials i Mètodes
Cerca de Selenoproteïnes homòlogues d' Homo sapiens:
1. Cerca de la seqüència de la selenoproteïna a buscar
2. Realització el BLAST (tBLASTn)
3. Elecció dels millors hits
4. Anàlisi de les potencials proteïnes homòlogues
5. Cerca de SECIS en les seqüències de les selenoproteïnes predites mitjançant homologia
Cerca de noves Selenoproteïnes en Phytophthora infestans: 1. Cerca de SECIS en tot el genoma
2. Cerca de selenoproteïnes potencials en tot el genoma
Cerca de Selenoproteïnes homòlogues d'Homo sapiens
En la cerca de selenoproteïnes en el genoma de Phytophthora infestans el primer que cal fer és trobar la seqüència genòmica d'aquest organisme. Hem trobat la seqüència en la pàgina web del Broad Institute
un centre involucrat amb projectes de recerca de les comunitats científiques de MIT i Hardvard. A partir d'aquí la nostra cerca ha estat basada en les possibilitats que ofereix aquesta pàgina web, tant a nivell de realitzar el BLAST, com altres opcions que hem utilitzat al llarg de la investigació.
A continuació s'explicaran de manera detallada els passos que hem anat seguint al llarg del procés de cerca de selenoproteïnes, ja conegudes en Homo sapiens, en Phytophthora infestans: Buscar la seqüència protèica de les seleneproteïnes: Hem buscat en la pàgina web del SelenoDB les proteïnes que ens interessen per fer la cerca; en aquest cas hem seleccionat i analitzat tant sols les selenoproteïnes, i maquinària relacionada, de l'espècie Homo sapiens, ja que es tracta l'espècie en la que s'han trobat més selenoproteïnes. El que s'utilitza en la cerca és la seqüència d'aminoàcids.
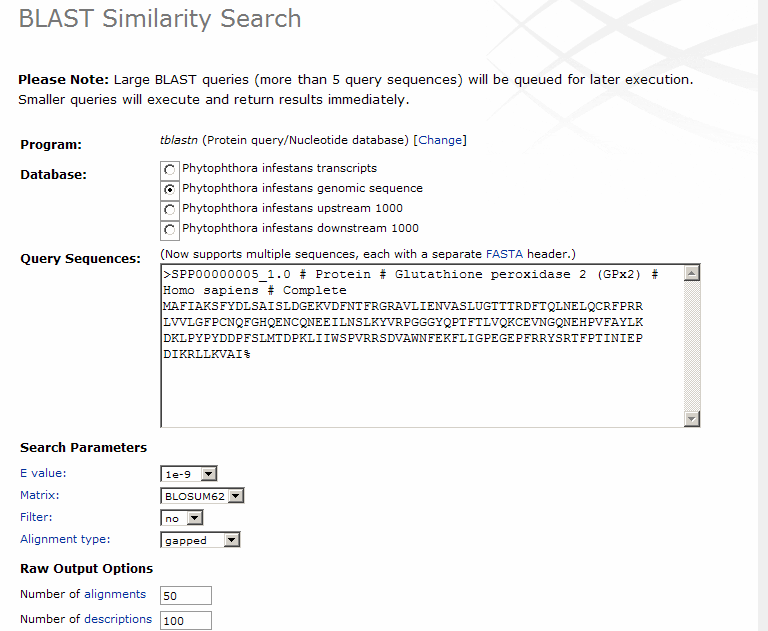
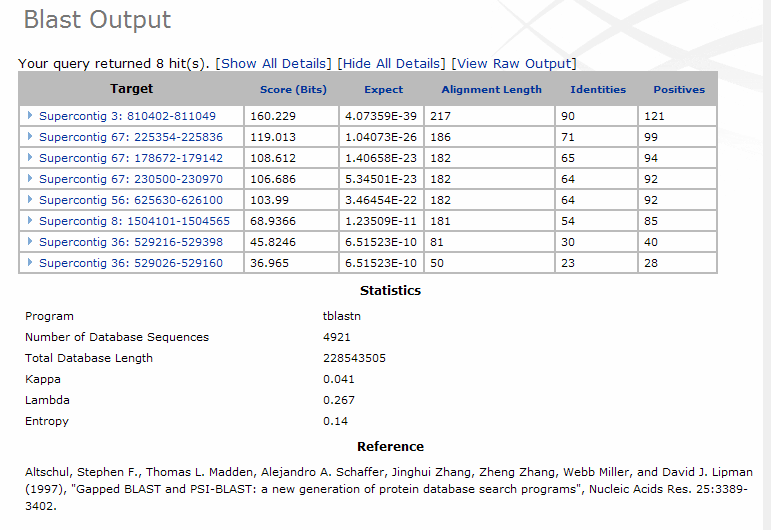
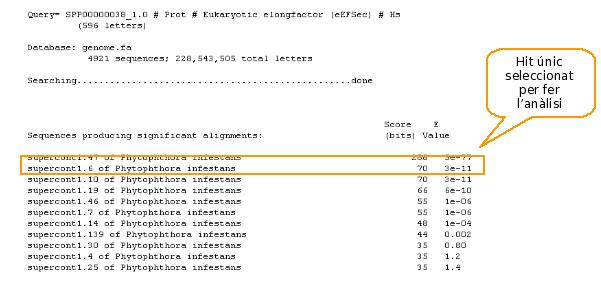
Realització del BLAST (Basic Local Alignment Search Tool): El següent pas ha estat realitzar el BLAST de les selenoproteïnes contra el genoma a analitzar. Aquest pas l'hem realitzat des de la pàgina web del Broad Institute; on hem cercat la base de dades del nostre organisme i hem seleccionat l'opció: BLAST Search i posteriorment tblastn (Protein query/Nucleotide database). A partir d'aquí hem seleccionat com a base de dades la Phytophthora infestans genomic sequence, i hem introduït la seqüència de la selenoproteïna humana que s'ha de cercar com a query sequence. Per tal de que els resultats obtinguts siguin significatius hem d'introduir alguns paràmetres de cerca (Search Parameters): E-value inferior a 1e-9 i no filter.
Els alineaments proposats en el BLAST del Broad Institute s'han comprovat realitzant un BLAST a través del terminal. Els resultats obtinguts en ambdós han estat comparats i finalment determinats els dos com a bons. Els passos posteriors han estat basats principalment en els resultats del Broad Institute, tot i que s'han anat comprovant amb els resultats del BLAST del terminal, veure taula de resultats. Elecció dels millors hits: Com a resultat del BLAST, tant del Broad Institute com del terminal, tenim un seguit de hits per a cada una de les selenoproteïnes humanes blastejades. En el nostre cas hem obtingut molts alineaments significatius (considerant significatius valors d'E-value < 1e-10), és per això que hem decidit fer certes restriccions a l'hora de seguir amb l'anàlisi. Quan una selenoproteïna X ha donat alineaments a diferents supercontigs del genoma hem aplicat les següents restriccions: tenint en compte els resultats obtinguts pel BLAST del terminal, escollirem sempre, pel posterior anàlisi, l'alineament amb el valor d'score més alt; i els hits següents els analitzarem només si el seu score és igual o més gran que la meitat de l'score més alt. Les consideracions anteriors només es tindran en compte sempre i quan els hits tingui un E-value < 1e-10.
Anàlisi de les potencials proteïnes homòlogues: el procés explicat a continuació s'ha realitzat amb tots els hits escollits de cada una de les selenoproteines analitzades:
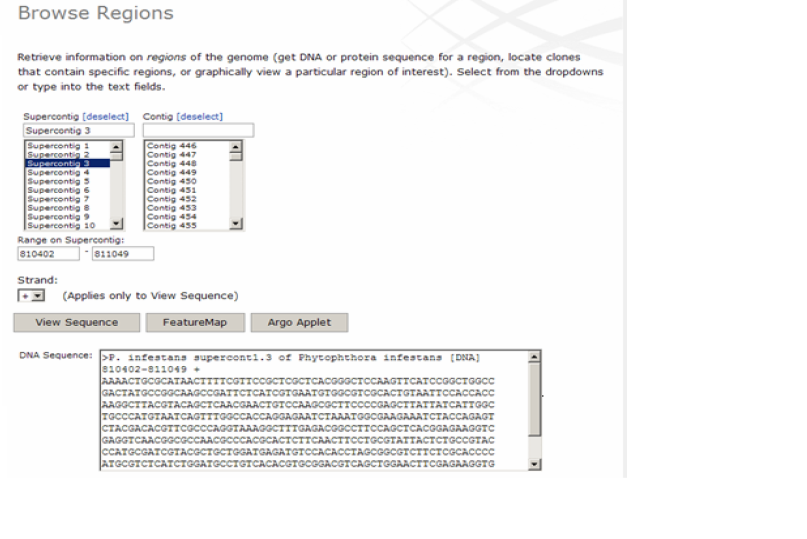
Cerca de la seqüència genòmica corresponent als aminoàcids alineats amb el BLAST. Aquest pas l'hem realitzat amb una opció de la pàgina web del Broad Institute, el Browse Regions, on s'introdueixen els valors aconseguits en l'output del BLAST que indiquen la regió nucleotídica corresponent a l'alineament, tot indicant el supercontig on es troba la seqüència on s'ha alineat la selenoproteïna.
Per saber si en la resta de l'anàlisi s'ha d'utilitzar l'strand positiu o el negatiu el que hem fet ha estat utilitzar la pàgina web ExPASy Proteomics Server per traduir la nostra seqüència a aminoàcids. Si la direcció obtinguda és 5'-3' utilitzarem l'strand positiu. En canvi si els aminoàcids resultants que equivalen als de Phytophthora infestans en l'alineament són els que corresponen a la direcció 3'-5' hem escollit l'strand negatiu en el Browse Region i hem utilitzat aquesta seqüència en la resta del procediment. Cerca de l'estructura gènica. Un cop determinada la regió a estudiar, regió potencialment corresponent al gen que codifica per una proteïna homòloga a la selenoproteïna humana, s'han de buscar els introns. Això es pot dur a terme utilitzant el programa genewise. Aquest programa l'hem utilitzat tant a nivell de pàgina web www.ebi.ac.uk/Wise2/index.html com pel Terminal. Aquesta última opció s'ha executat per obtenir els outputs en format GFF de la següent forma:
A l'hora d'utilitzar aquest programa s'ha de tenir en compte que té certes limitacions. El genewise utilitza, per trobar l'estructura gènica, la freqüència de codons en Homo sapiens, per tant pot ser que es produeixin errors en les prediccions degut al biaix de codons.
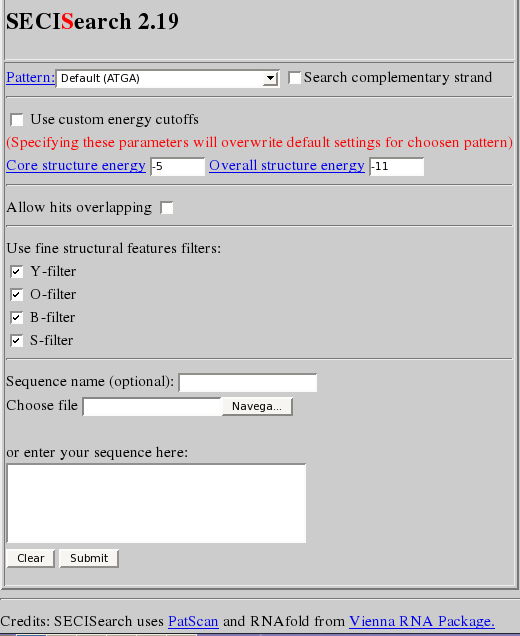
Cerca de SECIS en les seqüències de les selenoproteïnes predites mitjançant homologia: El SECISearch és un programa que et mostra possibles SECIS en una seqüència, utilitzant una conjunció de Patscan i RNAfolder. Els SECIS es caracteritzen per tractar-se d'unes estructures tridimensionals a la cadena de nucleòtids i que es troben properes al gen que codifica per a una selenoproteïna. El problema bàsic que tenen els SECIS és que no presenten gaire conservació a nivell de seqüència, però sí a nivell estructural. D'aquesta manera, SECISearch, i basant-se en Patscan, busca certs patrons característics de SECIS a la seqüència que nosaltres introduïm. A continuació determina l'estabilitat que presenten els SECIS predits, limitant el nombre de hits que nosaltres observem. Allarguem 1000 nucleòtids per banda a partir de la seqüència del gen (que inclou tant el codó de selenocisteïna com el de stop real), i n'estudiem la presencia de SECIS utilitzant totes les opcions de patrons disponibles: default (ATGA), default (GTGA), strict, loose (canonical), i loose (canonical and non-canonical). La resta de camps els deixem igual. Per no ometre possibles candidats, busquem també tots els patrons a la cadena complementària. Aquesta opció es selecciona clicant la opció que hi ha a la dreta dels patrons.
En el cas que s'obtinguin pocs o cap hit, i que aquests siguin poc fiables, anirem allargant cada cop més la seqüència en les dues direccions: 1000 bases més tant upstream com downstream respecte a la seqüència anterior. Per a evitar possibles confusions, crearem un fitxer diferent cada vegada, en el qual determinem l'increment gradual en el nombre de nucleòtids. Quan haguem allargat la seqüència original 5000 nucleòtids per cada banda, donarem per vàlids tots els resultats que obtinguem, ja que un possible SECIS a més distància del gen probablement es tracti d'un fals positiu. Seguidament, i després d'aconseguir les seqüències dels possibles SECIS, el que farem és buscar-los en la seqüència genòmica i localitzar on es troben respecte a la regió codificant del gen de la selenoproteïna predita. Aquest pas el realitzem per discernir alguns SECIS que donin positiu per estructura, però que la seva posició en el genoma els descarta com a possibles candidats. Cerca de noves Selenoproteïnes en Phytophthora infestans 1. Cerca d'elements SECIS en tot el genoma:
Per a realitzar una cerca general de possibles SECIS en tot el nostre genoma hem de fer una sèrie de passos previs, ja que la seqüència genòmica es massa gran com per analitzar-la tota d'un sol cop. I. Així doncs, comencem descarregant-nos el fitxer amb tot el genoma des del Broad Institute, o bé el copiem de /disc8/genomes/P.infestans/genome.fa que els professors de l'assignatura ens van facilitar. Tot seguit, hem de fragmentar el genoma en diferents parts. A través del terminal fem anar les comandes 'head' i 'tail' de la següent manera:
Aquestes comandes ens permeten dividir el genoma en dos fitxers diferents, anomenats part1.fa i part2.fa, tots dos de la mateixa grandària. El que hem fet és dividir el nombre total de línies del fitxer original per la meitat i escriure aquest número després de les comandes 'head' i 'tail'. Aquest pas l'anem realitzant consecutivament, cada cop dividint per dos la grandària del fitxer anterior.
II. Una vegada tenim preparats els fitxers del genoma fragmentat, carreguem cada arxiu al SECISearch. Per limitar el nombre de resultats i que els que obtenim siguin més fiables, apliquem únicament un patró estricte (Strict), enlloc de mirar cada un dels patrons disponibles. El que sí fem és fer dos cerques per a cada fragment: una a la cadena principal i una altra a la cadena complementària. Tot i que només busquem el patró estricte, no volem ometre cap possible resultat en no considerar la cadena complementària de la que introduïm al programa.
De tots els hits que obtinguem en descartarem tots els que presentin un COVE score inferior a 15, quedant-nos únicament amb els més significatius.
Durant la cerca de SECIS ens han sorgit una sèrie de problemes que val la pena mencionar. Després d'haver dividit el nostre genoma en 16 parts iguals i haver començat a analitzar-les a través de SECISearch la pàgina web on es troba el programa va deixar de funcionar. I tot seguit, un cop restablerta, el programa només acceptava seqüències de menys de 100000 nucleòtids. Creiem que consisteix en un límit establert pels propis programadors degut a l'alta afluència d'aquests últims dies. Davant d'aquest handicap, enlloc d'anar dividint els fragments en subseqüències cada cop més petites hem trobat molt més pràctic utilitzar la comanda 'fastasubseq' des del terminal. On path.fa és el fitxer que volem particionar, start és el número del nucleòtid on volem que comenci, i lenght és la llargada de la seqüència. El resultant el redireccionem a un nou fitxer. III. Com que utilitzant el SECISearch hem obtingut molts hits, hem procedit a utilitzar SECISearch a través del terminal. Aquesta versió diferent del SECISearch ha estat creada per en Charles Chapple a partir del programa original, de manera que es pot fer anar a través de comandes al terminal de Linux. Com que SECISearch utilitza Patscan i RNAFolder, aquests programes s'han d'instal·lar prèviament a l'ordinador on estem treballant. Els clars avantatges que suposa aquest mètode és que els diferents paràmetres són modificables. Així doncs, en els resultats, enlloc de fixar-nos en el COVE score, el qual ens dóna molts falsos positius i falsos negatius, el paràmetre més determinant és l'energia lliure de l'element SECIS predits.
IV. A partir d'aquí, hem agafat una a una les seqüències dels elements SECIS predits i les hem introduït al SECISearch disponible per internet. L'objectiu d'això és obtenir els COVE scores de tots aquests hits, en part per corroborar que es poden prendre com a resultats vàlids i per altra banda poder disposar d'un altre paràmetre restrictiu a l'hora de discernir falsos positius.
V. A partir dels elements SECIS potencials en que hem obtingut un SCORE significatiu (un valor superior a 15), introduïrem la seqüència que ens ha donat el SECISearch dins del genoma.
Tot i que en un principi teníem la intenció d'analitzar tots els SECIS potencials obtinguts a partir del programa de SECISearch, al final només ens hem quedat amb els 2 valors d'SCORE més elevats, degut a la manca de temps.
2. Cerca de selenoproteïnes potencials en tot el genoma:
I. Un cop hem obtingut la seqüència dels elements SECIS, hem amplificat fins a 6000 nucleòtids davant de la regió 5'. Això ho hem fet amb la intenció de buscar possibles selenoproteïnes relacionades amb els nostres elements SECIS potencials.
II. Tot seguit hem introduït tots aquests nucleòtids al programa GeneID per tal de que ens fes una predicció dels possibles gens continguts en aquesta regió. Per realitzar el GeneID, hem posat com a organisme similar Plasmodium, ja que igual que el nostre organisme, és un protista.
III. A partir dels resultats obtinguts del GeneID, hem mirat si el gen predit era possible dins de la nostra seqüència genòmica, i en el cas que aquest es trobés en el mateix strand en el que es troba l'element SECIS hi hem buscat un UGA (per poder predir si es podia tractar d'una selenoproteïna). En el cas de trobar-hi un UGA, l'hem valorat segons en quina regió es trobava.
IV. A continuació hem anat al Broad Institute, per veure les proteïnes que aquesta base de dades ha predit de la nostra regió d'interès. Si anem a l'apartat de Find Features i introduïm la nostra regió d'interès, aquest programa ens mostra proteïnes que ha predit mitjançant diferents ESTs, Blastx i geneID, entre d'altres. A partir d'aquestes proteïnes predites, hem escollit aquelles traduïdes a partir de DNA del mateix Strand que el nostre element SECIS i hem buscat si alguna d'elles contenia algun UGA que ens portés a deduir la possible presència d'alguna selenocisteïna.
V. Finalment, a partir de les regions EST que tenim del nostre organisme (també presents en la base de dades del Broad Institute), hem analitzat aquelles regions codificants localitzades en la nostra zona dinterès per trobar-hi, també, UGA.
És important, comentar, que per tal de trobar possibles selenoproteïnes, hauria estat interessant utilitzar altres programes per poder-les deduir. Per exemple l'InterPRO, per tal de buscar dominis en les selenoproteïnes del nostre genoma, i poder-les comparar a partir d'aquesta aplicació.
Resultats i discussió Resultats de la cerca de proteïnes homòlogues a Selenoproteïnes d'Homo sapiens A continuació mostrem la nostra taula de resultats. Hem englobat en famílies les selenoproteïnes o homòlogues de selenoproteïnes trobades en humà. Si cliquem en el quadre del costat, es mostra la seq;uuml;ència caracteritzada de les homòlogues a aquestes (tant si es tracta de selenoproteïnes o d'homòlogues) que hem trobat en el nostre organisme Phytosphora Infestans, així com una explicació de perquè hem deduït aquest resultat.
Hem diferenciat les nostres proteïnes en: A la taula també podem veure que les proteïnes humanes que no tenen cap proteïna homòloga en el nostre organisme, les hem deixat de color en blanc.
Finalment, per accedir al tBLASTn que hem obtingut de cada família de proteïnes, només és necessari clicar link contingut en el nom de la família. Resultats de la cerca de noves Selenoproteïnes A continuació es mostren dos resultats diferents després d'haver fet una cerca per trobar selenoproteïnes noves:
Aquest PDF conté els resultats dels potencials elements SECIS obtinguts a partir del SECISearch.
CONCLUSIONS Hem enfocat el nostre treball en la cerca de les homòlogues de les selenoproteïnes humanes i de la maquinària implicada en Phytophthora infestans. En aquest apartat farem un anàlisi de les diferents famílies homòlogues de selenoproteïnes humanes i quina informació ens aporten tots aquests resultats obtinguts sobre aquest organisme.
Per realitzar una cerca de selenoproteïnes en el genoma, és important primer de tot, saber si l'organisme presenta algun tipus de maquinària que li pugui permetre la síntesi de selenoproteïnes. És per això que hem buscat homòlegs en el nostre organisme de les dues proteïnes humanes, eEFSec i SBP2, implicades en aquest procés. En comparar-les amb el nostre genoma, només hem obtingut resultats positius en el cas d'eEFSec. Tot i així, això ja indica la possibilitat de que aquest organisme pugui presentar alguna selenoproteïna, tal i com ja esperàvem, ja que gairebé tots els organismes estudiats fins ara presenten alguna selenoproteïna. De totes maneres no descartem la presència d'altres proteïnes pertanyents a la maquinària pròpies de l'organisme o del regne dels protistes.
El resultat més significatiu de la nostra cerca, però, és la caracterització de dues selenoproteïnes homòlogues a la TR1 (Thioredoxin reductase 1) i TR2 humanes en Phytophthora infestans. Tot i que hem classificat les nostres proteïnes segons si s'assembla més a una TR o a l'altre, aquesta diferenciació no és del tot clara. El més probable és que les TR d'humà provinguin d'una sola TR, del mateix ancestre que les de Phytophthora infestans, després en els dos organismes s'han duplicat i han evolucionat de manera diferent. Un cop realitzat l'anàlisi per trobar les seqüències de la proteïna, hem fet la cerca d'elements SECIS per comprovar que realment aquestes eren selenoproteïnes. Tot i que els resultats no han tingut l'score suficient com per determinar-los com a significatius, la resta de resultats obtinguts en l'anàlisi (alineament perfecte de la U amb la C, patró comú amb les TR humanes...) ens porten a pensar que realment es tracta de selenoproteïnes. A més l'estructura dels SECIS, tot i tenir un score baix, són força determinants.
A més de selenoproteïnes també hem trobat una homòloga de TR3. En aquest cas no trobem que sigui una selenoproteïna ni una homòloga amb cisteïna però hem considerat aquests resultats perquè l'alineament del BLAST era molt significatiu. Això pot ser degut a una duplicació d'una de les selenoproteïnes i a la posterior adquisició d'una nova funció.
A part de les dues selenoproteïnes identificades, hem trobat moltes altres proteïnes homòlogues que no presenten selenocisteïna en la seva seqüència d'aminoàcids, sinó que tenen una cisteïna en aquesta posició, en la majoria de casos, o algun altre aminoàcid.
Uns dels resultats que hem trobat més interessants, i que han centrat, en part, la nostra atenció, ha estat la família de les GPx (Glutathione peroxidases). Aquesta família conté 8 proteïnes diferents, en Homo sapiens, que inclouen tant selenoproteïnes com proteïnes sense selenocisteïna. Realitzar l'anàlisi d'aquesta família de proteïnes no ha estat fàcil a causa de la gran quantitat de hits obtinguts en la realització del primer BLAST, ja que cada GPx presentava alineaments significatius en diferents supercontigs, i en una mateixa regió d'un mateix supercontig trobàvem homologies per diferents GPx. L'allau de resultats ha fet que ens prenguéssim l'estudi d'aquesta família amb molt deteniment, per això hem fet una cerca exhaustiva de cada proteïna en cada supercontig, i a partir d'aquí hem extret les conclusions, referides a escollir en cada regió de cada supercontig quina GPx humana n'és més homòloga. Els resultats obtinguts indiquen que Phytophtora infestans presenta proteïnes homòlogues a les GPx4, GPx8 i GPx2 humanes. Hem identificat inicialment tres proteïnes de P. infestans homòlogues a GPx4, després d'obtenir les seqüències d'aminoàcids hem vist que les obtingudes a partir dels supercontigs 56 i 67 són idèntiques. Aquest resultat ens pot portar a dos plantejaments diferents. El primer presentaria la possibilitat que aquestes dues proteïnes fossin resultat d'una duplicació del gen; mentre que la segona explicaria aquest fet per una mala seqüenciació del genoma o per un ensamblatge erroni. Creiem que la segona possibilitat és la més probable ja que ens hem trobat amb altres resultats que evidencien aquest fet, com per exemple els resultats obtinguts en el supercontig 8 .
Tal i com esperàvem, tenint en compte la simplicitat del genoma de P. infestans respecte del de Homo sapiens, hem trobat diferents selenoproteïnes en humans que en aquest organisme són homòlogues amb cisteïna. D'aquesta manera es confirma la hipòtesi que, en general, els organismes més complexes contenen més selenoproteïnes.
A partir de la cerca de potencials elements SECIS dins del nostre genoma, no hem trobat cap selenoproteïna. Això ha sigut degut, per una banda, a la manca de temps, que ha impedit que féssim la cerca amb més de dos elements SECIS potencials. Per altra banda, tot i que els elements que hem analitzat tenien un alt score, no hem aconseguit trobar cap proteïna que tingués un UGA que ens portés a pensar la presència de selenoproteïnes. És per això, que creiem que és important que es segueixen buscant possibles selenoproteïnes en el nostre genoma.
Una altra observació és que hem trobat resultats idèntics, tant pel que fa l'estructura com el valor d'energia lliure, en diferents supercontigs. Això ens torna a portar a la hipòtesi que el genoma està mal ensemblat.
A partir de tota la infromació que hem anat exposant podem concloure que el nostre genoma no està correctament ensemblat i que per tant, no és tan gran com pensàvem en un principi. Segurament la seqüències d'alguns supercontigs se solapen i per això hem trobat alguns resultats repetits durant la recerca. Aquest fet ens porta a suggerir que es necessària la correcta seqüenciació del genoma de Phytophthora infestans per poder fer una bona cerca de selenoproteïnes, o altres investigacions.
Degut a aquest mal ensemblatge la recerca ha estat més difícil de l'esperat, però per altra banda la pàgina web del Broad Institute, on està disponible el genoma de Phytophthora infestans i altra informació sobre l'organisme, ens ha facilitat la feina, sobretot a l'hora de buscar regions concretes del genoma per poder ser estudiades.
Bibliografia Recursos WEB:
Referències: Agraïments
$ genewise -gff fitxerproteina.fa fitxergenomic.fa > output.gff
Per poder fer la cerca d'introns i exons, el primer que cal és allargar la seqüència genòmica que hem trobat utilitzant el mateix Browse Region, afegint 1000 nucleòtids a l'inici de la seqüència i 1000 al final.
Els resultats obtinguts un cop executat el programa ens indiquen l'inici i el final dels introns, en el cas que n'hagi trobat, de la proteïna en la seqüència genòmica. A partir d'aquí hem eliminat aquestes seqüències intròniques del genoma de Phytophthora infestans i unit els exons per obtenir el cDNA de la proteïna i poder determinar la seqüència aminoacídica final de la proteïna homòloga.
A partir d'aquí també hem vist si realment els codons que codifiquen selenocisteïna o cisteïna es troben dins un exó, la qual cosa ens indicaria que estem davant d'una possible selenoproteïna o un homòleg de la humana.
Un cop hem trobat la seqüència gènica que codifica per a un homòleg de selenoproteïna humana en Phytophthora infestans, realitzem el següent procediment per obtenir els SECIS teòrics:
$ head -1432091 genoma.fa > part1.fa
$ tail -1432091 genoma.fa > part2.fa
$ fastasubseq path.fa start length > output.fa
Torna a l'inici
Taula de Resultats:
Familia Proteica
Proteïnes humanes
Homòlogues
eEFSec
eEFSec
maquinària
GPx1
no homòloga
GPx2
cisteïna
GPx3
no homòloga
GPx4
cisteïna
GPx
cisteïna
cisteïna
GPx5
no homòloga
GPx6
no homòloga
GPx7
no homòloga
GPx8
cisteïna
cisteïna
DI1
no homòloga
DI
DI2
no homòloga
DI3
no homòloga
MsrA
MsrA
cisteïna
SBP2
SBP2
no homòloga
SPS
SPS1
no homòloga
SPS2
cisteïna
Sel15
Sel15
no homòloga
SelH
SelH
no homòloga
SelI
SelI
cisteïna
cisteïna
SelK
SelK1
no homòloga
SelM
SelM1
no homòloga
SelN
SelN1
no homòloga
SelO
SelO
cisteïna
cisteïna
SelP
SelP1
no homòloga
SelR1
no homòloga
SelR
SelR2
no homòloga
SelR3
cisteïna
SelS
SelS1
no homòloga
SelT
SelT1
no homòloga
SelU1
no homòloga
SelU
SelU2
no homòloga
SelU3
no homòloga
SelV
SelV
no homòloga
SelW1
no homòloga
SelW
SelW2
no homòloga
SelW3
no homòloga
TR1
altres
TR
TR2
selenocisteïna
TR3
selenocisteïna
Torna a l'inici
Torna a l'inici
1-SelenoDB
2-Broad Institute
3-GeneWise
4-Exonerate
5-ExPASy
6-SECISearch
7-ClustalW
8-GeneId
Per la seva inestimable col·laboració i totes les hores dedicades, a en Charles.
A la Núria per ajudar-nos en aquells moments en els que no sabíem per on tirar.
A en Roderic per donar-nos l'oportunitat de descobrir les selenoproteïnes.
A tots els nostres companys per fer més amenes totes les hores compartides davant la pantalla.
I a tu, visitant, per dedicar un temps a llegir la nostra pàgina.
Gràcies!