
RESULTATS TR (supercontig 32)
Seqüència d'una proteïna SENSE selenocisteïna ni cisteïna homòloga a TR1 i TR2:

Seqüència de nucleòtids on es troba la proteïna:
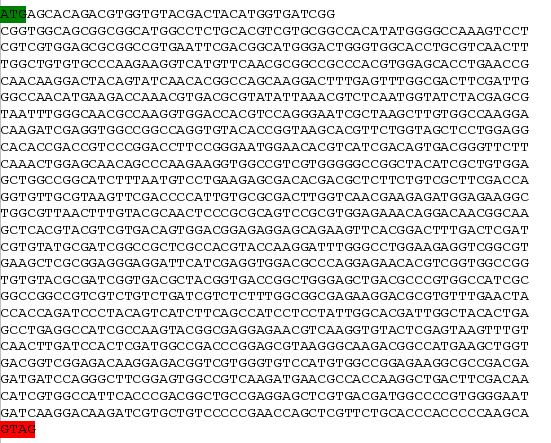
Discussió de la proteïna:
Aquesta proteïna és homòloga a TR1 i TR2, tot i que no presenta ni una selenoproteïna ni una selenocisteïna. Tot i aix´ l'hem analitzat perquè hem obtingut uns valors molt significatius en realitzar el Blast.

Quan hem realitzat el tBlastN, hem vist que els valors obtinguts del supercontig 32 eren molt semblants en TR1 i TR2, i molt significatius. Tot i que ni en l'alineament, ni en un posterior allargament dels aminoàcids hem vist presència d'alguna possible selenocisteïna ni de cap cisteïna, hem analitzat la proteïna perquè hem vist que tenia una alta semblança amb les proteïnes TR humanes.
TR1: Score = 244, Expect(3) = 1e-63
TR2: Score = 243, Expect(3) = 1e-63
TR3: Score = 228, Expect(3) = 9e-59
Quan hem realitzat el Genewise (resultat GFF), hem vist que la nostra proteïna no presenta cap intró. Això ho hem corroborat analitzant-la amb l'exonerate, i comprovant-ho amb els transcripts del Broad Institute (que es basen en EST i en prediccions del GeneId), on hem vist que la nostra seqüència és idèntica a aquesta.
Per comprovar que la nostra proteïna realment no presenta cap selenocisteïna ni cisteïna, hem amplificat la seqüència per la part 3', l'hem passat a aminoàcids, i hem comprovat que els stops obtinguts no estiguéssin sintetitzats per un codó (UGA). A més, hem mirat que no es seguís algun patró similar al que trobem a la selenoproteïna humana (UGU). Cap dels dos resultats ha sigut positiu, és per això que podem estar segurs que aquesta proteïna NO és homòloga ni és selenoproteïna. La gran semblança, però, que té respecte la proteïna humana, pot ser deguda a que sigui una duplicació de les selenoproteïnes de Phytosphthora Infestans que posteriorment ha perdut la funció.

