Selenoproteïna K
La Selenoproteïna K és una selenoproteïna que es troba anotada a selenoDB en Myotis lucifugus amb 4 variants: SPG00001515_2.0, 1516, 1517 i 1518. D'aquestes, només 1516 es troba mal anotada, la resta comencen per metionina. Al utilitzar qualsevol d'aquestes 3 com a query, obtenim dos hits, un a l'scaffold KB114370.1 i un a l'scaffold KB100898.1, en el sentit contrari a l'anotació. Per aquest motiu, vam decidir utilitzar només una d'aquestes querys.
Per a fer més fàcil la discussió, parlarem de SelK(+), pel primer scaffold i SelK (-), pel segon scaffold.
SelK (+)
Com ja hem dit, qualsevol de les entrades de SelK anotada per Myotis lucifugus ens dóna un blast, on el hit amb un e-value més petit és l'scaffold KB114370.1. Aquest es troba en el mateix sentit que l'anotació (+). El tcoffee té una puntuació de 100, que indica que les proteïnes són molt semblants.
L'exonerate i el genewise han predit tots dos una proteïna. Tant l'exonerate (raw score 429), com el genwise (score 201,98 bits), es prediu un gen sense seqüències intròniques. Tot i així, podem apreciar diferències notables entre les dues prediccions.
L'estructura predita mitjançant exonerate té un codó stop en la mateixa posició que la selenoproteïna de la query (l'avantpenúltim aminoàcid). La predicció per genewise no cobreix els 3 últims aminoàcids, probablement perquè interpreta la selenocisteïna com a codó stop. Deduïm així que la selenoproteïna està conservada.
Pel que fa a la predicció d'elements SECIS, el SECISearch 3 ens prediu un element SECIS a la posició 3', que ens valida la teoria que SelK és una selenoproteïna en Myotis davidii. D'altra banda, SEBLASTIAN ens confirma que en el nostre subseq trobem SelK, mostrant l'homologia del nostre scaffold amb la Selenoproteïna K de Canis lupus familiaris. Amb tota aquesta informació, podem predir l'estructura de la selenoproteïna:
SelK (-)
El segon hit del blast, tot i tenir un e-value més gran, també ens dona un valor prou baix com per considerar-lo. Es tracta de l'scaffold KB114370.1. Aquest es troba en el sentit contrari a l'anotació (-). El tcoffee té una puntuació de 98, que indica que les proteïna presenta gran homologia amb la nostra query.
L'exonerate i el genewise han predit tots dos una proteïna. L'exonerate prediu una proteïna amb 2 introns i 3 exons, amb una raw score de 405. El genewise, en canvi, prediu 3 introns i 4 exons, amb una score de 161.90.
Ambdues prediccions presenten un codó stop en la mateixa posició que la selenoproteïna de la query. Deduim així que la selenoproteïna està conservada.
Pel que fa a la predicció d'elements SECIS, el SECISearch 3 ens prediu un element SECIS a la posició 3', que ens valida la teoria que SelK és una selenoproteïna en Myotis davidii. D'altra banda, SEBLASTIAN ens confirma que en el nostre subseq trobem SelK, mostrant l'homologia del nostre scaffold amb la Selenoproteïna K de Homo sapiens. Amb tota aquesta informació, podem predir l'estructura de la selenoproteïna:
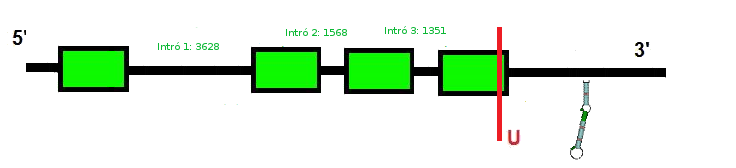
Donat que trobem dos variants de SelK, anotades en 2 scaffolds diferents, podem dir que aquesta està duplicada. No sabem si la duplicació és funcional o no, ja que podria tractar-se d'un pseudogen però, podem dir que la proteïna amb la selenocisteïna, està dues vegades en el genoma de Myotis davidii.
