Familia Gluthatione peroxidase (GPx)
Les Glutathione peroxidases (GPx) són una família d'isoenzims. En humans s'han trobat vuit GPx, i són aquestes vuit, o els seus homòlegs en Myotis lucifugus (si estàven ben anotats), les selenoproteïnes que hem utilitzat per identificar GPx en Myotis davidii. L'elevat grau d'homologia entre totes elles feia que els mateixos scaffolds donessin hits per totes les GPx. Hem usat la puntuació del T-coffee i la predicció del Seblastian per a discernir a quina de les GPx correspon cada scaffold.
Per a una navegació més còmode, podeu accedir a les diferents proteïnes d'aquesta família per mitjà dels següents enllaços: GPx1, GPx2, GPx3, GPx4, GPx5, GPx6, GPx7 i GPx8.
GPx1
Per la proteïna GPx1 vam usar una query d'Homo sapiens, ja que a Myotis lucifugus l'anotació era incorrecta (no començava per metionina).
En fer el blast, es van trobar múltiples hits. L'scaffold KB112367.1 era el que tenia l'e-value més baix.
L'exonerate i el genewise prediuen una proteïna no intrònica. L'exonerate té una raw score de 581, mentre que el genewise té un score de 273.53 bits.
Cap de les dues prediccions cobreixen el codó UGA, pel que no sabem si la selenocisteïna està conservada. Això pot ser degut a un problema en l'ensamblatge del nostre genoma.
Pel mateix motiu, l'anàlisi mitjançant Seblastian no troba cap resultat. El que si que podem predir és un element SECIS a 3' mitjançant SECISearch 3.
El t-coffee ens mostra un alineament molt bo; fet que, juntament amb els elements SECIS, ens permeten dir que la proteïna està ben conservada. D'altra banda, com que la selenocisteïna no està coberta, no podem dir si aquesta està conservada o no, i per tant si la GPx1 és funcional en Myotis davidii.
GPx2
Per la proteïna GPx2 vam usar una query de Myotis lucifugus, ja que estava correctament anotada a SelenoDB.
En fer el blast, vam tornar a trobar múltiples hits. L'scaffold KB104801.1 va ser el que tenia un e-value més petit, i un t-coffee amb la puntuació més alta, per això vam decidir que els hits en aquest scaffold corresponien a GPx2.
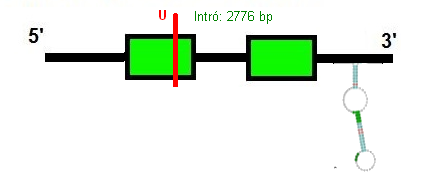
L'exonerate i el genewise prediuen tots dos una sola proteïna, amb una selenocisteïna a la posició 40 (igual que la GPx de Myotis lucifugus). Tots dos prediuen una proteïna de dos exons i un intró. L'exonerate té una raw score de 998, mentre que el genewise té una score de 437.21 bits.
Com que les proteïnes predites per ambdòs mètodes presenten un codó stop a la mateixa posició que la query, deduïm que la selenoproteïna està conservada. El fet que aquest scaffold sigui el que té un t-coffee amb puntuació més alta en comparar-lo amb GPx2 de Myotis lucifugus, ens ho confirma.
D'altra banda, el SECISearch 3 ens prediu un element SECIS a la posició 3', fet que respatlla la nostra teoria de que GPx2 està ben conservat en Myotis davidii. El Seblastian acaba de confirmar-ho, en donar-nos homologia del nostre scaffold amb la GPx2 de Bos taurus. Donat que a Myotis lucifugus també trobem GPx2, amb selenocisteïna, podem dir que es tracta d'una selenoproteïna present en l'ancestre comú d'aquests.
Amb tota la informació recopilada, podem predir com serà l'estructura de la nostra selenoproteïna:
GPx3
Per l'anàlisi de GPx3 vam usar com a query la GPx3 anotada a selenoDB per Homo sapiens, ja que, tot i que GPx3 també està anotada en Myotis lucifugus, l'anotació és incorrecta (no comença per metionina).
En fer el blast, i al comparar l'e-value i el tcoffee amb la resta d'scaffolds de GPx3, vam decidir que l'scaffold KB109247.1 corresponia era el més apropiat.
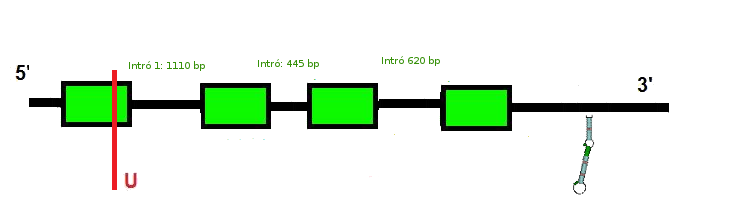
Tant l'exonerate com el genewise prediuen una proteïna amb un codó UGA a la posició 73. Ambdòs mètodes prediuen una proteïna de quatre exons i tres introns. L'exonerate té una raw score de 916, i l'score del genewise és de 381.15 bits.
L'inici de la proteïna no es troba en el nostre scaffold, probablement degut a algún problema en l'ensamblatge del genoma d'estudi.
Amb tota aquesta informació, i el tcoffee d'alta puntuació, que indica alta homologia, podem afirmar que la selenoproteïna està ben conservada.
Aquesta hipòtesi es confirma en realitzar l'anàlisi amb SECISearch3, que troba un element SECIS a 3' de la nostra proteïna. L'anàlisi mitjançant Seblastian acaba de confirmar-ho, donant homologia del nostre scaffold amb la precursora de GPx3 en Cricetulus griseus. Donat que en Myotis lucifugus també trobem GPx3, amb selenocisteïna, podem dir que es tracta d'una selenoproteïna que ja estava present en l'ancestre comú de les dues espècies de Myotis i d'humans.
Amb la informació obtinguda de l'anàlisi d'aquest scaffold podem predir com serà la nostra selenoproteïna:
GPx4
Per l'anàlisi de GPx4 vam usar com a query la GPx4 anotada a selenoDB per Homo sapiens, ja que, tot i que GPx4 també està anotada en Myotis lucifugus, l'anotació és incorrecta (no comença per metionina).
De tots els hits trobats amb el blast, mitjançant la comparació dels scaffolds amb la resta de GPx, vam decidir que GPx4 es troba a l'scaffold KB108036.1.
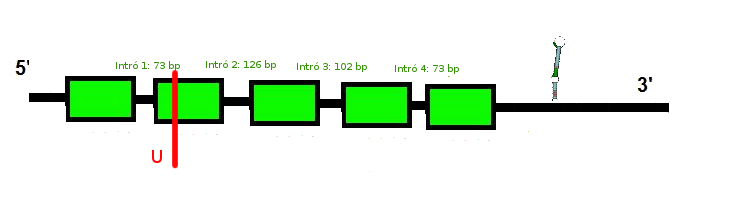
Tant l'exonerate com el genewise prediuen una proteïna amb cinc introns i sis exons. En les dues prediccions trobem un codó stop a la posició 73. El genewise té una score de 241.82 bits, mentre que el raw score de l'exonerate es de 713.
L'inici de la proteïna no es troba en el nostre scaffold, probablement degut a algun problema en l'ensamblatge del genoma d'estudi.
Com que tots dos mètodes prediuen la selenocisteïna a la mateixa posició que la query, i el tcoffee mostra una alta puntuació (alta homologia), podem afirmar que la selenoproteïna està ben conservada.
L'anàlisi amb SECISearch3 ens prediu un element SECIS a l'extrem 3' de la nostra proteïna. El Seblastian, d'altra banda troba homologies entre el nostre scaffold i la GPx4 de Capra hircus. Tot això ens confirma la hipòtesi de que GPx4 es troba conservada en Myotis davidii. Donat que també es troba conservat en Myotis lucifugus, podem dir que està present en l'ancestre comú de les dues espècies de Myotis i d'humans.
Basats en la predicció d'exonerate i genewise, podem hipotetitzar l'estructura de la nostra selenoproteïna:
GPx5
Per analitzar GPx5 hem usat la query de Myotis lucifugus. Aquesta està ben anotada, però no té codó UGA (no té selenocisteïna). Tot i amb això, la query humana tampoc conté selenocisteïna, pel que podem usar la de Myotis lucifugus.
De tots els hits trobats en el blast, comparant amb la resta d'scaffolds, el de KB101385.1 era el d'e-value més petit.
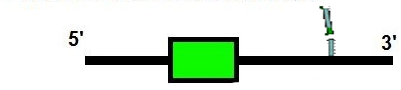
L'exonerate prediu dos segments proteics, amb un gap entre els aminoàcids 142 i 159. La raw score es de 157 pel primer segment i 135 pel segon. El genewise prediu una sola proteïna amb una score de 127.02 bits.
En fer l'anàlisi amb el t-coffee, trobem que l'inici de la proteïna no es troba en el nostre scaffold. A més, tenim el gap de l'exonerate de 17 bp. Tot i que trobem diferències entre les seqüències, la puntuació del tcoffee és de 98.
Per últim, l'anàlisi amb SECISearch ens prediu elements SECIS a l'extrem 3', que respatlla la teoria que aquesta proteïna es troba conservada. L'anàlisi amb Seblastian no prediu cap proteïna, ja que GPx5 no conté selenocisteïnes.
Així doncs, podem dir que la proteïna es troba conservada, tot i que no sabem si els canvis que presenta fan que sigui funcional o no. La proteïna predita pels nostres programes seria aquesta:
GPx6
Pel que fa a GPx6, la query que vam usar era d'Homo sapiens, ja que no hi havia GPx6 en Myotis lucifugus. Tots els hits que vam trobar, però, corresponien a altres proteïnes de la mateixa familia. Així doncs, hipotetitzem que no existeix GPx6 en el genoma de Myotis davidii. El fet que aquesta tampoc hi sigui en l'animal del mateix gènere ja anotat, el Myotis lucifugus fa sospitar que la pèrdua de GPx6 té lloc en un ancestre comú d'aquests.
GPx7
Per analitzar GPx7 hem usat la query d'Homo sapiens, ja que la de Myotis lucifugus no està ben anotada. Aquesta GPx no conté codó UGA (no té selenocisteïna).
De tots els hits trobats en el blast, comparant amb la resta de GPx, el de KB110890.1 era el d'e-value més petit, i en fer l'anàlisi amb el t-coffee, el que té més homologia. Aquest scaffold cobreix també DI1, tot i que en zones diferents, pel que no es superposen els exons.
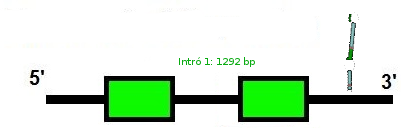
Tant l'exonerate com el genewise prediuen una proteïna amb dos exons i un sol intró. El genewise té una score de 328.60 bits, mentre que el raw score de l'exonerate es de 718.
L'inici de la proteïna no es troba en el nostre scaffold, probablement degut a algún problema en l'ensamblatge del genoma d'estudi.
L'anàlisi amb el t-coffee ens mostra alta homologia entre el nostre scaffold i GPx7, que indica que es troba ben conservada en Myotis davidii.
D'altra banda, el SECISearch3 ens prediu elements SECIS a 3', que respatlla la teoria que aquesta proteïna es troba conservada. L'anàlisi amb Seblastian no prediu cap proteïna, ja que GPx7 no conté selenocisteïnes.
Amb l'informació obtinguda de l'anàlisi d'aquest scaffold podem predir com serà GPx7:
GPx8
Per l'anàlisi de GPx8 hem usat la query d'Homo sapiens, ja que en Myotis lucifugus no està anotada. Aquesta GPx no conté codó UGA (no té selenocisteïna).
El hit amb menor e-value del blast, que no corresponia a cap altre GPx, era de l'scaffold KB105949.1.
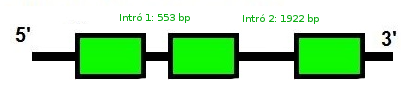
L'exonerate prediu una proteïna amb tres exons i 2 introns, amb una raw score de 658. El genewise, en canvi, prediu una proteïna amb dos exons i un intró de 17113 bp. L'score del genewise és de 277,07 bits.
El fet que el segon fragment, predit pel genewise estigui tan separat, fa sospitar d'una duplicació parcial de GPx8, amb un fragment que ha quedat com a pseudogen.
La nostra predicció no cobreix l'inici de la proteïna, probablement degut a algún problema en l'ensamblatge del genoma d'estudi.
L'anàlisi amb el t-coffee ens mostra alta homologia entre el nostre scaffold i GPx8, que indica que es troba ben conservada en Myotis davidii.
D'altra banda, el SECISearch3 no prediu elements SECIS. L'anàlisi amb Seblastian no prediu cap proteïna, ja que GPx8 no conté selenocisteïnes. Tot i això, no podem descartar que GPx8 estigui conservada.
Basats en l'estructura predita per exonerate, podem hipotetitzar l'estructura de GPx8:
Com s'ha pogut observar, encara que totes siguin proteïnes de la Gluthatione peroxidase, cadascuna d'elles és diferent de la resta, ja sigui tant perquè poden contenir o no selenocisteïnes, pels elements SECIS predits o si estan conservades en una altra espècie del mateix gènere (Myotis lucifugus).
