En aquest cas, vam seleccionar aquest BLAST perquè tenia un e-value bo i se'ns alineava la cisteïna de la query. Al fer l'anotació, el resultat del exonerate ens alineava una porció molt més petita que la que aconseguiem alinear amb el BLAST. Per això la primera proteïna que vam obtenir no era completa i el primer alineament era d'un tall molt petit de la proteïna query.
A continuació vam intentar utilitzar com a query una proteïna híbrida amb la part alineada en el BLAST i la part que no se'ns havia alineat, però vam obtenir el mateix resultat.
Finalment, vam agafar la seqüència de la proteïna alineada en el BLAST, i utilitzant el programa de traducció de la web d'EXPASY, vem mirar a que corresponien els codons que hi havien just on s'acabava aquesta proteïna. Aquí si que vam trobar un codó que podia correspondre a una selenocisteïna just allà on esperàvem que hi estigués. Per tant, ara vam fer servir la proteïna query junt amb la selenocisteïna i l'arginina que havíem trobat a la web just després de la proteïna que trobavem al BLAST i vam tornar-lo a fer obtenint el següent BLAST. Així vam poder confirmar que la selenocisteïna s'alineava bé.
Aleshores, per confirmar que la proteïna hi era, vem utilitzar com a query la proteïna que se'ns alineava en el BLAST, és a dir, que ja era la SelK de T.cruzi. Al continuar amb el procés vam obtenir un exonerate perfecte, a partir del qual obtenírem el cDNA complet de la SelK de T.cruzi el qual vam traduïr a proteïna.
Al fer l'alineament amb la nostra query ja podem concloure que T.cruzi té la SelK.
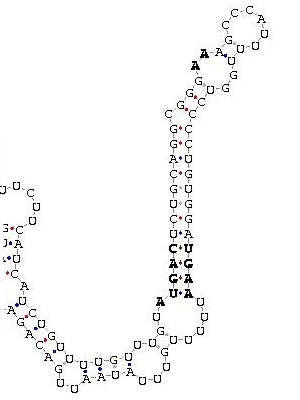
Element SECIS
Mitjançant el procés explicat a l'apartat de materials i mètodes vam trobar un element SECIS per aquesta proteïna amb un cove score de 13.80.
Torna a dalt
