
Estas selenoproteínas activan o inactivan hormonas tiroideas a través de la deodinación reductora. Las deiodinasas de yodotironinas se expresan en todos los tejidos. La tetrayodotironina (T4) se activa al eliminar un yodo para convertirse en T3. Además otras reacciones llevan a la reversión de T3 en T2, que es una forma inactiva. Es una proteína altamente expresada en la tiroides y puede contribuir de manera significativa a un aumento de la producción de la T3 en pacientes con la enfermedad de Graves y adenomas tiroideos. La proteína codificada por este gen es una oxidoredcutasa sensible al propiltiouracil que requiere tiol. Activa la hormona tiroidea convirtiendo la prohorma tiroxina (T4) a la forma bioactiva 3,3',5-triiodotironina (T3) mediante la deiodinación del anillo exterior. Asimismo, degrada las dos hormonas mediante la deiodinacinó del anillo interior. El splicing alternativo da lugar a múltiples tráncritos que codifican por diferentes isoformas. Las tres isoformas de DI contienen como mínimo una selenocisteína.
DI1
La proteína DI1 de Anolis carolinensis tiene 114 amino$aacute;cidos en su secuencia. Hemos predicho 3 genes de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestras 3 predicciones, la selenocisteína se ha alineado en todos ellas. El primer gen predicho se ha encontrado en el scaffold KN631148.1 entre las posiciones 10201-10545, con sólo un exón de 344 nucleótidos, que codifican para 114 aminoácidos. La segunda predicción se encuentra en el scaffold KN631311.1 entre las posiciones 23511-24524, con 2 exones que en total tienen 340 nucleótidos, los cuales codifican para 113 aminoácidos. El último gen predicho se sitúa en el scaffold KN637869.1 entre les posiciones 26908-27264, con un exón de 356 nucleótidos, que codifican para 119 aminoácidos. Como nuestros resultados sugieren, el genoma de Vipera berus contiene 3 parálogos del gen DI1.
Para la predicción de elementos SECIs, hemos obtenido 4 hits por el primer gen predicho. Tres de ellos se situaban en el frame correcto del scaffold y en la región 3' del gen. De éstos, sólo uno se encontraba a una distancia razonable del gen, localizado en las posiciones 11118-11196. En relación a la segunda predicción, hemos obtenido 5 hits, 2 que se encuentran en el frame positivo y downstream del gen, por lo que, hemos escogido el más cercano, localizado en las posiciones 22198 y 22266. Para la tercera predicción, hemos obtenido 4 hits, de los cuáles 2 estaban en la región 3' y en el frame correcto, por lo que hemos escogido el más cercano a nuestro gen (localización: 28700-28779).
.png)
.png)
.png)
DI2
La proteína DI2 de Anolis carolinensis tiene 226 aminoácidos en su secuencia. Hemos predicho 2 genes de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestras 2 predicciones, la selenocisteína se ha alineado en todos ellas. El primer gen predicho se ha encontrado en el scaffold KN637869.1 entre las posiciones 10735-27339, con sólo 2 exones de 793 nucleótidos, que codifican para 264 aminoácidos. La segunda predicción se encuentra en el scaffold KN631148.1 entre las posiciones 10177-10602, con 1 exón que en total tienen 425 nucleótidos, que codifican por 142 aminoácidos. De hecho, esta última predicción coincide y se solapa con la primera predicción del gen DI1, por lo tanto, la descartamos. En consecuencia, concluímos que el genoma de Vipera berus contiene 1 gen DI2, que es nuestra primera predicción.
Hemos obtenido 4 hits en la predicción de elementos SECIs, dos de los cuáles están en el frame correcto y en la región 3' del gen. De estos dos, sólo uno se encontraba a una distancia razonable del gen, por lo que ha sido nuestra elección (localització: 28700-28779).

Las glutatión peroxidasas (GPx) son una amplia familia de selenoproteínas conocidas. GPx es en general el nombre de una familia de enzimas con actividad peroxidasa, el rol biológico de las cuales es proteger contra el estrés oxidativo, mediante la reducción de los hidroperóxidos de los lípidos a los correspondientes alcoholes. Esta familia está formada por 8 homólogos (GPx1-GPx8).
GPx
La proteína GPx de Anolis carolinensis tiene 193 aminoácidos en la su secuencia. Hemos predicho 3 genes de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestras 3 predicciones, la selenocisteína se ha alineado sólo en la primera, aunque en las otras dos se alineó con una cisteína. El primer gen predicho se ha encontrado en el scaffold KN618059.1 entre las posiciones 62217-64867, con sólo 2 exones de 577 nucleótidos, que codifican por 192 aminoácidos. La segunda predicción se encuentra en el scaffold KN638969.1 entre las posiciones 43692-43964, con 1 exón que en total tienen 272 nucleótidos, que codifican para 91 aminoácidos. La tercera predicción se encuentra en el scaffold KN632563.1 entre las posiciones 92280-101503, con 2 exones que en total tienen 214 nucleótidos, que codifican para 71 aminoácidos. En consecuencia, descartamos les últimas dos predicciones, puesto que la longitud del gen es demasiado pequeña y sugerimos que el genoma de Vipera berus contiene 1 gen GPx.
Anolis carolinensis también presenta una GPx homóloga que contiene cisteína con 92 aminoácidos y, nuestra predicción en el genoma de Vipera berus se localiza en el scaffold KN632563.1 entre las posiciones 92175-92441, con 1 exón que en total tiene 266 nucleótidos, que codifican para 89 aminoácidos y la cisteína está alineada. Además, coincide con el primer exón de la tercera predicción de la GPx descrita en el parágrafo anterior.
Para la predicción del gen de la selenoproteína GPx, hemos obtenido 10 hits en la predicción SECIs, 3 de los cuales se encontraban en el frame correcto y en la región 3' del gen. De estos 3, sólo uno se encontraba a una distancia razonable del gen (localización: 61677-61745). Para la predicción del gen de la Gpx homóloga en cisteína, hemos obtenido 12 elementos SECIs, pero sólo uno cumplía nuestros criterios (localización: 90371-90444).


Gpx2
La proteína GPx2 de Anolis carolinensis té 189 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína se ha alineado. El gen predicho se ha encontrado en el scaffold KN633694.1 entre les posiciones 46754-52287, con 2 exones de 565 nucleótidos, que codifican para 188 aminoácidos. Nuestros resultados sugieren que el genoma de Vipera berus contiene 1 gen GPx2.
Hemos obtenido 6 hits en la predicción SECIs, 3 de los cuales se encontraban en el frame correcto y en la región 3' del gen. De estos 3, sólo uno se encontraba a una distancia razonable del gen (localización: 46492-46556)

GPx3
La proteína GPx3 de Anolis carolinensis tiene 191 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína se ha alineado. El gen predicho se ha encontrado en el scaffold KN638526.1 entre las posiciones 16865-20932, con 4 exones de 563 nucleótidos, que codifican para 188 aminoácidos. Nuestros resultados sugieren que el genoma de Vipera berus contiene 1 gen GPx3.
Hemos obtenido 2 hits en la predicción SECIs, sin embargo ninguno de ellos cumplía nuestros criterios, porque uno se encontraba en el otro frame y el otro no se situaba en la región 3' del gen.

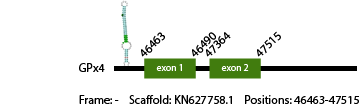
GPx4
La proteína GPx4 de Anolis carolinensis tiene; 64 aminoácidos en su secuencia y es una homóloga en cisteína. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus, sin la cisteína alineada. El gen predicho se ha encontrado en el scaffold KN627758.1 entre las posiciones 46463-47515, con 2 exones de 178 nucleótidos, que codifican para 59 aminoácidos. Nuestros resultados que el genoma de Vipera berus contiene1 gen GPx4.
Hemos obtenido 3 hits en la predicciones SECIs, de estas 3, sólo una se encontraba en el frame correcto y en la región 3' del gen, aunque se encuentra demasiado lejos del gen (localización: 42858-42932).
GPx8
Es una proteína de superficie.
La proteína GPx8 de Anolis carolinensis tiene 132 aminoácidos en la su secuencia y es una homóloga en cisteína. Hemos predicho 2 genes de este proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y las 2 predicciones, sólo en la primera la cisteína está alineada con la selenocisteína de la GPx8. El primer gen predicho se ha encontrado en el scaffold KN638969.1 entre las posiciones 43704-43967, con 1 exón de 263 nucleótidos, que codifican para 88 aminoácidos. El segundo gen predicho se ha encontrado en el scaffold KN635910.1 entre las posiciones 65237-65368, amb 1 exón de 131 nucleótidos, que codifican para 44 aminoácidos. Descartamos la última predicción y conlcuímos que el genoma de Vipera berus contiene 1 gen GPx8.
No se ha predicho ningú elemento SECIs en este gen.

Esta selenoproteína es una glycoproteína extracelular de unión a heparina asociada a las células endoteliales y con funciones antioxidantes. Hay diferentes variantes de splicing que codifican para diferentes isoformas.
SelP1
La selenoproteína SelP1 de Anolis carolinensis tiene 169 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína estaba alineada. El gen predicho se ha encontrado en el scaffold KN620947.1 entre las posiciones 816892-818519, con 3 exones de 498 nucleótidos, que codifican para 166 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SelP1.
Hemos obtenido 4 hits en la predicción de SECIs, 2 de los cuáles se encontraban en el frame correcto y en la región 3' del gen. De éstos, sólo uno se encontraba a una distancia razonable del gen (localización: 812208-812290).

SelP2
La selenoproteína SelP2 de Anolis carolinensis tiene 177 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína deAnolis carolinensis y nuestras predicciones, la selenocisteína está alineada. El gen predicho se ha encontrado en elscaffold KN623297.1 entre las posiciones 96916-99220, con 3 exones de 519 nucleótidos, que codifican para 173 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene1 gen SelP2.
Hemos obtenido 5 hits en la predicción de SECIs, 2 de los cuales se encontraban en el frame correcto y en la región 3' del gen. De estos, sólo uno se encontraba a una distancia razonable del gen (localización: 101813-101880).

Las reductasas de tioredoxina están involucradas en una serie de reacciones que llevan a la formación de enlaces disulfito reducidos. Las TRs reducen la tioredoxina tomando sus electrones y donándolos a una proteína que los transforma de la forma S2 (estado oxidado) a la SH2 (estado reducido). Este proceso es un mecanismo antioxidante.
La selenoproteína TR de Anolis carolinensis tiene 559 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus pero sin la selenocisteína alineada. El gen predicho se ha encontrado en el scaffold KN632363.1 entre las posiciones 26882-41340, con 12 exones de 1461 nucleótidos, que codifican para 487 aminoácidos. No obstante, descartamos esta predicción, porque se solapa con la de TR3, que contiene la selenocisteína alineada y más exones predichos, por lo tanto, sugerimos que la Vipera berus no presenta el gen TR.
TR3
La selenoproteína TR3 de Anolis carolinensis tiene 609 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína está alineada. El gen predicho se ha encontrado en el scaffold KN632363.1 entre les posiciones 9900-42843, con 16 exones de 1808 nucleótidos, que codifican para 608 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene1 gen TR3.
Hemos obtenido 4 hits en la predicción de SECIs, pero ninguna cumplia nuestros criterios.

Estudios con ratones sugieren que esta selonoproteína podría tener una función redox y podría estar involucrada en el control de calidad del plegamiento de proteínas.
La selenoproteína Sel15 de Anolis carolinensis tiene 137 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína estaba alineada. El gen predicho se ha encontrado en el scaffold KN622219.1 entre las posiciones 113416-132255, con 4 exones de 407 nucleótidos, que codifican para 136 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen Sel15.
Hemos obtenido 2 hits en la predicción de SECIs, pero ninguno cumplía nuestros criterios.

Su función es desconocida, sin embargo, es cree que podría ser responsable de la mayoría defectos del Selenio ingerido en la dieta. La sobreexpresión en neuronas tiene un efecto protector contra los efectos de los rayos UV. Muchos papers exponen que la su sobreexpresión en neuronas y otras células promueve la formación de mitocondrias.
La selenoproteína SelH de Anolis carolinensis tiene 79 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensisi nuestra predicción, la selenocisteína está alineada. El gen predicho se ha encontrado en el scaffold KN632251.1 entre las posiciones 178900-182678, con 2 exones de 232 nucleótidos, que codifican para 77 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene1 gen SelH.
Hemos obtenido 10 hits en la predicción de SECIs, 2 de los cuales se encontraban en el frame correcto y en la región 3' del gen. De estos, sólo uno se encontraba a una distancia razonable del gen (localización: 177376-177451).

Su función es desconocida.
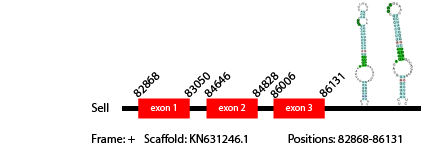
La selenoproteína SelI de Anolis carolinensis tiene 167 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína estaba alineada. El gen predicho se ha encontrado en el scaffold KN631246.1 entre las posiciones 82868-86131, con 3 exones de 489 nucleótidos, que codifican para 163 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SelI.
Hemos obtenido 5 hits en la predicción de SECIs, 2 de los cuales se encontraban en el frame correcto y en la región 3' del gen y a una distancia razonable del gen, por lo tanto hemos considerado los dos (localizaciones: 87910-87977, 86174-86255).
Esta selenoproteína se localiza en el retículo endoplasmático y es altamente expresada en el corazón, donde funciona como antioxidante.
La selenoproteína SelK de Anolis carolinensis tiene 93 aminoácidos en su secuencia. No hemos predicho ningún gen para a SelK en el genoma de Vipera berus.
Se expresa en muchos tejidos y su localización es perinuclear.
La selenoproteína SelM de Anolis carolinensis tiene 94 aminoácidos en su secuencia. No hemos predicho ningún gen para la SelM en el genoma de Vipera berus.
Se expresa en la membrana del reticulo endoplasmátic en varios tejidos. Mutaciones en este gen causen el fenotipo de la enfermedad multinicore y de la distrofia muscular congénita.
La selenoproteína SelN de Anolis carolinensis tiene 492 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína estaba alineada. El gen predicho se ha encontrado en el scaffold KN614981.1 entre las posiciones 26068-33905, con 7 exones de 878 nucleótidos, que codifican para 293 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SelN, aunque la predicción es demasiado corta.
Hemos obtenido 5 hits en la predicción SECIs, pero ninguno cumplía nuestros criteris.

Esta familia es la methionine sulfoxide reductase (Msr) que incluye enzimas de reparación que reducen residuos de metionina oxidados.
SelR1
La selenoproteína SelR1 de Anolis carolinensis tiene 94 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína estaba alineada. El gen predicho se ha encontrado en el scaffold KN635457.1 entre las posiciones 48530-49734, con 2 exones de 262 nucleótidos, que codifican para 87 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SelR1.
Hemos obtenido 4 hits en la predicción de SECIs, pero sólo 1 estaba en el frame correcto, en la región 3' del gen y se encontraba a una distancia razonable del gen (localitzacio: 50989-51066).

SelR2
Su función es desconocida.
La proteína SelR2 de Anolis carolinensis tiene 140 aminoácidos en su secuencia y es una homóloga en cisteína. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la cisteína está alineada con la selenocisteína de la proteína SelR1. El gen predicho se ha encontrado en el scaffold KN633105.1 entre las posiciones 32289-32474, con 1 exon de 185 nucleótidos, que codifican para 62 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene1 gen SelR2, aunque nuestro hit es demasiado corto.
Hemos obtenido 6 hits en la predicción SECIs, pero ninguno cumplía nuestros criterios.

SelR3
La proteína SelR3 de Anolis carolinensis tiene 184 aminoácidos en su secuencia y es una homóloga en cisteína. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la cisteína estaba alineada con la selenocisteína de la proteína SelR1. El gen predicho se ha encontrado en el scaffold KN628141.1 entre las posiciones 8521-67112, con 6 exones de 546 nucleótidos, que codifican para 182 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SelR3.
Hemos obtenido 2 hits en la predicción SECIs, pero ninguno cumplía nuestros criterios.

SelS tiene n papel importante en el control de la respuesta inflamatoria y está asociada con la enfermedad cardiovascular y diabetes[16].
La selenoproteína SelS de Anolis carolinensis tiene 164 aminoácidos en su secuencia. No hemos predicho ningún gen para la SelS en el genoma de Vipera berus.
Juntamente con SelW, SelH y Rdx12, la Sel T se ha propuesto como un nuevo miembro para la familia thioredoxin-like[17].
La selenoproteína SelT de Anolis carolinensis tiene 153 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína está alineada. El gen predicho se ha encontrado en el scaffold KN635297.1 entre las posiciones 59408-61861, con 4 exones de 455 nucleótidos, que codifican para 152 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SelT.
Hemos obtenido 5 hits en la predicción de SECIs, 3 de los cuales se encontraban en el frame correcto y en la región 3' del gen. De éstos, sólo uno se encontraba a una distancia razonable del gen (localización: 57993-58067).

SelU1
Su función es desconocida, se expresa, sin embargo, en hueso, cerebro, hígado y riñón.
La selenoproteína SelU1 de Anolis carolinensis tiene 233 aminoácidos en la su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. Además, al hacer el t-coffee entre la proteína de Anolis carolinensis y nuestra predicción, la selenocisteína está alineada. El gen predicho se ha encontrado en el scaffold KN635350.1 entre las posiciones 170571-180081, con 4 exones de 552 nucleótidos, que codifican para 184 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene1 gen SelU1.
Hemos obtenido 5 hits en la predicción SECIs, pero ninguno cumplía nuestros criterios.

SelU2
La función de la SelU2 es desconocida.
La proteína SelU2 de Anolis carolinensis tiene 225 aminoácidos en su secuencia y es una homóloga en cisteína. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus pero no contiene la cisteína alineada con la selenocisteína de la SelU1. El gen predicho se ha encontrado en el scaffold KN632497.1 entre las posiciones 8609-10556, con 2 exones de 256 nucleótidos, que codifican para 85 aminoácidos. Este resultado sugiere que el genoma de Vipera berus no contiene 1 gen SelU2.
Este gen codifica para una enzima que sintetiza selenofosfato a partir de selenio y ATP.
La selenoproteína SPS de Anolis carolinensis tiene 432 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus que contiene la selenocisteína alineada. El gen predicho se ha encontrado en el scaffold KN626389.1 entre las posiciones 80733-90051, con 7 exones de 1169 nucleótidos, que codifican para 390 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 1 gen SPS.

La proteína SPS1 es una proteína de la maquinaria relacionada con la síntesis de selenoproteínas. En Anolis carolinensis tiene 392 aminoácidos en su secuencia. Hemos predicho 2 genes de esta proteína en el genoma de Vipera berus. El primer gen predicho se ha encontrado en el scaffold KN636817.1 entre las posiciones 39126-51377, con 7 exones de 977 nucleótidos, que codifican para 326 aminoácidos en su secuencia. El segundo gen predicho se ha encontrado en el scaffold KN626389.1 entre las posiciones 80772-88750, y como se puede observar, corresponde con la predicción de la selenoproteína SPS. Este resultado sugiere que el genoma de Vipera berus contiene 1 copia del gen SPS1.
Sólo hemos encontrado un elemento SECIs que cumplía nuestros criteris (localización: 79442-79516). Para la proteína de la maquinaria, no se han predicho elementos SECIs, ya que no es una selenoproteína ni una homóloga.

Sel0 (1)
Su función es desconocida.
La proteína Sel0 (1) de Anolis carolinensis tiene 262 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El gen predicho se ha encontrado en el scaffold KN616814.1 entre las posiciones 126855-127061, con 1 exón de 206 nucleótidos, que codifican para 69 aminoácidos. En su secuencia. Este resultado sugiere que el genoma de Vipera berus no contiene el gen Sel0 (1), dado que la predicción es demasiado corta.
Sel0 (2)
Su función es desconocida.
La proteína Sel0 (2) de Anolis carolinensis tiene 66 aminoácidos en su secuencia y es una homóloga en cisteína. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El gen predicho se ha encontrado en el scaffold KN624046.1 entre las posiciones 121870-139588, con 9 exones de 256 nucleótidos, que codifican para 85 aminoácidos en su secuencia. Este resultado sugiere que el genoma de Vipera berus contiene el gen Sel0(2).
.png)
La Methionine Sulfide Reductase A es una enzima que realiza la reducción de la sulfoxid metionina a metionina. En estudios con humanos y animales, se han demostrado niveles elevados en riñón y tejido nervioso. Se ha propuesto que su función consiste en reparar daño oxidativo. Hay 3 tránscritos que codifican para diferentes isoformas de esta proteína.
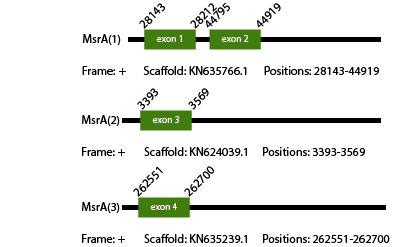
La proteína MsrA de Anolis carolinensis tiene 209 aminoácidos en su secuencia y es un homólogo que contiene cisteína. Teníamos 5 predicciones de nuestro gen que, por separado, eran demasiado pequeñas para formar un gen completo. Sin embargo, observando nuestros resultados del t-coffee para cada predicción quedaba claro que los 3 primeros hits son parte de la misma proteína, ya que mirando los alineamientos, donde terminaba una empezaba la otra. Por lo tanto, nuestro gen final está separado en 3 contigs, siguiendo el orden siguiente: KN635766.1 (posiciones: 28143-44919), KN624039.1 (posiciones: 3393-3569) i KN635239.1 (posiciones: 262551-262700). El gen tiene 4 exones, 518 nucleótidos y 173 aminoácidos. En consecuencia, concluímos que Vipera berus contiene un gen MsrA en su genoma
La SelW está altamente expresada en músculo esquelético y cardíaco. Podría estar relacionada en procesos antioxidantes, como la mayoría de selenoproteínas. Su función específica es desconocida.
La selenoproteína SelW de Anolis carolinensis tiene 48 aminoácidos en la su secuencia. No hemos predicho ningún gen per la SelW en el genoma de Vipera berus.
El Eukaryotic elongation factor tiene funciones de glutatión peroxidasa en la detoxificación del peróxido dehidrogen y es uno de los enzimas antioxidantes más importantes en humanos.
La proteína eEFsec es una proteína de la maquinaria relacionada con la síntesis de selenoproteínas. En el genoma de Anolis carolinensis hay 3 copias. La primera, eEFsec(1), tiene 215 aminoácidos en la su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El primer gen predicho se ha encontrado en el scaffold KN617171.1 entre las posiciones 36097-36741, con 1 exón de 644 nucleótidos, que codifican para 215 aminoácidos. La seguna, eEFsec(2) tiene 79 aminoácidos. No hemos predicho ningún gen per esta copia. La tercera, eEFsec(3) tiene 168 aminoácidos. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El primer gen predicho se ha encontrado en el scaffold KN617171.1 entre las posiciones 2901-10232, con 3 exones de 492 nucleótidos, que codifican per 164 aminoácidos. Este resultado sugiere que el genoma de Vipera berus contiene 2 copias del gen eEFsec.
.png)
.png)
SBP2 codifica para una proteína nuclear que funciona como una proteína de unión al SECIs. Mutaciones en este gen están asociadas con la reducción de la actividad de la deiodinasa tiroxina, una selenoproteína y con la alteración del metabolismo de la hormona tiroidea.
El genoma de Anolis carolinensis tiene 2 parálogos del gen SBP2. El primero, SBP2(1), tiene 1055 aminoacidos. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. La predicción se encuentra en el scaffold KN618877.1 entre las posiciones 581837-599039, con 15 exones que en total tienen 2997 nucleótidos, que codifican para 999 aminoácidos. El segundo, SBP2(2), tiene 854 aminocidos. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. La predicción se encuentra en el scaffold KN632477.1 entre las posiciones 82268-108573, con 14 exones que en total tienen 2170 nucleótidos, que codifican para 723 aminoácidos. Por lo tanto, hemos predicho las dos copias del gen SBP2. No se han predicho elementos SECIs, ya que no se trata de una selenoproteína ni de una homóloga.
.png)
.png)
La SecS cataliza el tercer paso de la síntesis del tRNa de la selenocisteína: la conversión de O-phosphoseryl-tRNA(Sec) a selenocysteinyl-tRNA(Sec).
La proteína Secs es una proteína de la maquinaria relacionada con la síntesis de selenoproteínas. En Anolis carolinensis tiene 466 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El gen predicho se ha encontrado en el scaffold KN615039.1 entre las posiciones 4537-76572, con 10 exones de 1299 nucleótidos, que codifican para 433 aminoácidos en su secuencia. Este resultado sugiere que el genoma de Vipera berus contiene1 gen SecS.

La quinasa Phosphoseryl-tRNA se encuentra muy conservada a lo largo de la evolución, lo que sugiere que tiene un rol importante en la biosíntesis de la selenoproteína o en su regulación.
La proteína PSTK es una proteína de la maquinaria relacionada con la síntesis de selenoproteínas. En Anolis carolinensis tiene 352 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El gen predicho se ha encontrado en el scaffold KN633361.1 entre las posiciones 47138-54632, con 6 exones de 1047 nucleótidos, que codifican para 349 aminoácidos en su secuencia. Este resultado sugiere que el genoma de Vipera berus contiene1 gen PSTK. No hemos predicho elemento SECIs, dado que, no es una selenoproteína ni una homóloga.

Esta proteína regula la expresión de selenoproteínas.
La proteína SECp43 es una proteína de la maquinaria relacionada con la síntesis de selenoproteínas. En Anolis carolinensis tiene 268 aminoácidos en su secuencia. Hemos predicho 1 gen de esta proteína en el genoma de Vipera berus. El gen predicho se ha encontrado en el scaffold KN636726.1 entre las posiciones 201915-204816, con 6 exones de 621 nucleótidos, que codifican para 207 aminoácidos en su secuencia. Este resultado sugiere que el genoma de Vipera berus contiene1 gen SECp43. No hemos predicho elemento SECIs, puesto que, no es una selenoproteína ni una homóloga.
