
Aquestes selenoproteïnes activen o inactiven hormones tiroidees mitjançant la deodinació reductora. Les deiodinases de iodotironina s'expressen en tots els teixits. La tetraiodotironina (T4) s'activa en eliminar un iode per convertir-se a T3. Tot i això, altres reaccions porten a la reversió de T3 a T2, la qual és una forma inactiva. És una proteïna altament expressada en la tiroides i pot contribuir de manera significativa a un augment de la producció de la T3 en pacients amb la malaltia de Graves i adenomes tiroideus. La proteïna codificada per aquest gen és una oxidoreductasa sensible a propiltiouracil que requereix tiol. Activa la hormona tiroidea convertint la prohorma tiroxina (T4) a la forma bioactiva 3,3',5-triiodotironina (T3) mitjançant la deiodinació de l'anell exterior. A més, degrada les dues hormones mitjançant la deiodinació de l'anell interior. L'splicing alternatiu dóna lloc a múltiples transcripts que codifiquen per diferents isoformes. Les tres isoformes de DI contenen com a mínim una selenocisteïna.
DI1
La proteïna DI1 d'Anolis carolinensis té 114 aminoàcids en la seva seqüència. Hem predit 3 gens d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i les nostres 3 prediccions, la selenocisteïna s'ha alineat en totes elles. El primer gen predit s'ha trobat en l'scaffold KN631148.1 entre les posicions 10201-10545, amb només un exó de 344 nucleòtids, que codifiquen per 114 aminoàcids. La segona predicció es troba en l'scaffold KN631311.1 entre les posicions 23511-24524, amb 2 exons que en total tenen 340 nucleòtids, els quals codifiquen per 113 aminoàcids. L'últim gen predit es situa en l'scaffold KN637869.1 entre les posicions 26908-27264, amb un exó de 356 nucleòtids, que codifiquen per 119 aminoàcids. Com els nostres resultats suggereixen, el genoma de Vipera berus conté 3 paràlegs del gen DI1.
Per a la predicció d'elements SECIs, hem obtingut 4 hits pel primer gen predit. Tres dels quals estaven en el correcte frame i en la regió 3'. D'aquestes, nomé una es trobava a a una distància correcta del gen per tant, l'hem escollit (localització 11118-11196). Pel que fa a la segona predicció, hem obtingut 5 hits, 2 dels quals es troben en el frame correcte i downstream per tant, hem escollit el més proper, localitzat en les posicions 22198 i 22266. Per la tercera predicció, hem obtingut 4 hits, dels quals 2 estaven en la regió 3' i en el frame correcte, per tant hem escollit el més proper al nostre gen (localització 28700-28779).
.png)
.png)
.png)
DI2
La proteïna DI2 d'Anolis carolinensis té 226 aminoàcids en la seva seqüència. Hem predit 2 gens d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i les nostres 2 prediccions, la selenocisteïna s'ha alineat en totes elles. El primer gen predit s'ha trobat en l'scaffold KN637869.1 entre les posicions 10735-27339, amb 2 exons de 793 nucleòtids, que codifiquen per 264 aminoàcids. La segona predicció es troba en l'scaffold KN631148.1 entre les posicions 10177-10602, amb 1 exó que en total té 425 nucleòtids, els quals codifiquen per 142 aminoàcids. De fet, aquesta última predicció coincideix i es solapa amb els nostre primer gen predit de DI1, per tant, el descartem. Així doncs, concloem que la Vipera berus conté 1 gen DI2, el qual és la nostra primera predicció.
Hem obtingut 4 hits en la predicció de SECIs, 2 dels quals es troben en el frame correcte i en la regió 3' del gen. D'aquests dos, només un estava a una distància correcta del gen (localització: 28700-28779).

Les glutatió peroxidasa (GPx) é una amplia família de selenoproteïnes conegudes. Gpx és en general el nom d'una família d'enzims amb activitat peroxidasa, el paper biològic dels quals és protegir contra l'estrès oxidatiu, mitjançant la reducció dels hidroperòxids dels lípds als corresponents alcohols. Aquesta família està formada per 8 homòlegs (GPx1-GPx8).
GPx
La proteïna GPx d'Anolis carolinensis té 193 aminoàcids en la seva seqüència. Hem predit 3 gens d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i les nostres 3 prediccions, la selenocisteïna s'ha alineat només en la primera, tot i que en les altres dues estava alineada amb una cisteïna. El primer gen predit s'ha trobat en l'scaffold KN618059.1 entre les posicions 62217-64867, amb només 2 exons de 577 nucleòtids, que codifiquen per 192 aminoàcids. La segona predicció es troba en l'scaffold KN638969.1 entre les posicions 43692-43964, amb 1 exó que en total té 272 nucleòtids, els quals codifiquen per 91 aminoàcids. La tercera predicció es troba en l'scaffold KN632563.1 entre les posicions 92280-101503, amb 2 exons que en total tenen 214 nucleòtids, els quals codifiquen per 71 aminoàcids. En conseqüència, descartem les últimes dues prediccions, ja que la llargada del gen és massa petita i suggerim que el genoma de Vipera berus conté 1 gen GPx.
Anolis carolinensis també presenta una GPx homòloga que conté cisteïna amb 92 aminoàcids i la nostra predicció en el genoma de Vipera berus es localitza en l'scaffold KN632563.1 entre les posicions 92175-92441, amb 1 exó que en total tenen 266 nucleòtids, que codifiquen per 89 aminoàcids i la cisteïna està alineada. A més, coincideix amb el primera exó de la tercera predicció de la GPx descrita en el paràgraf anterior.
Per la predicció del gen de la selenoproteïna GPx, hem obtingut 10 hits en la predicció SECIs, 3 dels quals es trobaven en el frame correcte i en la regió 3' del gen. D'aquests 3, només un es trobava a una distància raonable del gen (localització: 61677-61745). Per la predicció del gen de l'homòleg en cisteïna de GPx, hem obtingut 12 elements SECIs, però només un complia els nostres criteris (localització 90371-9444).


Gpx2
La proteïna GPx2 d'Anolis carolinensis té 189 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna s'ha alineat. El gen predit s'ha trobat en l'scaffold KN633694.1 entre les posicions 46754-52287, amb 2 exons de 565 nucleòtids, que codifiquen per 188 aminoàcids. Els nostres resultats suggereixen que el genoma de Vipera berus conté 1 gen GPx2.
Hem obtingut 6 hits en la predicció SECIs, 3 dels quals es trobaven en el frame correcte i en la regió 3' del gen. D'aquests 3, només un es trobava a una distància raonable del gen (localització: 46492-46556)

GPx3
La proteïna GPx3 d'Anolis carolinensis té 191 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna s'ha alineat. El gen predit s'ha trobat en l'scaffold KN638526.1 entre les posicions 16865-20932, amb 4 exons de 563 nucleòtids, que codifiquen per 188 aminoàcids. Els nostres resultats suggereixen que el genoma de Vipera berus conté 1 gen GPx3.
Hem obtingut 2 hits en la predicció SECIs, però cap d'ells complia els nostres criteris, perquè un es trobava en l'altre frame i l'altre no es situava en la regió 3' del gen.

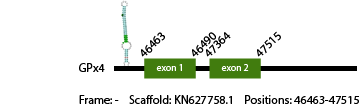
GPx4
La proteïna GPx4 d'Anolis carolinensis té 64 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus, sense la cisteïna alineada. El gen predit s'ha trobat en l'scaffold KN627758.1 entre les posicions 46463-47515, amb 2 exons de 178 nucleòtids, que codifiquen per 59 aminoàcids. Els nostres resultats suggereixen que el genoma de Vipera berus conté 1 gen GPx4.
Hem obtingut 3 hits en la predicció SECIs, D'aquests 3, només un es trobava en el frame correcte i en la regió 31 del gen, tot i que es trobi una mica massa lluny del gen (localització: 42858-42932).
GPx8
És una proteïna de superfície.
La proteïna GPx8 d'Anolis carolinensis té 132 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Hem predit 2 gens d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i les 2 prediccions, només en la primera la cisteïna està alineada amb la selenocisteïna de la GPx8. El primer gen predit s'ha trobat en l'scaffold KN638969.1 entre les posicions 43704-43967, amb 1 exó de 263 nucleòtids, que codifiquen per 88 aminoàcids. El segon gen predit s'ha trobat en l'scaffold KN635910.1 entre les posicions 65237-65368, amb 1 exó de 131 nucleòtids, que codifiquen per 44 aminoàcids. Descartem l'última predicció i concloem que el genoma de Vipera berus conté 1 gen GPx8.
No s'ha predit cap element SECIs en aquest gen.

Aquesta selenoproteïna és una glycoproteïna extracel•lular d'unió a heparina associada a les cèl•lules endotelials i amb funcions antioxidants. Hi ha diverses variants de splicing que codifiquen per diverses isoformes.
SelP1
La selenoproteïna SelP1 d'Anolis carolinensis té 169 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN620947.1 entre les posicions 816892-818519, amb 3 exons de 498 nucleòtids, que codifiquen per 166 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelP1.
Hem obtingut 4 hits en la predicció de SECIs, 2 dels quals es trobaven en el frame correcte i en la regió 3' del gen. D'aquests, només un es trobava a una distància raonable del gen (localització: 812208-812290).

SelP2
La selenoproteïna SelP2 d'Anolis carolinensis té 177 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN623297.1 entre les posicions 96916-99220, amb 3 exons de 519 nucleòtids, que codifiquen per 173 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelP2.
Hem obtingut 5 hits en la predicció de SECIs, 2 dels quals es trobaven en el frame correcte i en la regió 3' del gen. D'aquests, només un es trobava a una distància raonable del gen (localització: 101813-101880).

Les reductases de tioredoxina estan involucrades en una sèrie de reaccions que porten a la formació d'enllaços disulfit reduïts. Les TRs redueixen la tioredoxina prenent els seus electrons i donant-los a una proteïna que els transforma de la forma S2 (estat oxidat) a la SH2 (estat reduït). Aquest procés és un mecanisme antioxidant.
La selenoproteïna TR d'Anolis carolinensis té 559 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus però sense la selenocisteïna alineada. El gen predit s'ha trobat en l'scaffold KN632363.1 entre les posicions 26882-41340, amb 12 exons de 1461 nucleòtids, que codifiquen per 487 aminoàcids. Tot i axò, descartem aquesta predicció, ja que es solapa amb la de TR3, la qual conté la selenocisteïna alineada i més exons predits, per tant, suggerim que la Vipera berus no presenta el gen TR.
TR3
La selenoproteïna TR3 d'Anolis carolinensis té 609 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN632363.1 entre les posicions 9900-42843, amb 16 exons de 1808 nucleòtids, que codifiquen per 608 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen TR3.
Hem obtingut 4 hits en la predicció de SECIs, però cap complia els nostres criteris.

Estudis en ratolins suggereixen que aquesta selonoproteïna podria tenir una funció redox i podria estar involucrada en el control de qualitat del plegament de proteïnes.
La selenoproteïna Sel15 d'Anolis carolinensis té 137 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN622219.1 entre les posicions 113416-132255, amb 4 exons de 407 nucleòtids, que codifiquen per 136 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen Sel15.
Hem obtingut 2 hits en la predicció de SECIs, però cap complia els nostres criteris.

La seva funció és desconeguda, tot i això, es creu que podria ser responsable de la majoria d'efectes del Seleni ingerit en la dieta. La sobreexpressió en neurones té un efecte protector contra els efectes dels raigs UV. Molts papers reporten que la seva sobreexpressió en neurones i altres cèl•lules promou la formació de mitocòndries.
La selenoproteïna SelH d'Anolis carolinensis té 79 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN632251.1 entre les posicions 178900-182678, amb 2 exons de 232 nucleòtids, que codifiquen per 77 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelH.
Hem obtingut 10 hits en la predicció de SECIs, 2 dels quals es trobaven en el frame correcte i en la regió 3' del gen. D'aquests, només un es trobava a una distància raonable del gen (localització: 177376-177451).

La seva funció és desconeguda.
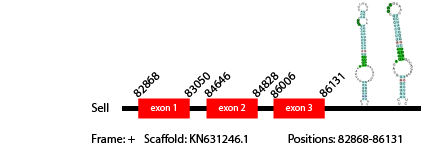
La selenoproteïna SelI d'Anolis carolinensis té 167 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN631246.1 entre les posicions 82868-86131, amb 3 exons de 489 nucleòtids, que codifiquen per 163 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelI.
Hem obtingut 5 hits en la predicció de SECIs, 2 dels quals es trobaven en el frame correcte i en la regió 3' del gen i es trobaven a una distància raonable del gen, per tant els hem considerat tots dos (localitzacions: 87910-87977, 86174-86255).
Aquesta selenoproteïna es localitza en el reticle endoplasmàtic i és altament expressada en el cor, on funciona com un antioxidant.
La selenoproteïna SelK d'Anolis carolinensis té 93 aminoàcids en la seva seqüència. No hem predit cap gen per la SelK en el genoma de Vipera berus.
S'expressa en molts teixits i la seva localització és perinuclear.
La selenoproteïna SelM d'Anolis carolinensis té 94 aminoàcids en la seva seqüència. No hem predit cap gen per la SelM en el genoma de Vipera berus.
S'expressa en la membrana del reticle endoplasmàtic en diversos teixits. Mutations en aquest gen causen el fenotip de la malaltia multinicore i la distròfia muscular congènita.
La selenoproteïna SelN d'Anolis carolinensis té 492 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN614981.1 entre les posicions 26068-33905, amb 7 exons de 878 nucleòtids, que codifiquen per 293 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelN, tot i que la predicció és massa curta.
Hem obtingut 5 hits en la predicció SECIs, però cap complia els nostres criteris.

Aquesta família és la methionine sulfoxide reductase (Msr) que inclou enzims de reparació que redueixen residus de metionina oxidats.
SelR1
La selenoproteïna SelR1 d'Anolis carolinensis té 94 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN635457.1 entre les posicions 48530-49734, amb 2 exons de 262 nucleòtids, que codifiquen per 87 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelR1.
Hem obtingut 4 hits en la predicció de SECIs, però només 1 estava en el frame correcte, en la regió 3' del gen i es trobaven a una distància raonable del gen (localització: 50989-51066).

SelR2
La seva funció és desconeguda.
La proteïna SelR2 d'Anolis carolinensis té 140 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la cisteïna està alineada amb la selenocisteïna de la proteïna SelR1. El gen predit s'ha trobat en l'scaffold KN633105.1 entre les posicions 32289-32474, amb 1 exó de 185 nucleòtids, que codifiquen per 62 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelR2, tot i que el nostre hit &eactue;s una mica massa curt.
Hem obtingut 6 hits en la predicció SECIs, però cap complia els nostres criteris.

SelR3
La proteïna SelR3 d'Anolis carolinensis té 184 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la cisteïna està alineada amb la selenocisteïna de la proteïna SelR1. El gen predit s'ha trobat en l'scaffold KN628141.1 entre les posicions 8521-67112, amb 6 exó de 546 nucleòtids, que codifiquen per 182 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelR3.
Hem obtingut 2 hits en la predicció SECIs, però cap complia els nostres criteris.

La SelS té un paper important en el control de la resposta inflamatòria i està associada a la malaltia cardiovascular i diabetis[16].
La selenoproteïna SelS d'Anolis carolinensis té 164 aminoàcids en la seva seqüència. No hem predit cap gen per la SelS en el genoma de Vipera berus .
Juntament amb la SelW, SelH i Rdx12, la Sel T s'ha proposat com un nou membre per a la família thioredoxin-like[17].
La selenoproteïna SelT d'Anolis carolinensis té 153 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN635297.1 entre les posicions 59408-61861, amb 4 exons de 455 nucleòtids, que codifiquen per 152 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelT.
Hem obtingut 5 hits en la predicció de SECIs, 3 dels quals es trobaven en el frame correcte i en la regió 3' del gen. D'aquests, només es trobava a una distància raonable del gen (localització: 57993-58067).

SelU1
La seva funció és desconeguda, tot i que s'expressa en os, cervell, fetge i ronyó.
La selenoproteïna SelU1 d'Anolis carolinensis té 233 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. A més, en fer el t-coffee entre la proteïna d'Anolis carolinensis i la nostra predicció, la selenocisteïna està alineada. El gen predit s'ha trobat en l'scaffold KN635350.1 entre les posicions 170571-180081, amb 4 exons de 552 nucleòtids, que codifiquen per 184 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SelU1.
Hem obtingut 5 hits en la predicció SECIs, però cap complia els nostres criteris.

SelU2
La funció de la SelU2 és desconeguda.
La proteïna SelU2 d'Anolis carolinensis té 225 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus però no conté la cisteïna alineada amb la selenocisteïna de la SelU1. El gen predit s'ha trobat en l'scaffold KN632497.1 entre les posicions 8609-10556, amb 2 exons de 256 nucleòtids, que codifiquen per 85 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus no conté 1 gen SelU2.
Aquesta gen codifica per un enzim que sintetitza selenofosfat a partir de seleni i ATP.
La selenoproteïna SPS d'Anolis carolinensis té 432 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus que conté la selenocisteïna alineada. El gen predit s'ha trobat en l'scaffold KN626389.1 entre les posicions 80733-90051, amb 7 exons de 1169 nucleòtids, que codifiquen per 390 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SPS.

La proteïna SPS1 és una proteïna de la maquinària relacionada amb la síntesis de selenoproteïnes. En Anolis carolinensis té 392 aminoàcids en la seva seqüència. Hem predit 2 gens d'aquesta proteïna en el genoma de Vipera berus. El primer gen predit s'ha trobat en l'scaffold KN636817.1 entre les posicions 39126-51377, amb 7 exons de 977 nucleòtids, que codifiquen per 326 aminoàcids en la seva seqüència. El segon gen predit s'ha trobat en l'scaffold KN626389.1 entre les posicions 80772-88750, i com es pot veure, correspon a la predicció de la selenoproteïna SPS. Aquest resultat suggereix que el genoma de Vipera berus conté 1 còpia del gen SPS1.
Només hem trobat un element SECIs que complia els nostres criteris (locatització: 79442-79516). Per la proteïna de la maquinària, no s'han predit element SECIs, ja que no és una selenoproteïna ni una homòloga.

Sel0(1)
La seva funció és desconeguda.
La proteïna Sel0(1) de Anolis carolinensis té 262 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El gen predit s'ha trobat en l'scaffold KN616814.1 entre les posicions 126855-127061, amb 1 exó de 206 nucleòtids, que codifiquen per 69 aminoàcids. en la seva seqüència. Aquest resultat suggereix que el genoma de Vipera berus no conté el gen Sel0(1), ja que la predicció és massa curta.
Sel0(2)
La seva funció és desconeguda.
La proteïna Sel0(2) de Anolis carolinensis té 66 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El gen predit s'ha trobat en l'scaffold KN624046.1 entre les posicions 121870-139588, amb 9 exons de 256 nucleòtids, que codifiquen per 85 aminoàcids. en la seva seqüència. Aquest resultat suggereix que el genoma de Vipera berus conté el gen Sel0(2).
.png)
La Methionine Sulfide Reductase A és un enzim que porta a terme la reducció de la sulfòxid metionina a metionina. En estudis humans i animals, s'han demostrat nivells elevats en ronyó i teixit nerviós. S'ha proposat que la seva funció consisteix en reparar dany oxidatiu. Hi ha 3 transcrits que codifiquen per diferents isoformes d'aquesta proteïna.
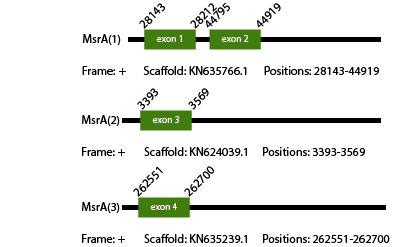
La proteïna de MsrA d'Anolis carolinensis té 209 aminoàcids en la seva seqüència i és una homòloga en cisteïna. Teníem 5 prediccions d'aquest gen que, per la seva part, eren massa petites per formar un gen complert. No obstant, mirant els resultats del t-coffee per cada predicció quedava clar que els tres primers hits són part de la mateixa proteïna, ja que observant l'alineament on acabava una començava l'altra. Per tant, el nostre gen final està separat en 3 contigs, seguint el següent ordre: KN635766.1 (posicions: 28143-44919), KN624039.1 (posicions: 3393-3569) i KN635239.1 (posicions: 262551-262700). El gen té 4 exons, 518 nucleòtids i 173 aminoàcids i, conseqüentment, podem concloure que Vipera berus conté un gen MsrA al seu genoma.
La SelW està altament expressada en múscul esquelètic i cardíac. Podria estar relacionada en processos antioxidatius, com la majoria de selenoproteïnes. La seva funció específica és desconeguda.
La selenoproteïna SelW d'Anolis carolinensis té 48 aminoàcids en la seva seqüència. No hem predit cap gen per la SelW en el genoma de Vipera berus.
El Eukaryotic elongation factor té funcions de glutatió peroxidasa de en la detoxificació del peròxid d'hidrogen i és un dels enzims antioxidants més importants en humans.
La proteïna eEFsec és una proteïna de la maquinària relacionada amb la síntesis de selenoproteïnes. En el genoma de Anolis carolinensis n'hi ha 3 còpies. La primera, eEFsec(1), té 215 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El primer gen predit s'ha trobat en l'scaffold KN617171.1 entre les posicions 36097-36741, amb 1 exó de 644 nucleòtids, que codifiquen per 215 aminoàcids. La segona, eEFsec(2) té 79 aminoàcids. No hem predit cap gen per aquesta còpia. La tercera, eEFsec(3) té 168 aminoàcids. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El primer gen predit s'ha trobat en l'scaffold KN617171.1 entre les posicions 2901-10232, amb 3 exons de 492 nucleòtids, que codifiquen per 164 aminoàcids. Aquest resultat suggereix que el genoma de Vipera berus conté 2 còpies del gen eEFsec.
.png)
.png)
SBP2 codifica per una proteïna nuclear que funciona com una proteïna d'unió al SECIs. Mutacions en aquest gen estan associats amb la reducció de l'activitat de la deiodinase tiroxina, una selenoporteïna i amb l'alteració del metabolisme de l'hormona tiroidea.
El genoma d'Anolis carolinensis té 2 paràlegs del gen SBP2. El primer, SBP2(1), té 1055 aminoàcids. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. La predicció es troba en l'scaffold KN618877.1 entre les posicions 581837-599039, amb 15 exons que en total tenen 2997 nucleòtids, que codifiquen per 999 aminoàcids. El segon, SBP2(2), té 854 aminoàcids. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. La predicció es troba en l'scaffold KN632477.1 entre les posicions 82268-108573, amb 14 exons que en total tenen 2170 nucleòtids, que codifiquen per 723 aminoàcids. Per tant, hem predit les dues copies del gen SBP2. No s'han predit elements SECIs, ja que no es tracta d'una seleneoproteïna ni d'una homòloga.
.png)
.png)
La SecS catalitza el 3r pas de la síntesi del tRNa de la selenocisteïna: la conversió de O-phosphoseryl-tRNA(Sec) a selenocysteinyl-tRNA(Sec).
La proteïna Secs és una proteïna de la maquinària relacionada amb la síntesis de selenoproteïnes. En Anolis carolinensis té 466 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El gen predit s'ha trobat en l'scaffold KN615039.1 entre les posicions 4537-76572, amb 10 exons de 1299 nucleòtids, que codifiquen per 433 aminoàcids en la seva seqüència. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SecS.

La quinasa Phosphoseryl-tRNA es troba molt conservada al llarg de l'evolució, cosa que suggereix que té un paper important en la biosíntesis de la selenoproteïna o en la seva regulació.
La proteïna PSTK és una proteïna de la maquinària relacionada amb la síntesis de selenoproteïnes. En Anolis carolinensis té 352 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El gen predit s'ha trobat en l'scaffold KN633361.1 entre les posicions 47138-54632, amb 6 exons de 1047 nucleòtids, que codifiquen per 349 aminoàcids en la seva seqüència. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen PSTK. No hem predit element SECIs, ja que no és una selenoproteïna ni una homòloga.

Aquesta proteïna regula l'expressió de selenoproteïnes.
La proteïna SECp43 és una proteïna de la maquinària relacionada amb la síntesis de selenoproteïnes. En Anolis carolinensis té 268 aminoàcids en la seva seqüència. Hem predit 1 gen d'aquesta proteïna en el genoma de Vipera berus. El gen predit s'ha trobat en l'scaffold KN636726.1 entre les posicions 201915-204816, amb 6 exons de 621 nucleòtids, que codifiquen per 207 aminoàcids en la seva seqüència. Aquest resultat suggereix que el genoma de Vipera berus conté 1 gen SECp43. No hem predit element SECIs, ja que no és una selenoproteïna ni una homòloga.
