Selenoproteïnes
Sel15, SelL, Fep15
Adell A, Agustín JM, Andrés C, Bartrolí M, Brasó M
Discussió Sel15
En aquesta part del treball explicarem amb detall els resultats obtinguts en l'apartat de resultats. Analitzarem els resultats de cada espècie per separat per tal que sigui més entenedor.
Després de realitzar el tBLASTn amb aquest genoma i les diferents queries, no s'ha obtingut cap hit significatiu ni tampoc cap alineament entre la U de la query i un codó stop o una cisteïna del genoma. Per això, es pot descartar que en A.laibachii hi hagi una proteïna homòloga a Sel15. Així doncs, no s'ha continuat amb l'anàlisi.
Torna a dalt
Després de realitzar el tBLASTn amb aquest genoma i les diferents queries, no s'ha obtingut cap hit significatiu ni tampoc cap alineament entre la U de la query i un codó stop o una cisteïna del genoma. Per això, es pot descartar que en A.rara hi hagi una proteïna homòloga a Sel15. Així doncs, no s'ha continuat amb l'anàlisi.
Torna a dalt
Després de realitzar el tBLASTn amb les diferents queries, s'ha obtingut un hit significatiu amb les següents queries:
Query E-value Alineació A. Anophagefferens 3x10-06 No D. Melanogaster 3x10-08 No E. Huxleyl 9x10-07 Valina D. Purpureum 8x10-05 Valina
En aquest hit es pot veure que la selenocisteïna de les queries s'alinea amb una proteïna que no conté cap codó stop o cisteïna alineada amb la Sec. Per aquest motiu, es pot afirmar que C.fasciculata no té cap homòleg de Sel15, i que aquest hit significatiu resulta de l'alineament amb una altra proteïna. Tot i això, també s'ha dut a terme l'Exonerate i el Genewise per veure si hi havia alineament de la Sec. Els resultats no han sigut satisfactoris.
Torna a dalt
Query E-value Alineació D. Purpureum 3x10-41 Stop
En fer tBLASTn amb la query de D.purpureum contra A.discoideum ha sortit un hit significatiu amb un E-value de 4x10-41 en el que s'alineava la Sec amb un codó stop.
En executar l'Exonerate apareix un resultat coherent amb l'observat al tBLASTn, ja que l'score és de 422 i la Selenocisteina (Unk) s'alinea amb un codó stop. Després de fer el T-Coffee i veure que l'score és de 97 i que la selenocisteïna de la queries s'alineava amb un gap de la proteïna predita, podem hipotetitzar que D. discodideum conté la Sel15. Per a corroborar que els resultats obtinguts eren fiables, també es va dur a terme el Genewise que predia una proteïna que conté la selenocisteïna i el corresponent T-Coffee, que concorda amb els resultats obtinguts amb l'Exonerate.
Seguidament, vam estudiar la maquinària necessària per a la síntesi de selenoproteïnes en aquest protist. Vam fer un tBLASTn amb seqüències diferents proteïnes de la maquinària com a query (EF-Sec, Pstk, SBP2, Sec p43, SLA/LP (SecS), SPS2 i tRNA) contra el genoma dels protists estudiats.
Després de fer el T-Coffee, vam concloure que D.discoideum conté en el seu genoma els gens que codifiquen per les següents proteïnes: SBP2, Sec p43, EF-Sec, Pstk i SLA/LP (SecS). Tot i així, nosaltres no vam trobar tRNAs de selenocisteïna en aquest genoma.
Un altre fet que dóna suport a l'afirmació que D. discoideum conté la Sel15, és la predicció d'un element SECIS a l'extrem 3' del gen de la Sel15. Aquest element SECIS està molt conservat (score = 25,07) i es troba a una distància menor de 4000 nucleòtids del gen.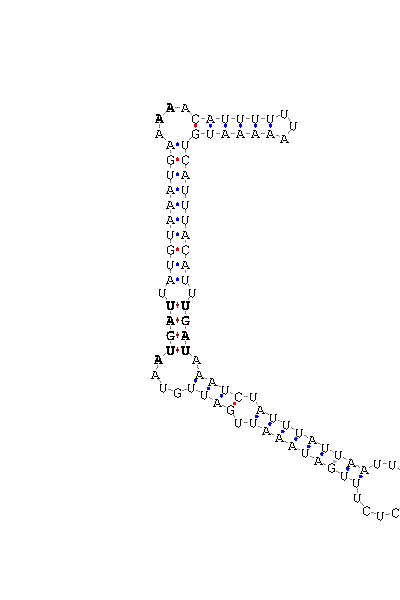
Un cop obtinguda, es va fer un pBLAST amb la base de dades NCBI utilitzant la proteïna predita com a query. Al fer-ho, vam trobar que l'agost del 2009 la Sel15 va ser descrita completament. Fins aleshores, s'havia confós la selenocisteïna amb un codó STOP i, per tant, la proteïna es descrivia truncada.
També hem vist que, l'any 2005, a la revista Nature es va descriure tot el genoma de D. discoideum i també es van descriure tRNAs de selenocisteïnes (que com ja hem esmentat, nosaltres no vam trobar).[9]
Degut a tots els resultats obtinguts, podem afirmar que aquest organisme té Sel15, tal i com nosaltres havíem predit.
Torna a dalt
S'ha realitzat el tBLASTn entre les diferents queries escollides i el genoma de D.fasciculatum i s'ha obtingut un hit significatiu en les següents queries: D.discoideum D. Purpureum, E. Huxleyl i G. gallus.
Query E-value Alineació D.discoideum 2x10-27 Stop D.Purpureum 2x10-33 Stop E. Huxleyl 1x10-15 Stop G.gallus 7x10-11 Stop
A partir d'aquests hits es van realitzar els corresponents Exonerates, Genewise i T-coffee:
- Exonerate: en tots dos casos la selenocisteïna de la query s'ha alineat amb un codó stop: D. discoideum i D. purpureum.
- Genewise: D. discoideum i D. Purpureum.
- Hem escollit els T-Coffee realitzats amb la proteïna predita pel Genewise, ja que donaven millors resultats que no pas els T-Coffee obtinguts amb l'exonerate (alineament de la selenocisteïna amb un gap i millor score): D. Discoideum i D. Purpureum
Obtinguts aquests resultats, vam decidir continuar la nostra investigació realitzant un BLASTp de la nostra proteïna predita pel Genewise contra la base de dades de proteïnes NCBI.
Degut a què els resultats de la cerca van donar lloc a dues proteïnes molt similars vam seguir l'estudi amb la proteïna predita amb la query D. discoideum. Tot i realitzar aquest estudi amb la proteïna predita pel Genewise amb la query D. discoideum, vam seguir tenint en compte la predita per l'Exonerate, que havia considerat com a intró una regió que el Genewise havia considerat com a exó, al mateix temps que també teníem en compte la predita per l'altra query (T-Coffee de les quatre proteïnes).
La seqüència que tenim a continuació correspon a la proteïna predita utilitzada per fer el BLASTp, contenint en negreta la part de la seqüència identificada com a part d'un intró per l'Exonerate i com a seqüència codificant pel Genewise:

Al fer el pBLAST entre la proteïna predita i la base de dades NCBI, trobem que dos dels hits més bons són proteïnes de D. Fasciculatum.
Hypothetical protein DFA_09870
La primera d'aquestes proteïnes correspon a l'hypothetical protein DFA_09870 [Dictyostelium fasciculatum]:
![]()
La nostra query amb aquesta altra proteïna té una identitat del 100%, però només el 49% de la query s'alinea (E-value = 2x10-39). Per aquest motiu, l'score que s'obté no és tan alt com esperaríem per un alineament amb un 100% d'identitat (score = 133).
Proteïna predita pel Genewise utilitzada com a query en el pBLAST amb la part alineada amb hypothetical protein DFA_09870 en color:

Com es pot veure només s'alinea la segona part de la seqüència, sense alinear la selenocisteïna. De fet, a la base de dades ja surt descrita com a proteïna que conté un domini redox possiblement pertanyent a Sel15 o SelM.
15 kDa selenoprotein
Curiosament, l'altra proteïna descrita a la base de dades NCBI de D. Fasciculatum que presenta gran identitat amb la nostra proteïna predita és 15 kDa selenoprotein [Dictyostelium fasciculatum], que s'alinea amb un 83% d'identitat en un 93% de la llargada de la proteïna (E-value = 8x10-28). Tot i aquests valors d'identitat, vam observar que hi havia una regió on les dues proteïnes són 100% idèntiques. Aquesta regió correspon a una part de la nostra proteïna predita que no coincideix amb la part que s'alinea amb Hypothetical protein DFA_09870 i que tampoc inclou la selenocisteïna.
Tot i que aquesta proteïna està classificada com a selenoproteïna de 15 kDa no conté cap selenocisteïna. A part, la identitat del 100% que presenta amb la nostra proteïna predita en un segment que precedeix la regió que conté la selenocisteïna, suggereix que la descripció de la proteïna no contempla part de la seqüència.
15 kDa selenoprotein [Dictyostelium fasciculatum]
![]()
Proteïna predita pel Genewise utilitzada com a query en el pBLAST amb la part alineada amb 15 kDa selenoprotein en color:

D'aquesta manera, la nostra proteïna predita s'alinea amb un 100% d'identitat amb dues proteïnes del mateix organisme classificades en la base de dades NCBI. A part, com es pot veure a baix, les dues parts alineades corresponen als dos extrems de la proteïna predita i no inclouen la regió on es troba la selenocisteïna:

Un cop vist aquest resultat, vam intentar aconseguir les seqüències nucleotídiques codificants d'aquestes dues proteïnes per tal de buscar la posició en què es troben dins el genoma de D. Fasciculatum. Aquestes seqüències no estan disponibles a la base de dades, de manera que vam decidir fer un tBLASTn amb les dues seqüències aminoacídiques d'aquestes proteïnes contra el genoma del protist.
Al fer aquests dos tBLASTn vàrem veure que en tots dos casos apareixia un hit significatiu amb un E-value molt baix en la mateixa regió del genoma (el mateix contig), que coincideix amb el contig en el que es troba el hit a partir del qual nosaltres hem predit la nostra proteïna en D. fasciculatum.
tBLASTn amb el que hem predit la nostra proteïna.
tBLASTn en el genoma de D. fasciculatum amb la proteïna 15 kDa selenoprotein.
tBLASTn en el genoma de D. fasciculatum amb la proteïna Hypothetical protein DFA_09870.
Seguidament, vam decidir utilitzar la comanda Fastasubseq per a traduir la regió del contig que engloba els dos hits que hem trobat, el de la 15 kDa selenoprotein i el de la Hypothetical protein DFA_09870.
A continuació, vam utilitzar el programa Fastatranslate per a traduir la seqüència nucleotídica obtinguda amb el Fastasubseq en les 6 pautes de lectura diferents per tal de trobar-hi les seqüències proteiques de què disposàvem: la seqüència proteica de 15 kDa selenoprotein, la de Hypothetical protein DFA_09870 i la de la nostra proteïna predita.
Els resultats obtinguts van ser:
La seqüència de Hypothetical protein DFA_09870 la vam trobar en la cadena revers, en la tercera pauta de lectura:
Hypothetical protein DFA_09870:
![]()
Situació en la traducció de la regió total:
[revcomp]:[translate(3)]

Pel que fa a la seqüència de 15 kDa selenoprotein, en vam trobar una part en la primera pauta de lectura de la cadena reversa i l'altra en la mateixa pauta de lectura i cadena en la que havíem trobat la proteïna Hypothetical protein DFA_09870 (regió marcada en color en la seqüència inferior).
15 kDa selenoprotein:
Situació en la traducció de la regió total:
[revcomp]:[translate(1)]

[revcomp]:[translate(3)]
Per últim, la nostra proteïna predita la vam trobar dividida en tres parts: una en la primera pauta de lectura, i l'altra, en la tercera (les dues en la cadena reversa):
Proteïna predita:

Situació en la traducció de la regió total:
[revcomp]:[translate(1)]

[revcomp]:[translate(3)]:

Com podem veure a continuació, la proteïna predita conté part de les seqüències de les altres dues proteïnes (en vermell la proteïna predita). A part, la regió que conté la selenocisteïna (en verd a les seqüències) queda entremig de les altres dues proteïnes:
[revcomp]:[translate(1)]

[revcomp]:[translate(3)]

Aquests resultats suggereixen que les dues proteïnes descrites com a hipotètiques o possibles homòlogues a Sel15 dins la base de dades NCBI, possiblement són una mateixa proteïna i que la proteïna total conté, a més, una regió entre una i altra on s'hi troba la selenocisteïna.
Per completar aquest anàlisi, hem identificat dins la regió del genoma de D. fasciculatum que inclou la codificació totes aquestes seqüències, els nucleòtids codificants per a cadascuna d'elles. Tenint en compte que les seqüències estan codificades en la cadena reversa, en groc apareix Hypothetical protein DFA_09870, en taronja 15 kDa selenoprotein i en vermell la proteïna predita pel Genewise.
>gi|328866594|gb|GL883026.1|:subseq(176875,715) Dictyostelium fasciculatum

En referència a l'existència d'elements SECIS, no se n'ha pogut predir cap mitjançant el programa SECISearch. Tot i així i tenint en compte que D. discoideum, D. purpureum i D. fasciculatum pertanyen tots tres al fílum de les amebes i, per tant, són filogenèticament propers, no és estranya la presència de Sel15 en tots tres genomes.
Amb totes les dades obtingudes, ens atrevim a dir que el genoma D. fasciculatum conté la selenoproteïna Sel15, la seqüència de la qual ha estat descrita a la base de dades NCBI com a dues proteïnes diferents i que la proteïna real conté, a més, una regió no descrita en aquesta base de dades que conté la selenocisteïna.
Torna a dalt
En el cas d'aquest genoma, no s'ha obtingut cap hit significatiu, i per tant, s'ha descartat la possibilitat de trobar un homòleg de Sel15 en F.cylindrus.
Torna a dalt
En el cas d'aquest genoma, no s'ha obtingut cap hit significatiu, i per tant, s'ha descartat la possibilitat de trobar un homòleg de Sel15 en G.niphandrodes.
Torna a dalt
En el cas d'aquest genoma, no s'ha obtingut cap hit significatiu, i per tant, s'ha descartat la possibilitat de trobar un homòleg de Sel15 en I.multifilis.
Torna a dalt
En aquest cas, amb el tBLASTn s'obté un hit significatiu amb dues queries: D.melanogaster i O.lucimarinus . Tot i així, la selenocisteïna de la query no es troba alineada amb cap codó stop o cisteïna del genoma. Per tant, es podria descartar la possibilitat de què en L.donovani hi hagi un homòleg de Sel15.
Malgrat saber-ho, hem seguit estudiant aquest protist i, per això, hem realitzat l'Exonerate, que, com esperàvem, no ha funcionat. També hem executat el Genewise i després el seu corresponent T-Coffee(D.melanogaster i O.lucimarinus ), que han donat scores força bons. Ara bé, la selenocisteïna de la query no s'alinea amb cap codó stop o cisteïna i, per tant, s'afirma que no es troba Sel15 en L.donovani.
Torna a dalt
En el cas d'aquest genoma, no s'ha obtingut cap hit significatiu, i per tant, s'ha descartat la possibilitat de trobar un homòleg de Sel15 en L.tarentolae.
Torna a dalt
En el cas d'aquest genoma, no s'ha obtingut cap hit significatiu, i per tant, s'ha descartat la possibilitat de trobar un homòleg de Sel15 en P.polycephalum.
Torna a dalt
En realitzar el tblastn s'ha obtingut un hit significatiu en les següents queries:
Query E-value Alineació A.anophagefferens 3x10-05 No C.elegans 4x10-05 No E. Huxleyl 4x10-08 Arginina X.tropicalis 8x10-09 Arginina
Tot i obtenir un hit significatiu, en cap d'ells la Sec s'alinea amb un codó stop o una cisteïna i, per tant, és molt probable que en aquest genoma no es trobi cap homòleg de Sel15. Tot i així, seguint amb la metodologia establerta, s'ha obtingut un Genewise amb X. tropicalis i el seu corresponent T-Coffee, amb un score acceptable (88). En aquest T-Coffee, però, la selenocisteïna es troba alineada amb una arginina, de manera que es pot reafirmar que no hi ha cap homòleg de Sel15 en P.capsici.
Torna a dalt
En el cas d'aquest genoma, no s'ha obtingut cap hit significatiu, i per tant, s'ha descartat la possibilitat de trobar un homòleg de Sel15 en S.arctica.
Torna a dalt
En realitzar el tblastn s'ha obtingut un hit significatiu en un parell de queries:
Query E-value Alineació D.discoideum 8x10-05 Lisina D.purpureum 3x10-04 Lisina
Aquest hit no té un bon alineament de la selenocisteïna, i per tant, el més probable és que aquest genoma no contingui cap Sel15. Aquesta hipòtesis es pot reafirmar veient que l'Exonerate no dóna cap resultat i que tot i que el Genewise sí, la selenocisteïna de la query no es troba alineada amb cap codó stop o amb cap cisteïna. Per tant, podem dir que T.congolense no conté cap homòleg de Sel15.
Torna a dalt
