Discussió
Introducció
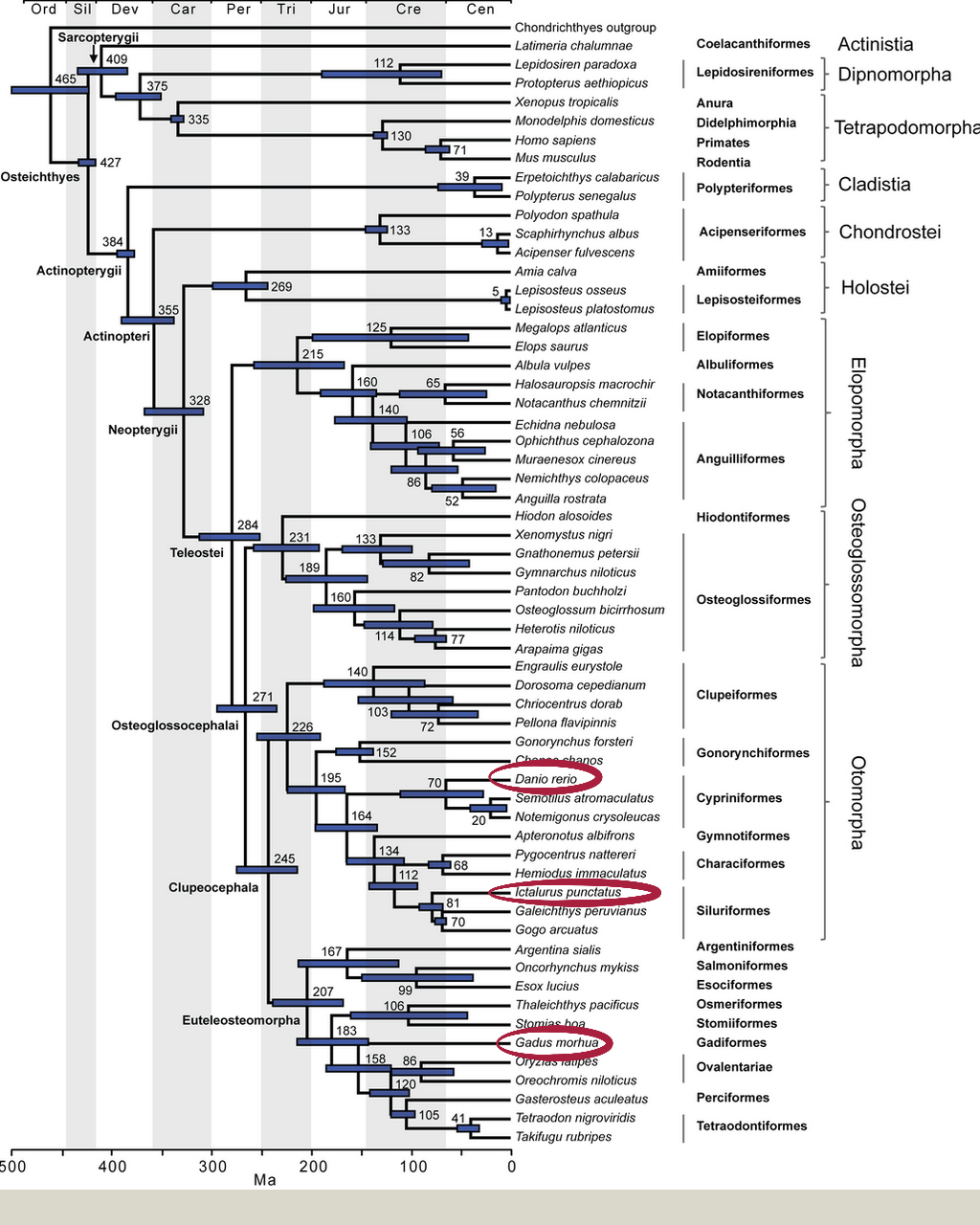
Les selenoproteïnes d’Ictalurus punctatus (peix gat americà) es van caracteritzar a partir de l’anàlisi de la seva homologia amb les selenoproteïnes de Danio rerio (peix zebra) i d’Homo sapiens (humà). L’elecció de Danio rerio es va fer gràcies a la seva proximitat filogenètica -tots dos pertanyen a la classe Actinopterygii- i perquè, al ser un model animal en peixos, esperàvem que la seva caracterització del genoma estigués ben feta. A més a més, Danio rerio és el peix ossi que presenta un selenoproteoma més gran, amb un total de 36 selenoproteïnes (més 8 homòlogues en cisteïna i 8 proteïnes de maquinària) extretes de les bases de dades SelenoDB i UniProt. Tot i això en alguns casos vam utilitzar Gadus morhua (bacallà comú) -que també està aprop filogenèticament- abans de confirmar que algunes selenoproteïnes no estaven presents en el nostre peix (Sel J i Sel L).
Els peixos ossis són el llinatge amb el major número de selenoproteïnes i conté algunes selenoproteïnes exclusives com SelE (Fep15) -que va sorgir d’una duplicació de SelM-, SelL i Sel J. En l’evolució dels vertebrats, a mesura que es va donar la colonització del medi terrestre el número de selenoproteïnes es va anar reduint. Alguns gens es van perdre i d’altres van reemplaçar la selenocisteïna (Sec) per una cisteïna (Cys). És per això que en els mamífers trobem el menor número de selenoproteïnes (25), reforçant la idea que els animals superiors han reduït la utilització de Sec. Tot i això algunes selenoproteïnes com GPx5 i GPx6 només es troben en mamífers placentaris i, per tant, no es van trobar en el nostre organisme.
Per altra banda, els peixos ossis presenten algunes selenoproteïnes duplicades com GPx1b, GPx3b, GPx4b, DI3b, SelT1b. En Danio rerio per exemple, hi ha duplicacions que també vam trobar en Ictalurus punctatus com SelT (T1, T1b i T2) i Sel W2 (SelWa i SelWb) i d’altres com SelO (SelO1 i SelO2) que no estaven duplicades en el nostre organisme. Altres duplicacions en Danio rerio com DI3 (DI3a i DI3b) simplement no es van identificar en Ictalurus punctatus.
A continuació s’expliquen àmpliament els resultats obtinguts en el nostre anàlisi d’acord amb la informació bibliogràfica trobada dels peixos ossis en general i de Danio rerio. Tot i això algunes interpretacions han sigut difícils de realitzar ja que no hi ha prou recerca bibliogràfica que recolzés els nostres resultats. Falta doncs, una major recerca del selenoproteoma dels vertebrats, i en especial dels peixos. [28]

Selenoproteïnes identificades en Ictalurus punctatus
SEL 15
La selenoproteïna 15 (Sel15) és una de les proteïnes ancestrals que es troba en tots els vertebrats i que, per tant, també està present en Danio rerio i Ictalurus punctatus. La seqüència de Sel 15 es va obtenir de la base de dades UniProt ja que no estava ben anotada a SelenoDB. Es va trobar un scaffold CM004430.1 i amb el t-coffee es va comprovar que es donava un alineament correcte. La proteïna predita conté un residu Sec i un element SECIS a la regió 3’UTR. Per tant, Sel15 està present en Ictalurus punctatus.
SEL M i SELENO E (Fep15)
La selenoproteïna M (SelM) s’expressa principalment al cervell on se l’ha relacionat amb efectes neuroprotectors. Recents estudis han determinat el seu rol com a enzim antioxidant per la seva implicació en la regulació de l’alliberament de calci del reticle endoplasmàtic en resposta a H2O2. Així doncs, s'ha corroborat la seva funció protectora envers el dany oxidatiu cerebral. [26]
Per altre banda, la selenoproteïna E (Fish selenoprotein 15 o Fep15), és una proteïna de la família Sep15 que només es troba en peixos, i que es creu que va sorgir d’una duplicació de SelM. Fep15 és una selenoproteïna resident al reticle endoplasmàtic de la qual encara no es coneix la funció, tot i això la seva estreta distribució suggereix que té una funció especialitzada que és diferent de Sep15 i SelM. [27]
En Ictalurus punctatus vam trobar les dues proteïnes fent servir com a genoma de referència el de Danio rerio.
L’anàlisi del blast de SelE va mostrar un scaffold per aquesta proteïna en Ictalurus punctatus (CM004435.1) i es va obtenir un alineament correcte. Es va veure que presentava un residu Sec (codificat pel codó TGA) en la seva seqüència i un element SECIS en l’extrem 3’UTR.
En l’anàlisi de la proteïna SelM -seqüència obtinguda d’UniProt- es va trobar el mateix scaffold (CM004435.1) que per Fep15, malgrat que els dos gens es trobaven en posicions diferents (14754818-14759539 per SelM i 3592794-3594767 per SelE). Aquests resultats reforcen la idea que Fep15 va sorgir d’una duplicació de SelM. En ambdues proteïnes es va caracteritzar bé el seu element SECIS.
Família Glutatió peroxidases (GPx)
La família de les glutatió peroxidades (GPx) està formada per enzims que presenten diferents funcions en l'organisme, però la funció més important en la qual participen és en la protecció de l'organisme enfront el dany oxidatiu. [28]
Aquests enzims catalitzen la reducció del peròxid o lipoperòxid utilitzant com a agent reductor el glutatió reduït. [29]
La família GPx és la família de selenoproteïnes més gran dels vertebrats i està implicada en múltiples processos. En mamífers, GPx està formada per 8 proteïnes homòlogues, de les quals cinc són selenoproteïnes (GPx1-4 i GPx6) i les altres han evolucionat a partir d’aquestes. Però, en canvi, en els peixos ossis no trobem GPx5 i GPx6 (van aparèixer en l’origen dels mamífers placentaris a partir d’una duplicació en tàndem de GPx3) [29] [30]. Aquest fet també es pot veure en la imatge de l’evolució del selenoproteoma dels vertebrats en la introducció.
En el nostre anàlisi es van poder identificar bé la resta de les proteïnes d’Ictalurus punctatus: GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, GPx7 i GPx8. Val a dir que totes les proteïnes d’aquesta família es van analitzar manualment perquè l’alt grau d’homologia trobat en el blast dificultava l’assignació proteïna-scaffold. En els peixos ossis, se sap que GPx1b, GPx3b i GPx4b s’han generat a partir de duplicacions igual que en la resta de peixos [28]. En realitzar el blasts vam trobar que moltes de les parelles (p.ex: GPx1a-GPx1b) compartien molts hits amb % d’identitat i e-value bons (Blast conjunt Família GPx). També es va observar que GPx7 i GPx8 tenien un alt grau de similitud. L’e-value més gran per GPx7 corresponia al segon millor scaffold o hit per GPx8 i viceversa. Els resultats concordaven amb la bibliografia: se sap que la Cys present en ambdues proteïnes va evolucionar a partir de la GPx4, fet que va succeir abans de la separació de peixos i mamífers. [28]
Pel que fa als elements SECIS, es van trobar en totes les GPx excepte en la GPx3b, en la GPx7 i en la GPx8. Aquestes dues últimes no presenten cap Sec en la seva seqüència proteica i per tant no es va poder trobar cap element SECIS en l’extrem 3’ UTR (tal i com indica la bibliografia).
En el cas de la proteïna Gpx3b el fet que no aparegui element SECIS es podria deure a errors de computació o problemes d’ensamblatge ja que al tractar-se d’una selenoproteïna hauria de tenir SECIS a l’extrem 3’UTR.
Família iodotironina deionidases (DI)
La família iodotironina deionidases (DI) està formada per enzims que catalitzen la pèrdua d'àtoms de iode de les hormones tiroidees, així doncs, aquests enzims regulen tant l'activació com la inactivació d'aquestes hormones. En el peix zebra (Danio rerio), igual que en els mamífers, hi ha tres proteïnes DI (DI1, DI2 i DI3) que mostren homologia intrafamiliar. En tots els peixos ossis s’ha observat que DI3 està duplicada, donant com a resultats les proteïnes DI3a i DI3b [31], [32]. La seqüència de DI1 es va obtenir de la base de dades UniProt ja que estava mal anotada en SelenoDB. Les altres es van obtenir de SelenoDB.
En l’anàlisi de la família, es van obtenir dos scaffolds (CM004438.1 i CM004422.1) per a totes les proteïnes (D1, D2, D3a i D3b). Basant-nos en les seves identitats i e-values es va analitzar l’scaffold CM004438.1 per a la proteïna DI1 i el CM004422.1 per DI2. En ambdues proteïnes vam obtenir el seu element SECIS corresponent. A partir d’aquests resultats es conclou que DI1 i DI2 estan presents en Ictalurus punctatus i que s’ha produït la pèrdua del gen DI3.
Msr (MsrA i MsrB)
Les Metionina sulfoxi reductases (Msr) són enzims dependents de tiol que catalitzen la conversió de sulfoxi-metionina (Met(O)) a metionina (Met). La majoria de les Msr són proteïnes homòlogues en cisteïna on la selenocisteïna (Sec) ha estat substituïda per una cisteïna (Cys) en la seqüència proteica. Dins d’aquesta família trobem tres proteïnes: MsrA, MsrB i fRMsr, sent MsrA i MsrB les principals. Ambdues actuen de forma sinèrgica i simultània per assegurar una bona reducció de les Met(O), tenint així un paper molt important en la defensa cel·lular contra l’estrès oxidatiu i en la longevitat dels organismes. [33]
En l’anàlisi de la família es van obtenir els següents resultats. En el cas de la MsrA, es va observar que Danio rerio presenta dues proteïnes MsrA (MrsA 1 i MsrA 2). Al blastejar aquestes dues seqüències proteiques contra el genoma d’Ictalurus punctatus, es van obtenir en ambdues seqüències un mateix scaffold (CM004422.1) amb identitats i e-values molt semblants en totes dues. A partir d’aquests resultats es va hipotetitzar la existència d’una única proteïna MsrA en Ictalurus punctatus, a diferència de la duplicació present en Danio rerio.
La proteïna trobada es va poder alinear correctament i no es va trobar cap element SECIS tal i com indica la bibliografia (proteïna homòloga en cisteïna).
Pel que fa a la MsrB, es va observar que Danio rerio presenta quatre proteïnes MsrB (MsrB1a, MsrB1b, MsrB2 i MsrB3). Al fer el blast conjunt de totes les proteïnes contra el genoma d’Ictalurus punctatus es va observar la presència de dos scaffolds en les proteïnes MsrB1a i MsrB1b (CM004415.1 i CM004426.1) que no es trobaven en les proteïnes Msrb2 i MsrB3. En canvi, es van identificar dos scaffolds nous (CM004436.1 i CM004432.1) en les proteïnes MsrB2 i MsrB3. Partint d’això, es van analitzar els scaffolds amb major identitat i e-values elevats en cada una de les proteïnes i es van poder obtenir alineaments correctes en tots els casos. A més a més, en MsrB1a/1b es van identificar les selenocisteïnes i els elements SECIS en 3’UTR, mentre que en MsrB2 i MsrB3 -al tractar-se de proteïnes homòlogues en cisteïna- no es van trobar ni Sec ni elements SECIS. Amb això, es pot dir que en Ictalurus punctatus la proteïna MsrB està present en quatre formes proteiques diferents.
SEL H
La selenoproteïna H (SelH) és una proteïna present en el genoma ancestral. Estudis d’immunoprecipitació de la cromatina han mostrat que SelH s’ajunta específicament a seqüències que contenen elements heat shock i de resposta a l’estrès [34]. A més a més, té activitat glutatió peroxidasa [35] i està implicada en la regulació de la transcripció d’un grup de gens implicats en la síntesi de novo del glutatió i la detoxificació en fase II.
Es va trobar en Ictalurus punctatus fent servir com a genoma de referència el de Danio rerio. Presentava un scaffold (CM004421.1) i el seu element SECIS es va identificar correctament.
SEL I
La selenoproteïna I (SelI) és una selenoproteïna ancestral que es troba només en els vertebrats [36]. És una proteïna transmembrana que conté un domini altament conservat CDP-alcohol fosfatidiltransferasa, que està present en la colina (CHPT1) i en la colina/etanolamina (CEPT1) fosfotransferasa. CHPT1 i CEPT1 catalitzen l’últim pas en la síntesi de novo dels dos fosfolípids principals a través de la transferència dels grups de fosfocolina i fosfoetanolamina a diacilglicerol des de CDP-colina i CDP-etanolamina, respectivament [37].
Vam trobar que SelI també està present en Ictalurus punctatus. Es va blastejar contra el genoma humà ja que en Danio rerio estava mal anotada tan en SelenoDB com en UniProt. L’element SECIS es va identificar correctament.
SEL S i SEL K
Les selenoproteïnes S i K (SelS i SelK) són dues proteïnes ancestrals que es poden agrupar dins la mateixa família ja que, tot i no presentar similaritat de seqüència, tenen un domini transmembrana en l’extrem NH2-terminal, presència d’un segment ric en glicina (G-rich), i aminoàcids carregats positivament. A més, presenten una localització característica dels residus Sec en l’extrem COOH-terminal [38]. Anàlisis de genòmica comparativa han revelat que la família SelK/SelS és la família més estesa entre els eucariotes, i que està present des dels organismes unicel·lulars fins als éssers humans.
D’aquestes dues proteïnes només es va poder identificar com a selenoproteïna en Ictalurus punctatus la SelK utilitzant el genoma de Danio rerio com a referència, també es va trobar el seu element SECIS en la regio 3’UTR. Pel que fa a SelS, es va obtenir un alineament correcte indicant que la proteïna està present en aquesta espècie, però enlloc del residu Sec la proteïna presenta en la seva seqüència una Arg. Amb això es pot concloure que en el selenoproteoma del Ictalurus punctatus només trobem la SelK i que la SelS ha evolucionat passant a ser una proteïna sense Sec.
SEL N
La selenoproteïna N (SelN) és una glicoproteïna de membrana que s’expressa en concentracions elevades durant el desenvolupament embrionari i en menor mesura en els teixits adults, incloent el múscul esquelètic [39]. Estudis amb Danio rerio com a animal model han revelat que SelN és necessària per les primeres etapes de desenvolupament del múscul -ja que s’unix al receptor de rianodina (RYR)- i per la diferenciació d’aquest peix. [40] [41] [42]
També és una de les selenoproteïnes ancestrals que es va trobar en Ictalurus punctatus blastejant contra Danio rerio. L’element SECIS també es va identificar correctament.
SELENO O
La família de SELENO O és una familia ancestral de selenoproteïnes present en tots els vertebrats. Tot i que la selenoproteïna O (SelO) es coneix des de fa més d’una dècada [36] no s’ha estudiat la seva estructura o caractertització bioquímica. S'han detectat homòlegs de SelO en una àmplia varietat d'espècies, incloent bacteris, llevats, animals i plantes. SelO conté un residu Sec en l’antepenúltima posició en l'extrem COOH-terminal de la proteïna. No obstant, la majoria dels homòlegs contenen un residu Cys enlloc de Sec (les seqüències de SelO que contenen Sec estan presents només en vertebrats) [42]. En vertebrats, SelO presenta un pèptid de direccionament mitocondrial i un domini de proteïna quinasa [43], fent que es localitzin en el mitocondri i s’expressin en diferents teixits. SelO és una selenoproteina activa mitocondrial redox amb activitat fosfotransferasa en el seu centre actiu (on presente el residu Sec). La família SelO està formada per dues isoformes, SelO1 i SelO2 en els peixos ossis (es pot veure en la imatge de l’evolució del selenoproteoma dels vertebrats en la introducció).
Al analitzar ambdues proteïnes contra el genoma d’Ictalurus punctatus: SelO1 (blast contra Danio rerio) i SelO2 (blast contra el genoma humà), es va observar un alineament correcte només en el cas de la proteïna SelO1. D’aquesta es va identificar una selenocisteïna en la seva seqüència i un element SECIS en 3’UTR. Per altra banda, l’alineament obtingut de SelO2 era molt pobre. Això podria ser degut a la curta llargada del scaffold que apareixia en el blast, donant lloc a un alineament ínfim i fent impossible la identificació de Sec i d’elements SECIS. Com a conclusió, els nostres resultats confirmen la pèrdua de la duplicació present en altres llinatges. Ictalurus punctatus només presenta SELO1.
SELENO P
La selenoproteïna P (SelP) és una de les selenoproteïnes ancestrals present en tots els vertebrats i és una proteïna important per les funcions cerebrals ja que manté la homeostasi de Se al cervell. [44] [45]
En Danio rerio, SelP està formada per la SelP1 i la SelP2. Estudis en aquest peix han mostrat que durant el seu desenvolupament SelP1 s’expressa sobretot en el cor, en el cervell i en els ronyons; mentre que SelP2 predomina a nivell hepàtic. Amb això han conclòs que la seva transcripció en peixos adults es dóna en el cervell (SelP1) i en altres teixits (SelP2) [46]. SelP és la única proteïna amb múltiples residus i això es pot comprovar en veure la seqüència de Danio rerio i d’Ictalurus punctatus alineada (SelP1 en presenta 15 i SelP2 en presenta 1). En Ictalarus punctatus es van trobar totes dues proteïnes SelP. En SelP1 es va trobar l’scaffold CM004418.1 mentre que en SelP2 el CM004433.1. Es va observar que d’aquestes 17 selenocisteïnes presents en Danio rerio, dues s’han convertit en cisteïna en la seqüència proteica de la proteïna SP1 en Ictalurus punctatus i dues s'han convertit en arginina i glicina, tenint en total només 13 residus Sec en aquesta espècie.
En ambdues proteïnes es van trobar els seus elements SECIS corresponents, tots dos de grau A. Concordant amb la bibliografia, SelP1 presenta dos SECIS (un entre 198688-198758 i l’altre entre 199211-199280), ambdós situats en l’extrem 3’UTR i propers.
Família SELENO T
La selenoproteïna T (SelT) pertany a la família de selenoproteïnes amb característiques redox (SELENOT). Els membres d'aquesta família tenen un plegament del tipus thioredoxin-like i estan caracteritzats per la presència del domini conservat Cys-x-x-Sec. SelT es localitza al reticle endoplasmàtic i a l'aparell de Golgi principalment. Presenta un paper important en l'adhesió cel·lular i promou l'expressió de diferents gens que codifiquen per oxidoreductases. [47]
En Danio rerio trobem la presència de tres selenoproteïnes (T1, T1b i T2) que també es van identificar correctament en Ictalurus punctatus. En analitzar el blast es van identificar 3 scaffolds presents en les 3 proteïnes. A més, vam observar un alt grau d’homologia entre T1 i T1b, el que confirmava que hi ha hagut una duplicació de T1 que ha originat T1b. En aquestes proteïnes vam trobar els scaffolds CM004417.1 i CM004420.1, que també estaven presents a T2, però T2 presentava un nou scaffold (CM004421.1) amb un grau d’identitat i e-value més elevats. Amb això vam poder concloure que en Ictalurus punctatus estan presents les tres proteïnes de la família. També es va confirmar per la presència, en les tres, de l’element SECIS en l’extrem 3’UTR.
Família SELENO U
La selenoproteïna U (SelU) es va trobar primer en peixos i posteriorment es va observar que també estava present en ocells i en organismes eucariotes com Chlamydomonas reinhardtii . [48] [49]
La família SELENO U està formada per tres membres (SelU1, SelU2 i SelU3), però només SelU1 forma part del selenoproteoma ancestral. La seva funció roman desconeguda però el seu domini Prx-like2 recolza la idea que potser pertanyen a la superfamília tioredoxina. [50]
Múltiples alineaments i anàlisis filogenètics mostren que les proteïnes d’aquesta família estan àmpliament esteses i conservades en tots els vertebrats incloent peixos, amfibis, ocells i mamífers [50]. Tot i això altres estudis han mostrat que hi ha diferències en la família, en concret els mamífers presenten proteïnes amb la forma Cys mentre que els peixos presenten la forma Sec. Es creu que potser les formes Cys dels mamífers van evolucionar a partir de les formes dels peixos (conversió evolutiva de Sec per Cys, procés que contribueix a la reducció del selenoproteoma) però encara no hi ha evidències que determinin aquest esdeveniment per SelU2 i SelU3.
Danio rerio té les selenoproteïnes U1a, U2 i U3. En un primer moment les tres proteïnes de la família es van analitzar per Danio rerio, però com U2 i U3 estaven mal anotades a SelenoDB es va decidir analitzar les selenoproteïnes agafant com a referència el genoma humà (que presenta homòlogues en cisteïna).
En l’anàlisi de SelU1a es van obtenir dos scaffolds CM004416.1 i CM004426.1 amb e-values i identitats molt bones. Observant els alineaments, es va confirmar que en Ictalurus punctatus es produeix una duplicació que origina SelU1a i SelU1b ja que no es van veure similituds de seqüència entre ambdues proteïnes. Els seus elements SECIS es van identificar correctament.
L’anàlisi de SelU2 i SelU3 contra humà va mostrar els mateixos scaffolds que per Danio rerio corroborant l’existència d’aquestes proteïnes.
Així doncs en Ictalurus punctatus trobem SelU1a, SelU1b, SelU2 i SelU3.
Família SELENO W
La selenoproteïna W (SelW) és la selenoproteïna més petita que conté una selenocisteïna (Sec) en el motiu conservat Cis-X-X-Sec (10kDa). Es troba al citosol i s’expressa en diferents teixits, tot i que és més abundant en el múscul esquelètic i en el cervell dels mamífers [51]. És important degut al seu paper com a antioxidant en el metabolisme cel·lular. [52] [53]
A partir d’estudis, s’ha observat la presència de diverses proteïnes homòlogues de SelW en vertebrats no-mamífers. En concret, en peixos s’han identificat diverses còpies de la proteïna SelW2. En Danio rerio s’observen dues copies de SelW2 (SelW2a i SelW2b), ambdues selenoproteïnes localitzades en forma de tàndem en el cromosoma 3. Per contra, s’ha pogut observar la pèrdua de la proteïna SelW1 en diferents espècies de peixos com és el cas de Percomorpha. [52]
Al fer l’anàlisis de comparació de les seqüències proteiques de la família SelW presents en Danio rerio contra el genoma d’Ictalurus punctatus, es van poder obtenir resultats concordants amb la bibliografia anteriorment descrita. No es va poder identificar cap proteïna SelW1 per Ictalurus punctatus. Pel que fa a les proteïnes SelW2a i SelW2b, es van observar dos scaffolds (CM004441.1 i CM004415.1) i es van poder obtenir els alineaments correctes. També es van identificar els elements SECIS en posició 3’UTR. Amb això es confirma la duplicació de la proteïna SelW2 (SelW2a i SelW2b) en el genoma d’Ictalurus punctatus, fet comú en la majoria d’espècies de peixos.
Família tioredoxin reductasa (TR o TXNRD)
La família tioredoxina reductases (TR) són proteïnes que controlen l’estat redox de les tioredoxines, que alhora regulen l’estat redox dels processos cel·lulars. En mamífers trobem tres proteïnes (TR1, TR2 i TR3) però en els peixos ossis només s’han descrit dues: TR2 i TR3 [52]. Tot i això es va investigar si TR1 podria estar en Ictalurus punctatus a partir de la seqüència de TR1 humana. Al fer el blast es va trobar un scaffold (CM004424.1) que va donar lloc a un alineament correcte i també es va poder identificar un element SECIS a 3’ UTR.
Pel que fa TR2 i TR3, en el nostre anàlisi vam trobar una scaffold per la TR2 (CM004435.1) però, en canvi, vam trobar dos per la TR3 (CM004424.1 i CM004423.1). TR2 es va identificar correctament i es va trobar el seu element SECIS de grau A. En TR3 es descarta la duplicació de la proteïna ja que l’scaffold CM004424.1 es troba present en TR1. Quan es va analitzar l’scaffold CM004423.1 per veure si estava present en Ictalurus punctatus, es va obtenir un alineament molt pobre sense cap resultat de selenocisteïna en el genoma d’aquesta espècie (veure t-coffee). A més, no es va poder identificar l’element SECIS.
Per tant, aquests resultats confirmen en Ictalurus punctatus la presència de les proteïnes TR1 i TR2 i la pèrdua de la proteïna TR3.
Proteïnes de maquinària
SEPHS o SPS
La selenofosfat sintasa (SEPHS o SPS) és la única proteïna de maquinària de biosíntesi de Sec que alhora pot ser també una selenoproteïna [54], de manera que té dos gens paràlegs: SEPHS1 i SEPHS2. Un estudi recent mostra que els gens de SPS1 es van originar a partir d’una sèrie de duplicacions del gen SPS2. L’estudi mostra com, en els gens de la família de SPS, duplicacions paral·leles i subfuncionalitzacions convergents han donat com a resultat la segregació dels diferents loci de les funcions realitzades inicialment per un sol gen. [55]
És una proteïna que està conservada des dels bacteris fins els humans amb un 30% d’identitat [56] i que també es va identificar en Ictalurus punctatus. Ambdues proteïnes es van caracteritzar a partir de Danio rerio reforçant la informació de la bibliografia consultada: SEPHS1 és la selenoproteïna, en la qual també es va trobar l’element SECIS, mentre que SEPHS2 és la proteïna de maquinària.
Phosphoseryl-tRNA kinase (PSTK)
La fosfoseril-tRNA quinasa (PSTK) és una proteïna de maquinària conservada en eucariotes i arqueobacteris. Catalitza la reacció de seril-tRNA[Ser]Sec a la forma fosforilada fent servir una molècula d’ATP. Està formada per dos dominis independents connectats, el domini catalític N-terminal (NTD) i el domini C-terminal (CTD). La seva acció és essencial per la codificació específica UGA de les selenocisteïnes. [57]
PSTK es va analitzar fent servir com a referència el genoma humà ja que en Danio rerio estava mal anotada. La proteïna es va alinear correctament amb el genoma d’Ictalurus punctatus el que es confirma la seva presència en el nostre nostre peix.
SECIS binding protein 2 (SBP2)
Mentre que Selb s'uneix directament a SECIS i promou la descodificació sense ajuda (en procariotes), eEFSec només pot fer-ho en presència d'un factor de proteïna auxiliar exclusivament eucariota que és el SECIS Binding Protein 2 (SBP2).
SBP2 es va analitzar fent servir com a referència el genoma humà ja que en Danio rerio estava mal anotada. Es van obtenir dos scaffolds que coincidien amb els assignats per Danio rerio (CM004417.1 per SBP2.1 i CM004435.1 per SBP2.2). Es pot concloure, per tant, que en Ictalurus punctatus estan presents totes dues proteïnes: SBP2.1 i SBP2.2.
Selenocysteine synthase (SecS)
En els eucariotes i els arqueobacteris, la selenocisteïna sintasa (SecS) catalitza la reacció terminal de la selenocisteïna, en concret converteix O-fosfo-L-seril-tRNA[Ser]Sec a selenocisteil-tRNA[Ser]Sec gràcies a l’ús del selenofosfat com a donador de seleni. [58]
Estudis recents mostren que mutacions en aquesta proteïna poden poden provocar transtorns neurològics, d’aparició precoç, en diferents poblacions humanes. [59]
En el genoma d’Ictalurus punctatus es va poder predir SecS. No es va trobar element SECIS a la regió 3’UTR.
tRNA selenocysteine 1 associated protein (SECp43)
La proteïna SECp43, juntament amb la proteina SEPHS, forma part del complex proteic selenocisteïna sintasa. Aquest complex permet la incorporació del selenofosfat a l’aminoàcid. Així doncs, SECp43 és essencial per tal d’incorporar Sec-tRNASec a les selenoproteïnes de nova creació. Danio rerio presenta dues proteïnes SECp43 (SECp43 1 i SECp43 2). Quan vam blastejar aquestes dues seqüències proteiques contra el genoma d’Ictalurus punctatus, vam obtenir 3 scaffolds per les dues proteïnes: CM004437, CM004433 i CM004436. L’scaffold CM004433 es va assignar a SECp43 1 ja que presentava una identitat, e-value i llargada elevats, i es va poder alinear correctament. Els scaffolds per la SECp43 2 no es van considerar ja que presentaven llargades molt curtes i e-values i identitats mitjanes, donant lloc a alineaments ínfims. Aquest fet ens porta a concloure que en Ictalurus punctatus només hi ha una sola proteïna SECp43, a diferència de Danio rerio que la té duplicada.
Eukaryotic elongation factor (eEFsec)
El factor d’elongació eucariòtic o EFsec és una GTPasa que uneix Sec-tRNASec amb gran afinitat i especificitat i que té un paper molt important durant la descodificació mitjançant el lliurament de Sec-tRNASec al lloc de la traducció en resposta a un codó in-frame UGA [60]. El seu equivalent en procariotes es SelB. [61] [62]
Es va poder predir eEFsec en el genoma d’Ictalurus punctatus. No es va trobar element SECIS a la regió 3’UTR.

Selenoproteines no identificades en Ictalurus punctatus
SELENO J i SELENO L
Tot i que aquestes dues proteïnes -proteïnes ancestrals- es troben presents en Danio rerio, no es van trobar hits o scaffolds significatius per la nostra espècie en cap de les dues proteïnes (e-values elevats: 8,2 i 1,9) (blast SELENO J i blast SELENO L). Es va prosseguir a intentar buscar homologia de seqüències amb l’espècie Garus morhua, que es troba pròxima filogenèticament, però tampoc es van obtenir hits. Amb això es pot concloure que en l’espècie Ictalurus punctatus aquestes dues selenoproteïnes no estan presents.