Resultats i Discussió de F. cylindrus
La cerca amb tBlastn de la query d'Emiliana huxleyi al genoma de F.cylindrus ha predit 9 hits significatius (E-value < 1.10-4) en scaffolds diferents.
Hit 1
Tant Exonerate com Genewise prediuen un únic alineament. És directe i consta de 3 exons i 2 introns. A la tercera de les regions codificants, la selenocisteïna de la query s'alinea amb un residu de cisteïna. Genewise sols prediu l'exó principal. L'alineament de la seqüència peptídica amb Tcoffee detecta una regió d'elevada similitud que engloba la totalitat de la query. El Blastp realitzat amb la base de dades del NCBI retorna la pròpia seqüència query, dos precursors de EhSEP2, una selenoproteïna, i un gran nombre de proteïnes de la família de les Tiorredoxina-Disulfit Isomerases (TDI). No s'han trobat elements SeCIS amb SECISearch. Els bons resultats del Tcoffee i del Blastp permeten concloure que el primer dels nou hits és un gen homòleg a EhSEP2.
Hit 2
La predicció amb Exonerate retorna dos alineaments, mentre que Genewise sols reporta el primer, lleugerament més curt.
El primer alineament és directe i conté 2 exons i 1 intró. A la regió final del primer exó es troba un codó de parada TGA que s'alinea amb la selenocisteïna de la query. Així, molt probablement es tracta d'una selenoproteïna homòloga a EhSEP2 de E.huxleyi. El Tcoffee mostra un alineament molt bo i extens entre la seqüència proteica del hit i la query. El Blastp amb NCBI també retorna la query, els precursors d'EhSEP2, la selenoproteïna, i un gran nombre de proteïnes del tipus TDI.
El segon alineament és revers i presenta 3 exons i 2 introns. La seqüència consens de les TDI queda separada pel primer intró, de tal manera que el primer residu del segon exó correspon a la selenocisteïna de la query, la qual s'alinea amb una cisteïna. A diferència de l'alineament anterior, aquest es tractaria d'un homòleg amb cisteïna de EhSEP2. L'alineament obtingut amb Tcoffee també és molt bo, i mostra una extensa zona d'alta similitud, malgrat és lleugerament menor que en el cas anterior. Els alineaments reportats per NCBI inclouen també tots els mencionats anteriorment.
S'ha trobat un element SeCIS situat a 282 posicions downstream del final del primer alineament. Els dos alineaments no se solapen i són significatius, de manera que es tracta de dues còpies homòlogues a EhSEP2 situades en un mateix scaffold. La primera correspon a una selenoproteïna, fet reforçat per la presència d'un element SeCIS a 3' de l'alineament, i la segona a un homòleg amb cisteïna.
Hit 3
Exonerate prediu dos alineaments dins el mateix scaffold, i Genewise sols obté el primer, malgrat de menor longitud.
Aquest alineament és directe i conté 2 exons i 1 intró. Destaca la presència d'un codó de parada TGA a la regió final del primer exó, posició que s'alinea amb la selenocisteïna de la query. Molt probablement es tractarà doncs d'una selenoproteïna. L'alineament amb Tcoffee engloba pràcticament la totalitat de la seqüència query i la similitud és molt alta. El Blastp amb NCBI retorna resultats similars als anteriors, detectant la pròpia query, els dos precursors d'EhSEP2 i altres proteïnes de tipus TDI.
El segon alineament és revers, amb 3 exons i 2 introns. La seqüència consens de les TDI queda separada pel primer intró, i la selenocisteïna de la query s'alinea amb una cisteïna. L'alineament proteic amb Tcoffee mostra una regió d'elevada similitud relativament extensa, i NCBI reporta diverses proteïnes de la família TDI.
S'ha trobat un element SeCIS situat a 283bp downstream del final del primer alineament. Els dos alineaments no són solapants i són significatius. Es pot concloure que es tracta de dos gens situades dins un mateix scaffold i que són homòlogs a EhSEP2. El primer correspon a una selenoproteïna, fet reforçat per la presència d'un element SeCIS downstream de l'alineament, mentre que el segon és un homòleg amb cisteïna.
Hit 4
Un únic alineament ha estat predit tant per Exonerate com per Genewise en aquest scaffold. És directe, amb 2 exons i 1 intró. A la regió central del primer exó es troba la selenocisteïna de la query alineada amb un residu de cisteïna. L'alineament predit per Tcoffee és molt bo amb un fragment d'alta similitud que inclou pràcticament la totalitat de la query. NCBI retorna nombroses proteïnes de la família TDI. No s'han trobat elements SeCIS amb SECISearch. Els resultats obtinguts permeten concloure que l'alineament és òptim i que, amb força seguretat, es tracta d'un gen homòleg d'EhSEP2 amb cisteïna.
Hit 5
Exonerate prediu un únic alineament, revers i format per 4 exons i 3 introns. La selenocisteïna de la query queda alineada al tercer exó amb un codó de parada TGA. Molt probablement es tracta doncs d'una selenoproteïna homòloga a EhSEP2. L'alineament predit amb Genewise sols contempla el fragment posterior al codó de parada i, per tant, no conté la selenocisteïna. L'alineament del Tcoffee s'estén al llarg de la major part de la query amb una similitud molt alta. NCBI retorna múltiples proteïnes de tipus TDI. No s'han trobat elements SeCIS amb SECISearch. La gran similitud entre la seqüència proteica i la query, junt amb els resultats del Blastp amb NCBI fan pensar que l'alineament predit és significatiu i es tracta d'una selenoproteïna homòloga a EhSEP2. Si aquesta hipòtesi fos correcta, s'hauria d'haver detectat un element SeCIS a l'extrem 3' del gen, no obstant, el programa per detectar aquests elements té poca sensibilitat i sovint pot donar falsos negatius. Com a conseqüència, l'absència de l'element SeCIS no és una evidència suficient per descartar l'homologia detectada entre ambdues seqüències.
Hit 6
La predicció amb Exonerate retorna un sol alineament. Aquest és directe i, igual que en el cas anterior, conté 4 exons i 3 introns amb un codó de parada TGA alineat amb la selenocisteïna de la query al tercer exó. Igualment, Genewise sols prediu l'alineament passat el codó de parada. El Tcoffee és molt bo i extens, i NCBI retorna els diverses proteïnes de la família TDI. Malgrat no s'ha predit cap element SeCIS, l'alta homologia entre la seqüència proteica del hit i la query, junt amb els resultats del NCBI, permeten concloure que, amb molta probabilitat, es tracta d'una selenoproteïna homòloga d'EhSEP2.
Hit 7
Tant Exonerate com Genewise prediuen un sol alineament directe, amb un únic exó que conté cisteïna a la posició alineada amb la selenoptroteïna de la query. Dos dels aminoàcids del patró PWCGH estan canviats (PWCVW). El resultat de Tcoffee és molt bo, malgrat de menor llargada que en els casos anteriors. El Blastp realitzat amb NCBI retorna si nombroses proteïnes de la família de les TDI. L'alta similitud detectada amb Tcoffee i els resultats del Blastp permeten concloure que el hit obtingut és probablement un homòleg amb cisteïna d'EhSEP2.
Hit 8
Ambdós, Exonerate i Genewise, prediuen un únic alineament revers, amb 2 exons i 1 intró, en el que un residu de cisteïna s'alinea amb la selenocisteïna de la query al primer exó. Tcoffee mostra una regió considerable d'alta similitud, i NCBI retorna diverses proteïnes del tipus TDI en diferents organismes. No s'han trobat elements SeCIS. Per tant, és força probable que aquest hit sigui un gen homòleg amb cisteïna d'EhSEP2.
Hit 9
El hit predit amb tBlastn no ha donat lloc a cap alineament òptim, ni amb Exonerate ni amb Genewise. Per tant, es descarta que aquest gen sigui homòleg a EhSEP2.
Conclusió
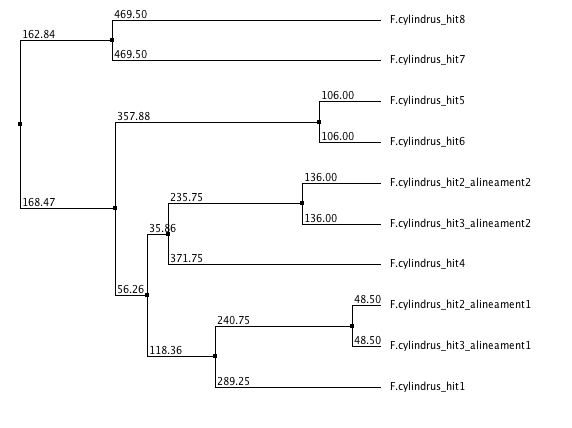
El genoma de F.cylindrus conté gens homòlegs a EhSEP2. Se n'han detectat un total de 10 repartits en 8 scaffolds diferents. Quatre d'aquests gens són selenoproteïnes, i la resta són homòlegs amb cisteïna. S'ha observat que la seqüència del hit 5 i 6 és idèntica, amb tan sols un aminoàcid diferent, fet que planteja la hipòtesis d'una duplicació relativament recent d'un dels gens. Els primers alineaments del hit 2 i 3 són també extremadament similars i ambdós contenen selenocisteïna. De la mateixa manera, el segon alineament d'ambdós hits també són molt similars, i són homòlegs amb cisteïna. Una hipòtesi per explicar aquests resultats és que un gen homòleg original (ja sigui una de les selenoproteïnes o un dels homòlegs amb cisteïna), va duplicar-se a una regió relativament propera dins del mateix scaffold. Posteriorment, va tenir lloc una duplicació conjunta de les dues còpies a un scaffold diferent.