

 |  |  |  |  |  |  |
| INTRODUCCIÓN | OBJETIVOS | MATERIALES | RESULTADOS | DISCUSIÓN | AGRADECIMIENTOS | REFERENCIAS |

A continuación explicaremos el resultado para cada una de las selenoproteínas estudiadas.
1 dna:chromosome chromosome:CHIMP1A:1:85694382:85729748:1
Exonerate sólo fue capaz de predecir tres exones en esta región genómica por lo que decidimos hacer un BLAT en UCSC Browser de aquella región de la selenoproteína humana para la cual no habíamos conseguido alineación. Ahora esta región del genoma de chimpancé ya tenía el mismo número de exones que la selenoproteína humana.
Para visualizar la salida del Exonerate pulse aquí.
GFF Para visualizar la información genómica en formato gff pulse aquí.
Secis No hemos encontrado.
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.
Fig.1. Alineamiento global de la selenoproteína 15-kDa

Tabla 1. Anotación genómica de la selenoproteína 15-kDa.
DI1
En este caso el output del programa encontró tres hits que alineaban con la Sec y decidimos quedarnos con aquel contig que tenía un e-value menor para predecir exones mediante Exonerate.
1 dna:chromosome chromosome:CHIMP1A:1:52330546:52345697:1
Exonerate sólo fue capaz de predecir tres exones en esta región genómica por lo que decidimos ampliar la región genómica para ver si era capaz de predecir toda la selenoproteína. En este caso el Exonerate fué capaz de predecir toda la selenoproteína.
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis
Fig.2.1. Estructura del elemento Secis de la selenoproteína DI1
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es casi idéntico y además las dos Sec alinean perfectamente.
Fig.2.2. Alineamiento global de la selenoproteína DI1

Tabla 2. Anotación genómica de la selenoproteína DI1.
DI2
El output del programa encontró tres hits que alineaban con la Sec. Decidimos quedarnos con el contig del cromosoma 14 puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
14 dna:chromosome chromosome:CHIMP1A:14:79802385:79839743:1
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína ya que con el Exonerate fuimos capaces de encontrar todos los exones para esta región.
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis
Fig.3.1. Estructura del elemento Secis de la selenoproteína DI2
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es casi idéntico y además las dos Sec alinean perfectamente.
Fig.3.2. Alineamiento global de la selenoproteína DI2

Tabla 3. Anotación genómica de la selenoproteína DI2.
DI3
El output del programa encontró tres hits que alineaban con la Sec. Decidimos quedarnos con el contig del cromosoma 14 puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
14 dna:chromosome chromosome:CHIMP1A:14:101706382:101716454:1
Exonerate en este caso el tBLASTn predijo la misma región que exonerate (a partir del aminoácido 80). A pesar de haber utilizado métodos como el BLAT no fuimos capaces de predecir el extremo 3' de la selenoproteína. El Exonerate no fue capaz de predecir ningún intrón.
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis
Fig.4.1. Estructura del elemento Secis de la Selenoproteína DI3
ClustalW mediante clustalw se puede ver visualmente como los primeros 80 aminoácidos no encajan, aunque la Sec sí.
Fig.4.2. Alineamiento global de la selenoproteína DI3

Tabla 4. Anotación genómica de la selenoproteína DI3.
Exonerate en este caso el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína ya que con el Exonerate fuimos capaces de encontrar los 2 exones que contiene la selenoproteína. Exonerate, en este caso, ha aportado información que no deducía tBLASTn.
Fig.5.1. Estructura del elemento Secis de la selenoproteína GPx1. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.
Fig.5.2. Alineamiento global de la selenoproteína GPx1. Tabla 5. Anotación genómica de la selenoproteína GPx1.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína ya que con el Exonerate fuimos capaces de encontrar los dos exones de la selenoproteína. Exonerate, en este caso, ha aportado información que no deducía tBLASTn.
Fig.6.1. Estructura del elemento Secis de la selenoproteína GPx2. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es prácticamente idéntico y además las dos Sec alinean perfectamente.
Fig.6.2. Alineamiento global de la selenoproteína GPx2. Tabla 6. Anotación genómica de la selenoproteína GPx2.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína ya que con el Exonerate fuimos capaces de encontrar los cinco exones de la selenoproteína. Además Exonerate ha aportado información que no deducía tBLASTn.
Fig.7.1. Estructura del elemento Secis de la selenoproteína GPx3. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.
Fig.7.2. Alineamiento global de la selenoproteína GPx3. Tabla 7. Anotación genómica de la selenoproteína GPx3.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína ya que con el Exonerate fuimos capaces de encontrar los siete exones de la selenoproteína.
Fig.8.1. Estructura del elemento Secis de la selenoproteína GPx4. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.
Fig.8. Alineamiento global de la selenoproteína GPx4. Tabla 8. Anotación genómica de la selenoproteína GPx4.
Exonerate hace dos predicciones de genes pero sólo una de ellas (la segunda) tiene todos los exones de la selenoproteína.
Fig.9. Alineamiento global de la selenoproteína GPx6. Tabla 9. Anotación genómica de la selenoproteína GPx6.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína, pero no contiene el mismo número de exones que tiene la selenoproteína humana.
Fig.10.1. Estructura del elemento Secis de la selenoproteína SelH. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es prácticamente idéntico y además ambas Sec alinean perfectamente.
Fig.10.2. Alineamiento global de la selenoproteína SelH. Tabla 10. Anotación genómica de la selenoproteína SelH.
Exonerate la predicción de Exonerate era muy corta y por tanto poco creible. Entonces miramos el resultado del tBLASTn para buscar una mejor predicción aunque no tuviera la alineación de la selenocisteína. El tBLASTn alineaba en diferentes regiones del mismo cromosoma así ampliamos la región problema. Aun así no encontramos todos los exones de la selenoproteína por lo que decidimos hacer un BLAT UCSC Browser con la región que no alineaba. Esta región si que contenía todos los exones aunque la anotación era diferente a la del Ensembl ya que situaba a la proteína en el cromosoma 12 en vez de en el cromosoma 2A.
Fig.11.1. Estructura del elemento Secis de la selenoproteína SelI. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.
Fig.11.2. Alineamiento global de la selenoproteína SelI. Tabla 11. Anotación genómica de la selenoproteína SelI.
Exonerate con la región del cromosoma predicha pudimos predecir toda la región que codificaba para la selenoprotéina a pesar de que presentara un menor número de exones que la selenoproteína humana.
Fig.12.1. Estructura del elemento secis de la selenoproteína SelK ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.
Fig.12.2. Alineamiento global de la selenoproteína SelK Tabla 12. Anotación genómica de la selenoproteína SelK.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína, sin embargo Exonerate es capaz de predecir una pequeña región que no predecía tBLASTn.
Fig.13. Alineamiento global de la selenoproteína SelM. Tabla 13. Anotación genómica de la selenoproteína SelM.
Exonerate el tBLASTn predijo una posible región dónde se podría encontrar la selenoproteína, pero a la hora de predecir los exones vemos como una pequeña región queda sin predecir. Y a pesar de intentar métodos comentados anteriormente no conseguimos anotar esa región. Aún así, Exonerate consiguió predecir 11 exones de los 12 que tiene la selenoproteína humana.
Fig.14.1.Estructura del elemento Secis de la selenoproteína SelN. ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es prácticamente idéntico y la Sec alinea perfectamente.
Fig.14.2. Alineamiento global de la selenoproteína SelN. Tabla 14. Anotación genómica de la selenoproteína SelN.
Exonerate al utilizar el Exonerate a pesar de encontrar dos posibles genes, no fue capaz de predecir la última región de la posible selenoproteína, por lo que decidimos intentar encontrarlo mediante un BLAT, que en esta ocasión no funcionó. El resultado no es muy bueno ya que además de no encontrar la Sec la región predicha para ser la posible selenoproteína de Pan troglodytes tiene un codón STOP en la posición 408.
Fig.15. Alineamiento global de la selenoproteína SelO. Tabla 15. Anotación genómica de la selenoproteína SelO.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína ya que con el Exonerate fuimos capaces de encontrar los cuatro exones en esta región. Para que la predicción fuera correcta, era necesario omitir algunos nucleótidos (#).
Fig.16.1. Estructura del elemento Secis de la selenoproteína SelP ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es casi idéntico y además las 10 Sec alinean perfectamente, nueve con Sec y una con Cys.
Fig.16.2. Alineamiento global de la selenoproteína SelP Tabla 16. Anotación genómica de la selenoproteína SelP.
Exonerate el tBLASTn predijo la región correcta dónde se encontraba la posible selenoproteína al completo aunque carecía de exones y para que la predicción fuera correcta, era necesario omitir algunos nucleótidos (#).
Fig.17. Alineamiento global de la selenoproteína SelR Tabla 17. Anotación genómica de la selenoproteína SelR.
Exonerate el tBLASTn predijo prácticamente la misma región predicha por el Exonerate. El Exonerate fue capaz de predecir los 6 exones que tiene la selenoproteína humana, pero para que la predicción fuera correcta, era necesario omitir un nucleótido de la secuencia de Pan troglodytes (#).
Fig.18.1. Estructura del elemento Secis de la selenoproteína SelS ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es casi idéntico encajando incluso la Sec.
Fig.18.2. Alineamiento global de la selenoproteína SelS Tabla 18. Anotación genómica de la selenoproteína SelS.
Exonerate para este cromosoma el Exonerate predecía todo el gen pero esta predicción carecía de intrones por lo que decicimos ampliar la búsqueda en el siguiente contig con valor más alto, el contig del cromosoma 3. Ahora el Exonerate predecía 4 de los 6 exones que tiene la selenoproteína humana pero faltaba por predecir el extremo 3' de ésta (30 aminoácidos). Para intentar buscarlos ampliamos el contig obtenido mediante Ensembl e hicimos un BLAT, pero ninguno de los dos métodos funcionó. Finalmente decidimos quedarnos con la predicción del contig del cromosoma 3 que predecía intrones pero carecía del extremo 3'.
Fig.19.1.Estructura del elemento Secis de la selenoproteína SelT ClustalW mediante clustalw se puede ver visualmente como el extremo 3' carece de alineamiento, sin embargo tras le Sec, que alinea correctamente, se puede ver conservación.
Fig.19.2. Alineamiento global de la selenoproteína SelT Tabla 19. Anotación genómica de la selenoproteína SelT.
GeneWise en este caso en especial al utilizar Exonerate para predecir los exones, nos quedaba de tal forma que la Sec alineaba con una Pro por lo que por consejo de nuestro tutor utilizamos GeneWise como programa para predecir exones. El GeneWise si que mostraba el alineamiento de la Sec y era capaz de predecir 5 de los 6 exones de la selenoproteína humana.
Fig.20.1. Estructura del elemento Secis de la selenoproteína SelV ClustalW mediante clustalw se puede ver visualmente como el extremo 3' carece de alineamiento, sin embargo tras le Sec, que alinea correctamente, se puede ver conservación.
Fig.20.2. Alineamiento global de la selenoproteína SelV Tabla 20. Anotación genómica de la selenoproteína SelV.
Exonerate Exonerate fue capaz de ampliar la región predicha por el tBLASTn, pero únicamente predijo 2 exones para la posible selenoproteína, en vez de los 6 que tiene la selenoproteína humana.
Fig.21. Alineamiento global de la selenoproteína SelW Tabla 21. Anotación genómica de la selenoproteína SelW.
Exonerate el tBLASTn no fue capaz de prededir el extremo 5' de la selenoproteína pero el Exonerate si que consiguió predecirlo. El resultado es dudoso ya que no aparece ningún intrón.
Fig.22.1. Estructura del elemento Secis de la selenoproteína SPS2 ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es prácticamente idéntico encajando incluso la Sec.
Fig.22.2. Alineamiento global de la selenoproteína SPS2 Tabla 22. Anotación genómica de la selenoproteína SPS2.
Exonerate el tBLASTn predijo la posible región donde se encontraba la selenoproteína pero eso sí, ampliando el contig en 300000 pb en el extremo 3'. El Exonerate fue capaz de predecir 13 de los 15 exones que tiene la selenoproteína humana.
Fig.23.1. Estructura del elemento Secis de la selenoproteína TR1 ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es casi idéntico encajando incluso la Sec.
Fig.23.2. Alineamiento global de la selenoproteína TR1 Tabla 23. Anotación genómica de la selenoproteína TR1.
Exonerate las predicciones que ofreía para el contig del cromosoma 12 no eran demasiado buenas por lo que nos quedamos con la del cromosoma 3. La predicción del cromosoma 3 no era completa asique ampliamos la región del contig. Ahora si que era capaz de predecir los 16 exones que tiene la selenoproteína humana.
Fig.24.1. Estructura del elemento Secis de la selenoproteína TR2 ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es casi idéntico encajando incluso la Sec.
Fig.24.2. Alineamiento global de la selenoproteína TR2 Tabla 24. Anotación genómica de la selenoproteína TR2.
A pesar de haber intentado anotar la TR3 en el genoma de Pan troglodytes no ha sido posible, probablemente a la alta homología que tienen las selenoproteínas de la familia TR3.
GPx1
El output del programa encontró siete hits que alineaban con la Sec y decidimos quedarnos con el contig del cromosoma 2_random puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
2_random dna:chromosome chromosome:CHIMP1A:2_random:38844896:38848789:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

GPx2
En el caso de la GPx2 el programa también encontró siete hits que alineaban con la Sec y decidimos quedarnos con el contig del cromosoma 14 puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
14 dna:chromosome chromosome:CHIMP1A:14:64069426:64090994:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

GPx3
En el caso de GPx3 el programa encontró nueve hits que alineaban con la Sec y decidimos quedarnos con el contig del cromosoma 5 puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
5 dna:chromosome chromosome:CHIMP1A:5:157302916:157344864:1
Para visualizar la salida del exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

GPx4
En el caso de GPx4 el programa encontró siete hits que alineaban con la Sec y decidimos quedarnos con el contig del cromosoma 20_random puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
20_random dna:chromosome chromosome:CHIMP1A:20_random:285439:290276:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

GPx6
Para la GPx6 el programa encontró nueve hits que alineaban con la Sec y decidimos quedarnos con el contig del cromosoma 6 puesto que tenía un e-value menor y por tanto era el más adecuado para predecir exones mediante Exonerate.
6 dna:chromosome chromosome:CHIMP1A:6:29055823:29086017:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis no hemos encontrado.
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es prácticamente idéntico y además ambas Sec alinean perfectamente.

SelH
Para la SelH el programa encontró un único hit que alineaba con la Sec y por tanto es el que utilizamos para predecir exones mediante Exonerate.
11 dna:chromosome chromosome:CHIMP1A:11:58163732:58185099:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelI
Para la SelI el programa encontró un único hit que alineaba con la Sec y por tanto es el que utilizamos para predecir exones mediante Exonerate.
2A dna:chromosome chromosome:CHIMP1A:2A:27438271:27449311:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis
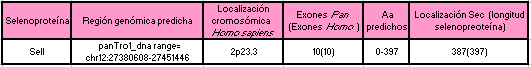
SelK
Para SelK el output del programa no encontró ningún hit que alineara con la Sec por lo que decidimos mirar el mejor alineamiento del tBLASTn aunque no apareciera la alineación de la Sec. Lo más probable es que tBLASTn no reconociera la Sec ya que ésta se encuentra en la antepenúltima posición y por tanto la reconoce como un codón STOP.
3 dna:chromosome chromosome:CHIMP1A:3:55516355:55589234:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelM
Para SelM el programa encontró un único hit que alineaba con la Sec y por tanto es el que utilizamos para predecir exones mediante Exonerate ya que además tienía un e-value considerablemente bajo.
22 dna:chromosome chromosome:CHIMP1A:22:29920327:29929990:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis No hemos encontrado.
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es del 100%.

SelN
Para SelN el programa encontró un único hit que alineaba con la Sec y por tanto es el que utilizamos para predecir exones mediante Exonerate ya que además tenía un e-value considerablemente bajo.
1 dna:chromosome chromosome:CHIMP1A:1:26490458:26509883:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelO
Para SelO el programa encontró un único hit que alineaba con la Sec pero era un fragmento muy corto y por tanto decidimos mirar el output del tBLASTn para quedarnos con un alineamiento mejor (cromosoma 22) aunque no tuviera la Sec.
22 dna:chromosome chromosome:CHIMP1A:22:49385946:49398004:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis no hemos encontrado.
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas es bastante similar pero se ve que la secuencia de Pan troglodytes está aún por terminar de secuenciar y además no somos capaces encontra la alineación de la Sec.

SelP
El output del programa encontró tres hits que alineaban la Sec. Los tres hits correspondían a la misma región genómica, ya que la SelP tiene 10 Sec y por tanto el tBLASTn es capaz de predecir la alineación para más de una Sec.
5 dna:chromosome chromosome:CHIMP1A:5:73670788:73706411:1
Para visualizar la salida del exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelR
El output del programa encontró dos hits que alineaban la Sec pero ambos pertenecín a la misma región genómica, al cromosoma 19.
19 dna:chromosome chromosome:CHIMP1A:19:41379379:41392780:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis No hemos encontrado.
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad de ambas selenoproteínas elevado pero no idéntico. Ambas Sec alinean perfectamente.

SelS
El output del programa no encontró ningún hit que alineaba la Sec, por lo que decidimos mirar el mejor alineamiento del tBLASTn aunque no tuviera la Sec dentro del alineamiento. Lo más probable es que tBLASTn no reconozca la Sec porque se encuentra en la penúltima posición y por tanto la reconoce como un codón STOP.
15 dna:chromosome chromosome:CHIMP1A:15:100806218:100823418:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelT
El output del programa encontró tres hits que alineaban la Sec. Decidimos quedarnos con aquel que tenía un e-value inferior para predecir el número de exones mediante el Exonerate.
9 dna:chromosome chromosome:CHIMP1A:9:6171787:6194306:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelV
El output del programa encontró tres hits que alineaban la Sec. Decidimos quedarnos con aquel que tenía un e-value inferior para predecir el número de exones mediante el Exonerate.
19 dna:chromosome chromosome:CHIMP1A:19:41684716:41697800:1
Para visualizar la salida del GeneWise pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

SelW
El output del programa encontró dos hits que alineaban la Sec. Decidimos quedarnos con aquel que tenía un e-value inferior para predecir el número de exones mediante el Exonerate.
1 dna:chromosome chromosome:CHIMP1A:1:31916368:31922588:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis No hemos encontrado.
ClustalW mediante clustalw se puede ver visualmente como el porcentage de identidad entre ambas es prácticamente idéntico y además ambas Sec alinean.

SPS2
El output del programa encontró dos hits que contenían la Sec y nos quedamos con aquel que presentaba un e-value menor que correspondía al cromosoma 16.
16 dna:chromosome chromosome:CHIMP1A:16:31106628:31132333:1
Para visualizar la salida del exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

TR1
El output del programa encontró dos hits que contenían la Sec pero tras intentar predecir la Selenoproteína con aquel que tenía un e-value menor vimos que el resultado no era bueno por lo que finalmente nos quedamos con aquel que tenía un e-value superior pero un porcentage de identidad también superior.
12 dna:chromosome chromosome:CHIMP1A:12:105672694:106000000:1
Para visualizar la salida del exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

TR2
El output del programa no encontró ningún hit que alineaba la Sec al igual que ocurrí con la SelK y ls SelS, por lo que decidimos mirar los dos mejores alineamientos del tBLASTn aunque no tuvieran la Sec dentro del alineamiento. Lo más probable es que tBLASTn no reconozca la Sec porque se encuentra en la penúltima posición y por tanto la reconoce como un codón STOP.
12 dna:chromosome chromosome:CHIMP1A:12:105672694:105705631:1
3 dna:chromosome chromosome:CHIMP1A:3:129670265:129679313:1
Para visualizar la salida del Exonerate pulse aquí
GFF Para visualizar la información genómica en formato gff pulse aquí
Secis

TR3
El output del programa encontró un único hit que alineaba la Sec, y era el hit que había predicho para la para la TR2.
3 dna:chromosome chromosome:CHIMP1A:3:129670265:129679313:1
María de las Heras && Amaia Mugica