El principal objectiu del treball és identificar i anotar les selenoproteïnes, les proteïnes homòlogues de cisteïna i les de maquinària presents al genoma de Taeniopygia guttata . Els resultats els hem obtingut a partir d’un anàlisi bioinformàtic i d’aproximacions computacionals a partir de la comparació de les proteïnes anotades al SelenoDB de Gallus gallus .
Les seloproteïnes en general presenten una alta homologia, d’aquesta manera ha estat possible predir la majoria d’elles a l’espècie estudiada. Al genoma de Gallus gallus hi ha 37 selenoproteïnes mentre que en Taeniopygia guttata n’hem predit 31.
Cal comentar que les selenoproteïnes de l’espècie estudiada es troben anotades al SelenoDB, però hem cregut que és preferible comparar el genoma donat amb el genoma de Gallus gallus , ja que és una espècie molt estudiada i el genoma està més ben anotat. D’aquesta manera es poden comparar els resultats obtinguts amb les proteïnes anotades al SelenoDB de l’espècie problema.
A continuació, us mostrem una taula a mode resum per il·lustrar els resultats obtinguts.
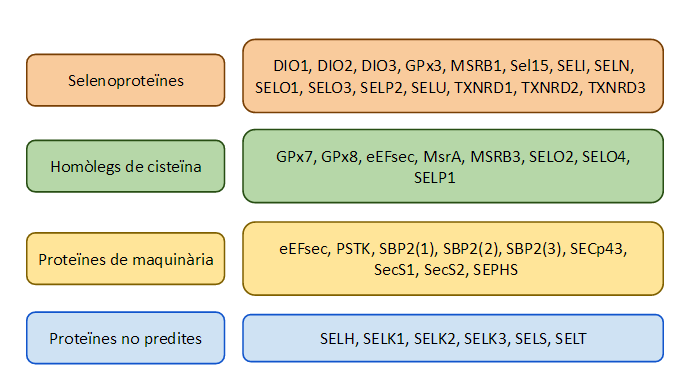
Com podem observar, hem pogut predir la majoria de selenoproteïnes del genoma de Taeniopygia guttata a partir del genoma de Gallus gallus i la majoria de les isoformes. A més a més en l’anotació del SelenoDB de l’espècie estudiada en certes ocasions apareixen més isoformes de les que hem predit en aquest treball i apareixen també algunes selenoproteïnes que nosaltres no hem predit com SelH, SelK, SelS, SelT i també apareixen més isoformes que les que hem predit com per exemple a la família de les GPx i de les DIO.
Una de les limitacions que ens hem trobat durant la realització del treball, és que el genoma de Taeniopygia guttata té anotades algunes proteïnes que no hem pogut predir perquè no estan presents al genoma de Gallus gallus .
Hem fet l’anàlisi el més acurat possible utilitzant les eines i coneixements que ens ha proporcionat l’assignatura.



