Introducción
| 1. Cebus capucinus imitator |
|---|
| 1.1 Descripción |
| 1.2 Biología y comportamiento |
| 1.3 Alimentación |
| 1.4 Conservación |
| 2. Selenoproteínas |
| 2.1 ¿Qué son? |
| 2.2 Formación de las selenoproteínas |
| 2.3 Anotación |
Cebus capucinus imitator, es un mono del nuevo mundo que se encuentra en América Central y América del Sur así como en el Sur de Argentina. También es conocido como mono capuchino o carablanca. Normalmente ocupan los bosques húmedos del Caribe y Panamá y los bosques de hoja caduca en la costa del Océano Pacífico.Cebus capucinus imitator se encuentra en el reino Animalia, fílum Chordata, clase Mammalia, orden Primates, família Cebidae, género Cebus,especie C.capucinus, subespecie C.capucinus imitator.[1]
| Reino: | Animalia |
|---|---|
| Fílum: | Chordataa |
| Clase: | Mamífero |
| Orden: | Primates |
| Familia: | Cebide |
| Género: | Cebus |
| Especie: | C. capucinus |
| Subespecie: | C. capucinus imitator |
El mono capuchino se identifica por su pelaje negro o marrón con manchas blancas en la cara, cuello y pecho. Mide de 32 a 48cm de largo desde la cabeza hasta la base de la cola y su cola es tan larga como el mismo cuerpo midiendo de 34 a 48cm. Su peso, oscila entre los 1,4 y los 3,4 kg. Otra de sus características es que presenta unos pulgares bien definidos, una cola semi-prénsil muchas veces enrrollada y pelaje por todo su cuerpo.[2]

Como la mayoría de los monos del nuevo mundo, son animales diurnos que viven en los árboles. Su comportamiento se basa en la busca de comida durante el dia y en el descanso durante la noche, durmiendo entre las ramas de los árboles.
Estos monos viven normalmente en grupos de entre 10-30 individuos en el bosque aunque fácilmente se pueden adaptar a los lugares donde viven los humanos.
Son animales teritoriales que marcan un territorio suficiente para encontrar alimentos para todo el grupo. Este marcaje lo realizan con orina y mediante la defensa contra intrusos.
El capuchino es básicamente omnívoro, principalmente se alimenta de frutas y insectos. Normalmente encuentran los alimentos en la corteza de los árboles, en las hojas, rompiendo ramas secas o utilizando piedras para romper frutos duros.
El 20% de los alimentos que comsumen son de origen animal, el 65% son frutas y el 15% vegetales.
Su pico de actividad alimentaria diaria se lleva a cabo de las 5am a las 9am y de 3pm a 5pm.

La conservación del capuchino de cara blanca se considera una precaución menor desde el punto de vista de la UICN. A pesar de ello, en ocasiones pueden verse amenazados por la caza para el mercado de mascotas y por la deforestación.
El Capuchino, se puede adaptar fácilmente a la fragmentación del bosque mejor que otras especies por su capacidad de vivir en una amplia variedad de bosques y explotar una ámplia variedad de alimentos.
(Para más información consultar Cebus capucinus imitator-Viquipèdia)
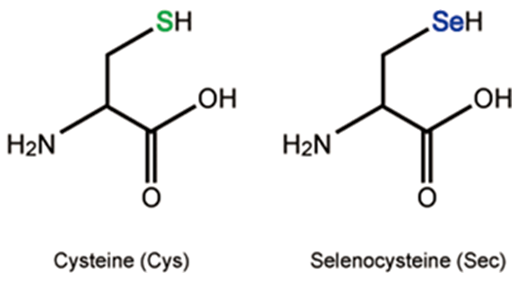
Las selenoproteínas son proteínas que contienen el aminoácido selenocisteína (Sec) en su estructura, el aminoácido número 21 en la lista de aminoácidos que conforman proteínas. Las selenocisteínas tienen una estructura parecida a las cisteínas pero con un átomo de selenio en vez de azufre.
Para incorporar este aminoácido necesitamos el codón UGA (codón que se suele interpretar como un STOP), junto con la estructura secundária SECIS (selenocystein insertion sequence) en el extremo 3’. El aminoácido de selenocisteína resultante se representa mediante una U.
Además del codón UGA y la estructura SECIS, para la formación de las selenocisteínas, es necesaria la maquinaria adecuada formada por las proteínas:
- SPS1 y SPS2 (Selenofosfat Syntase 1 and 2).
- eEFSec (Selenocysteine specific elongation factor) encargado de la incorporación de Sec a las selenoproteínas.
- SBP2 (SECIS Binding Protein 2) también encargado de la incorporación de Sec junto con EFsec.
- tRNASec: tRNA específico de selenocisteína.
- Ribosomal protein L30: proteína ribosomica implicada en la unión a los elementos SECIS.
- Sec43p: formado por SCP2 / tRNAsec / eEFSec, pero con función desconocida.
- Dried (SLA / LP):convierte el tRNASec en Sec-tRNASec.
- PSTK (Phosphoseryl tRNA kinase): fosforil·la Ser-tRNASec.
Para incorporar Sec a la secuéncia necesitamos la fosforalización de tRNASec mediante PSTK para que SPS2 sea capaz de preparar el selenio para la incorporación. Cuando de haya formado el sec-tRNAsec por la incorporación del selenio SBP2 se une a SECIS y al mismo tiempo EFsec. De esta forma, conseguimos que finalmente las proteínas de la maquinaria puedan seleccionar el tRNA donde se encuentra el codón UGA y así poder incorporar la selenocisteína correctamente.
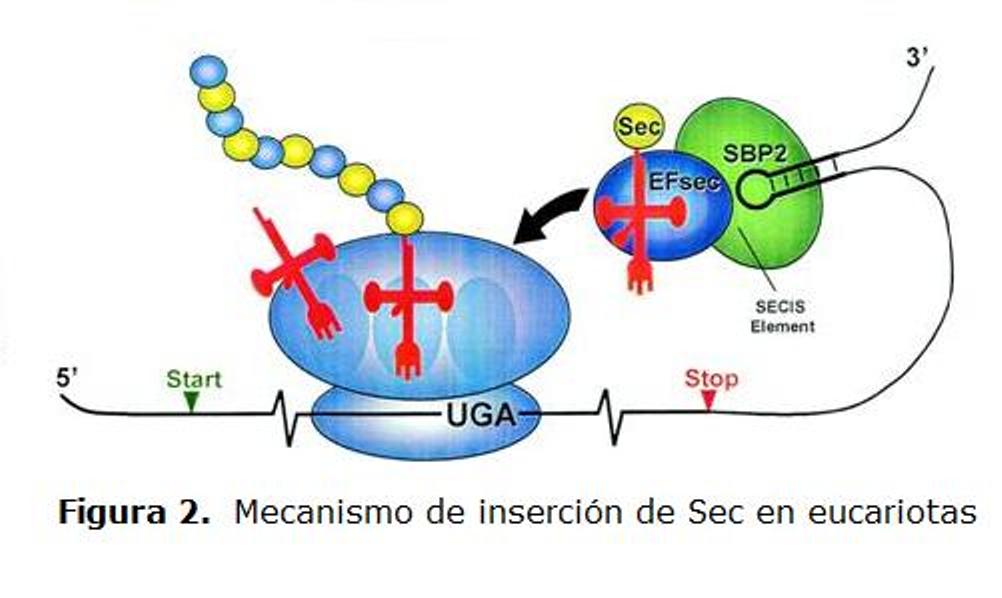
Este proceso está altamente conservado y se encuentra en numerosas espécies lo que nos hace pensar que las selenoproteínas tienen una función importante. Se conocen unas 25 selenoproteínas en mamífero pero el número de selenoproteínas varía según la especie. Además, los ortólogos de las selenoproteínas muchas veces son cisteínas.
Debido a que las selenocisteínas están codificadas por el codón UGA, muchas veces estas son mal anotadas como codones STOP. Este hecho, provocará cambios en la anotación del genoma. Por ello, es necesario compararlo con secuencias de selenoproteinas ya anotadas como por ejemplo las del genoma humano. Además de la identificación de estas regiones también es necesario encontrar los elementos SECIS en la región 3’ del gen candidato.
El problema de las anotaciones es que muchas veces estas regiones no están del todo conservadas en diferentes especies. Por suerte, la estructura tridimensional si que está conservada y nos permite una mejor comparación entre especies para proceder a la buena anotación de las selenocisteínas y con ello del genoma.
