
INTRODUCCIÓN
¿QUÉ SON LAS SELENOPROTEÍNAS?
El Selenio es un elemento poco abundante en la naturaleza, pero constituye un nutriente esencial para animales, microorganismos y otros eucariotas; de hecho se encuentra en los tres dominios de la vida: Archea, Eubacteria y Eucariota. En los organismos, forma parte del aminoácido Selenocisteína (SeCys), el cual es considerado el aminoácido 21 y se denomina con la letra U. Las proteínas que contienen este aminoácido en su secuencia son denominadas selenoproteínas. Este mecanismo se ha desarrollado para evitar que el selenio se encuentre en su forma libre, y así evitar la intoxicación por selenio. El número de selenoproteínas es diferente según el taxón. Por ejemplo, se han descrito 30 selenoproteínas en peces, 25 en humanos y 3 en Drosophila melanogaster.
La mayoría de las selenoproteínas intervienen en reacciones redox, debido a la alta reactividad del átomo de selenio situado en su centro catalítico, y por tanto tienen una posible capacidad protectora antioxidante.
El aminoácido SeCys es idéntico a una cisteína pero sustituye el radical de azufre (HS) por uno de selenio (HSe).
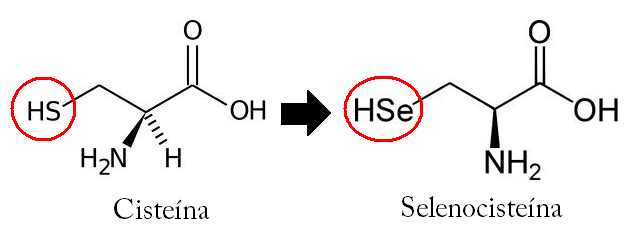
El selenio y el azufre tienen propiedades químicas similares, y por esta razón a veces se encuentran homólogos de selenoproteínas que contienen el aminoácido cisteína en el lugar del SeCys. Se ha observado que la mayoría de estos eventos ocurren en la raíz del árbol filogenético, aunque también se observa que en general las selenoproteínas están conservadas entre los vertebrados. También se han encontrado organismos que no presentan selenoproteínas, así como algunos duplicaciones ǵenicas que dan lugar a homólogos en cisteína dentro de una misma especie.
......................................................................................................................................................................................................................................................
SÍNTESIS DE SELENOPROTEÍNAS
En las selenoproteínas el aminoácido selenocisteína está codificado por el codon UGA, el cual es un codón que típicamente da lugar a un codon stop. Esto provoca que la mayoría de selenoproteínas estén mal anotadas. Durante la síntesis de las selenoproteínas el codon UGA sufre una recodificación específica a cargo del elemento SECIS (SElenoCysteine Insertion Sequence), el cual es una estructura tridimensional localizada en la región 3'UTR del mRNA de los genes que codifican para selenoproteínas en eucariotas y archea.
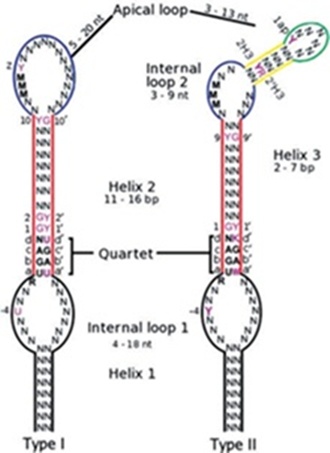
Fig 1. Secuencia consenso para el elemento SECIS de eucariota. Extraído de Chapple CE, Guigó R, Krol A. SECISaln, a web-based tool for the creation of structure-based alignments of eukaryotic SECIS elements. Bioinformatics.2009 Mar;25(5):674-5.
El elemento SECIS tiene una estructura de stem-loop que permite la incorporación del tRNA específico para selenocisteína. Esto ocurre en aproximadamente 1 de cada 1000 proteínas humanas con el codón UGA. Este elemento recluta una proteína llamada SeCis Binding Protein (SBP2), que se une y recluta un factor de elongación expecífico de selenoproteínas (EFsec). Este factor es el encargado de selecionar el tRNA específico de SeCys, determinando la incorporación de la SeCys. La síntesis de selenoproteínas, por otra parte, también requiere de una familia proteica que participa en la catálisis del tRNA específico de SeCys, que ocurre a partir del tRNA específico de serina, así como de otros factores que participan en la traducción. Por tanto, la síntesis de selenoproteínas requiere de una maquinaria compleja y específica compuesta por muchas proteínas. A pesar de esta complejidad, se han observado pocos casos de pérdida de esta familia en la historia evolutiva.
......................................................................................................................................................................................................................................................
ANOTACIÓN Y PREDICCIÓN DE SELENOPROTEÍNAS
No existe ningún método exacto para predecir la localización de selenoproteínas en un genoma, ya que la búsqueda es muy inespecífica y depende de programas de predicción de genes. La predicción de elementos SECIS depende de la búsqueda de secuencias que se puedan plegar dando lugar a la estructura 3D típica de este elemento, pero para hacer una predicción eficiente de la localización de las selenoproteínas hay que superponer la información obtenida de la predicción de elementos SECis con análisis de estructuras génicas, formas alternativas de splicing, regiones no traducidas, y pseudogenes.
......................................................................................................................................................................................................................................................
EL SELENOPROTEOMA DE VERTEBRADOS
Se han detectado selenoproteínas en humano, Drosophila melanogaster, y un gran número de protistas, pero faltan muchos genomas de vertebrados que han sido secuenciados recientemente y cuyas selenoproteínas no han sido correctamente anotadas. Se han conseguido caracterizar algunas regiones del selenoproteoma de vertebrados pero aún faltan muchos organismos por anotar. El rápido y continuo desarrollo de las tecnologías de secuenciación han hecho posible el estudio de cientos de nuevos genomas, y suponen una herramienta disponible para el análisis del selenoproteoma.
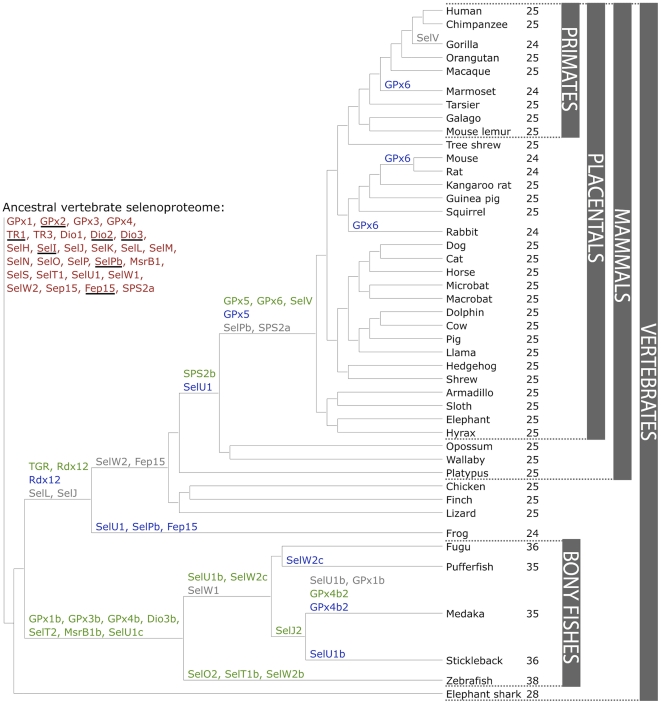
Fig 2. Evolution of the vertebrate selenoproteome. Extraído de
Mariotti M, Ridge PG, Zhang Y, Lobanov AV, Pringle TH, Guigo R, et al.Composition and evolution of the vertebrate and mammalian selenoproteomes. PLoS One. 2012;7(3):e33066.
Usando las predicciones de selenoproteínas que presentamos en este trabajo y observando el selenoproteoma de los vertebrados, se puede predecir automáticamente los puntos del árbol filogenético en los cuales se han producido duplicaciones, pérdidas o conversiones de selenoproteínas. Por tanto, las selenoproteínas también son una herramienta para estudiar procesos evolutivos. En este caso, la historia de las selenoproteínas en mamíferos añadirían nuevos puntos de vista a la especiación que ha ocurrido dentro de este grupo de organismos. En este trabajo presentamos un método para identificar las selenoproteínas de Meleagris gallopavo a partir de la secuencia genómica. Este método es reproducible y aplicable a otros genomas secuenciados recientemente, y se basa en la búsqueda de proteínas homólogas en el genoma de un organismo cercano, y en la aplicación de métodos bioinformáticos para la predicción de nuevas selenoproteínas. Presentamos también un análisis de los resultados encontrados, haciendo especial énfasis en la historia filogenética de esta familia de proteínas.
......................................................................................................................................................................................................................................................
Meleagris gallopavo
Meleagris gallopavo, o pavo, es un ave de la familia de las Phasinidae, dentro del orden Galliformes. Se trata de una especie con interés en la biología evolutiva debido a que ha estado sometida a una intensa selección artificial las últimas décadas. Su genoma ha sido secuenciado recientemente mediante técnicas de Next Generation Sequencing, y el análisis posterior ha puesto de manifiesto las diversas áreas del genoma que presentan mayor divergencia, como los genes del sistema inmune. Los próximos análisis del genoma de esta especie podrían poner de manifiesto variantes genéticas que determinen rasgos económicamente importantes, así como la influencia de la selección artificial en el genoma de los animales.
......................................................................................................................................................................................................................................................
