Les Selenoproteïnes
1. Què són les selenoproteïnes?
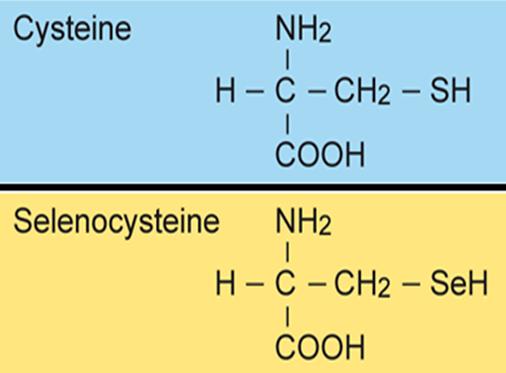
Les selenoproteïnes són proteïnes que incorporen selenocisteïna (Sec o U) en la seva seqüència i estan presents en els tres dominis de la vida (archae, eubacteria, eukarya). La seva funció no es coneix massa, però es creu que són enzims que catalitzen reaccions d’oxidoreducció i que, per tant, tenen un paper antioxidant. La selenocisteïna és l'aminoàcid 21 i és semblant a la cisteïna, de fet, l’única diferència que hi ha entre ambdós és que la selenocisteïna en lloc de tenir un àtom de sulfur en té un de seleni.
El seleni (Se) és un element traça el qual és essencial per la majoria d’éssers vius de manera que un dèficit d'aquest oligonutrient pot provocar greus malalties; cal destacar però, que un excés de seleni pot tenir efectes tòxics.
Les selenoproteïnes són la forma principal en què el seleni es presenta en els organismes, per tant, podríem dir que les selenoproteïnes són també essencials per la vida fet que es podria relacionar amb la seva possible acció com a antioxidants
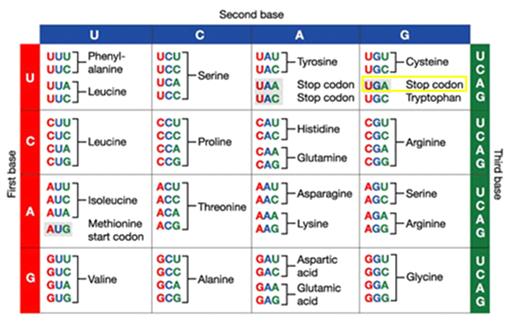
Si ens fixem en el codi genètic, veurem fàcilment que tot els codons codifiquen per un aminoàcid concret, llavors quin codó codifica per la selenocisteïna? el codó stop UGA.
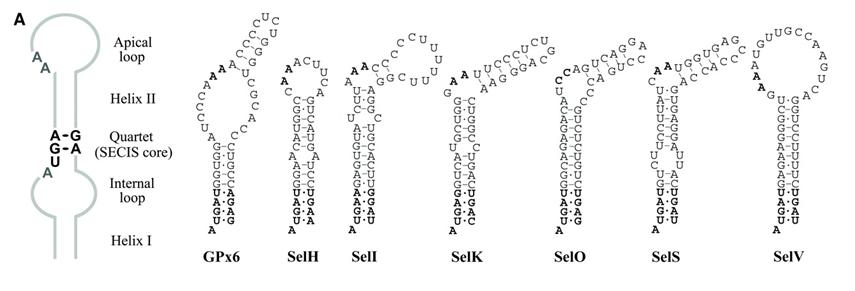
Aquest doble ús del codó UGA comporta un problema principal: com la maquinària cel·lular pot decidir quin dels 2 significats té un codó UGA? Hi ha un mecanisme que permet a la cèl·lula decidir com interpretar el codó UGA: els elements SECIS, els quals són un motiu estructural 3D situat a l’extrem 3’UTR del mRNA d’una selenoproteïna.
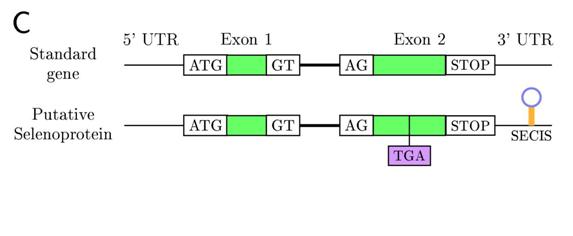
Per tant, una selenoproteïna té els següents elements indispensables:
- Codó UGA dins del open reading frame
- Element SECIS a la regió 3'UTR (en eucariotes i archae) i adjacents al codó UGA en procariotes
- Regió codificant (exons) up i down-stream del codó UGA
Hi ha una gran varietat d’elements SECIS en funció de l’estructura:
2. Síntesis de les selenoproteïnes
Per sintetitzar les selenoproteïnes necessitem molècules que s'encarreguin de la síntesis del aminoàcid com ara:
- SPS1 (Selenofosfat Sintasa 1): s'encarrega del reciclatge de la selenocisteïna i la recuperació del seleni.
- SPS2 (Selenofosfat Sintasa 2): genera la font de seleni necessari per la síntesi de la selenocisteïna, ho fa en forma de selenomonofosfat.
- SLA/LP: proteïna sintasa que participa en el pas final de la síntesi de selenocisteïna.
- SecS: sintetasa important en la síntesis del sectRNA.
També calen molècules per incorporar Sec a les selenoproteïnes:
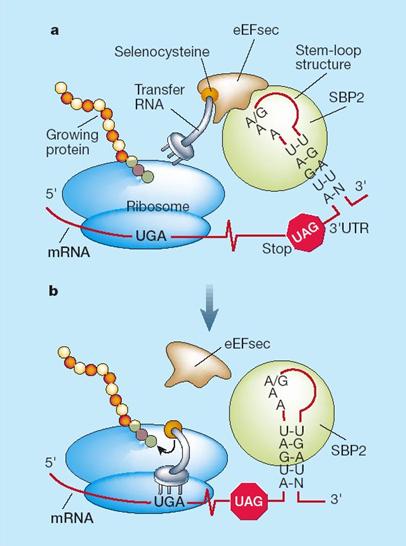
- SBP2 (SECIS Binding Protein 2): és un dels elements clau en la incorporació de la selenocisteína ja que fa de pont entre l'element SECIS i el complex EFSec-selenocisteil-tRNAsec en el ribosoma.
- EFsec: factor d’elongació específic per selenoproteïnes i que té com a funció conduir el tRNAsec amb la selenocisteïna al ribosoma. És l'homòleg al factor d'elongació EF2 d'eucariotes
- tRNAsec: tRNA específic per selenocisteïna que condueix aquest aminoàcid al ribosoma.
- PSTK: quinasa que fosforila tRNAsec.
Finalment també són necessaris els Elements SECIS.
Totes aquestes molècules estan involucrades en el procés de síntesis de selenoproteïnes.
Quan un ribosoma arriba a un codó UGA, normalment l’interpreta com un codó stop i acaba la traducció del mRNA. Ara bé, si darrere d’aquest UGA hi ha un element SECIS, aquest recluta a SBP2. Llavors, SBP2 s'uneix al factor d’elongació Efsec que apropa el tRNAsec (i la selenocisteïna) al ribosoma. En conseqüència, el ribosoma interpreta el UGA com una selenocisteïna, incorpora aquest aminoàcid i continua la traducció fins al següent codó stop que es trobi.
Tornar a l' inici
3. Identificació de les selenoproteïnes
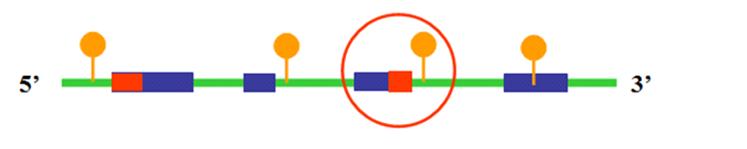
La identificació de les selenoproteïnes no és una tasca fàcil principalment degut al caràcter dual del codó UGA. Tot i aquest doble ús del codó UGA, hi ha una gran tendència a interpretar-lo com a stop de manera que en moltes bases de dades podrem trobar seqüències que acaben en punts erronis, és a dir, UGAs que han sigut interpretats com el final de la proteïna quan en realitat codifica per una selenocisteïna i la proteïna continua fins el següent stop
Per identificar nomes selenoproteïnes en un genoma concret normalment es fan dues cerques:
- Cerca d'elements SECIS amb PatScan
- Cerca de les regions codificants amb geneid
- Cerca dels TGAs
Allà on observem una regió codificant seguida d'un codó TGA i posteriorment un element SECIS serà un bon candidat a ser una nova selenoproteïna
Un cop hem identificat una selenoproteïna, ens pot interessar mirar quines espècies la tenen al seu genoma. De fet aquesta web té com a objectiu final explicar com hem realitzat aquesta tasca en les següents espècies:
A.taiwanensis, C.merolae, C.muris, C.owczarzaki, C.parvum, E.siliculosus, H.arabidopsidis, N.gruberi, P.pallidum, P.tricornutum, P.ultimum, S.parasitica, T.trahens
Sent les proteines que hem cercat: Lmsel1, Sel4, SelT
4. Famílies de selenoproteïnes estudiades
Lmsel1
Malgrat que la via de síntesis de la selenociteïna ha estat identificada en molts organismes, fins fa poc encara no s'havia identificat a Kinetoplastida Leishmania. Recentment ha aparegut una evidència consistent de la presència d'una via de síntesis de selenocisteïna en aquesta espècie ja que s'han trobat certes proves d'això. Una d'elles és el fet que s'han trobat seqüències codificants per elements SECIS en el genoma de Leishmania major i Leishmania infantum, també s'ha vist que hi ha incorporació de 75Se a les proteïnes de Leishmania, i que hi ha una presència destacada del tRNASec en aquesta espècie. A més a més, també s'han trobat altres gens importants per la síntesis de selenocisteína com ara els que codifiquen per SelB, SelD, PSTK i Secp43.
Tot això ha permès acabar descobrint la selenoproteïna Lmsel1 en Leishmania, seqüència de la qual emparada en el present projecte pot descarregar aquí.
Sel4
Plasmodium és una genere de protistes en la qual s'han descrit diverses selenoproteïnes, entre elles Sel4. Aquesta està formada per dos exons i, en aquesta i totes les selnoproteïnes de plasmodium, el codó UGA codificant per la selenocisteïna i els elements SECIS estan presents en l'últim exó tot i que la distància netr el UGA i els SECIS sí que varia
La seqüència de la selenoproteina Sel4 emparada en el present projecte pot descarregar aquí.
SelT
SelT és una selenoproteïna petita la funció de la qual no està definida, però sí que se sap que conté un motiu redox N-terminal CXXU. A diferencia de les dos selenoproteïnes anteriors, SelT està present en diverses espècies diferents entre sí, totes elles descarregables aquí.