Conclusions
El nostre objectiu en el present projecte ha estat determinar la presència o absència de 3 famílies de selenoproteïnes en un conjunt de 13 organismes protists.
Un cop analitzats els resultats obtinguts, hem determinat que cap dels organismes candidats contenia cap selenoproteïna de la família Lmsel1 ni Sel4.En canvi, sí que hem trobat bastants indicis que podrien confirmar que els genomes de P. tricornutum, S. parasitica, E. siliculosus i T. trahens contenen la selenoproteïna T. De fet, la presència d’elements SECIS en tots aquests genomes i de molts dels elements de la maquinària necessària per la síntesis de selenoproteïnes, ens reafirma la nostra hipòtesi. Per altra banda, volem destacar que en C. parvum i C. muris hem trobat homòlegs en cisteïna.
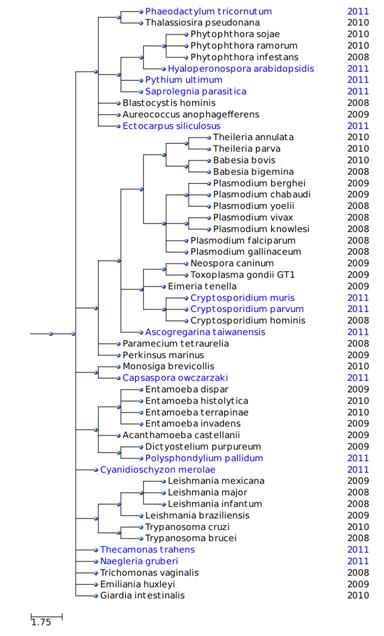
A més d’arribar a aquestes conclusions hem volgut pensar si li podríem donar una explicació evolutiva als nostres resultats, dit d’una altra manera, hem volgut saber si els resultats obtinguts concorden amb les relacions evolutives establertes en l’arbre filogenètic que mostrem i per fer-ho hem comparat els nostres resultats amb els d’anys anteriors.
En el cas de P.tricornutum, hem observat que l’espècie més propera, T.pseudonana, també presenta la selenoproteïna T al seu genoma fet que, degut a la proximitat evolutiva de ambdues espècies, consolida la fiabilitat del nostres resultats.
Per a S.parasitica, el seu veí més immediat en l’arbre és P.ultimum. Tal i com es pot veure en l’apartat de resultats, hem determinat que no conté la selenoproteïna T ja que no hem trobat cap hit significatiu en el tBLASTn. Com que hem utilitzat 6 queries, creiem que és coherent afirmar que P.ultimum podria haver perdut SelT.
El tercer organisme estudiat per a SelT és E.siliculosus el qual només hem pogut comparar amb A.anophagefferens. Aquest protist posseeix un homòleg en cisteïna, cosa que no encaixa amb el nostre resultat. Això ens portaria a pensar que l’ancestre comú entre els organismes P.tricornutum, S.parasitica i E.siliculosus tenia SelT i que qualsevol espècie pertanyent a aquest grup, com A.anophagefferens, que no presenti la selenoproteina seria degut a la seva pèrdua recent.
El quart organisme és T.trahens el qual és un dels genomes en els que hem trobat SelT. Observant l’arbre filogenètic, veiem que aquesta espècie es troba evolutivament molt a prop de l’ancestre més antic de l’arbre el qual creiem que presentava SelT degut a la relativa abundància de SelT en les diferents branques. Hem arribat a aquesta suposició perquè és molt més fàcil que l’ancestre tingués SelT i posteriorment algunes espècies l’haguessin perdut que a l’inversa.
Respecte els homòlegs en cisteïna trobats en C.muris i C.pardum, serien dos exemples de espècies que han canviat respecte l’ancestre comú. Mirant l’arbre, podem veure que és interessant mirar si el tercer element del gènere, C.cryptosporidium, també conté un homòleg en cisteïna per SelT per tal de corroborar una mica més la nostra hipòtesis. Buscant en treballs anteriors, hem vist que efectivament es tractava d’un altre homòleg en cisteïna per SelT.
Tot i que no tenim molts organismes sobre els que poder extreure conclusions fiables, hem intentat entendre també les relacions evolutives de les diverses proteïnes que conformen la maquinaria.
El primer element de maquinària analitzat ha estat eEFSec, el qual es troba present en tots els protists que tenien selenoproteïna T, excepte en C.muris. Creiem que el que ha passat és que el hit que hem utilitzat (el de millor e-value) en realitat ens ha servit per trobar el factor d’elongació 1-alfa, una proteïna que probablement comparteix un domini amb la proteïna eEFSec. Per poder afirmar això, hauríem d’haver estudiat altres hits significatius que potser ens haguessin dut al resultat esperat.
Pel que fa SPS2, només l’hem trobat en 2 protists (S.parasitica i T.trahens). Aquests dos organismes estan evolutivament força allunyats, fet que ens fa pensar que en els casos en que no hem trobat aquesta proteïna són deguts a un problema de metodologia, és a dir, com en el cas anterior, hauríem d’haver estudiat altres hits.
En el cas de SecS, l’hem observat en tots els genomes excepte en C. muris i C. parvum, dos homòlegs en cisteïna. Aquest resultat té sentit ja que SecS es troba involucrada en la síntesi del tRNAsec i per tant només aquells organismes que tinguin en el seu proteoma una selenoproteïna la necessitaran.
Per acabar, PSTK ha estat observada en P. tricornutum i S. parasítica. Considerem que aquest resultat és força coherent ja que aquests dos protists es troben propers en l’arbre filogenètic; però llavors prer aquesta mateixa raó, hauríem d’haver trobar PSTK en E. siliculosus, tot i això no ha estat així. Seguint amb el mateix raonament, la resta de genomes que expressen SelT en la branca evolutiva oposada, podrien haver perdut PSTK o haver evolucionat diferent.
Finalment ens agradaria remarcar el fet de que no hem analitzat tots els hits degut a una manca de temps, ja que anàvem desenvolupant el shell script a mesura que avançàvem en el projecte manualment. Un altre fet que ens agradaria destacar és que aquest treball ens ha fet pensar molt en la importància de la bioinformàtica en el món de la recerca, doncs cada cop es generen més dades en els experiments i és necessaria l'existència d'eines que ens permetin manipular tanta informació. En el nostre cas concret però, pensem que, per confirmar els resultats obtinguts in silico, caldria realitzar assajos in vitro perquè cal contrastar els resultats per les dues vies
.