Alba Bach (alba.bach01@campus.upf.es)
Luis Benjumea (luisfernando.benjumea01@campus.upf.es)
| INTRODUCCIÓ | ESTRUCTURA | ORTÒLEGS | EXPRESSIÓ | PROMOTORS | FUNCIÓ | CONCLUSIONS | BIBLIOGRAFIA |
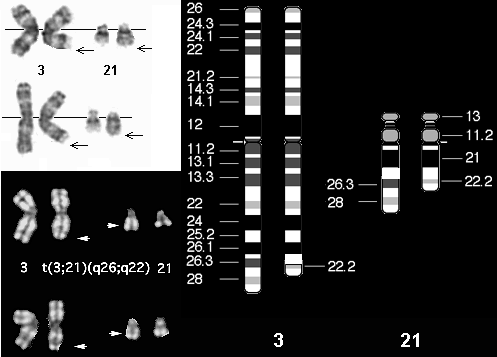
A partir de la seqüència proteica que es mostra a continuació, es va realitzar un blastp al NCBI. El resultat ens va indicar que es tractava de la proteïna de fusió AML1-EVI1. Seguidament, es va buscar la seqüència corresponent a aquests gens a ENSEMBL i UCSC. Es va veure que la seqüència donada contenia els tres primers exons del gen AML1, tota la seqüència codificant del gen EVI1, a més d'una regió UTR corresponent a l'exó 2 i una part de l'exó 3 també del gen EVI1. Sorprenentment, la seqüència entre els dos gens (113 aa), no corresponia a cap d'aquests dos. Al realitzar un blastp d'aquesta seqüència desconeguda es va descobrir una tercera proteïna anomenada MDS1. Concretament l'exó 3 de la proteïna en qüestió. A partir d'aquest punt, la proteïna de fusió va a passar a ser AML1/MDS1/EVI1 (AME).
MRIPVDASTSRRFTPPSTALSPGKMSEALPLGAPDAGAALAGKLRSGDRSMVEVLADHPGELVRTDSPNFLCSVLPTHWRCNKTLPIAF KVVALGDVPDGTLVTVMAGNDENYSAELRNATAAMKNQVARFNDLRFVGRSGRGKSFTLTITVFTNPPQVATYHRAIKITVDGPREPRN NECVYGNYPEIPLEEMPDADGVASTPSLNIQEPCSPATSSEAFTPKEGSPYKAPIYIPDDIPIPAEFELRESNMPGAGLGIWTKRKIEV GEKFGPYVGEQRSNLKDPSYGWEILDEFYNVKFCIDASQPDVGSWLKYIRFAGCYDQHNLVACQINDQIFYRVVADIAPGEELLLFMKS EDYPHETMAPDIHEERQYRCEDCDQLFESKAELADHQKFPCSTPHSAFSMVEEDFQQKLESENDLQEIHTIQECKECDQVFPDLQSLEK HMLSHTEEREYKCDQCPKAFNWKSNLIRHQMSHDSGKHYECENCAKVFTDPSNLQRHIRSQHVGARAHACPECGKTFATSSGLKQHKHI HSSVKPFICEVCHKSYTQFSNLCRHKRMHADCRTQIKCKDCGQMFSTTSSLNKHRRFCEGKNHFAAGGFFGQGISLPGTPAMDKTSMVN MSHANPGLADYFGANRHPAGLTFPTAPGFSFSFPGLFPSGLYHRPPLIPASSPVKGLSSTEQTNKSQSPLMTHPQILPATQDILKALSK HPSVGDNKPVELQPERSSEERPFEKISDQSESSDLDDVSTPSGSDLETTSGSDLESDIESDKEKFKENGKMFKDKVSPLQNLASINNKK EYSNHSIFSPSLEEQTAVSGAVNDSIKAIASIAEKYFGSTGLVGLQDKKVGALPYPSMFPLPFFPAFSQSMYPFPDRDLRSLPLKMEPQ SPGEVKKLQKGSSESPFDLTTKRKDEKPLTPVPSKPPVTPATSQDQPLDLSMGSRSRASGTKLTEPRKNHVFGGKKGSNVESRPASDGS LQHARPTPFFMDPIYRVEKRKLTDPLEALKEKYLRPSPGFLFHPQMSAIENMAEKLESFSALKPEASELLQSVPSMFNFRAPPNALPEN LLRKGKERYTCRYCGKIFPRSANLTRHLRTHTGEQPYRCKYCDRSFSISSNLQRHVRNIHNKEKPFKCHLCDRCFGQQTNLDRHLKKHE NGNMSGTATSSPHSELESTGAILDDKEDAYFTEIRNFIGNSNHGSQSPRNVEERMNGSHFKDEKALVTSQNSDLLDDEEVEDEVLLDEE DEDNDITGKTGKEPVTSNLHEGNPEDDYEETSALEMSCKTSPVRYKEEEYKSGLSALDHIRHFTDSLKMRKMEDNQYSEAELSSFSTSH VPEELKQPLHRKSKSQAYAMMLSLSDKESLHSTSHSSSNVWHSMARAAAESSAIQSISHV
SEQÜÈNCIA PROTEICA: proteína de fusió AME.
FIGURA 1. Representació de la proteïna de fusió: En aquesta imatge hi ha representada la unió dels tres gens involucrats en la translocació. S'hi pot veure els diferents exons que conformaran la proteïna de fusió, així com, les diferents regions que s'uneixen i els nucleótids dels extrems exonals. Si mireu atentament es veu la corresponència dels colors de la seqüència proteica amb als colors del dibuix. També es troben diferenciats les parts no codificants d'EVI1 amb diverses entonacions de verd. El codó que codifica per la N marcada de vermell es forma a partir de l'útim nucleòtid de l'exó 3 d'AML1 i els dos primers nucleòtid de l'exó 3 de MDS1.
FIGURA 2. Esquema del procés de fusió resultant de la translocació cromosòmica. En primer lloc podem veure com, per splicing alternatiu, el fragment resultant de la fusió entre EVI1 i MDS1 provoca l'aparició d'un nou domini PR. Aquesta proteïna (MDS1/EVI1) es pot traduïr de forma natural. De manera que quan es produeix la translocació, la meitat N-terminal d'AML1 amb un domini runt s'afegeix a aquesta estructura.
| RUNX1/AML1 | MDS1 | EVI1 |
| Nom oficial | Runt - related transcription factor 1 (acute myeloid leukemia 1; aml1 oncogene) | Altres noms | AML1; CBFA2; AMLCR1; PEBP2aB; AML1-EVI-1 |
| Localització | Cromosoma 21 (21q22.3) |
| Resum | El CBF (core binding domain) és un factor de transcripció que s'uneix a l'element central de molts enhancers i promotors. La proteïna codificada per aquest gen representa la subunitat alfa de CBF i es pensa que està involucrada en el desenvolupament normal de la hematopoesis. Les translocacions cromosòmiques que afecten a aquest gen estan associades a diferents tipus de leucèmia. | Transcrits | Segons la base de dades UCSC el gen té 2 transcrits que codifiquen per diferents isoformes (isoforma a i isoforma b). Per altra banda, segons la base de dades Ensembl el gen té 5 transcrits que també codifiquen per diferents isoformes. Un dels transcrits de UCSC és igual a un dels transcrits de Ensembl. Concretament, aquest transcrit es correspon a la variant més llarga. De manera que a partir d'aquest hem comparat la resta de transcrits. CCDS13639.1 |
| Transcrit ID | Número d'exons | Llargada del transcrit (bps) | Llargada del transcrit (aa) |
| NP_001745/ENST00000300305 | 8 | 6.215 bps | 480 aa |
| NP_001001890 | 6 | 7.267 bps | 453 aa |
| ENST00000325074 | 7 | 5.734 bps | 468 aa |
| ENST00000342083 | 4 | 2.145 bps | 188 aa |
| ENST00000344691 | 8 | 2.997 bps | 472 aa |
| ENST00000358356 | 5 | 2.722 bps | 250 aa |
Taula 1. Característiques de cada un dels transcrits pel gen RUNX1. Si cliqueu en el seu ID podreu veure detalladament l'estructura proteica i nucleotídica del transcrit.
Clustalw alignment transcrits AML1: Clustalw de les proteïnes obtingudes a partir dels diferents transcrits. Si obriu el fitxer podreu observar que hi ha una part comuna al mig que es repeteix a tots els transcrits. Mentre que el principi i el final varien d'un transcrit a l'altre.
| Nom oficial | Myelodysplasia syndrome 1 | Altres noms | PRDM3; MDS1-EVI1 |
| Localització | Cromosoma 3 (3q26.2) |
| Resum | La proteïna codificada pel gen MDS1 és un factor de transcripció de funció encara desconeguda. S'ha vist que s'expressa com un únic transcrit o com un transcrit unit al gen EVI1. | Transcrits | Segons la base de dades de l'ENSEMBL el gen MDS1 presenta un transcrit diferent del transcrit que descriu VEGA. Només mencionem el transcrit de VEGA ja que és el que ens permet explicar millor la proteïna de fusió. |
| Transcrit ID | Número d'exons | Llargada del transcrit (bps) | Llargada del transcrit (aa) |
| OTTHUMT00000267016 | 5 | 1.285 bps | 169 aa |
Taula 2. Característiques del transcrit pel gen MDS1. Si cliqueu en el seu ID podreu veure detalladament l'estructura proteica i nucleotídica del transcrit.
| Nom oficial | Ecotropic viral integration site 1 | Altres noms | PRDM3; MDS1-EVI1; AML1-EVI-1; MGC163392 |
| Localització | Cromosoma 3 (3q26.2) |
| Resum | El gen EVI1 s'ha vist que s'expressa com un únic transcrit o com un transcrit unit al gen MDS1. MDS1 és un gen petit, que es troba 300-400kb upstream del primer exó d'EVI1. La fusió de MDS1-EVI1 es produeix per un splicing entre l'exó dos de MDS1 cap a l'exó 3 de EVI1. EVI1 té 1051 aminoàcids i és una proteïna d'unió al DNA de 145kDa. Presenta dos dominis amb set i tres sets de repeticions del motiu zinc finger, un domini de repressió entre els dos sets de zinc finger, i un domini acídic a la regió C-terminal. La tranducció de MDS1-EVI1 afageix 188 aminoàcids upstream de l'inici de traducció del tercer exó. 63 aa deriven de la regió no codificant de l'exó 2 i part de la l'exó 3 d'EVI1, i els 125 aa restans provenen del gen MDS1. | Transcrits | Segons la base de dades de ENSEMBL i SWISS-PROT el gen EVI1 presenta dues isoformes o transcrits diferents produits per splicing alternatiu. La primera isoforma codifica per la variant més llarga mentre que la segona isoforma codifica per una variant petita. CCDS3205.1 |
| Transcrit ID | Número d'exons | Llargada del transcrit (bps) | Llargada del transcrit (aa) |
| ENST00000264674 | 16 | 3.572 bps | 1.051 aa |
| ENST00000343372 | 15 | 5.722 bps | 166 aa |
Taula 3. Característiques de cada un dels transcrits pel gen EVI1. Si cliqueu en el seu ID podreu veure detalladament l'estructura proteica i nucleotídica del transcrit.
Desprès de caracteritzar l'estructura genòmica es va realitzar un estudi de l'homologia de cada un dels gens en altres espècies. A partir de la base de dades de l'ENSEMBL, es va obtenir la seqüència proteica i nucleotídica de cada un dels ortòlegs així com el seu % d'homologia. En el cas del gen EVI1, la base de dades de l'ENSEMBL no mostrava cap gen ortòleg. Per això es van buscar a la base de dades de l'UCSC on vam obtenir resultats positius. Una vegada recopilats tots els transcrits, es va realitzar la construcció dels arbres filogenètics per tenir una imatge mès visual de l'homologia de cada gen entre les diferents espècies. Per dur a terme aquestes construccions es van fer servir diverses eines informàtiques com Clustalw, Bioedit i Mega.
| RUNX1/AML1 | MDS1 | EVI1 |
Ortòlegs del gen RUNX1
| ESPÈCIE | GENEID | % HOMOLOGIA |
 Aedes aegypti |
AAEL006160 | 52 |
 Anopheles gambiae |
ENSANGG00000019555 | 71 |
 Bos taurus |
ENSBTAG00000004742 | 98 |
 Caenorhabditis elegans |
B0414.2 | 29 |
 Canis familiaris |
ENSCAFG00000009596 | 97 |
 Cavia porcellus |
ENSCPOG00000002684 | 94 |
 Ciona intestinalis |
ENSCING00000002253 | 27 |
 Ciona savignyi |
ENSCSAVG00000004072 | 64 |
 Dasypus novemcinctus |
ENSDNOG00000015041 | 46 |
 Drosophila melanogaster |
CG15455 | 35 |
 Echinops telfairi |
ENSETEG00000011313 | 86 |
 Felis catus |
ENSFCAG00000006761 | 89 |
 Gallus gallus |
ENSGALG00000016022 | 77 |
 Gasterosteus aculeatus |
ENSGACG00000015276 | 48 |
 Loxodonta africana |
ENSLAFG00000004290 | 74 |
 Macaca mulatta |
ENSMMUG00000001649 | 99 |
 Monodelphis domestica |
ENSMODG00000021051 | 90 |
 Mus musculus |
ENSMUSG00000022952 | 95 |
 Ornithorhynchus anatinus |
ENSOANG00000011162 | 65 |
 Oryctolagus cuniculus |
ENSOCUG00000013731 | 98 |
 Oryzias latipes |
ENSORLG00000020699 | 56 |
 Pan troglodytes |
ENSPTRG00000013883 | 100 |
 Rattus norvegicus |
ENSRNOG00000001704 | 94 |
 Takifugu rubripes |
SINFRUG00000149207 | 55 |
 Tetraodon nigroviridis |
GSTENG00034595001 | 80 |
 Tupaia belangeri |
ENSTBEG00000000192 | 77 |
 Xenopus tropicalis |
ENSXETG00000014140 | 84 |
Taula 4. Llista de les diferents espècies que presenten ortòlegs pel gen RUNX1 humà. Per cada un d'aquests ortòlegs s'indica el seu transcrit ID, amb un enllaç a la pàgina d'on s'han obtingut i el percentatge d'homologia.
Arbre filogenètic dels ortòlegs del gen RUNX1
Arbre 1. Representació filogenètica dels ortòlegs de RUNX1. Blau: mamífers. Lila: aus i amfibis. Verd: peixos. Blau fosc i marró: insectes. Blau cel: nemàtodes. Vermell: ascidies (cordats).
El gen RUNX1 humà que codifica per un factor de transcripció amb un domini runt presenta una gran varietat de gens ortòlegs en la majoria d'espècies del regne animal. Els gens ortòlegs amb una homologia més elevada corresponen evidentment a mamífers, com Pan troglodytes (100%), Macaca mulatta (99%) i Mus musculus (95%) entre d'altres. Per altra banda, l'homologia es prou elevada en aus i peixos, i mitjana-baixa en invertebrats. Respecte a insectes, s'ha comprovat que presenta homologia amb el gen pair-rule de Drosophila. Segons tots els resultats observats, es pot afirmar que es un gen força conservat al llarg de l'evolució, fet que indica que té un paper molt rellevant en el funcionament cel·lular.
Ortòlegs del gen MDS1
| ESPÈCIE | GENEID | % HOMOLOGIA |
 Bos taurus |
ENSBTAG00000020168 | 93 |
 Canis familiaris |
ENSCAFG00000024240 | 90 |
 Cavia porcellus |
ENSCPOG00000012256 | 91 |
 Dasypus novemcinctus |
ENSDNOG00000017489 | 92 |
 Erinaceus europaeus |
ENSEEUG00000010532 | 89 |
 Felis catus |
ENSFCAG00000004548 | 95 |
 Gasterosteus aculeatus |
ENSGACG00000007274 | 34 |
 Loxodonta africana |
ENSLAFG00000010308 | 93 |
 Macaca mulatta |
ENSMMUG00000031567 | 99 |
 Monodelphis domestica |
ENSMODG00000024658 | 77 |
 Mus musculus |
ENSMUSG00000051636 | 87 |
 Ornithorhynchus anatinus |
ENSOANG00000007584 | 71 |
 Rattus norvegicus |
ENSRNOG00000011640 | 82 |
 Tetraodon nigroviridis |
GSTENG00003821001 | 22 |
 Tupaia belangeri |
ENSTBEG00000009672 | 93 |
 Xenopus tropicalis |
ENSXETG00000022835 | 46 |
Taula 5. Llista de les diferents espècies que presenten ortòlegs pel gen MDS1 humà. Per cada un d'aquests ortòlegs s'indica el seu transcrit ID, amb un enllaç a la pàgina d'on s'han obtingut i el percentatge d'homologia.
Arbre filogenètic dels ortòlegs del gen MDS1
Arbre 2. Representació filogenètica dels ortòlegs de MDS1. Blau i lila: mamífers. Blau fosc: amfibis. Verd i marrò: peixos.
El gen MDS1 humà no presenta tants gens ortòlegs com RUNX1, però en presenta a la majoria d'espècies del regne animal, menys en invertebrats. Els gens ortòlegs amb una homologia més elevada corresponen evidentment a mamífers, com Macaca mulatta (99%), Felis catus (95%) i Mus musculus (87%), entre d'altres. Per altra banda, l'homologia es mitjana-baixa en peixos i amfibis. Segons els resultats observats i la informació obtinguda, el gen MDS1 humà no té una funció clara però tot i així ha de ser important en alguna funció cel·lular perquè s'hagi mantingut al llarg de l'evolució.
Ortòlegs del gen EVI1
| ESPÈCIE | GENEID | % HOMOLOGIA |
 Mus musculus |
AJ001482 | 92 |
 Xenopus laevis |
AY939893 | 78 |
 Sus scrofa |
AK232131 | 93 |
Taula 6. Llista de les diferents espècies que presenten ortòlegs pel gen EVI1 humà. Per cada un d'aquests ortòlegs s'indica el seu transcrit ID, amb un enllaç a la pàgina d'on s'han obtingut i el percentatge d'homologia.
El gen EVI1 humà gairebé no presenta ortòlegs. Fet evident tenint en compte que es tracta d'un lloc d'integració ecotròpica viral. Nomès es van obtenir tres ortòlegs pel gen EVI1 a partir de la base de dades de l'UCSC, Mus musculus (92%), Xenopus laevis (78%) i Sus scrofa (93%). En diversos estudis es destaca l'elevat grau d'homologia entre els ortòlegs d'humà i ratolí que mantenen conservats els dos dominis zinc-finger d'aquesta proteïna. Per altre banda, en la base de dades HomoloGene (Discover Homologs) de l'NCBI es van trobar possibles gens ortòlegs d'EVI1 en Canis familiaris, Ratus norvegicus, Drosophila melanogaster i Anopheles gambiae. Curiosament també s'ha trobat en aquesta base de dades un gen homòleg similar a la proteina de fusió AML1-EVI1 en Gallus gallus (LOC424997) i Pan troglodytes (LOC460837). En el cas de Gallus gallus presenta una similaritat del 84% a nivell nucleotídic i del 89% a nivell d'aminoàcids.
La caracterització de l'expressió dels diferents gens es va determinar a partir dels microarrays presents en la base de dades de l'UCSC. Aquesta informació es va comparar amb la referida a diversos articles i fonts digitals com Swiss-Prot. També es va contrastar tota la informació amb les imatges disponibes del VisiGene.
El gen RUNX1 presenta una expressió en quasibé tots els teixits excepte al cervell. Els seus nivells més elevats es troben en timus, en medul·la òssia i en sang perifèrica. Una de les principals funcions d'AML1 és el desenvolupament de cèl·lules hematopoètiques, de manera que és normal que es trobi tant expressat en aquest teixits com també en malalties de la sang (leucèmies).
Pel que fa al gen MDS1, els resultats representats en els chips de microarrays indiquen una alta expressió en el sistema nerviòs, que és comuna en els tres tipus de chips. Però aquests resultats són poc concluents ja que no es disposen de cites bibliogràfiques que esmentin la comprovació d'aquesta expressió.
Taula 7. GNF Expression Atlas 2 Data from U133A and GNF1H Chips
Taula 8. Normal Human Tissue cDNA Microarrays
Taula 9. GNF Expression Atlas 1 Human Data on Affy U95 Chips
Per caracteritzar la regió promotora de cada un dels gens estudiats, es va fer un PROGRAMA PERL basat en la identificació de possibles factors de transcripció a partir d'una seqüència promotora. Aquest programa llegeix un fitxer amb matrius de factors de transcripció (FTs) i un altre fitxer amb una seqüència d'una regió promotora en format FASTA. Posteriorment, mostrarà la llista dels FTs proporcionats amb un p-value per cada FT que ens indicarà la probabilitat de rebutjar erròniament la hipòtesi inicial de que el factor no s'uneix. A partir de les dades obtingudes, se seleccionarà un conjunt de factors de transcripció amb una alta probabilitat d'actuar sobre la seqüència promotora. Per contrastar i ampliar els resultats, es va fer sevir el pograma PROMO per la detecció de factors de transcripció regulables coneguts (filtres: FTs humans i llocs d'unió a FTs humans). Un cop recopilada tota la informació s'han realitzat diverses taules per les regions promotores de cada un dels diferents gens.
| RUNX1/AML1 | EVI1 | MDS1 |
FTs de les seqüències promotores del gen AML1
El gen AML1, com s'ha vist a l'apartat de l'estrucutra genòmica, presenta diversos transcrits. Conseqüentment hi ha 3 regions promotores diferents per les quals s'ha fet l'estudi per separat.
| PROMOTOR 1 AML1 (PROGRAMA) | ||||||
| FT | S* | SEQUENCIA | POSICIÓ | P-VALUE | ||
| AP-1 | 3,9515 | TGAGTCA | 784-790 | 0 | 0 | 0 |
| RXR-α | 3,1437 | TGAACC | 1062-1067 | 0 | 0 | 0 |
Taula 10. Resultats del programa perl. Es mostren els FTs més significatius amb una S* elevada i un p-value inferior a 0,05.
| PROMOTOR 1 AML1 (PROMO) | |||||||
| FT | DISSIMILARITAT | SEQUENCIA | POSICIÓ | RE equally | RE query | ||
| AP-1 | 0,3489 | ACTGAGTCA | 781-789 | 0,05035 | 0,05698 | ||
| RXR-α | 3,3959 | TGAACCCA | 1061-1068 | 0,06714 | 0,07157 | ||
Taula 11. Resultats del PROMO pels FTs identificats amb el nostre programa.
| POSSIBLES FACTORS DE TRANSCRIPCIÓ REGIÓ PROMOTORA 1 D'AML1 (PROMO) | |||||||
| FT | APARICIONS | FT | APARICIONS | FT | APARICIONS | FT | APARICIONS |
| AP-1 | 1 | AR | 1 | c-Ets-2 | 2 | c-Jun | 2 |
| Elk-1 | 1 | GR | 1 | HNF-3α | 2 | HOXD10 | 2 |
| HOXD9 | 2 | IRF-1 | 3 | LEF-1 | 1 | MAZ | 1 |
| MITF | 1 | NF-1 | 1 | NF-AT1 | 2 | NF-AT2 | 1 |
| NF-Y | 1 | PEA3 | 1 | PR A | 1 | PR B | 1 |
| PXR-1:RXR-α | 2 | RAR-β | 1 | SRY | 1 | TBP | 1 |
| TCF-4E | 1 | WT1 I | 13 | WT1 I-KTS | 13 | WT1 I-del2 | 13 |
Taula 12. Resultats del PROMO. S'indica els FTs i el nombre d'aparicions de cada un. Per descartar FTs poc probables, només s'han considerat com significatius aquells que presentaven una dissimilaritat inferior a 5 i un RE equally inferior a 0,1.
Els resultats obtinguts en el nostre programa coincideixen amb els resultats obtinguts en el PROMO. Els FTs AP-1 i RXR-α són FTs amb un alt potencial d'actuar sobre la seqüència promotora 1 d'AML1. Com que es tracta de la seqüència promotora del trascrit principal, presenta, a part dels dos anteriors, molts d'altres possibles FTs.
| PROMOTOR 2 AML1 (PROGRAMA) | |||||||
| FT | S* | SEQUENCIA | POSICIÓ | P-VALUE | |||
| HIF-1 | 3,8872 | AAAGCACGT | 971-979 | 0,01 | 0,01 | 0,02 | |
Taula 13. Resultats del programa perl. Es mostren els FTs més significatius amb una S* elevada i un p-value inferior a 0,05.
| PROMOTOR 2 AML1 (PROMO) | |||||||
| FT | DISSIMILARITAT | SEQUENCIA | POSICIÓ | RE equally | RE query | ||
| HIF-1 | 2,0085 | AAAGCACGT | 970-978 | 0,07133 | 0,05854 | ||
Taula 14. Resultats del PROMO pels FTs identificats amb el nostre programa.
| POSSIBLES FACTORS DE TRANSCRIPCIÓ REGIÓ PROMOTORA 2 D'AML1 (PROMO) | |||||||
| FT | APARICIONS | FT | APARICIONS | FT | APARICIONS | FT | APARICIONS |
| AhR:Arnt | 1 | c-Ets-1 | 1 | c-Ets-2 | 2 | E2F-1 | 2 |
| EBF | 1 | ENKTF-1 | 1 | GCF | 5 | HIF-1 | 1 |
| NF-1 | 1 | NF-AT1 | 1 | Sp1 | 7 | WT1 | 1 |
| PROMOTOR 3 AML1 (PROGRAMA) | |||||||
| FT | S* | SEQUENCIA | POSICIÓ | P-VALUE | |||
| RXR-α | 3,1415 | TGAACT | 106-111 | 0 | 0 | 0 | |
Taula 16. Resultats del programa perl. Es mostren els FTs més significatius amb una S* elevada i un p-value inferior a 0,05.
| PROMOTOR 3 AML1 (PROMO) | |||||||
| FT | DISSIMILARITAT | SEQUENCIA | POSICIÓ | RE equally | RE query | ||
| PXR-RXR-α | 0,9417 | TGAACTCG | 105-112 | 0,06714 | 0,01945 | ||
Taula 17. Resultats del PROMO pels FTs identificats amb el nostre programa.
| POSSIBLES FACTORS DE TRANSCRIPCIÓ REGIÓ PROMOTORA 3 D'AML1 (PROMO) | |||||||
| FT | APARICIONS | FT | APARICIONS | FT | APARICIONS | FT | APARICIONS |
| AhR | 1 | AR | 1 | EBF | 1 | Elk-1 | 1 |
| GCF | 3 | MAZ | 1 | p53 | 1 | PXR-1:RXR-α | 1 |
| RXR-α | 2 | Sp1 | 7 | Sp3 | 1 | STAT1β | 1 |
Taula 18. Resultats del PROMO. S'indica els FTs i el nombre d'aparicions de cada un. Per descartar FTs poc probables, només s'han considerat com significatius aquells que presentaven una dissimilaritat inferior a 5 i un RE equally inferior a 0,1.
Els resultats obtinguts en el nostre programa coincideixen amb els resultats obtinguts en el PROMO. El FT RXR-α té un alt potencial d'actuar sobre la seüència promotora 3 d'AML1. Com es pot apreciar a la taula 18, aquesta regió promotora tampoc pertany al transcrit principal i per tant presenta també menys possibles FTs en comparació amb la seqüència promotora 1.
FTs de la seqüència promotora del gen EVI1
| PROMOTOR EVI1 (PROGRAMA) | |||||||
| FT | S* | SEQUENCIA | POSICIÓ | P-VALUE | |||
| NF-AT1 | 3,8645 | GGAAAAA | 242-248 | 0 | 0 | 0 | |
| RXR-α | 3,1415 | TGAACC | 203-208 | 0 | 0 | 0 | |
Taula 19. Resultats del programa perl. Es mostren els FTs més significatius amb una S* elevada i un p-value inferior a 0,05.
| PROMOTOR EVI1 (PROMO) | |||||||
| FT | DISSIMILARITAT | SEQUENCIA | POSICIÓ | RE equally | RE query | ||
| NF-AT1 | 4,8235 | TGGAAATCGA | 394-403 | 0,04196 | 0,04134 | ||
| RXR-α | 4.0904 | TGAACCAG | 202-209 | 0,06714 | 0,06678 | ||
Taula 20. Resultats del PROMO pels FTs identificats amb el nostre programa.
| POSSIBLES FACTORS DE TRANSCRIPCIÓ REGIÓ PROMOTORA D'EVI1 (PROMO) | |||||||
| FT | APARICIONS | FT | APARICIONS | FT | APARICIONS | FT | APARICIONS |
| AR | 2 | c-Ets-2 | 4 | ETF | 1 | FOXO4 | 1 |
| HNF-3α | 4 | HOXD10 | 1 | TCF-4E | 1 | IRF-1 | 3 |
| LEF-1 | 1 | NF-AT1 | 1 | Pax-5 | 1 | PXR-1:RXR-α | 1 |
| RAR-β | 1 | Sp1 | 1 | STAT1β | 1 | T3R-β1 | 1 |
| PROMOTOR MDS1 (PROGRAMA) | |||||||
| FT | S* | SEQUENCIA | POSICIÓ | P-VALUE | |||
| NF-AT1 | 3,4474 | GGAAAAA | 406-412 | 0 | 0 | 0 | |
| NF-kappaβ | 4,1635 | GGAATTCCC | 38-46 | 0 | 0 | 0,02 | |
Taula 22. Resultats del programa perl. Es mostren els FTs més significatius amb una S* elevada i un p-value inferior a 0,05.
| PROMOTOR MDS1 (PROMO) | |||||||
| FT | DISSIMILARITAT | SEQUENCIA | POSICIÓ | RE equally | RE query | ||
| NF-AT1 | 0,6490 | GGAAAGAAT | 344-352 | 0,01259 | 0,01422 | ||
Taula 23. Resultats del PROMO pels FTs identificats amb el nostre programa.
| POSSIBLES FACTORS DE TRANSCRIPCIÓ REGIÓ PROMOTORA D'MDS1 (PROMO) | |||||||
| FT | APARICIONS | FT | APARICIONS | FT | APARICIONS | FT | APARICIONS |
| AR | 1 | c-Ets-2 | 2 | E2F | 1 | ENKTF-1 | 1 |
| GCF | 2 | HNF-1B | 1 | HNF-1C | 1 | HOXD10 | 1 |
| HOXD9 | 1 | IRF-1 | 2 | LEF-1 | 1 | MAZ | 2 |
| NF-1 | 1 | NF-AT1 | 1 | Pax-5 | 1 | PEA3 | 2 |
| PXR-1:RXR-α | 1 | Sp1 | 2 | SRY | 1 | TCF-4 | 1 |
Taula 24. Resultats del PROMO. S'indica els FTs i el nombre d'aparicions de cada un. Per descartar FTs poc probables, només s'han considerat com significatius aquells que presentaven una dissimilaritat inferior a 5 i un RE equally inferior a 0,1.
Per últim es va caracteritzar la regió promotora del gen MDS1. En aquest cas, es va observar que un dels FTs que ens indicava el programa perl, NF-Kappaβ, no apareixia en la llista de possibles FTs del PROMO. També vam obtenir un altre FT al programa perl, NF-AT-1. Aquest últim si que estava present a la llista del PROMO, però com hem comentat anteriorment, la matriu per aquest FT es diferent als dos programes utilitzats.
Altres funcions de RUNX1:
El gen MDS1 codifica per un factor de transcripció, però la seva funció encara està per determinar. La seva importància ve donada pel fet que en la translocació, MDS1 queda situat entremig d'AML1 i EVI1 fent que acabi formant part de la proteïna de fusió.
En referència al gen EVI1, en humans no s'ha determinat la funció que tindria a les cèl·lules, però s'ha comprovat que correspon a un lloc d'integració vírica ecotròpica. Tot i així s'ha vist que en Rattus norvegicus si que tindria diverses funcions com ara la regulació de la proliferació cel·lular i la regulació positiva de la transcripció a partir del promotor de la RNA pol II. Cal dir però, que aquestes funcions es basen en prediccions.
Com ja hem dit abans, la proteïna de fusió (AME) dóna lloc a una malaltia anomenada leucèmia mieloide crònica. Aquesta proteïna actua com una oncoproteïna multi-funcional, ocasionant desordres a les cèl·lules hematopoètiques i provocant una transformació maligna en aquestes. S'han identificat 4 mecanismes on AME hi esta involucrada:
La proteïna de fusió AME originada de la translocació t(3;21)(q26;q22) està formada pels tres primers exons d'AML1 (transcrits 2,4,5 o 6), l'exó tres de MDS1, exó dos i part del tres no codificants d'EVI1, i tots els exons codificants també d'EVI1 (transcrit 1). S'ha vist que algunes espècies presenten un gen d'homologia amb la proteïna AML1-EVI1, encara que aquesta proteïna sigui tïpica d'una translocació humana. Els tres gens es transcriuen de forma reversa de manera que al produïr-se la translocació permet que aquests gens es puguin transcriure un darrera l'altre formant la proteïna de fusió. Els gens MDS1 i EVI1 quedan sotmesos sota el control de la regió promotora d'AML1. En conseqüència, l'expressió de la proteïna de fusió queda restringida als teixits on AML1 s'expressa característicament. Com s'ha vist, els teixits on s'expressa AML1 de forma abundant són medul·la òssia, sang perifèrica i timus, ja que la funció principal d'AML1 és el desenvolupament de les cèl·lules hematopoètiques. Per tant, l'acció d'AME repercutirà sobre aquests teixits, donant lloc a anomalies de la sang, com la leucèmia mieloide crònica.
1. Fears, S., Mathieu, C., Zeleznik-Le, N., Huang, S., Rowley, J.D., Nucifora, G. (1996) Intergenic splicing of MDS1 and EVI1 occurs in normal tissues as well as in myeloid leukemia and produces a new member of the PR domain family. Proc. Natl. Acad. Sci. 93, p. 1642-1647.
2. Mitani, K. (2004) Molecular mechanisms of leukemogenesis by AML1/EVI1. Oncogene 23, p. 4263-4269.
3. Cuenco, G.M., Nucifora, G., Ren, R. (2000) Human AML1/MDS1/EVI1 fusion protein induces an acute myelogenous leukemia (AML) in mice: A model for human AML PNAS. 97, p. 1760-1765.
4. Mitani, k., Ogawa, S., Tanaka, t., Miyoshi, H., Kurokawa, M., Mano, H., Yazaki, Y., Ohki, M., Hirai, H. (1994) Generation of the AML 1- EVI- 1 fusion gene in the t(3;21)(q26;q22) causes blastic crisis in chronic myelocytic leukemia. The EMBO Journal. 13, p. 504-510.
5. Tanaka, t., Mitani, K., Kurokawa, M., Ogawa, S., Tanaka, K., Nishida, J., Yazaki, Y., Shibata, Y., Hirai, H. (1995) Dual Functions of the AML1/Evi-1 Chimeric Protein in the Mechanism of Leukemogenesis in t(3;21) Leukemias. Molecular and Cellular Biology. 15, p. 2383-2392.
6. Izutsu, K., Kurokawa, M., Imai, Y., Ichikawa, M., Asai, T., Maki, K., Mitani, K., Hirai, H. (2002) The t(3;21) fusion product, AML1/Evi-1 blocks AML1-induced transactivation by recruiting CtBP. Oncogene 21, p. 2695-2703.
7. Messeguer, X., Escudero, R., Farré, D., Nuñez, O., Martínez, J., Mar Albà, M. (2002) "PROMO: detection of known transcription regulatory elements using species-tailored searches". Bioinformatics 18, p. 333-334.
8. Farré, D., Roset, R., Huerta, M., Adsuara, J.E., Roselló, L., Mar Albà, M., Messeguer, X. (2003) "Identification of patterns in biological sequences at the ALGGEN server: PROMO and MALGEN". Nucleic Acids Res, 31, p. 3651-3653.
9. www.ncbi.nlm.nih.gov
10. www.ensembl.org
11. www.genome.ucsc.edu
12. www.ebi.ac.uk/clustalw
13. www.mbio.ncsu.edu/BioEdit/bioedit
14. www.megasoftware.net
15. www1.imim.es/courses/BioinformaticaUPF