DISCUSIÓ
Per tal de predir les selenoproteïnes, les proteïnes homòlogues cisteïna i les de la maquinària del genoma de Callorhinus ursinus s’ha realitzat una comparació amb el genoma d’Homo sapiens ja que les seves selenoproteïnes estan correctament anotades.
Hem de dir que en el cas de les famílies de selenoproteïnes iodothyronine deiodinase (DI), glutathione peroxidase (GPx) i thioredoxin reductase (TR), s’ha tingut en compte la complexitat que tenen i per això s’han estudiat amb més precisió, analitzant-los detalladament. A més, encara que la meitat de les selenoproteïnes dels mamífers no tenen una funció molt coneguda, s’ha trobat que moltes tenen una funció redox, i moltes altres estan involucrats en la prevenció del càncer, la modulació de l’envelliment i en la resposta immune.
Tenint en compte això i el que s’ha vist a la literatura, moltes selenoproteïnes tenen funcions similars entre elles. Tant la selenoproteina M com la 15, la W i la T tenen un motif redox comú; CXXU on la U indica una selenocisteïna. Per altre banda, les selenoproteïnes residuals del reticle endoplasmàtic (ER) contribueixen a moltes funcions essencials com ara la regulació de l'homeòstasi del Ca2 +, sent la SelT, SelN i SelM les que tenen un paper important en aquesta funció. Pel que respecte al control de qualitat plegable de proteïnes i la resposta a la proteïna no desenvolupada, s’ocupen las Selenoproteïna de 15 kDa i SelS [11].
A més a més, s’ha vist que hi ha moltes selenoproteïnes conservades a l’evolució dels vertebrats: GPx1-4, TR1, TR3, DI1, DI2, DI3, SelH, SelI, SelK, SelM, SelN, SelO, SelP, MsrB1, SelS, SelT, SelW1, Sel15 [1]. En canvi, algunes selenoproteïnes només es troben en certs llinatges, alguns es van perdre durant l'evolució d'un llinatge en particular i altres van substituir el seu residu Sec per a un residu Cys.
Basant-nos en tot això, a continuació es descriurem i discutirem exhaustivament els resultats de l’anàlisi de cada proteïna i representem els gens que codifiquen per a cada una d’aquestes. Cal destacar que les posicions dels exons i del SECIS en les imatges estan expressades en valors relatius, mentre que les presentades en la taula, son els absoluts.
La família de les proteïnes iodotironina deionidasa (DIO) està formada per tres enzims (DI1, DI2 i DI3) que catalitzen la deiodinació de les hormones tiroidees, i que per tant, regulen tant l’activació (DI1 i DI2) com la inactivació (DI3) d’aquestes hormones [1].
Callorhinus Ursinus, com en tots els mamífers, té les tres DIO i totes tres contenen selenocisteïna, les quals mostren homologia entre elles. Per tant, aquestes proteïnes són paràlogues [1].
Al cercar les proteïnes d’aquesta família en el genoma de Callorhinus ursinus s’observa que el blast obtingut per a les tres proteïnes dóna com a resultat els tres mateixos scaffolds per a cada una d’elles (KZ836243.1, KZ836245.1 i KZ836210.1). A més, les posicions en cada scaffold son similars. Els valors del e-value en DI3 son molt diferents entre ells, sent el de l’scaffold KZ836243.1 molt superior (2.06e-170). En el cas de DI2, passa el mateix, l’e-value de l’scaffold KZ836210.1 és molt més elevat que el dels altres dos scaffolds (1.30e-120). Finalment, en DI1, els e-value son molt similars en tots tres scaffolds. Com que les posicions que cobreixen els hits en el genoma son molt similars en les tres proteïnes, s’han descartat aquells scaffolds on es va predir DI2 i DI3. Per tant, només queda l’scaffold KZ836245.1. Per tal de confirmar que DI1 està codificada en aquest scaffold, s’ha vist en els resultats del T-coffee, on l’score és molt bo.
Per acabar l’anàlisi s’ha fet un arbre filogenètic, gràcies al qual es podrà confirmar si les proteïnes s’han predit correctament. Efectivament, tal i com mostra la imatge (Figura 1), les queries es troben properes a les proteïnes predites corresponents, per tant es pot considerar que la predicció és correcte.
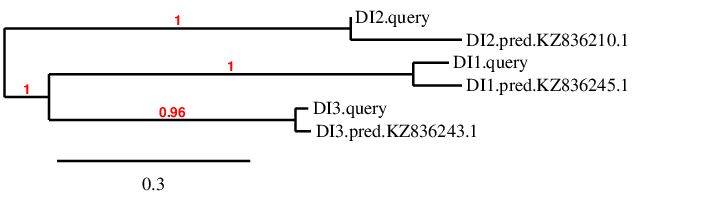
Figura 1: Arbre filogenètic de la família DIO
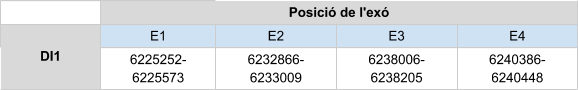
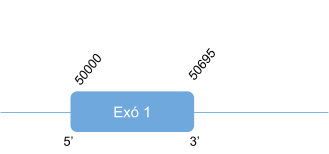
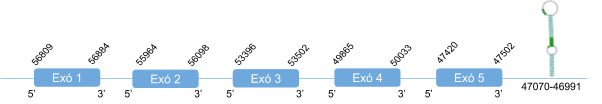
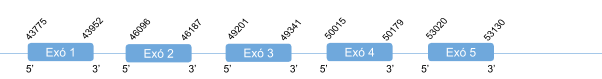
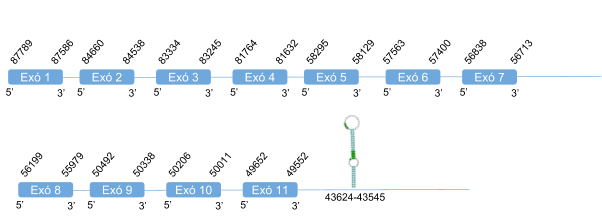
Com s’ha dit anteriorment, DI1 es troba en l’scaffold KZ836245.1, entre les posicions 6225252 i 6240448en la cadena positiva (+). El gen que la codifica conté 4 exons, i té la selenocisteïna en l’exó 2 i un element SECIS a l’extrem 3’ UTR.
DI1 té una llargada de 249 aminoàcids i comença per metionina.
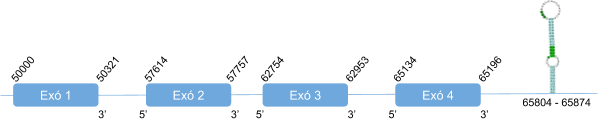
Posicions en valors absoluts:
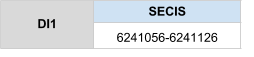
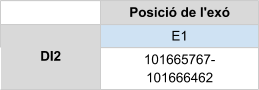
DI2 es troba a l’scaffold KZ836210.1 entre les posicions 101665767 i 101666462de la cadena positiva (+), com s’ha mencionat anteriorment. El gen conté només un exó, en el qual es troba la selenocisteïna, però en aquest cas no s’ha trobat cap element SECIS en l’extrem 3’ UTR. Com que es tracta d’una selenoproteïna, hauria de tenir aquest SECIS, pel que s’ha arribat a la conclusió de que el fet de que no en presenti cap, sigui degut a la utilització d’un output de Fastasubseq massa curt, o bé que el programa no sigui suficientment sensible. En el SelenoDB veiem que DI2 humana sí que mostra SECIS en l’extrem 3’ UTR.
La proteïna conté 254 aminoàcids i no comença per metionina. En l’alineament amb T-coffee s’observa que a l’inici de la seqüència hi ha un gap, fet que pot ser degut a causa d’una deleció en el genoma de Callorhinus ursiuns, o més probablement que aquest no estigui anotat correctament.
Aquesta proteïna conté dos codons UGA, que és un tret característica de DI2. En un estudi anterior en un sistema de cultiu cel·lular es va trobar que el segon UGA podia insertar selenocisteïna quan el primer codó UGA estava mutat [1].
Posicions en valors absoluts:
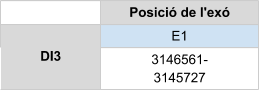
Per altra banda, DI3 es troba en el scaffold KZ836243.1 entre les posicions 3145727 i 3144930 a la cadena negativa (-).El gen conté només un exó en el qual es troba la selenocisteïna. En aquesta proteïna s’ha predit un element SECIS en l’extrem 3’ UTR.
Aquesta proteïna està formada per 278 aminoàcids i comença per metionina.
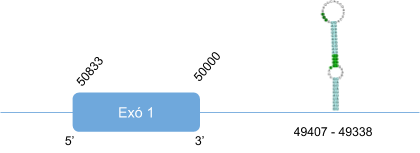
Posicions en valors absoluts:

La família de les proteïnes glutatió peroxidases (GPx) és la família més gran que es troba en els vertebrats. Està formada per 8 proteïnes homòlogues en mamífers (GPx1, GPx2, GPx3, GPx4, GPx5, GPx6, GPx7 i GPx8). Van ser les primeres proteïnes humanes que es va demostrar que incorporen seleni en forma de selenocisteïna en el centre catalític. Estan implicades en l’activitat antioxidant del seleni i en molts altres processos fisiològics [3].
En humans, el genoma que hem agafat de referència, trobem que GPx1, 2, 3, 4, i 6 són selenoproteïnes i GPx5, 7 i 8 en canvi, són proteïnes homòlogues en cisteïnes. GPx5 i GPx6 són les proteïnes d’aquesta família que més tard han aparegut, sent el resultat d’una duplicació en tàndem de GPx3 als mamífers placentaris [1].
En Callorhinus ursinus hem trobat que GPx1, 2, 3 i 4 són selenoproteïnes i GPx5, 6, 7, i 8 homòlogues en cisteïna.
Aquesta família és molt gran i hi ha un elevat grau d’homologia. Observem que hi ha tres grups d’evolució: GPx1/GPx2, GPx3/GPx5/GPx6 i GPx4/GPx7/GPx8 [1]. Coneixent això, al analitzar el Blast veiem que apareixen hits en els mateixos scaffolds i en posicions similars per a cada grup d’evolució. Tots els e-values majoritàriament son bons. Per tant, per assignar un scaffold a cada proteïna s’ha tingut en compte la llargada del hit, comprovant que aquesta és similar a la llargada de la query obtinguda del SelenoDB.
Per acabar de definir l’scaffold s’ha estudiat més d’un en cada proteïna amb l’objectiu final d’escollir la millor predicció. A més a més s’ha realitzat un arbre filogenètic que mostra la proximitat filogenètica que hi ha entre les proteïnes d’Homo sapiens i Callorhinus ursinus.
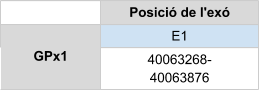
Per GPx1 s’ha analitzat l’scaffold KZ836298.1 entre les posicions 40063307 i 40063876, el KZ836247.1 entre les posicions 3028598 i 3029136 i per últim el scaffold KZ836246.1 entre les posicions 3028734 i 3029272. Després de l’estudi fet en aquests tres scaffolds es pot concloure que aquesta proteïna es troba predita a l’scaffold KZ836298.1, a la cadena positiva (+) ja que és el que cobreix major part de la query i a més a més el T-coffee dóna un score de 1000 i l’alineament és bo tot i que no comença per metionina. El gen conté un únic exó en el qual es troba la selenocisteïna i a més a més a l’extrem 3’ UTR d’aquest gen hi ha un element SECIS.
Aquesta proteïna està formada per 203 aminoàcids i no comença per metionina, com ja s’ha mencionat anteriorment.
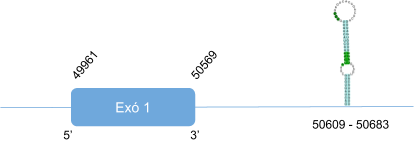
Posicions en valors absoluts:

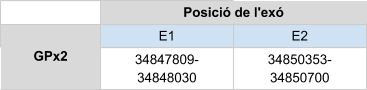
En GPx2 s’han analitzat dos scaffolds: KZ836298.1 entre les posicions 40063307 i 40063858 i KZ836243.1 entre les posicions 34847809-34850700. Finalment s’ha determinat que aquesta proteïna es troba a l’scaffold KZ836243.1, a la cadena positiva (+), s’ha confirmat amb el T-coffee amb un score de 1000 i l’alineament és molt bo.
El gen que codifica per GPx2 està format per 2 exons, la selenocisteïna està localitzada al primer exó. A més a més, s’ha predit un element SECIS a l’extrem 3’ UTR.
Aquesta proteïna està formada per 190 aminoàcids i comença per metionina.
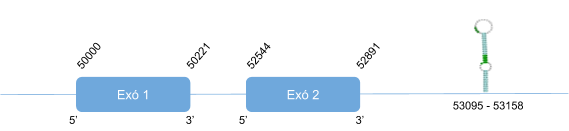
Posicions en valors absoluts:

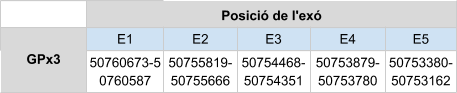
En GPx3 s’ha analitzat dos scaffolds: KZ836298.1 entre les posicions 40063310 i 40063855 i el KZ836236.1 entre les posicions 50753162 i 50750502. Finalment s’ha determinat que aquesta proteïna es troba a l’scaffoldKZ836236.1, a la cadena negativa (-), ja que és on es van trobar millors resultats, un score de 1000 i un molt bon alineament s’observa amb T-coffee. El gen està format per 5 exons i la selenocisteïna es troba al segon exó. Mitjançant Seblastian s’ha predit un element SECIS que es troba a l’extrem 3’ UTR de la cadena negativa.
Aquesta proteïna està constituïda per 226 aminoàcids i comença per metionina.
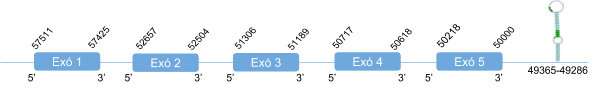
Posicions en valors absoluts:

Per GPx4 s’ha analitzat 2 scaffolds: KZ836507.1 entre les posicions 4478301 i 4477269 i el KZ836403.1 entre les posicions 2396432 i 2396856. Finalment s’ha determinat que aquesta proteïna es troba a l’scaffoldKZ836507.1, a la cadena negativa (-), l’score del T-coffee és de 1000 i l’alineament molt bo. El gen està format per 7 exons, la selenocisteïna es troba al exó 3. S’ha predit un element SECIS a l’extrem 3’ UTR de la cadena negativa.
Aquesta proteïna està formada per 197 aminoàcids i comença per metionina.
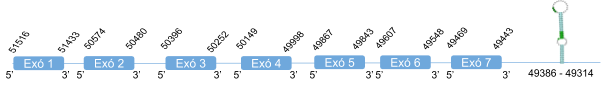
Posicions en valors absoluts:
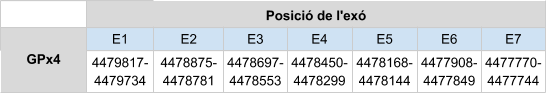

Per GPx5 s’ha analitzat els scaffolds següents: KZ836298.1 entre les posicions 40063310 i 40063861, el KZ836418.1 entre 469409 i 474150, el KZ836236.1 entre 50750529 i 50753162 i per últim l’scaffold KZ836243.1 entre les posicions 34847827 i 34850691. Després d’un anàlisis exhaustiu s’ha definit que GPx5 es troba a l’scaffold KZ836418.1, a la cadena positiva (+), ja que s’ha obtingut bons resultats i a l’alineament amb el T-coffee amb un score de 999. En Callorhinus ursinus és una proteïna homòloga en cisteïna igual que en Homo sapiens. El gen que codifica per aquesta proteïna està format per 4 exons i no s’han predit elements SECIS.
Aquesta proteïna està formada per 221 aminoàcids i no comença per metionina. A l’inici hi ha un gap que pot ser degut a una mala anotació del genoma o una deleció en el genoma de Callorhinus ursinus.
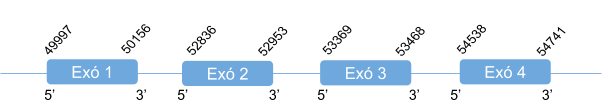
Posicions en valors absoluts:
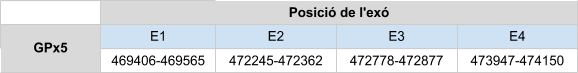
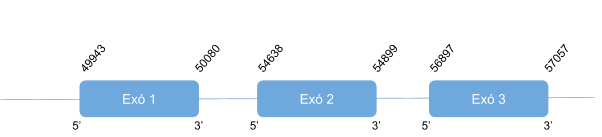
Per GPx6 s’han analitzat els scaffolds següents: KZ836298.1entre les posicions 40063310 i 40063310, el KZ836418.1 entre434356 i 436625 i per últim el KZ836236.1 entre 50750529 i 50753162. Després d’un anàlisis detallat s’ha definit que GPx6 es troba a l’scaffold KZ836418.1, a la cadena negativa (-), ja que és el que obté un score més alt mitjançant T-coffe (999). En l’alineament observem que la selenocisteïna de la GPx6 humana s’alinea amb una cisteïna, per tant es pot concloure que en Callorhinus ursinus s’ha perdut la selenocisteïna.
El gen que codifica per aquesta proteïna està format per 4 exons. No s’han predit elements SECIS en l’extrem 3’ UTR de la cadena negativa.
En Callorhinus ursinus aquesta proteïna homòloga en cisteïna està formada per 221 aminoàcids i no comença per metionina.
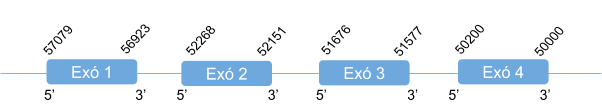
Posicions en valors absoluts:
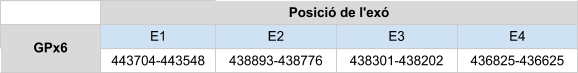
En aquest cas només s’ha analitzat l’scaffold KZ836245.1 entre les posicions 5078362 i 5085419 a la cadena positiva (+). A diferencia de les altres GPx només s’ha analitzat un scaffold ja que els altres tenen un valor d’e-value molt més gran. Amb el resultat del T-coffee (score = 997 i un bon alineament) i de l’arbre filogenètic s’ha pogut confirmar que GPx7 es troba a aquest scaffold.
El gen que codifica per aquesta proteïna està format per tres exons, no té present cap element SECIS al extrem 3’ UTR. Tot i així, el Seblastian prediu un element SECIS a la cadena negativa, però com ja s’ha mencionat en altres casos aquest sistema no és gaire sensible ja que és molt automàtic. A més a més aquest es troba a la cadena contraria que el gen per tant es pot rebutjar.
És una proteïna homòloga en cisteïna formada per 187 aminoàcids i comença per metionina.
Posicions en valors absoluts:
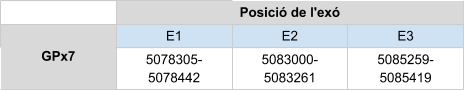
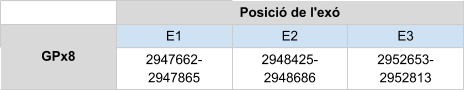
Finalment, per la proteïna GPx8, igual que en el cas anterior, només s’ha analitzat un scaffold, KZ836345.1 entre les posicions 2947662 i 2952813 de la cadena positiva (+). S’ha analitzat només aquest ja que té un e-value molt inferior comparat amb la resta i a més, és el que té una major longitud. Igual que amb GPx7, s’ha confirmat amb els resultats del T-coffee (score = 1000 i un bon alineament) i l’arbre filogenètic.
El gen que codifica per GPx8 està format per 3 exons, no es prediuen SECIS en aquesta cadena, tot i que el programa SECISearch en detecta a la cadena negativa, però es rebutja ja que està ubicat a la cadena contraria d’on es troba el gen.
És una proteïna homòloga en cisteïna formada per 209 aminoàcids i comença per metionina.
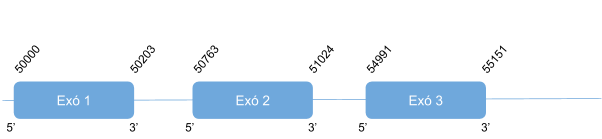
Posicions en valors absoluts:
Després de l’anàlisi exhaustiu de la família de les GPx i de predir-les, s’ha realitzat un arbre filogenètic, com s’ha mencionat anteriorment. Com es pot observar a la imatge (Figura 2) totes les proteïnes predites es troben properes a la de referencia, és a dir, a la proteïna humana, exceptuant GPx5. Això pot ser degut a una predicció errònia o que aquesta ha divergit molt respecte la proteïna humana.
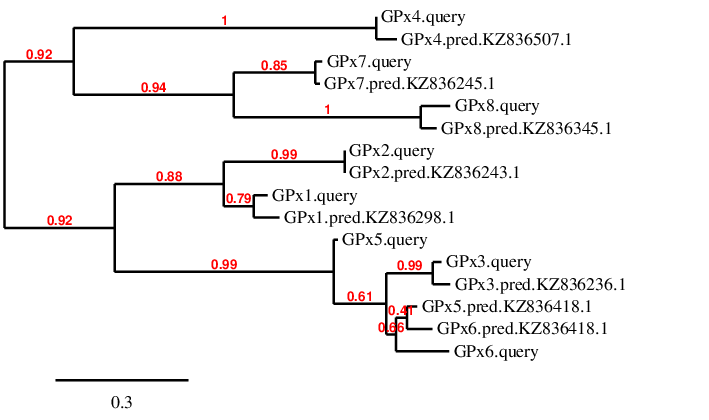
Figura 2: Arbre filogenètic de la família GPx
És una de les selenoproteïnes que està més conservada en vertebrats i forma part de la família Se15. S'expressa en molt teixits, sent en el cervell, pulmó, testicles, ronyó, fetge i tiroides on major expressió té. A més, s’ha vist que la seva expressió es troba incrementada en el càncer de colon, i disminuïda en el càncer de fetge i pròstata. A la cèl·lula es localitza al reticle endoplasmàtic i interacciona amb la UDP-glucosa. Per tant, basant-nos en això, es pot suposar que està molt involucrada en el control de qualitat del plegament de les glicoproteïnes. S’ha vist també que pot tenir funció antioxidant, tot i que la seva funció específica encara no es coneix [3].
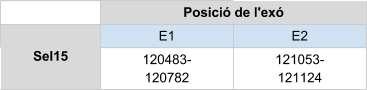
En fer l’alineament de la query d’Homo sapiens amb el genoma del Callorhinus ursinus s’han donat diversos hits. Seguidament s’ha analitzat aquell hit amb millor e-value i els resultats obtinguts són molt bons. Per aquesta raó s’ha determinat que Sel15 es troba a l’scaffold KZ836410.1, entre les posicions 120483 i 120794de la cadena positiva (+). El gen que codifica per Sel15 està format per dos exons, sent el primer el que conté la selenocisteïna alineada amb la de Sel15 humana. També s’ha vist que li apareix una altra selenocisteïna en el segon exó alineada amb arginina. Mitjançant Seblastian s’ha predit un element SECIS en l’extrem 3’UTR.
Aquesta proteïna té una llargada de 124 aminoàcids i comença per metionina.
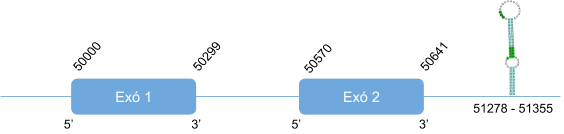
Posicions en valors absoluts:

Es tracta d’una selenoproteïna que es troba en tots els vertebrats. És la més gran que es troba en els mamífers i a més a més té molts ortòlegs en altres organismes. És una proteïna mitocondrial involucrada en les interaccions redox a través del seu motiu CXXU [5].
En fer l’alineament amb Homo sapiens s’han donat hits en 4 scaffolds. S’ha analitzat l’scaffold amb l’e-value més petit (KZ836294.1) i s’ha vist que l’score obtingut amb el T-coffee és baix. A més, l’alineació conté molts gaps. Posteriorment, s’ha analitzat l’scaffold KZ836293.1, ja que el valor del e-value és molt bo i a més, tots els hits que s’han donat cobreixen gairebé tota la seqüència de la query. En aquest cas, els resultats del T-coffee són millors, tot i que la seqüència segueix tenint alguns gaps. Tot i això, s’ha determinat que SelO es troba a l’scaffoldKZ836293.1, entre les posicions 125249 i 112518de la cadena negativa (-). El gen que codifica per SelO està format per 10 exons, conté la selenocisteïna a l’últim exó i un element SECIS en l’extrem 3’UTR. Els resultats obtinguts del Seblastian confirmen la presència d’aquests elements.
En Callorhinus ursinus, aquesta proteïna té una llargada de 677 aminoàcids i no comença per metionina. El principi de la seqüència s’alinea amb un gap força petit.
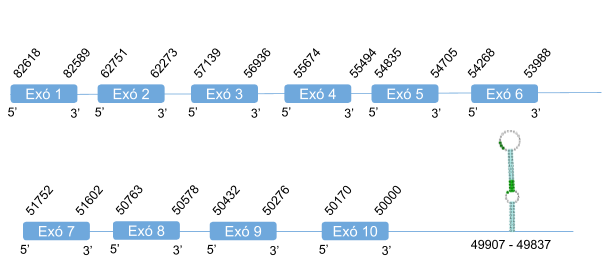
Posicions en valors absoluts:
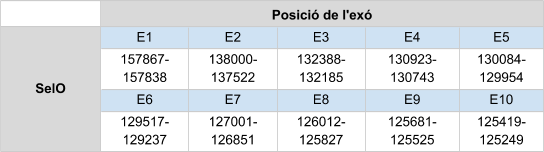

La SelR és una proteïna que conté una selenocisteïna i està present en tots els mamífers. En diversos genomes, els gens de SelR i MsrA estan fusionats o agrupats, i els seus patrons d'expressió suggereixen un paper d'ambdues proteïnes en la protecció contra l'estrès oxidatiu.
SelR forma part d’una via de reducció de sulfòxid de metionina, i la seva funció és la protecció contra l'estrès oxidatiu i/o la regulació redox dels processos cel·lulars. El seu component principal, el “peptide metionine sulfoxide reductase” (MsrA), catalitza la reducció de sulfòxid de metionina dependent de thioredoxin. Tanmateix, encara que MsrA explica la major part de l'activitat cel·lular de metionina sulfòxid reductasa, el paper d'aquesta proteïna com a únic reductor del sulfòxid de metionina del pèptid contrasta amb dades experimentals.
Hi ha tres tipus de selenoproteïnes R: SelR1, SelR2 i SelR3.
Per l’alineament amb Homo sapiens s’han donat hits en tres scaffolds, dels quals s’ha analitzat el que té un e-value més bo (6.53e-24), el KZ836278.1. Els resultats obtinguts amb T-coffe són bons ja que presenta un score de 999. S’ha determinat que SelR1 es troba a l’scaffold KZ836278.1, entre les posicions 653498 i 653677 de la cadena positiva (+). El gen que codifica per SelR1 està format per tres exons, sent l’últim exó el que conté la selenocisteïna, i en l’extrem 3’UTR té un element SECIS. Els resultats obtinguts al Seblastian ho confirmen, indicant així que es tracta d’una selenoproteïna.
En Callorhinus ursinus, aquesta proteïna té una llargada de 116 aminoàcids i comença per metionina.
Posicions en valors absoluts:
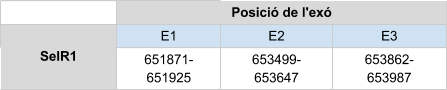

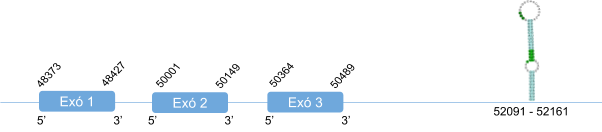
En l’alineament amb Homo sapiens només s’han donat hits en tres scaffold diferents, dels quals s’ha analitzat el que té un e-value més petit (6.18e-76), el KZ836211.1.
SelR1 es troba a l’scaffold KZ836211.1, entre les posicions 822395 i 822796 de la cadena positiva (+), fet que es corrobora amb els bons resultats obtinguts amb T-coffee. El gen que codifica per SelR2 està format per un exó. Aquesta proteïna és homòloga en cisteïna, per tant no conté cap selenocisteïna. Tot i això, s’ha predit un element SECIS a l’extrem 3’ UTR mitjançant Seblastian. Això pot ser degut a que anteriorment fos una selenoproteïna que ha perdut el seleni que forma la selenocisteïna, però manté l’element SECIS. Els resultats obtinguts al Seblastian corroboren aquests resultats.
En Callorhinus ursinus, aquesta proteïna té una llargada de 199 aminoàcids i no comença per metionina i el principi de la seqüència s’alinea amb un gap.
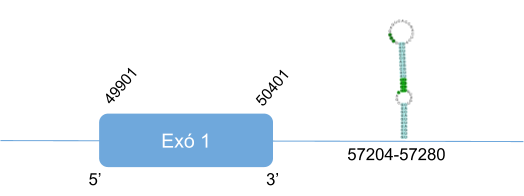
Posicions en valors absoluts:
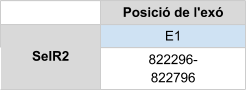

Al fer l’alineament amb Homo sapiens s’han donat hits en set scaffolds. S’ha analitzat el hit amb un e-value menor de l’scaffold KZ836211.1, i no s’han obtingut resultats gaire bons. Amb el T-coffee s’ha obtingut un score baix i a més, presenta gaps a l’inici, al mig i al final de la seqüència. Aquest és el motiu pel qual es rebutja i s’analitza l’scaffold KZ836297.1.
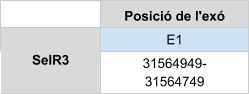
SelR3 es troba a l’scaffold KZ836297.1, entre les posicions 31564557 i 31564165de la cadena negativa (-). L’score obtingut de l’alineament amb T-coffee és bo, tot i que veiem un gran gap a l’inici i al final del alineament.
SelR3 es troba a l’scaffold KZ836297.1, entre les posicions 31564557 i 31564165de la cadena negativa (-). L’score obtingut de l’alineament amb T-coffee és bo, tot i que veiem un gran gap a l’inici i al final del alineament.
El gen que codifica per SelR3 està format per un exó. Aquesta proteïna és homòloga en cisteïna, pel que no conté selenocisteïna. Mitjançant el programa Seblastian s’obté que la seqüència conté un element SECIS a l’extrem 3’ UTR de la cadena positiva, però com que aquesta proteïna es troba codificada a la cadena negativa es rebutja.
En Callorhinus ursinus, aquesta proteïna té una llargada de 192 aminoàcids, no comença per metionina i el principi de la seqüència s’alinea amb un gap.
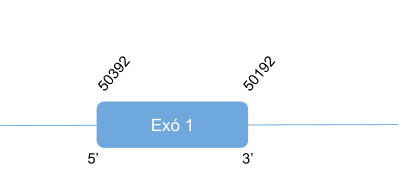
Posicions en valors absoluts:
Les reductases de sulfòxid de metionina s’encarreguen de reparar els residus oxidats de metionina en proteïnes i sulfòxids lliures de metionina. Estan formades per dues famílies MsrA i MsrB. MsrA s’encarrega de reduir les formes S del sulfòxid de metionina, mentre que MsrB redueix les formes R.
Al fer l’alineament amb Homo sapienss’han trobat diversos hits en un mateix scaffold(KZ836264.1), dels quals s’ha analitzat el primer, ja que és el que menor e-value té. En l’alineament del T-coffee s’ha obtingut un bon score (998), però tot i així, s’observa que no comença per metionina i a l’inici de la seqüència hi ha un gap molt gran. Això pot ser degut a que al agafar només el primer hit s’hagi desestimat part de la proteïna, ja que analitzant posteriorment el blast s’ha vist que aquell hit cobreix només uns 100 aminoàcids de la query.
El gen predit està format per un únic exó i no conté cap selenocisteïna. MsrA és una proteïna homòloga en cisteïna, formada per 235 aminoàcids.
Posicions en valors absoluts:
L'expressió d’aquesta proteïna és induïda en resposta a l'estrès del reticle endoplasmàtic. És una selenoproteïna que es troba conservada en vertebrats i forma part dels components del sistema de degradació proteica associada al reticle endoplasmàtic (ERAD). Aquest sistema protegeix de l’acumulació de proteïnes mal plegades, transportant-les del reticle endoplasmàtic al proteosoma [3]. A nivell fisiològic, l'expressió de SelS augmenta notablement en el dejuni (en el fetge dels animals diabètics). Diversos informes també mostren una associació entre la desregulació de SelS i els trastorns metabòlics. A més, s’ha proposat que SelS pot servir com una reductasa dependent de thioredoxinen experiments in vitro [6].
En aquest cas, en el resultat del blast es veu que aquesta proteïna dóna múltiples hits en dos scaffoldsdiferents. Primer s’ha analitzat aquell que té un menor e-value, (KZ836305.1). Els resultats obtinguts del T-coffee no són gaire bons, pel que s’ha procedit a analitzat el segon scaffold(KZ836305.1) el qual mostra un major percentatge d’identitat. Amb aquest segon scaffoldel resultat del T-coffee és molt bo (score = 997). El gen que codifica per aquesta proteïna està format per 5 exons i es troba a la cadena negativa (-). La selenocisteïna es troba en l’últim exó i s’ha predit un element SECIS en l’extrem 3’ UTR.
SelS està formada per 190 aminoàcids i comença per metionina.
Posicions en valors absoluts:
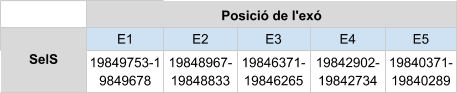

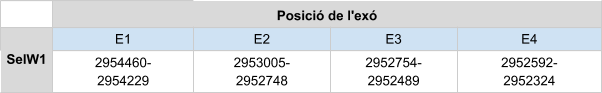
Sel V és la selenoproteïna menys conservada en els mamífers. És probable que aquesta selenoproteïna sorgeixi per la duplicació de la SelW, una selenoproteïna petita (85 a 88 aminoàcids) identificada per primera vegada en ovelles que pateixen deficiència de seleni. Ambdues es troben al citosol, i encara que la funció de les dues proteïnes no es coneix, se sap que Sel V només s’expressa en els testicles, en canvi, Sel W s’expressa en diferents òrgans com el múscul, cor, melsa i cervell. La literatura diu que totes dues tenen la mateixa estructura. Ambdues estan codificades per un gen de 6 exons, sent l’exó 1 on hi ha més diferències. Es creu que això és degut a que la SelV incorpora una seqüència en l’extrem N-terminal [1].
Per aquesta proteïna s’ha trobat dos possibles hits a un únic scaffold, KZ836394.1, a la cadena positiva (+). Amb el T-coffee s’obté un score més baix (971), comparat amb els scores obtinguts de les altres proteïnes. La selenocisteïna de la query s’alinea amb una serina, tot i així comença per metionina. Amb l’Exonerate veiem que el gen predit està format per 6 exons. A més a més, no es prediuen SECIS a la cadena positiva a l’extrem 3’ UTR.
SelV esta formada doncs per 349 aminoàcids.
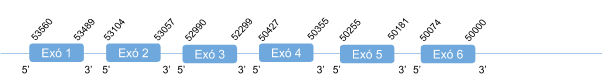
Posicions en valors absoluts:
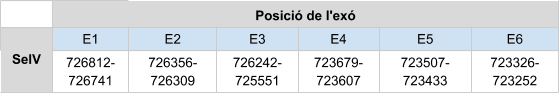
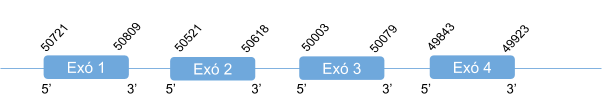
Aquesta proteïna és predita a l’scaffold KZ836356.1, a la cadena negativa (-). El gen està format per 4 exons, sent el primer on es troba la selenocisteïna, i té un element SECIS a l’extrem 3’ UTR.
La proteïna està constituïda per 87 aminoàcids i comença per metionina.
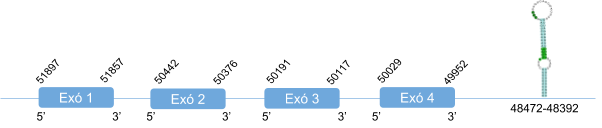
Posicions en valors absoluts:

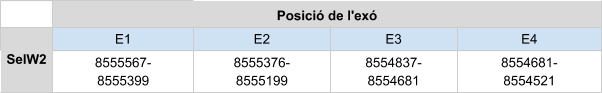
SelW2 es troba a l'scaffold KZ836445.1, a la cadena negativa (-). El gen està format per 4 exons, no conté selenocisteïnes, i tampoc elements SECIS. És una proteïna homòloga en cisteïna, com s’ha vist en els mamífers, aquesta proteïna va perdre el seleni que forma la selenocisteïna donant lloc a cisteïna, s’hipotitza que això va passar abans de la separació dels amfibis, ja que els amfibis mantenen SelW2 com a selenoproteïna.
La proteïna està formada per 115 aminoàcids.
Posicions en valors absoluts:

SelH és una selenoproteïna ancestral que es troba en tots els vertebrats, tot i així la seva funció segueix sent desconeguda. Es considera una oxidoreductasa nucleolar amb funció antioxidant i està relacionada amb la regulació de gens [3].
SelH presenta certes similituds amb altres selenoproteïnes dels mamífers (SelW, SelT, SelM i SelV), una d’elles és la conservació d’un fragment de la seqüència d’aminoàcids que consisteix en Cys-X-X-Sec [7]. A les nostres proteïnes predites s’observa que aquesta similitud només es troba a SelH, SelM i SelT.
Al alinear SelH humana amb el genoma de Callorhinus ursinus s’han donat dos hits, en dos scaffolds diferents. S’ha analitzat aquell que té millor e-value, el qual es troba en l’scaffold KZ836402.1. Com que els resultats obtinguts són bons (score del t-coffee va ser 1000), s’ha determinat que la selenoproteïna H es localitza a l’scaffoldKZ836402.1 a la cadena positiva (+), entre les posicions 32957821 i 32958114. El gen està format per un únic exó en el que es troba la selenocisteïna. S’ha predit un element SECI a l’extrem 3’UTR.
Aquesta proteïna en Callorhinus ursinus està formada per 122 aminoàcids i comença per metionina.
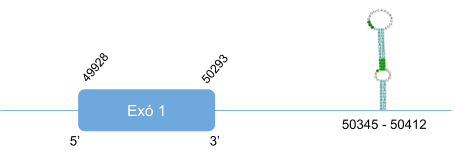
Posicions en valors absoluts:


La selenoproteïna I és de les últimes proteïnes que ha estat descrita i es troba a tots els vertebrats. És una selenoproteïna transmembrana que conté un domini CDP-alcohol fosfatidiltransfersa, el qual està altament conservat. A més, està involucrada en el manteniment de les membranes vesiculars, en la regulació del metabolisme lipídic i en el plegament de proteïnes [1].
En el blast de SelI s’han obtingut múltiples hits en tres scaffolds diferents. S’ha analitzat aquell el que té un e-value menor, és a dir, el KZ836237.1. Els resultats del T-coffee mostren un bon alineament amb un score de 1000. Per aquesta raó es va determinar que aquesta proteïna es troba a l’scaffoldKZ836237.1 a la cadena positiva (+), entre les posicions 21643854-21644126. El gen que codifica per la SelI està format per 10 exons i la selenocisteïna es troba a l’últim. S’ha predit un element SECIS a l’extrem 3’ UTR.
En Callorhinus ursinus aquesta proteïna té 397 aminoàcids i comença per metionina.
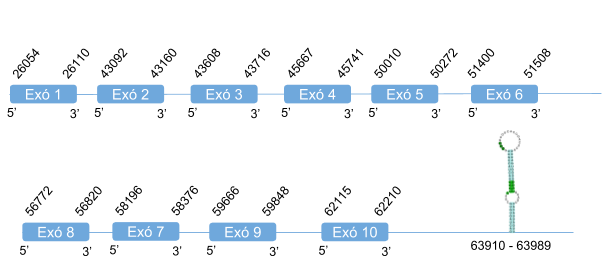
Posicions en valors absoluts:


La Selenoproteïna K és una proteïna que es troba a tots els vertebrats. Es localitza al reticle endoplasmàtic i a la membrana plasmàtica, i també revela components de degradació associada al reticle endoplasmàtic (ERAD). El seu gen conté un element funcional de resposta a l'estrès del reticle endoplasmàtic, i la seva expressió està regulada per condicions que indueixen l'acumulació de proteïnes mal contigües al reticle endoplasmàtic [3, 8].
L’expressió de SelK predomina al cor i al múscul esquelètic. A la cèl·lula es troba al reticle endoplasmàtic, on manté l’homeòstasi, i a la membrana plasmàtica. Una sobre expressió de SelK disminueix els nivells intracel·lulars de ROS i protegeix els cardiomiocits de l’estrès oxidatiu. Es considera que té una funció antioxidant al cor [3].
Els resultats del blast de SelK mostren la presència hits en tres scaffolds diferents. S’ha analitzat l’scaffold de millor e-value que a més a més és el que major longitud té. Els resultats obtinguts amb el T-coffee són molt bons, l’alineament és complet i l’score obtingut és de 1000. Per tant, SelK es troba a l’scaffold KZ836306.1, entre les posicions 44273330 i 44273088 de la cadena negativa (-).
El gen que codifica per SelK està format per un únic exó, en el qual es troba la selenocisteïna. Mitjançant el programa Seblastian es prediu que la seqüència conté un element SECIS a l’extrem 3’ UTR de la cadena negativa.
En Callorhinus ursinus, aquesta proteïna té una llargada de 94 aminoàcids i comença per metionina.
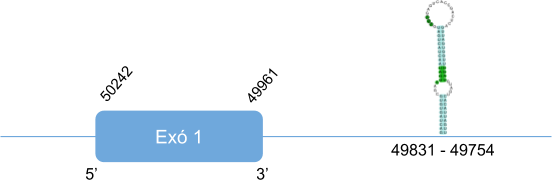
Posicions en valors absoluts:
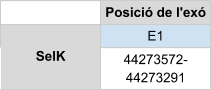

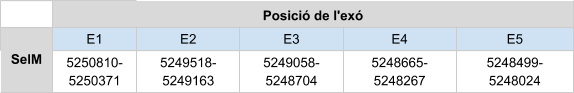
La selenoproteïna M es troba a tots els vertebrats. Comparteix un 31% d'identitat de seqüència amb la Sel15 en animals [9]. Conté el motiu C-XX-U [3]. És similar a la proteïna tioredoxina que es troba al reticle endoplasmàtic i té com a funció regular l’homeòstasi de les reaccions redox. A més, s’ha vist que la selenoproteïna M pot tenir un paper important en la protecció contra el dany oxidatiu del cervell i pot funcionar potencialment en la regulació del calci. Per tant, es troba altament expressada al cervell tenint propietats neuroprotectores [9].
Al alinear SelM humana amb el genoma de Callorhinus ursinus només es va donar un hit en el scaffold KZ836407.1. SelM es troba a l’scaffold KZ836407.1, entre les posicions 5248344 i 5248024 de la cadena negativa (-).Els resultats obtinguts amb el T-coffee són bons, així com l’alineament i l’score (995), tot i que hi ha un petit gap a l’exó 1.
El gen que codifica per SelM està format per cinc exons i la selenocisteïna es troba al segon. Els resultats obtinguts al Seblastian indiquen que hi ha un element SECIS a l’extrem 3’ UTR però en canvi, no pot predir la selenoproteïna. Això pot ser degut a que Seblastian és un programa que té una base de dades que conté seqüències d’espècies molt diferents i pot ser que al fer l’alineament no predigui bé la proteïna de Callorhinus ursinus.
En Callorhinus ursinus, aquesta proteïna té una llargada de 145 aminoàcids i comença per metionina.
Posicions en valors absoluts:

La selenoproteïna N també es troba en tots els vertebrats i és l’única que està directament relacionada amb una malaltia. És una glicoproteïna ubicada a la membrana del reticle endoplasmàtic relacionada amb el desenvolupament embrionari i el múscul esquelètic. A nivell cel·lular, dades recents suggereixen que SelN participa en l’homeòstasi oxidativa i de calci, amb un paper potencial en la regulació de l'activitat del receptor de ryanodina [3, 10].
La seva deficiència causa diversos trastorns neuromusculars hereditaris denominats col·lectivament com miopaties relacionades amb SEPN1. Aquestes es caracteritzen per la presència d'atròfia muscular generalitzada, la escoliosi severa, la insuficiència respiratòria i la debilitat muscular que afecta sobretot als músculs axials i condueix a la rigidesa de la columna vertebral [3, 10].
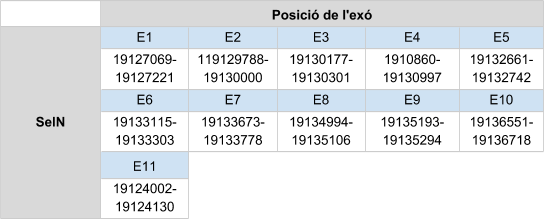
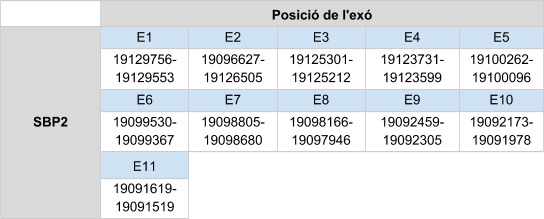
SelN presenta diferents hits en un mateix scaffold, dels quals s’ha analitzat el primer hit, ja que és el que té millor e-value. L’scaffolden qüestió és KZ836402.1 i es troba entre les posicions 19129788 i 19130126 de la cadena positiva (+).El gen està format per 11 exons, la selenocisteïna es troba al exó 7. S’ha predit un element SECIS al extrem 3’ UTR.
Aquesta proteïna en Callorhinus ursinusestà formada per 591 aminoàcids, no comença per metionina i a l’inici de la proteïna hi ha un gap bastant gran. Aquest gap pot ser degut a una mala anotació del genoma de Callorhinus ursinuso a una deleció en aquesta espècie.
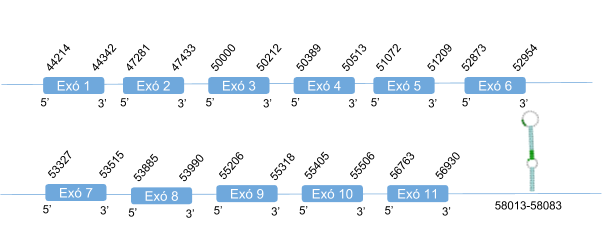
Posicions en valors absoluts:

La selenoproteïna P és la segona selenoproteïna més abundant que trobem al plasma, després de GPx3. Aquesta és secretada pel fetge al plasma en forma glicosilada, encara que la seva expressió es detecta en tots els teixits. El paper principal de SelP és el transport i lliurament de seleni als teixits [3].
El domini Nt de SelP és semblant a un domini enzimàtic, ja que la selenocisteïnaes troba en un motiu redoxUxxC. En canvi el domini Ct, és un mitjà de transport segur del selenireactiu pel cos. A més a més s'ha vist que és una proteïna que s'uneix a heparina, ja que té dues regions riques en histidines. És capaç d'unir-se a diferents metalls pesants com el mercuri. Es creu que la selenoproteïna P només s'expressa en vertebrats. S'ha vist que els testicles i el cervell tenen receptors per selenoproteïna P i que regulen el seu mecanisme per obtenir seleni quan hi ha una deficiència [3].
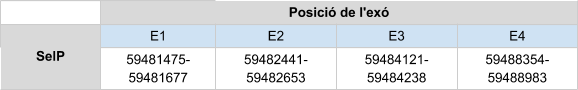
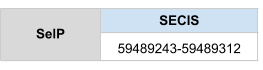
Al fer l’alineament, SelP ha donat diferents hits en un mateix scaffold, dels quals només s’ha analitzat el primer hit ja que és el que té un e-value més bo. A més, és el hit que té una major longitud. S’ha determinat que SelP es troba a l’scaffold KZ836236.1, entre les posicions 59488354 i 59488965, a la cadena positiva (+).El gen està format per 4 exons, tot i que hi ha un petit gap en el exó 4. Té 15 selenocisteïnes, a diferència de la SelP humana que tan sols conté 10. A aquest gen se li ha predit un element SECIS al extrem 3’ UTR de la cadena positiva.
La selenoproteïna en Callorhinus ursinusestà formada per 389 aminoàcids i comença per metionina.
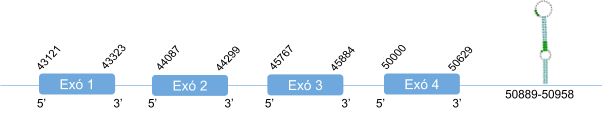
Posicions en valors absoluts:
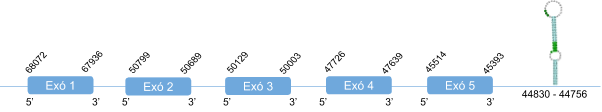
La selenoproteïna T és una proteïna similar a la tioredoxina i està regulada pel polipèptid tòpic de polipèptids adenilat ciclasa (PACAP) de neuropéptids tròfics en cèl·lules adrenomedular diferenciadores però no madures. A més, s’expressa de forma abundant però transitòria durant l’ontogènesi cerebral. S’ha realitzat un estudi que relaciona la deficiència d’aquesta proteïna amb anormalitats al desenvolupament neuronal i comportament hiperactiu en ratolins [11].
SelT conté un motiu conservat anomenat thioredoxin-like fold (CXXU) que es caracteritza per la presència d'un residu de cisteïna (C) a la zona de Sec (U), separat per altres dos aminoàcids (X). Aquest motiu confereix una important activitat redox [2].
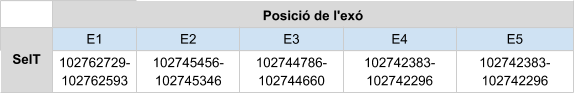
En el Blast de SelT es van obtenir diferents hits en un mateix scaffold,KZ836210.1. Es va analitzar només el primer hit, ja que era el que tenia milor e-value i a és a més el que cobria major part de la query. Els resultats obtinguts van ser bons, l’score obtingut amb el t-coffee és de 1000 i l’alineament és bo. Per aquesta raó s’ha definit que SelT es troba enl’scaffold KZ836210.1, entre les posicions 102744657 i 102744526, a la cadena negativa (-).El gen està format per cinc exons,i la selenocisteïna es troba al segon exó. Els resultatsobtinguts al Seblastian indiquen la presència d’un element SECIS a l’extrem 3’UTR.
La proteïna en Callorhinus ursinusestà formada per 195 aminoàcids i comença per metionina.
Posicions en valors absoluts:

La família de proteïnes SelU s’expressa als ossos, al fetge i al ronyó. Té un motiu conservat similar a l'existent en altres selenoproteïnes reguladores de redox que depenen de tiol; no obstant això la seva funció és desconeguda [2]. En els mamífers SelU1, SelU2, SelU3 són homòlogues en cisteïna, de manera que no són selenoproteïnes, hi ha un reemplaçament de selenocisteïna per cisteïna [1].
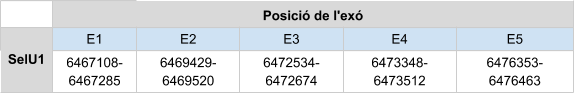
Al alinear la SelU1 humana amb el genoma de Callorhinus ursinuses van donar diversos hits en tres scaffoldsdiferents. Es va analitzar el hit que donava millor e-value, el qual es troba en l’scaffoldKZ836358.1. Els resultats obtinguts van ser bons, l’score obtingut amb el T-coffee va ser de 1000 i l’alineament entre la proteïna predita i la de referència va ser molt bo. Per tant,SelU1 es troba a l’scaffoldKZ836358.1, entre les posicions 6473333-6473512a la cadena positiva (+). El gen està format per cinc exons. És una proteïna homòloga en cisteïna, per tant no conté cap selenocisteïna. Els resultatsobtinguts al Seblastian no prediuen cap element SECIS.
La proteïna en Callorhinus ursinusestà formada per 229 aminoàcids i comença per metionina.
Posicions en valors absoluts:

En el blast es van obtenir diferents hits en un mateix scaffold,KZ836374.1. Es va analitzar aquell hit que tenir un valor de e-value menor i els resultatsque es van obtenir van ser bons. L’score resultat de l’alineament amb T-coffee va ser de 998 i l’alineament molt bo tot i presentar un gap de dos aminoàcids a l’inici de la seqüència. Per aquesta raó es va determinar que SelU2 es troba a l’scaffoldKZ836374.1, entre les posicions 6941623 i 6941231a la cadena negativa (-). El gen està format per sis exons. És una proteïna homòloga en cisteïna, per tant no conté cap selenocisteïna. Els resultatsobtinguts al Seblastian indiquen que no hi ha elements SECIS.
La proteïna en Callorhinus ursinusestà formada per 226 aminoàcids i no comença per metionina.
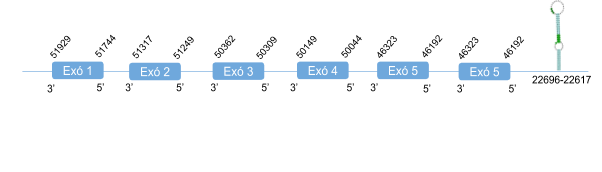
Posicions en valors absoluts:


En SelU3 només es va donar un hit, en l’scaffoldKZ836402.1. Al alinear amb T-coffee la seqüencia predita de SelU3 amb la de referencia es va obtenir un score de 992, i es va veure que en el primer exó i al final de la seqüència hi havia un gap. Tot i això es va determirar que la proteïna SelU3 es troba enel scaffold KZ836402.1, entre les posicions 1343859 i 1344065 a la cadena positiva (+). El gen està format per sis exons. És una proteïna homòloga en cisteïna. Mitjançant el programa Seblastian s’obté que conté elements SECIS a la seqüència. Però com que els elements SECIS predits es troben a la cadena negativa i aquesta proteïna és codificada a la cadena positiva, aquests no es tenen en compte.
Aquesta proteïna és homòloga en cisteïna en humans, però en Callorhinus ursinuss’ha trobat que conté una selenocisteïna codificada en l’últim exó, que s’alinea amb una arginina.
La proteïna en Callorhinus ursinusestà formada per 231 aminoàcids.
Posicions en valors absoluts:

La família Thioredoxin reductasa (TR) està formada per tres proteïnes (TR1, TR2 i TR3). Són una família de oxidoreductases de nucleòtids-disulfuro de piridina que contenen seleni amb identitat de mecanisme i seqüència, incloent un lloc catalític conservat -Cys-Val-As-Val-Gly-Cys-redox, a glutatió reductases. Són enzims que pertanyen a la família de la flavoproteïna del nucleòtid-disulfur de piridina oxidoreductasas que inclouen lipoamida deshidrogenasa, glutatió reductasa i reductasa de ió mercúric. Aquestes són encarregades de la regulació de les reaccions redox que es donen als processos cel·lulars. Tenen una funció enfront l'estrès oxidatiu i el metabolisme del Seleni [12].
TR1, es tracta d’una selenoproteïna, que està sobre expressada en molts tumors malignes. És un enzim antioxidant crític que catalitza la reducció de tioredoxina depenent de NADPH per regular l'homeòstasi redox cel·lular. S’ha associat amb la proliferació cel·lular, apoptosi i transformació. A més, està regulat en moltes malalties humanes i funciona com un factor pronòstic per a molts tumors, com ara carcinomes escamosos de cèl·lules orals, càncer de pulmó, càncer de mama i astrocitomes [13].
En el cas del TR2, es tracta de una proteïna que es troba al mitocondri [1].
Per altre banda, TR3, és un flavenoenzim que conté selenocisteïna, que redueix les thioredoxins, així com altres substrats, i té un paper clau en l’homeòstasi de les reaccions redox [1].
En la nostra espècie, Callorhinus Ursinus, totes tres són selenoproteïnes, igual que en la resta de mamífers.
Al ser una família es va observar que hi ha molta homologia entre les tres proteïnes, ja que en el blast de cada TR es donen hits en els mateixos scaffolds i en posicions similars. A més, totes els e-values són bons. S’han analitzat diversos scaffolds per a cada proteïna i s’ha pogut determinar l’scaffold que correspon a cadascuna d’elles, gràcies a la comparació entre els T-coffee i l’arbre filogenètic (Figura 3).
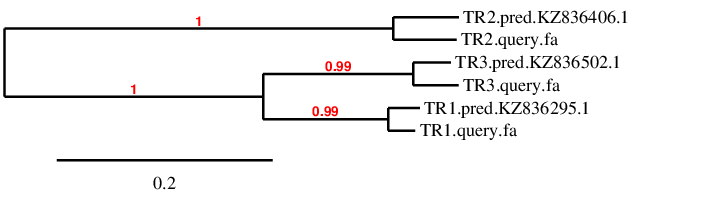
Figura 3: Arbre filogenètic de la família TR
Per aquesta primera proteïna TR s’han analitzat els següents scaffolds: KZ836295.1 de la posició 2405251 a 2426165, KZ836292.1 de la posició 2404239 a 2432662 i el KZ836236.1 de la posició 5925926 a 5927218. Finalment es determina que TR1 es troba a l’scaffold KZ836295.1, entre les posicions 2405251 i 2426165 a la cadena negativa (-), ja que és l’scaffold on s’obtenen millor resultats. L’score obtingut a partir de l’alineament amb T-coffee és de 1000 i l’alineament dels aminoàcids és molt bo.
El gen que codifica per aquesta proteïna té 13 exons, sent l’últim el que es troba la selenocisteïna. Conté un element SECIS en l’extrem 3’ UTR de la cadena negativa.
TR1 està formada per 499 aminoàcids i comença per metionina.
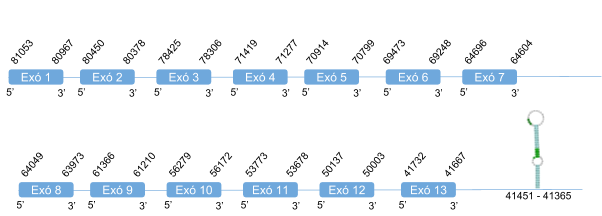
Posicions en valors absoluts:

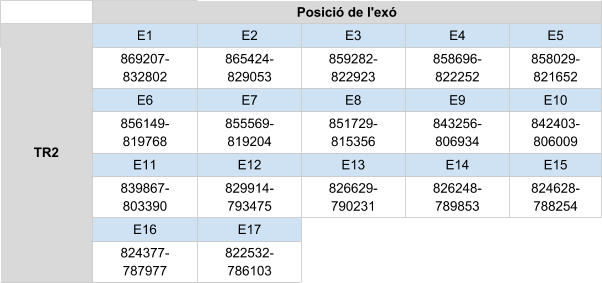
Per TR2 s’han analitzat els scaffolds KZ836237.1 (entre la posició 41561141 i 41562465), el KZ836236.1 (entre la posició 5925971 5927275) i KZ836406.1 (entre la posició 786203 i 822506). Finalment, s’afirma que la proteïna es localitza a l’scaffoldKZ836406.1 entre les posicions 786203 i 822506 de la cadena negativa (-), ja que els resultats obtinguts són molt bons, és a dir, l’score obtingut amb el T-coffee és de 995 i l'alineament entre els aminoàcids és molt bo.
El gen està format per 17 exons, la selenocisteïna es localitza a l’últim i té un element SECIS a l'extrem 3’ UTR de la cadena negativa.
La proteïna està formada per 525 aminoàcids. S’observa que els dos primers aminoàcids de la seqüència predita són V i un gap, el tercer és una metionina, per tant es considera que aquesta proteïna comença per metionina.
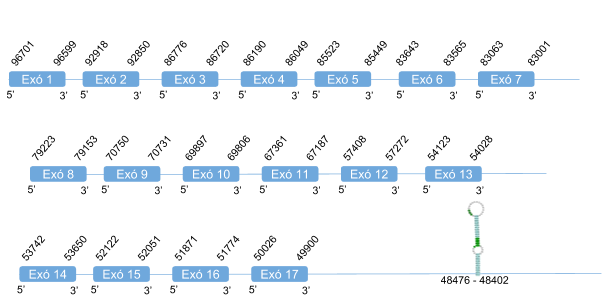
Posicions en valors absoluts:
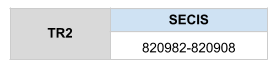
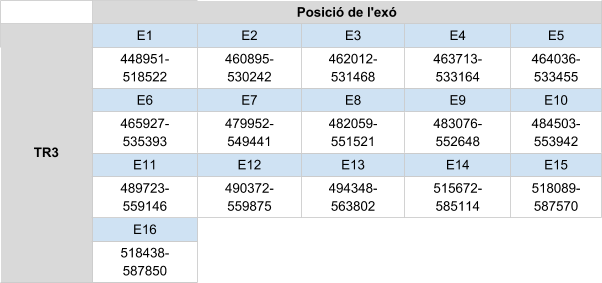
Finalment, per TR3 s’han analitzat dos scaffolds: KZ836237.1 en la posició 41561269-41562459 i KZ836502.1 en la posició 448879-518226. Després l’estudi, es va poder determinar que TR3 es troba a l’scaffoldKZ836502.1 entre les posicions 448879-518226 a la cadena positiva (+), ja que els resultats obtingut amb el T-coffee són bons. L’score és de 998 i l’alineament bo, tot i que en l’inici de l’alineament trobem un gap força gran.
El gen que codifica per TR3 té 16 exons, i la selenocisteïna es troba en l’últim exó. Es prediu un element SECIS a l’extrem 3’ UTR de la cadena positiva.
La proteïna TR3 està formada per 755 aminoàcids, i no comença amb metionina sino que com ja s’ha mencionat, comença amb un gran gap. Aquesta gap pot ser degut a una mala anotació o una deleció en el genoma de Callorhinus ursinus o perquè l’inici de la proteïna es troba codificada en un altra scaffold.
Finalment, si ens fixem en la imatge (Figura 3) observem que les proteïnes predites es troben properes filogenèticament a la proteïna de referència corresponent.
Posicions en valors absoluts:
La síntesi de selenoproteïna es basa en selenocistein-tRNASec (Sec-tRNASec), seqüència d'inserció de selenocisteïna (SECIS) i altres elements de mRNA de selenoproteïna, un factor proteic in-transSECIS binding protein2(SBP2) i eEFSec. La proteïna eEFsec (factor d’elongació eucariòtic) és una proteïna de la maquinària de la síntesis de les selenoproteïnes. És una GTPasa que interacciona amb el complex format pel SECIS i la SBP2, promovent la unió del tRNA-Sec al ribosoma [3].
A l’alinear la seqüencia proteica de eEFsec humana amb el genoma humà es van donar molts hits en diferents scaffolds. Es va analitzar aquell que va donar lloc a l’e-value menor, KZ836298.1, a més a més era el que cobria major part de la query. Els resultats que es van obtenir eren bons, l’score obtingut amb el T-coffee és de 999. Tot i tenir un score molt bo l’alineament mostra un gap molt gran a l’inici de la seqüència i un petit gap al final. Tot i això eEFsec es va predir en aquest scaffold.
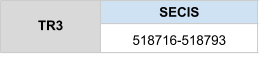
eEFsec es troba l’scafolld KZ836298, entre les posicions 925333-925992, a la cadena positiva (+). És una proteïna homòleg en cisteïna, és a dir, no conté selenocisteïna, ha perdut el seleni convertint-se en una cisteïna. El gen que codifica per aquesta proteïna està format per dos exons. Seblastian va predir un element SECIS entre els dos exons, per tant pot ser rebutjat, no es troba a l’extrem 3’ UTR de la cadena positiva.
eEFsec està formada per 596 aminoàcids i no comença per metionina. La presència d’aquest gap tant gran a l’inci de la seqüència pot indicar una gran deleció en el genoma de Callorhinus ursinus o una mala anotació, però també pot ser degut a que la part inicial d’aquesta proteïna es troba codificada a un altra scaffold i no s’ha tingut en compte.

Posicions en valors absoluts:
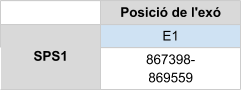
SBP2 és un factor auxiliar de la proteïna eEFsec, exclusiu d’eucariotes. Aquesta proteïna està associada de manera estable als ribosomes i conté un domini RNAL7Ae, el qual uneix els elements SECIS amb una gran afinitat i especificitat [3].
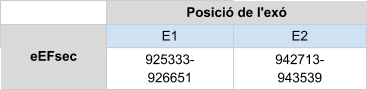
Al alinear el genoma de Callorhinus ursinusamb la proteïna humana SBP2 es van donar diferents hits en tres scaffoldsdiferents. Primer es va analitzar l’scaffoldamb millor e-value (KZ836331.1), i el resultat obtingut amb el T-coffee no és massa bo. Això pot ser degut a que el hit cobreix només una petita part de la query, tot i que el percentatge d’identitat és elevat. Posteriorment, s’ha analitzat el mateix scaffold, però aquest cop tenint en compte tots els hits que s’han donat a la cadena negativa (-). El resultat obtingut del T-coffee és molt bo (score = 994).
El gen està format per 11 exons. Aquesta proteïna és homòloga en cisteïna, per tant no conté selenocisteïna. Tot i així s’ha predit un element SECIS a l’extrem 3’ UTR, lo qual pot ser degut a que anteriorment fos una selenoproteïna que ha perdut el seleni que forma la selenocisteïna, però que manté l’element SECIS. Encara que com el SECIS es troba a més de 20000 parells de bases, aquest es rebutjaria.
Aquesta proteïna homòloga en cisteïna està formada per 856 aminoàcids, i no comença per metionina. En el T-coffee s’observa que hi ha un gran gap a l’inici de la seqüència predita. Això pot ser degut a una deleció en el genoma o bé una anotació errònia.
Posicions en valors absoluts:
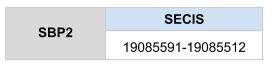
La funció d’aquesta proteïna consisteix en generar el donador de seleni (Se) necessari per a la biosíntesis de la selenocisteïna. És l’única proteïna de la maquinaria que pot ser una selenoproteïna. La selenofosfat sintetasa té dos gens paràlegs, SPS1 i SPS2. Estudis recents han demostrat que SPS1 sorgeix de duplicacions del gen SPS2 [1].
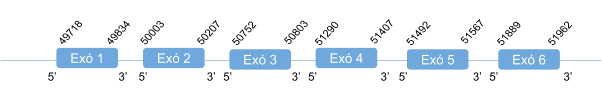
SPS2 és una selenoproteïna que està present en tots els vertebrats, per tant, Callorhinus ursiuns també la té. En els mamífers el gen que codifica per aquesta proteïna està format per un únic exó [1].
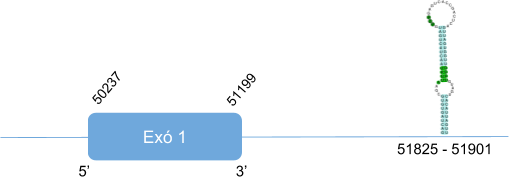
En l’alineament de la query de SPS1, d’Homo sapiens, i el genoma de Callohrinus ursinuss’han donat hits en quatre scaffoldsdiferents. S’ha analitzat el que donava un e-value menor, és a dir, l’scaffoldKZ836392.1. Posteriorment s’ha vist amb el T-coffee que no es realitza un molt bon alineament, i a més a més, es superposa amb la predicció de SPS2.
Per tant, s’analitza l’scaffoldKZ836391.1 entre les posicions 867161 i 868360, a la cadena positiva (+), el qual cobreix gairebé tota la llargada de la query i té un percentatge d’identitat molt bo. En l’anàlisi amb T-coffee s’obtenen bons resultats (score=997), però per contra, la predicció no comença amb metionina i a l’inici hi ha un gap. La causa d’aquest gap pot ser deguda a una deleció en el genoma de Callorhinus ursinuso una mala anotació d’aquest.
El gen que codifica per SPS1 només té un exó i no conté selenocisteïna, ja que és un homòleg en cisteïna. Malgrat això, s’ha predit un element SECIS a l’extrem 3’ UTR, fet que pot estar degut a una pèrdua de la selenocisteïna però que manté el element SECIS.
La proteïna està constituïda per 392 aminoàcids.
Posicions en valors absoluts:

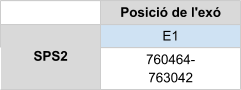
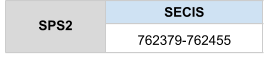
En l’alineament de la seqüència proteica de SPS2 humana amb el genoma de Callorhinus ursinus s’han donat hits en quatre scaffolds diferents. Es va analitzar l’scaffold amb e-value menor, KZ836392.1, i que a més a més era el que cobria major part de la query. Els resultats que es van obtenir a l’alinear la seqüencia proteica predita en Callorhinus usinus amb la query de referencia van ser molt bons, l’score obtingut amb el T-coffee va ser de 998 i l’alineament era bo. Per aquesta raó es va definir que SPS2 està ubicada a l’scaffold KZ836392.1, entre les posicions 760536 i 761774a la cadena positiva (+). S’ha predit un element SECIS a l’extrem 3’ UTR.
La proteïna està constituïda per 448 aminoàcids i comença per metionina.
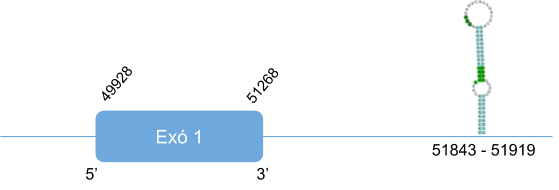
Posicions en valors absoluts: