CONCLUSIONS
Aquest treball té com a objectiu principal identificar i anotar les selenoproteïnes, les proteïnes de maquinària associades i les proteïnes homòlogues en cisteïna presents al genoma de Callorhinus ursinus. Els resultats s’han obtingut a partir de la comparació del genoma d’aquest ós marí amb les proteïnes de la base de dades de SelenoDB de l’Homo sapiens.
A nivell filogenètic, Canis lupus és una de les espècies més propera a Callorhinus ursinus, però la predicció i anotació de les seves selenoproteïnes es troba incomplerta. Per tant s’ha escollit a l’Homo sapiens com a espècie de referència, ja que és el mamífer que té les selenoproteïnes més ben anotades. A més a més tant Callorhinus ursinus, com l’Homo sapiens són mamífers placentaris, de manera que les proteïnes es troben molt ben conservades. S’han pogut predir la majoria de las selenoproteïnes en la nostra espècie, pel fet que els mamífers placentaris són unes espècies que estan molt conservades.
Per agilitzar el análisis i poder obtenir uns resultats òptims, s’ha creat un programa automatitzat en llenguatge bash per els següents programes: tblastn, Fastafetch, Fastasubseq, exonerate, Fastaseqfromgff, Fastatranslate i T-coffee. A més, s’ha fet ús del Seblastian i SECISearch3 per poder comprovar la presència d’elements SECIS a la proteína predita, ja que són essencials per la síntesis de las selenoproteïnas. A més a més, s’ha utilitzat un altre programa per aquelles families complexes, per poder analitzat un a un els scaffolds d’interès de cada proteïna. Tot i així, en el tblastn, s’han posat diferent condicions a la comanda per optimitzar la obtenció del hits.
Després de l'anàlisis exhaustiu del resultats i discutir-los es va determinar que Callorhinus ursinus tenia un total de 17 selenoproteïnes que comencen per metionina i per tant tenen l’anotació completa i 4 que tenen l’anotació incompleta, 4 homòlogues en cisteïna amb anotació completa i 5 amb anotació incompleta, 2 possibles selenoproteïnes amb anotació completa i 2 amb anotació incompleta i per últim 1 proteïna de maquinària amb anotació completa i 3 amb anotació incompleta.
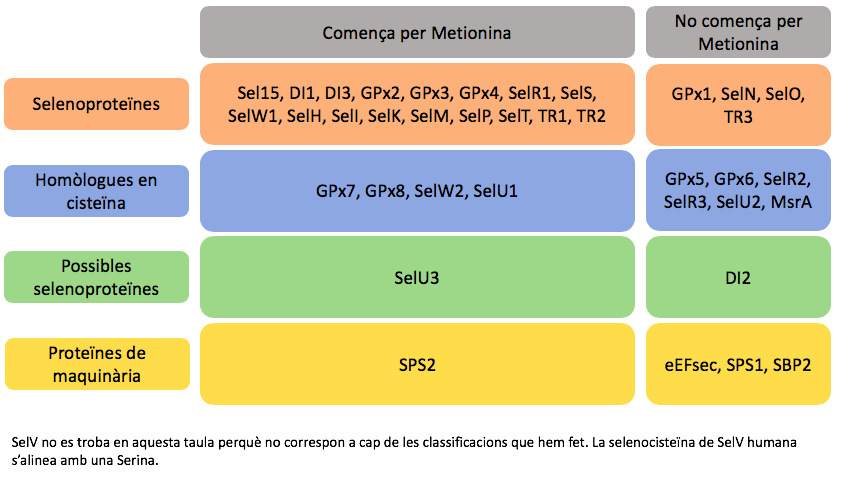
Totes les selenoproteïnes predites estan conservades i corresponen també a selenoproteïnes en l’Homo sapiens. Tot i això una possible selenoproteïna que s’ha predit és SelU3, la qual és homòloga en cisteïna en l’humà. El que observem, com ja s’ha mencionat en la discussió, és que una arginina humana s’alinea amb una selenocisteïna.
Respecte les possibles selenoproteïnes que no podem assegurar degut a que no estan ben anotades, trobem entre elles SelV. Aquesta segons la literatura [8] és una selenoproteïna que va sorgir d’una duplicació de SelW i que és la menys conservada en els mamífers. Això coincideix amb els nostres resultats ja que el grau de semblança entre les seqüències alineades és molt baix.
Respectes les proteïnes que s’han caracteritzat com homòlogues en cisteïna, la gran majoria es conserven respecte l’Homo sapiens. Exceptuant GPx6, la qual en humà és una selenoproteïna. S’hipotitza que en algun moment de l’evolució de Callorhinus ursinus es va donar aquest canvi, de selenocisteïna a cisteïna.
El procediment realitzat per a l’obtenció dels resultats i l’anàlisi d’aquest, presenta algunes limitacions. Les primeres limitacions que ens vam trobar van ser relacionades amb els pocs coneixements que teniem sobre la programació i les selenoproteïnes. En segon lloc, els resultats que s’han obtingut s’ha fet a partir de l’estudi de l’homologia amb selenoproteïnes anotades en altres espècies. Per tant, no podem detectar selenoproteïnes en el nostre genoma, el de Callorhinus ursinus, que no hagin estat anotades en el de referència. A més a més, com a proteïnes de referència es van utilitzar les de l’Homo sapiens, una espècie no gaire propera a Callorhinus ursinus, però que té les selenoproteïnes molt ben anotades.
El nostre mètode de treball es va basar en escollir de manera automàtica mitjançant un programa, el hit que tenia millor e-value. Aquest mètode ens va permetre superar la limitació del temps, ja que no disposavem del temps suficient per aprendre i programar com fer l’automatització per tal de que s’analitzessin tots els scaffolds. Aquest sistema ens va limitar la detecció de duplicacions d’algunes proteïnes i tampoc ens ha permès detectar si alguna proteïna es trobava dividida en dos scaffolds. Hagués estat més acurat estudiar més scaffolds i tenir en compte tots els paràmetres que ens proporciona el Blast. Tot i això, quan els resultats obtinguts no eren adecuats vam analitzar altres scaffolds amb un programa alternatiu. Per altre banda, un altre punt a tenir en compte és la subjectivitat alhora d’escollir el hit, en funció dels valors adequats de l’e-value, la llargada del hit i percentatge de similitud.
Tot i així, aquest treball ens ha permès aprendre més sobre les selenoproteïnes i la programació, tant de l’html com de bash.