En aquest treball comptàvem amb una base de dades a la qual hi ha els scaffolds d’una espècie que ha estat seqüenciada aquest mateix any 2018: el Xiphophorus hellerii. L'objectiu d’aquest treball és predir i anotar les selenoproteïnes, els homòlegs amb cisteïna i la maquinària implicada en el processament de les selenoproteïnes que presenta l’espècie que estem analitzant. Hem predit i anotat aquestes proteïnes del X. hellerii alineant i buscant homologies amb una altra espècie propera que ja les té notades: l’espècie Danio rerio, àmpliament estudiada. Aquesta ens aporta dos beneficis: per una banda, té gran proximitat filogenètica amb l’espècie estudiada, i per altra constitueix un model d’estudi ben establert. Per comparar les selenoproteïnes una a una, hem utilitzat la base de dades selenoDB versió 2.0, on hi ha anotades les selenoproteïnes del Danio rerio.
El fet d’utilitzar com a model un altre peix facilita la tasca, ja que els peixos presenten particularitats genòmiques, com duplicacions, i hi trobem més selenoproteïnes de les que es troben en altres espècies com l’Homo sapiens (humà) o el Mus musculus (ratolí).
Hem fet un anàlisi d’un total de 50 proteïnes (entre les quals hi ha selenoproteïnes, homòlegs en cisteïna i maquinaria implicada al processament de les selenoproteïnes), dividides en 26 famílies. Per fer la discussió hem valorat els següents paràmetres:
- Nombre d’scaffolds als qual vam obtenir una predicció significativa
- Nombre d’exons
- Longitud de la proteïna predita
- Percentatge d’identitat: hem fet servir un programa creat per nosaltres amb Perl que ens ha predit el nombre d’aminoàcids modificats.
- Puntuació de l’aliniament amb el T-coffee
- Inici de l’alineament amb metionina o no
- Alineament amb selenoproteïna amb el Seblastian
- Predicció d’una estructura 3D de mRNA associat a selenoproteïna amb el SECISearch3: només s’han tingut en compte aquells que es troben a la regió 3’UTR de la mateixa cadena.
Hem dividit la discussió en dos parts: selenoproteïnes, i homòlegs amb cisteïna i maquinària. En cas de no comentar explícitament la presència d’una modificació de les selenocisteïnes, indiquem que aquest aminoàcid s’ha conservat a la seqüència predita. Per suportar visualment la predicció dels gens hem elaborat unes taules on figura la localització de cada exó, i amb un color diferent es veu l’exó on es troba la selenocisteïna.
SELENOPROTEÏNES
Sel15
Aquesta selenoproteïna, també coneguda com a selenoproteïna 15 que es troba conservada a tots els vertebrats. Té un pes de 15 kD i està localitzada al reticle endoplasmàtic. Té un domini tiorredoxina i es pensa que pot tenir una funció redox. La seva funció principal és garantir un plegament correcte de les glicoproteïnes. Consta de 4 exons i té un element SECIS.

Nosaltres hem predit una selenoproteïna homòloga al Xiphophorus hellerii situada a l’scaffold QPIH01000031.1, posició 2933324-2936882 (cadena -) i consta de 4 exons. Aquesta té una longitud de 146 aminoàcids i la puntuació de l'alineament amb el T-coffee és de 1000. El percentatge d’aminoàcids canviats és un 20%, tenint en compte els gaps. No comença per metionina (tampoc la notació del Danio rerio) i al començament de l’aliniament observem que hi falta un fragment, probablement degut a que la predicció no ha sigut molt acurada en aquesta regió. Presenta un element SECIS grau A que valida la nostra predicció com a selenoproteïna.
SELENOE
Aquesta selenoproteïna només està present en peixos. Està distantment relacionada amb la proteïna sel15 i per això també és coneguda com a Fep15 (Fish selenoprotein 15). Al igual que la sel15, també està present al reticle, però la seva funció encara no està del tot clara. Té 4 exons i presenta un element SECIS.

Al X. hellerii hem trobat una proteïna homòloga de 3 exons localitzada a l’scaffold CM010386.1, posició 11685291-11696937 (+) i amb un SECIS de grau A. La longitud d’aquesta és de 138 aminoàcids i la puntuació de l’aliniament de 999. No comença per metionina perquè la predicció no comença amb l’inici de la query, que sí comença amb aquest aminoàcid. El percentatge d’aminoàcids canviats és 40%.
FAMÍLIA GPx
La família de les glutatió peroxidases és el conjunt de selenoproteïnes i homòlegs més gran dels vertebrats. Són enzims amb una funció principalment destinada a combatre el dany oxidatiu produït en múltiples processos metabòlics, el manteniment de la homeòstasi redox i la senyalització de la senyalització per H2O2. Això ho fan reduint el peròxid d’hidrogen (H2O2) i altres hidroperòxids orgànics mitjançant la donació d’electrons per part del glutatió (GSH). Consta de 7 selenoproteïnes: 5 (Gpx 1-4) amb duplicacions a les GPx1,3 i 4 al Danio rerio i 2 (Gpx7-8) homòlegs amb cisteïna, que probablement es van duplicar en algun moment i van anar divergint fins perdre la selenocisteïna.
Nosaltres hem pogut predir totes aquestes proteïnes, però vam obtenir molts hits amb bons E-values en cada cas. Per tal de seleccionar el millor hit per cada selenoproteïna vam realitzar un arbre filogenètic amb les Gpx del Xiphophorus hellerii i el Danio rerio, i així vam poder assegurar-nos que els scaffolds seleccionats eren correctes.
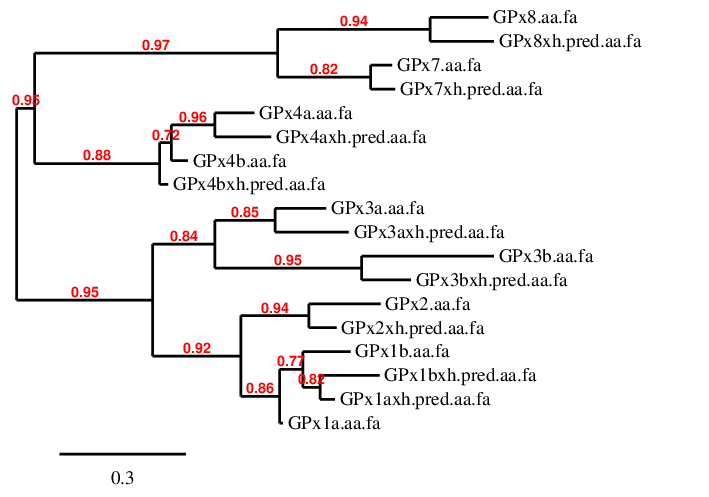
GPx1a
Aquesta selenoproteïna (Gpx1) es troba duplicada en el genoma de Danio rerio. Consta de 2 exons al Danio rerio i presenta un element SECIS.

En la nostra predicció del Xiphophorus hellerii vam trobar una proteïna homòloga de 190 aa de longitud i només un 22% d’aquests s’han modificat. La seva localització és a l’scaffold CM010394.1, posició 12274991-12276069 (-), amb 2 exons i un element secis de grau A. El Seblastian també ha sigut capaç de predir un alineament amb una selenoproteïna. Només la query comença per metionina, perquè la nostra predicció no s’ha pogut estendre fins el començament de la proteïna, però la puntuació de l’aliniament amb el T-coffee és de 998.
GPx1b
Aquesta selenoproteïna al Danio rerio consta de 2 exons i presenta un element SECIS.

Al Xiphophorus hellerii hem trobat una homologia situada al mateix scaffold que l’anterior (CM010394.1), però ara en posició 15404926-15405861 (+). També presenta 2 exons i un element SECIS de grau A. Comença per metionina, igual que la query, ja que tenen la mateixa llargada, i la seva puntuació al T-coffee és de 1000. Veiem un 21% d’aminoàcids modificats.
GPx2
Aquesta selenoproteïna, també coneguda com a glutatió peroxidasa intestinal, està formada per 2 exons al Danio rerio i presenta dos elements SECIS.

Vam trobar una homologia al Xiphophorus hellerii, situada a l’scaffold CM010393.1, posició 21007004-21007736 (-) que té una llargada de 191 aminoàcids el 23% modificats respecte del Danio rerio, repartits en 2 exons. Comença per metionina i té exactament la mateixa longitud que la query, de fet presenta una puntuació de 1000 a l’aliniament del T-coffee.
GPx3a
Danio rerio presenta dues selenoproteïnes GPx3. La GPxa presenta 5 exons i un element SECIS.

Al Xiphophorus helleriii hem predit una proteïna de 222 aminoàcids de longitud a l’scaffold CM010397.1, posició 10073847-10075835 (-). Les dues es troben a la cadena reverse, En aquest cas presenta 4 exons. Tot i que presenta un 40% d’aminoàcids modificats, la puntuació de l’aliniament en el T-coffee és de 998. És més curta que la query, ja que l’aliniament no ha ha arribat fins a cap dels dos extrems, potser en aquests gaps hi havia l’exó de diferència que observem entre la notació del Danio rerio i la nostra predicció. No comença en metionina pel fet que no comença al començament de la proteïna del Danio rerio. El SECISearch3 ha predit un element SECIS de grau A.
GPx3b
Aquesta proteïna, codificada en la cadena forward, es composa de 4 exons i té un element SECIS, segons el SelenoDB.

En En la nostra predicció al Xiophophorus hellerii hem trobat una homologia a l’scaffold CM010384.1, posició 8315501-8317319 (+). També consta de 4 exons i una longitud de 178 aminoàcids. D’aquests, trobem un 36,5% modificats al Xiphophorus hellerii respecte del Danio rerio contant els gaps. La puntuació de l'alineament del T-coffee és de 1000, però trobem que la query s’ha alineat amb gaps a l’extrem terminal. Les dues comencen per metionina. Tot i que al selenoDB hi ha anotat un element SECIS al Danio rerio, el SECISearch3 no ha sigut capaç de trobar cap a la seqüència del X. hellerii.
GPx4a
Al Danio rerio aquesta proteïna es composa de 4 exons i presenta un element SECIS.

En la nostra predicció trobem una proteïna homòloga a l’scaffold CM010380.1, posició 20047163-20048260 (+), que presenta 3 exons, amb una llargada de 113 aminoàcids, sense gaps, i una puntuació de 1000 a l'alineament del T-coffee. Veiem un 20% d’aminoàcids canviats i ambdues proteïnes comencen per F. A més, el Seblastian ha pogut fer un alineament amb una selenoproteïna i el SECISearch 3 ha trobat un element SECIS de grau A.
GPx4b
Al Danio rerio aquesta proteïna es composa de 6 o 7 exons (en funció de la isoforma) i presenta un element SECIS.

Al Xiphophorus hellerii hem trobat una proteïna homòloga a aquesta a l’scaffold CM010380.1, posició 18822399-18823643 (+), amb 4 exons. Té una longitud de 130 aminoàcids (1 menys que el Danio rerio) i només un 20% d’aquests han patit una modificació. L’aliniament amb el T-coffee té una puntuació de 999. No comença amb metionina, però tampoc ho fa la del Danio rerio. El Seblastian ha pogut fer un alineament amb una selenoproteïna i el SECISearch ha trobat un element secis de grau A.
FAMÍLIA DIO
La família de les iodotironina deiodinases (DIO) està formada per 3 proteïnes (DIO1-3) implicades en el metabolisme de les hormones tiroïdals: catalitzen la activació i inactivació d’aquestes hormones. Els peixos ossis presenten una duplicació de la proteïna DIO3, trobant les proteïnes DIO3a i DIO3b.
En aquesta família (igual que a les Gpx) com que hi ha homologia intra-familiar, hem trobat hits amb bons E-values a diferents scaffolds per cada proteïna. Per determinar quin scaffold pertany a cada proteïna vam realitzar un arbre filogenètic que relaciona la predicció feta en cada scaffod amb la proteïna homòloga corresponent al Danio rerio
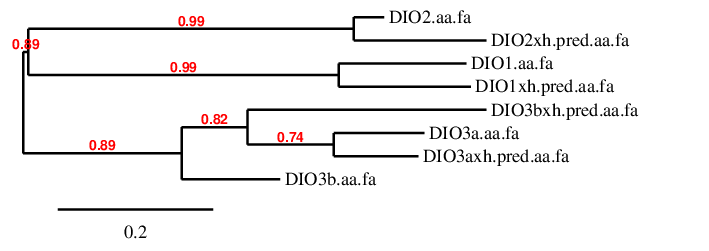
DIO1
DIO1 és una proteïna de membrana de 4 exons codificada a la cadena reversa. Bioquímicament és una iodotironina deiodinasa. Les seves funcions són activar la prohormona tiroxina (T4) per iodació de l’anell extern i passar-la a forma T3 al fetge, i també degradar les dues anteriors per desiodació de l’anell intern. No s’ha trobat en peixos pulmonats. Aquesta selenoproteïna presenta dos SECIS.

Hem pogut fer una predicció d’una proteïna homòloga a X. hellerii a l’scaffold CM010383.1, posició 17199477-17201178 (-) amb 3 exons i un element SECIS de grau A. Té una longitud de 226 aminoàcids, igual que la de Danio rerio, i presenta un 40% d’aminoàcids modificats. Tant la proteïna predita com la query comencen per leucina.
DIO2
Aquesta proteïna també controla l’activitat de les hormones tiroïdals als diferents teixits. La seva funció és la mateixa que la del DIO1. Té tres 3 exons i presenta un element SECIS.

La nostra predicció d’aquesta proteïna es troba a l’scaffold CM010393.1, posició 19019999-19024709 (-), té una longitud de 266 aminoàcids i consta de 2 exons. Comença, igual que la del Danio rerio, amb metionina i el percentatge d’aminoàcids modificats és 27%. La seva puntuació de l'alineament del T-coffee és 986. Tot i que trobem 3 elements SECIS, només un d’ells es troba a l’extrem 3’UTR de la cadena forward. El Seblastian no ha pogut predir una selenoproteïna, però a la nostra predicció sí que trobem, igual que a la query, una selenocisteïna.
Si parem atenció a l’output del T-coffee trobem alguns gaps intercalats a l’aliniament, tant a la proteïna query com a la predita. Probablement hi ha hagut una translocació d’uns 6-7 aminoàcids. Hipotetitzem això perquè a una part hi ha un gap d’aquesta longitud a la predicció i poc després el trobem a la query. Hipotetitzem que hi ha hagut una regió, que es va inserir en un lloc diferent dins de la mateixa proteïna i a partir d’allí va començar a divergir. Com la trobem dins d’una família de proteïnes, considerem que pot haver-se modificat perquè en aquesta família hi ha moltes proteïnes homologues amb funcions semblants i fins i tot redundants (com és el cas de la DIO1) i per tant pot haver adquirit una nova funció, o potser la seva funció ha quedat inactivada i ara es troba en forma de pseudogen. Potser això també afecta a l’estructura dels exons, i per aquest motiu veiem que en la proteïna del Danio rerio hi ha 3 exons i en la nostra predicció només 2.
DIO3a-b
S’ha observat una duplicació de DIO3 en tots els peixos ossis, donant dues proteïnes (DIO3a i DIO3b). La seva funció és la de deiodar i inactivar els metabòlits T3 i T4. Consten de dos exons. Segons el SelenoDB, la proteïna DIO3a no presenta cap element SECIS però la DIO3b en sí que en presenta un.

A la nostra predicció de la proteïna DIO3a només trobem un exó. Aquestes dues proteïnes es troben molt properes al genoma, de fet, nosaltres les hem trobat en el mateix scaffold (CM010389.1) i la mateixa cadena (reverse). La localització de DIO3a al genoma és 5274023-5274790 (-), la longitud en la seqüència proteica és de 267 i el percentatge d’aminoàcids modificats és d’un 28%. L’aliniament del T-coffee presenta una puntuació de 980 i el Seblastian ha predit un element SECIS de grau A. No comença per metionina, ja que és lleugerament més curta que la proteïna de Danio rerio: comença uns 7 aminoàcids després i n’acaba 1 abans. Suposem que això és degut a que la predicció no ha estat molt acurada als extrems.

Pel que fa a la proteïna DIO3b, hem trobat una proteïna homòloga a l’scaffold CM010389.1, posició 2463779-2464399 (-), d’una longitud de 245 aminoàcids, amb un percentatge de 50% aa modificats. La puntuació de l’aliniament és de 988 i el Seblastian no ha pogut predir cap element SECIS. Veiem que aquest alineament no és molt bo, trobem molts gaps i al principi falta una regió gran.
Igual que en el cas del Danio rerio, trobem SECIS a una de les proteïnes (DIO3b) i no a l’altra (DIO3a). Pero en aquest cas veiem que el SECIS està al DIOI3a. També hem trobat que a l’alineament del DIO3b hi ha molts gaps i regions que no es troben. Podem hipotetitzar que, com que els dos es troben al mateix scaffold, potser s’han confós a l’hora de fer les prediccions i s’ha predit la DIO3a en la posició de la DIO3b i a l'inrevés (tot i que a l’arbre filogenètic ha sortit coherent en relació amb el Danio rerio).
En el cas de la proteïna DIO3b pensem que s’ha perdut un exó a l’inici ja que concordaria amb el fet que a la original hi ha 2 exons i en aquesta hem predit només 1. A més, com que la funció d’aquestes dues proteïnes és la mateixa, és factible que una d’elles (DIO3b), al modificar la seva estructura d’exons, hagi modificat la seva funció o l’hagi perdut o es quedi en forma de pseudogen.
SELENOH
Aquesta selenoproteïna (també anomenada selenoproteïna M o Birthday selenoprotein) es troba localitzada al nucli i té activitat peroxidasa (antioxidant). Consta de 1 o 2 exons (en funció de la isoforma) i presenta un element SECIS.

L’homologia trobada consta de 3 exons, ubicada a l’scaffold CM010397.1, posició 3980050-3982040 (-). Té una longitud de 127 aminoàcids, amb un 46% d’ells modificats. No comença per metionina i és lleugerament més curta que la query. Comença amb un alineament no molt bo de tres aminoàcids (només hi ha un match), i després trobem que falta un fragment de 8 aminoàcids a la nostra predicció. A partir d’aquest punt sí que hi ha un bon alineament; tot i que la identitat no és molt alta, els aminoàcids que han estat substituits s’assemblen bioquímicament. El Seblastian ha fet un alineament amb selenoproteïna i el SECISearch3 ha trobat un element SECIS a l’extrem 3’UTR.
SELENOI
Tot i que no es coneix la funció concreta, SELENOI s’ha vist associada a membranes, tant d’orgànuls (reticle endoplasmàtic i aparell de Golgi) com la membrana cel·lular, fent de transferasa al procés de biosíntesi dels fosfolípids. És una proteïna de 10 exons amb un element SECIS.
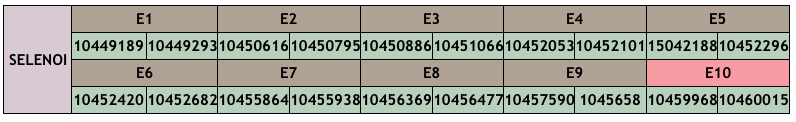
Quan vam fer el tBLASTn d’aquesta proteïna al genoma del Xiphophorus hellerii vam trobar 3 scaffolds que donaven hits amb bons E-values: CM010389.1, CM010391.1 i CM010398.1. Al fer els alineaments amb el T-coffee i avaluar la identitat dels aminoàcids entre la predicció i la query i la puntuació de l’alineament, vam arribar a la conclusió que l’únic scaffold al qual trobem una bona homologia és el CM010389.1, posició 10449189-10460015 (-), on trobem els 10 exons que conté la proteïna del Danio rerio (als altres dos només en trobavem 2 exons). La longitud de la proteïna predita és de 397 aminoàcids (igual que la notació d’aquesta proteïna al SelenoDB), amb un 20% d’ells diferents dels de la query. Ambdues comencen per tirosina (Tyr). El Seblastian ha predit una selenoproteïna que comença per metionina i el SECISearch3 ha trobat un element SECIS de grau A.
SELENOJ
Aquesta proteïna no es troba en altres vertebrats i, a diferència de la majoria de les selenoproteïnes, s’ha determinat que té una funció estructural.
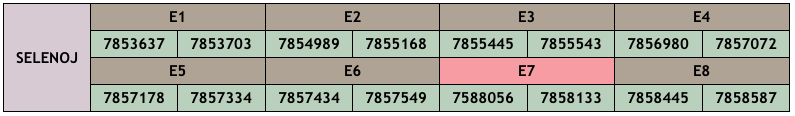
Hem trobat hits en 3 scaffolds diferents: CM010395.1, CM010380.1 i CM010384.1; tots tres amb una longitud al voltant dels 343 aminoàcids i amb 8, 9 i 2 exons respectivament.
Tot i que l’scaffold amb un E-value més estadísticament significatiu és el CM010395.1, hem comprovat l’scaffold CM010380.1 ja que mostrava més hits significatius i no només ens ha donat un score major (999 vs. 994), sinó que hi havia un nombre menor de gaps i es mantenia l’inici en metionina.
Hem comprovat també el 3r scaffold (CM010384.1) i, tot i que l’score és de 999 (igual que al 2n scaffold) i el nombre d’aminoàcids canviats és també molt superior (82,74% vs. 20,76%), trobem un canvi molt interessant: Hem observat un alineament d’una selenocisteïna amb una cisteïna i per tant, podríem hipotetitzar que el X. hellerii ha evolucionat fent aquest canvi. Tot i això, observem una selenocisteïna en la nostra seqüència predita uns aminoàcids desplaçada i per tant, descartem la hipòtesi que hi ha hagut una pèrdua d’aquesta selenoproteïna.
L’scaffold CM010380.1 es troba a la posició 21613050-21615865 (-) i presenta un element SECIS a l’extrem 3’UTR de grau A, fet que valida la nostra predicció com a selenoproteïna.
SELENO K
Aquesta selenoproteïna (també anomenada selenoproteïna G) està localitzada a la membrana del reticle endoplasmàtic i es troba altament expressada el cor, on es creu que podría funcionar com antioxidant. Té 5 exons, 4 d’ells codificants, i presenta un element SECIS.

Al genoma del Xiphophorus helleri vam trobar un hit significatiu a l’scaffold CM010375.1, posició 8808974-8810614 (+). Al fer la predicció vam obtenir una proteïna de 4 exons, amb una longitud de 109 aminoàcids i un 60% d’ells modificats. Tot i que la puntuació del T-coffee és bona (955) i ambdues proteïnes comencen en metionina, no hem obtingut un bon alineament: hi ha molts gaps i molts missmatches. No podem fer hipòtesis sobre una posible evolució. Hem fet una comprovació de la qualitat realitzant una nova predicció amb el Genewise i ens ha sortit igual, probablement la predicció no hagi estat bona. El Seblastian no ha pogut fer cap predicció de selenoproteïna, tot i que el SECISearch3 sí que ha trobat un element SECIS de grau B a l’extrem 3’UTR.
SELENOL
Aquesta proteïna pertany a la subfamília SelL, dins d’una superfamília de tiorredoxines (Trx). Es composa d’11 exons i té una distribució filogenètica poc usual, només present en organismes aquàtics, conseqüentment, els únics vertebrats als quals es troba són els peixos. S’ha inclós dins aquesta superfamília pel fet de posseir un domini amb un plegament thioredoxin-like. Presenta un element SECIS en Danio rerio.
El plegament de tiorredoxina es forma per la presència de dues cisteïnes separades per dos aminoàcids qualsevol. En aquesta superfamília s’han substituit les cisteïnes per selenocisteïnes (SxxS), que produeixen el plegament thiorredoxin-like, estructura implicada en procesos redox.

En el cas de la SELENOL del Xiphophorus hellerii hem apreciat que aquest domini no s’ha conservat: la seqüència d’aa predita és WxxS. Això és important des del punt de vista de la funció, ja que si no es conserva aquest domini, probablement la funció tampoc estarà conservada. A més, és interessant que l’aminoàcid que ha substituït la cisteïna -un aminoàcid petit- és un triptòfan -l’aminoàcid més gran i amb menor taxa de substitució-. L'única propietat bioquímica que aquests dos aminoàcids tenen en comú és que són hidrofòbics.
La proteïna homòloga trobada se situa l’scaffold QPIH01000029.1, posició 6475243-6478731 (+). Té una longitud de 302 aminoàcids i 4 exons. L’aliniament del T-coffee presenta una puntuació de 995. El percentatge d’aminoàcids canviats és molt alt, un 71%, això és degut, sobretot, a la gran quantitat de gaps que trobem, sobretot al principi de la proteïna. La query de Danio rerio comença per metionina però aquesta no, degut a que falta una regió del començament. Probablement s’han perdut exons, ja sigui al fer la predicció o en l’evolució d’aquest peix. S’ha predit tant una selenoproteïna com un element SECIS gràcies al Seblastian i el SECISearch3.
SELENOM
Aquesta selenoproteïna es troba al reticle endoplasmàtic i participa en la formació d’enllaços disulfur. Conté un domini redox que es pensa que és regulat per una selenocisteïna.

El gen d’aquesta selenoproteïna es situa a la posició 15339676-15345368 (-) i conté 5 exons. La nostra seqüència predita té una longitud de 127 aminoàcids amb un percentatge de 26,77% aminoàcids canviats respecte a la query corresponent al Danio rerio, fet que ens fa pensar que està bastant conservada. Malgrat que la nostra predicció és una mica més curta que la query (hi ha un petit gap de 13 aminoàcids a l’inici), té un score de 1000, indicant de que existeix una homologia molt bona entre ambdós seqüències.A més, el Seblastian ens ha pogut fer un alineament amb una selenoproteïna i el SECISearch3 ens ha predit elements SECIS de grau A, confirmant-nos que el X. hellerii conté aquesta selenoproteïna.
SELENON
Aquesta selenoproteïna de 12 exons es troba localitzada a la membrana del reticle endoplasmàtic de tots els teixits. Es desconeix la seva funció exacta.
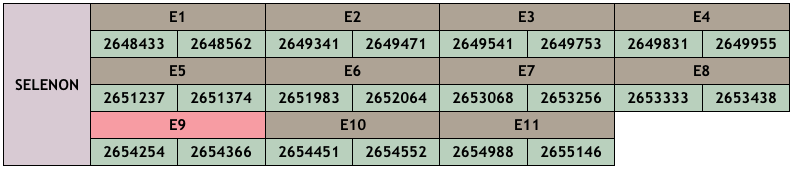
La proteïna homòloga que nosaltres hem trobat es localitza a l’scaffold CM010396.1, posició 2648433-2655146 (+) i conté 11 exons. Té una longitud de 557 aminoàcids i un 69% d’identitat. La puntuació de l’aliniament del T-coffee és de 999. Veiem que al començament de l'alineament falta una àmplia regió que no es troba a la nostra predicció, això quadra amb el fet que la proteïna de Danio rerio conté 12 exons i a la nostra predicció hem trobat només 11. Probablement aquesta àmplia regió del començament correspon a l’exó de diferència que observem en aquesta proteïna. El Seblastian ha predit un alineament amb una selenoproteïna que comença amb Metionina i el SECISearch3 ha trobat un element SECIS de grau A a l’extrem 3’UTR.
FAMÍLIA SELENOO
Aquesta família està formada per dues selenoproteïnes en Zebrafish: SELENOO1 i SELENOO2. Estan localitzades als mitocondris de la majoria d’organismes (animals, plantes, bacteris i llevats) i la seva funció és desconeguda.
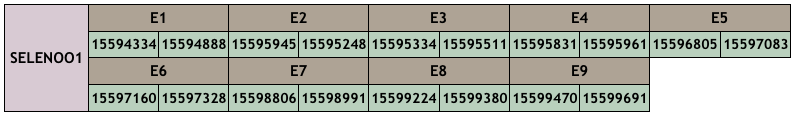
En la SELENOO1, que té 9 exons, hem trobat hits amb E-values significatius a l’scaffold QPIH01000032.1, a la posició 15594334-15599691 (+) de longitud 787 però ens donava un score molt baix (831). Hem canviat l’scaffold (CM010391.1) i al fer-ho ha incrementat l’score 200 punts.
QPIH01000032.1
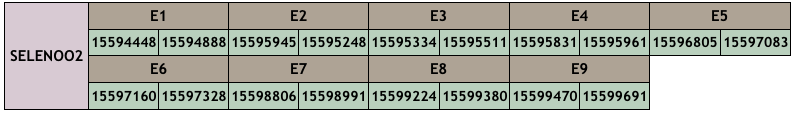
CM01391.1
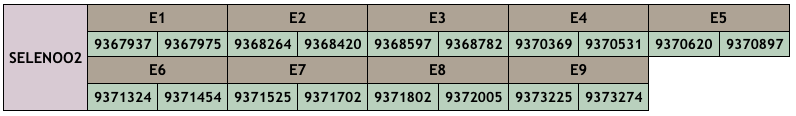
Pel que fa a la SELENOO2, que també té 9 exons, l’scaffold amb un millor E-value era el mateix que el primer que havíem analitzat del SELENOO1, però ens donava un score de 680. A l’analitzar el segon millor scaffold amb hits significatius, hem trobat que era el mateix que el que havíem triat del SELENOO1 (CM010391.1). En analitzar la puntuació d’aquest segon scaffold hem pogut observar un increment de 300 punts.
Hem descobert que la SELENOO2 és la mateixa proteïna que la SELENOO1 i, tot i que la seva seqüència en alguns exons és diferent, coincideixen en el número d’exons (9) i en la seva major part en seqüència i localització.
Al Danio rerio la SELENOO2 i la SELENOO1 són gens diferents però observem que al X. hellerii són 2 isoformes del mateix gen, és a dir, són proteïnes paràlogues que tenen un origen comú i alguns exons diferents.
Pel que fa a l’alineament amb el T-coffee, a la SELENOO1 trobem Sec en ambdós seqüències però no s’alineen, mentre que en SELENOO2 no observem cap selenocisteïna en la nostra predicció.
Tot i que el SECISearch3 sí que ha trobat un element SECIS de grau B a l’extrem 3’UTR, el Seblastian no ha pogut fer cap predicció de selenoproteïna.
SELENOP1
La primera isoforma de la proteïna (SELENOP1) de Danio rerio conté 4 exons codificants que no presenten element SECIS.

A Xiphophorus hellerii s’ha trobat una proteïna homòloga a l’scaffold CM010386.1, posició 11356279-11357667 (+), amb una puntuació d’aliniament de 963, i vé codificada per 3 exons. Consta de 368 aminoàcids, dels quals el 75% són diferents en comparació al genoma de referència, un percentatge força alt que ens fa sospitar d’un error a la predicció o que aquesta proteïna s’ha modificat molt al llarg de l’evolució d’ambdues espècies. Sí que comença per metionina i és més curta que la query. Després de fer el Seblastian no s’ha pogut predir cap alineament amb una selenoproteïna, però el SECISearch3 sí que ha predit dos elements SECIS a la cadena forward (+), tot i que ambdós són de grau B.
SELENOP2
La segona isoforma de la proteïna (SELENOP2) de Danio rerio també conté 4 exons que, a més, presenten dos elements SECIS a la cadena forward (+).

Al genoma de l’espècie que estem estudiant, hem pogut obtenir un proteïna homòloga a l’scaffold CM010380.1, posició 23063771-23065044 (+), amb una puntuació d’aliniament de 999, codificada per 3 exons. La proteïna SELENOP2 consta de 327 aminoàcids, i d’aquests el 67% s’han modificat quan s’ha comparat amb el genoma de Danio rerio. Encara que el peix zebra sí que comença per metionina, la seqüència de X. hellerii no s’alinea fins després d’un gap petit. A més d’aquest gap a l’inici, també hi ha un altre gap molt llarg al final, i aquest probablement és degut a la pèrdua d’un exó, cosa que coincideix amb la diferència d’exons entre la proteïna de Danio rerio i la precisió, o a un error en la predicció. El Seblastian sí que ha estat capaç de predir un alineament amb una selenoproteïna, i a més el SECISearch ha predit un element SECIS a la cadena forward (+) de grau A
FAMÍLIA MSRB1
Aquesta família, tal com el nom indica, té la funció de reduir la forma R de la sulfoximetionina a metionina. Està formada per selenoproteïnes i per homòlogues amb cisteïna, les quals es troben a una varietat de teixits tant adults com fetals i es localitzen al nucli de la cèl·lula i al citosol.
MSRB1a
La proteïna MSRB1a de Danio rerio està codificada per un gen que presenta 4 exons codificants i un element SECIS.

Per una altra banda, a l’espècie que estem estudiant, s’ha trobat una proteïna homòloga en cisteïna a l’scaffold CM010390.1, posició 1472471-1474052 (+), amb una puntuació d’aliniament de 1000, i està codificada per 3 exons, un menys que al peix zebra.Consta de 110 aminoàcids amb un 24% d’aquests modificats en comparació al genoma de referència. Comença també per metionina i és més curta que la query, ja que al final de la predicció hi ha un gap de 4 aminoàcids. Encara que són pocs, la falta d’aquests pot estar relacionada amb el possible exó perdut al llarg dels anys.
El Seblastian ha trobat una predicció d’un alineament amb una selenoproteïna, i a més amb el SECISearch hem pogut predir un element SECIS de grau A a la cadena forward (+).
MSRB1b
La proteïna MSRB1b de Danio rerio està codificada per un gen que presenta 3 exons codificants i un element SECIS.

Al genoma de Xiphophorus hellerii s’ha trobat una proteïna homòloga en cisteïna a l’scaffold CM010384.1, posició 3504926-3504015 (-), amb una puntuació d’aliniament de 1000, i ve codificada per 3 exons també. La llargada d’aquesta proteïna és de 104 aminoàcids, i d’aquests només el 26% són diferents en comparació al genoma de referència. A més, comença per metionina i té la mateixa llargada que la query, que juntament amb el percentatge anterior mostra que la seqüència ha estat força conservada al llarg del temps. Per últim, amb el Seblastian no ha estat possible predir un alineament amb una selenoproteïna, encara que el SECISearch sí ha predit un element SECIS de grau B a la cadena reverse (-).
SELENOS
La proteïna SELENOS (SelS, SEPS o VCP-interacting membrane protein) és una proteïna transmembrana que es localitza al reticle endoplasmàtic, i participa activament al procés de degradació de proteïnes que s’han plegat malament dins d’aquest orgànul. A més també exerceix un paper en el control de la producció de citoquines i, per tant, en el control de la inflamació. Té una isoforma que no conté Sec perquè no presenta l’element SECIS degut a que es tracta d’una variant del transcript amb un splicing alternatiu. Consta de 6 exons que codifiquen per a 190 aminoàcids amb un element SECIS.
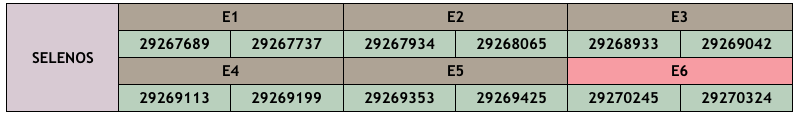
Al genoma de Xiphophorus hellerii, per altra banda, s’ha trobat una proteïna homòloga a l’scaffold CM010378.1, posició 29267689-29270324 (+), amb una puntuació alineament de 951, que ve codificada per 6 exons també. Està composta per 191 aminoàcids, un nombre gairebé igual a la proteïna de Danio rerio, amb un total del 43% dels aminoàcids modificats en comparació amb aquest. No comença per metionina, al contrari de la query, i aquesta és 11 aminoàcids més llarga. Això té sentit perquè la predicció que fa el programa no és perfecta.Per últim, el Seblastian ha predit un alineament amb una selenoproteïna i apart s’ha predit un element SECIS, el qual té un grau A i es troba a la cadena forward (+).
FAMÍLIA SELENOT
Aquesta família està formada per selenoproteïnes molt conservades en tots els vertebrats ja que s’encarreguen de protegir les neurones dopaminèrgiques davant l’estrès oxidatiu i la mort cel·lular gràcies a la seva funció oxidoreductasa.
SELENOT1

La SELENOT1, està localitzada a la posició 16282407-16288237 (+), té 5 exons, una longitud de 179 aminoàcids i únicament un 18,43% d’aminoàcids canviats respecte la query, fet que ens indica que hi ha hagut poca divergència. Ambdós comencen per metionina i, tot i que l’score és de 977, s’observa una homologia molt bona al T-coffee.
SELENOT1b

En la SELENOT1b, l’E-value més baix correspon a un hit (QPIH01000030.1) que en realitat pertany a la proteïna SELENOT1. L’scaffold que hem triat de la SELENOT1b (CM010380.1) està localitzat a la posició 16286545-16286682 (+) i la nostra predicció proteica té una longitud de 194 aminoàcids, amb un 32% d’aminoàcids canviats i un score de 998. La predicció no comença en metionina, fet que ens fa pensar que li falta un exó ja que observem un gap bastant gran a l’inici de la seqüència.A més, aquesta selenoproteïna coincideix amb l’últim fragment de la SELENOT2 i per tant concloem que es superposen amb T2.
SELENOT2

El gen de la SELENOT2 es localitza a la posició 6159894-6163565 (+) i té 5 exons. La nostra seqüència predita té una longitud de 210 aminoàcids, amb un 37% d’aminoàcids canviats i un score de 991. En el T-coffee no només observem una homologia molt bona, sinó que ambdós comencen per metionina i acaben al mateix punt.
En les 3 selenoproteïnes hem obtingut elements SECIS de grau A amb el SECISearch3 i a més, el Seblastian ens les ha pogut alinear amb selenoproteïnes, fet que ens confirma que, encara que SELENOT1b es superposi a SELENOT2, es troben en el genoma del X. hellerii.
FAMÍLIA SELENOU
La família de selenoproteïnes SelU s’ha restringit al llarg de la evolució sobretot a peixos i aus, encara que també es troben a algunes espècies d’organismes unicel·lulars eucariotes. En canvi, en mamífers, com per exemple humans, trobem homòlogues amb cisteïna en comptes de selenocisteïna. Les proteïnes d’aquesta família contenen un motiu UxxC (U per selenocisteïna i C per cisteïna) al mateix lloc on hi ha un motiu CxxC a les proteïnes humanes homòlogues, de les quals sí que en coneixem més. Per això es pot plantejar la idea de què ambdues comparteixen funcions similars, i en aquest cas seria participació en la regulació redox a l’interior cel·lular.
SELENOU1a
El gen SELENOU1a de Danio rerio presenta diversos transcripts, dels quals ens hi fixarem en el que té 6 exons i codifica per a una proteïna de 212 aminoàcids amb un SECIS.
Al genoma de X. hellerii, per altra banda hem trobat una proteïna homòloga a dos scaffolds:

La primera d’elles està a l’scaffold CM010396.1, posició 7301407-7302953 (+), amb una puntuació d’aliniament de 999, codificada per 5 exons.SELENOU1a té una llargada de 222 aminoàcids, i d’aquests el 29% són diferents. No presenta metionina, i potser s’ha perdut un exó a l’inici del gen.S’ha pogut predir una selenoproteïna i un SECIS de grau A que es troba a la cadena forward (+).

La segona d’elles es troba a l’scaffold CM010384.1, posició 14514886-14516459 (-), amb una puntuació d’aliniament de 1000, i està codificada per 5 exons.Consta de 222 aminoàcids, dels quals el 38% estan modificats. No comença per metionina ja que, encara que hi ha una major homologia, comença amb un gap de 12 aminoàcids on es pot haver perdut un exó. S’ha predit un alineament amb una selenoproteïna a més d’un SECIS de grau A que també es troba a la cadena forward (+).
FAMÍLIA SELENOW
La família de proteïnes SELENOW es caracteritza per ser les selenoproteïnes més petites que contenen l’aminoàcid Sec, encara que la seva funció encara no hi és clara. S’expressa a diversos teixits, tant a organismes procariotes com a eucariotes, però es troba en major quantitat al múscul esquelètic de mamífers. Presenta 3 isoformes, de les quals vam seleccionar només la SELENOW2 -que comença amb metionina- , perquè la SELENOW1 no donava cap hit significatiu (segurament degut a la longitud) i la SELENOW3 era més curta que seleccionada (i a més, hem detectat que estan superposades).
SELENOW2
La proteïna SELENOW2 a Danio rerio vé codificada per 4 exons, presenta 94 aminoàcids i un SECIS.

A X. hellerii s’ha trobat una proteïna homòloga a l’scaffold CM010390.1, posició 10142819-10145708 (-), amb una puntuació d’aliniament de 1000 i conté també 4 exons.Consta de 95 aminoàcids i d’aquests, el 22% es troben modificats en comparació al de referència. Comença per metionina i té la mateixa llargada que la query. El Seblastian ha predit dues selenoproteïnes diferents, ambdues comencen per metionina i presenten un element SECIS de grau A cadascuna que suposem que corresponen a les dues isoformes (W2-3).
FAMÍLIA TXNRD
TXNRD2
La proteïna TXNRD2, coneguda com a Tioredoxina Reductasa 2, és un enzim dimèric NADPH-dependent que conté FAD i que catalitza la reducció del centre actiu disulfit de la tioredoxina i altres substrats. TXNRD2 conté 17 exons i codifica per a una proteïna d’una llargada de 515 aminoàcids. Encara no s’ha trobat un SECIS.
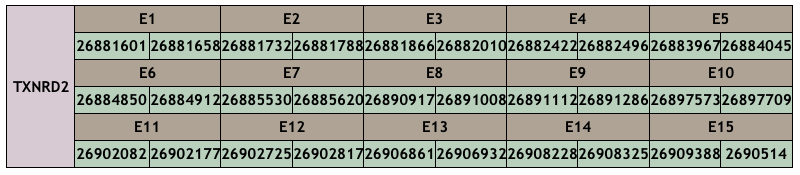
A X. hellerii hem trobat una proteïna homòloga a l’scaffold CM010386.1, posició 26881601-26909514 (+), amb una puntuació d’alineament de 998, i presenta 15 exons.Aquesta proteïna conté 486 aminoàcids, on s’han vist modificats el 20% d’ells en comparació amb el genoma que hem agafat com a referència. No comença per metionina, i és igual de llarga que la query. No ha estat possible fer una predicció d’un alineament amb una selenoproteïna. Sí que s’han pogut predir dos SECIS, pero cap dels dos és vàlid.
És possible que a aquesta proteïna hagi perdut un exó al llarg de la seva història evolutiva i per això presenta menys exons i aminoàcids que la proteïna de Danio rerio, o potser la nostra predicció no és tan bona encara que haguem obtingut una puntuació alta al T-coffee.
TXNRD3
TXNRD3, també coneguda com a tioredoxina reductasa 3 és una proteïna que pertany a la família de les oxireductases del nucleòtid de piridina. Per tant, la seva funció principal consisteix en catalitzar la reducció de tioredoxina, i al mateix temps participa a la defensa contra l’estrès oxidatiu. Aquesta proteïna conté un domini glutaredoxina N-terminal adicional que els altres isozims de la mateixa família no presenten, i consta de 16 exons. A més, presenta un element SECIS.
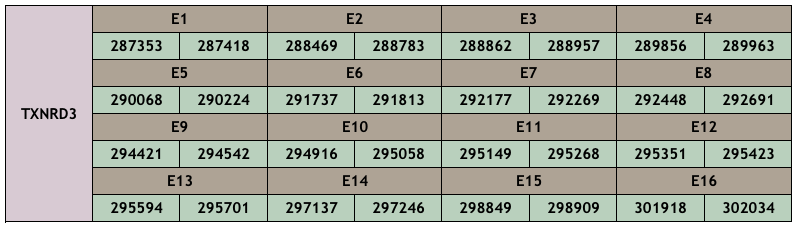
A X. hellerii hem aconseguit trobar una proteïna homòloga a QPIH01000031.1, a la posició 287353-302034 (-), amb una puntuació d’aliniament de 999, i té 16 exons.Presenta un total de 611 aminoàcids, dels quals un 20% estan modificats en comparació amb el de referència. A més, també comença per metionina i té un llargada similar a la de la query. El Seblastian no ha estat capaç de predir cap alineament amb una selenoproteïna. Tot i que s’han trobat dos SECIS, un d’ells queda descartat perquè es troba a la cadena forward, mentre que l’altre es troba al 3’UTR de la reverse i a més és de grau A.
MAQUINÀRIA I HOMÒLEGS AMB CISTEÏNA
eEFsec
Tot i que aquesta proteïna també és coneguda com a selenoproteïna B, no és en realitat una selenoproteïna, sinó un factor d’elongació eucariòtic, un tRNA específic per a selenocisteïna. Participa en la detoxificació del peròxid d’hidrogen amb la seva funció de glutatió peroxidasa. Consta de 7 exons i no presenta cap element SECIS.
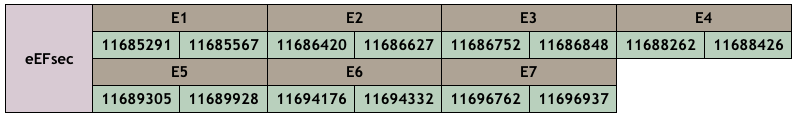
Nosaltres hem fet una predicció al Xiphophorus hellerii d’una proteïna amb una longitud de 579 residus i una puntuació de 990 a l’alineament realitzat pel T-coffee. Es localitza a l’scaffold CM010394.1, posició 11685291-11696937 (+). No comença per metionina perquè la predicció no ha arribat fins el començament i la nostra predicció és una mica més curta. El percentatge d’aminoàcids canviats és un 24%. Tot i que la proteïna del Danio rerio no presenta cap SECIS, el Seblastian ha predit un SECIS de grau B.
GPx7
Aquesta proteïna (també anomenada GPx6 de vegades) tot i pertànyer a la família de les glutatió peroxidases, no és una selenoproteïna sinó un homòleg amb cisteïna. Consta de 3 exons i no té cap element SECIS.

Hem trobat una homologia al genoma del Xiphophorus hellerii, a l’scaffold CM010383.1, posició 30565348-30567174 (+), amb 2 exons. Té una longitud de 147 aminoàcids (igual que la notació del Danio rerio al SelenoDB) amb un 27% modificats. Ambdues comencen per Q. El Seblastian no ha predit cap selenoproteïna i els dos SECIS que s’han trobat no són vàlids.
GPx8
Igual que l’anterior, aquesta és un homòleg amb cisteïna de la família de les glutatió peroxidases. Es composa de 3 exons i no presenta cap element SECIS.

Al Xiphophorus helleriii hem trobat un homòleg de 3 exons a l’scaffold CM010386.1, posició 5404930-5406775 (+).Té una longitud de 135 aminoàcids (igual que la notada al SelenoDB) amb un 23,5% d’ells modificats. Ambdues comencen per R. El SECISearch 3 ha predit un element SECIS que ha quedat descartat per trobar-se a la cadena reversa.
MsrA
Aquesta proteïna catalitza la reducció enzimàtica de de sulfoxi-metionina a metionina. En alguns organismes unicel·lulars eucariotes i bacteris anaerobis, MsrA conté una selenocisteïna al centre catalític d’aquests enzims redox que els confereix avantatges catalítics. Tot i això, en vertebrats s’ha vist que ha degenerat a cisteïna.

El gen que codifica per aquesta proteïna homòloga en cisteïna té 4 exons, es localitza a la posició 7596286-7607098 (-) i la seva longitud en la nostra predicció de la seqüència proteica és de 238 aminoàcids, amb un score de 999 i un percentatge d’aminoàcids canviats de 61,76%. En el nostre cas, no trobem ni a la query (Danio rerio) ni a la seqüència que hem predit del Xiphophorus hellerii cap Seci, encara que el SECISearch3 ens prediu un element SECIS, aquest no és vàlid per no trobar-se a la regió 3’ UTR. Per tant, reafirmem que s’ha produït una degeneració a cisteïna, tal com especifica la literatura quan la descriu en els vertebrats.
Cap de les dues seqüències comença per metionina però s’inicia la seva predicció per una arginina. Tot i això, la predicció no és molt acurada als extrems ja que observem grans gaps tant a l’inici com al final de la predicció.
PSTK
Aquesta quinasa és una proteïna de maquinària que està implicada en la catalització de la fosforilació de seril-tRNA[Ser]Sec mitjançant ATP i gràcies a la seva funció es pot produir la codificació específica UGA de les selenocisteïnes.

El gent que codifica per aquesta proteïna conté 2 exons i es situa a la posició 11875083-11875398 (-). Ni en la query ni en la predicció hem trobat cap selenocisteïna i el SECISearch 3 no ens ha predit cap element SECIS. La longitud és de 205 aminoàcids i és homòloga a la nostra predicció amb un score de 999. El percentatge d’aminoàcids canviats respecte al Danio rerio és de 41%. Cap de les dues seqüències comença per metionina, fet que ens fa pensar que la predicció està incompleta i per tant requeriria un anàlisi alternatiu en un futur amb una altra espècie per arribar a tenir una predicció més acurada i fiable.
SBP2
Aquesta proteïna de maquinària es troba exclusivament en eucariotes i s’encarrega, tal com indica el nom (SECIs binding protein 2), de la unió als elements SECIS i de codificar el codó UGA per a selenocisteïna.
CM010378.1

CM010386.1

En la nostra predicció al Xiphophorus hellerii, hem obtingut hits en 2 scaffolds però ni a l’scaffold CM010378.1 ni a l’scaffold CM010386.1 la predicció coincideix completament amb la proteïna (veiem un gap gran tant al principi com al final). La longitud d’ambdós scaffolds és de 985 i 982 respectivament i cal remarcar que observem una millora en l’score quan analitzem el 2n scaffold (CM010386.1): passa de 973 a 996, però aquest canvi no es tradueix en una millor homologia (veiem una homologia més bona en l’scaffold CM010378.1). Tanmateix, hem comprovat si ambdós scaffolds es superposen i no és el cas.
Cal destacar que en ambdós scaffolds tant la nostra predicció com la query comencen amb metionina i el percentatge d’aminoàcids modificats és 81,99 i 93,99% en cadascun, fet que ens fa pensar que és una proteïna que ha divergit molt. Tot i que s’ha predit un element SECIS, aquest no és vàlid per no trobar-se a la prosició 3’ UTR. Hi ha hagut una homologia perquè el seu centre actiu s’ha conservat però tot i això, no considerem que sigui una selenoproteïna sinó una proteïna homòloga en cisteïna.
SecS
Aquesta proteïna forma part de la maquinària biosíntesi de les selenocisteïnes, concretament participa en la conversió de O-fosfo-L-seril-tRNA[Ser]Sec a selenocisteil-tRNA[Ser]Sec gràcies a l’ús del selenofosfat com a donador de seleni, és a dir, és l’encarregada d’unir la selenocisteïna al tRNA que les incorpora a les selenoproteïnes. Tot i participar en aquesta funció tan essencial, no conté selenoproteïnes ni a la query ni a la nostra predicció, fet que ens indica que és una proteïna homòloga en cisteïna.
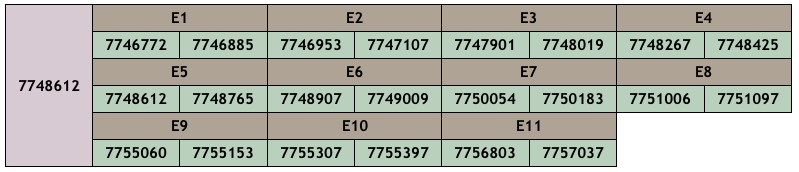
La seqüència predita del X. hellerii té una longitud de 490 aminoàcids i un score de 999 amb 17,95% aminoàcids canviats respecte la query, fet que ens mostra que hi hagut poca divergència. Observant el T-coffee veiem que s’estableix una homologia molt bona entre la query i la nostra predicció: ambdós comencen per metionina i acaben pràcticament en el mateix punt: la predicció acaba 8 aminoàcids abans però és segurament causa d’imperfeccions a l’hora de predir els extrems. Es localitza a la posició 7746772-7757037 (+), consta de 11 exons i el Seblastian no ha predit cap selenoproteïna en la nostra seqüència. El SECISearch3 ha predit un element SECIS, però aquest queda descartat perquè no es troba a la regió 3’UTR.
SEPHS2
Aquesta selenoproteïna està implicada en la síntesi del selenofosfat encarregat de donar el seleni per sintetitzar la selenocisteïna.
QPIH01000032.1
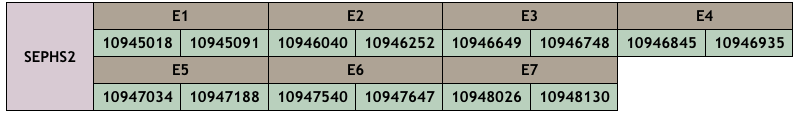
CM010375.1
En aquesta proteïna hem trobat homologies significatives en dos scaffolds: QPIH01000032.1, amb longitud 447, i CM010375.1, amb longitud 451.
Encara que l’E-value del primer scaffold (QPIH01000032.1) era més baix, hem comprovat l’scaffold CM010375.1 ja que mostrava un gran nombre de hits significatius i tot i que ens ha donat un score molt menor (966 vs. 1000), hi ha un nombre molt menor de gaps, ambdós comencen per metionina i les selenocisteïnes s’alineen entre la query i la predicció. A més, observem una segona selenocisteïna a la predicció que s’alinea amb una arginina a la query. Tanmateix, hem observat un menor nombre d’aminoàcids canviats al 2n scaffold (CM010375.1): 31,26% vs. 50,11%, fet que ens indica que és més probable que estiguin més propers al D. rerio evolutivament.
El gen que codifica per l’scaffold QPIH01000032.1 està localitzat a la posició 10945018-10948130 (-), té 7 exons i s’ha predit un element SECIS de grau B, però no és vàlid per no trobar-se a la regió 3’ UTR. Tot i així, aquesta proteïna no conté cap selenocisteïna, ja que es tracta d’un homòleg de cisteïna. Aquesta fet fa pensar que segurament al llarg de l’evolució la Sec s’ha reemplaçat per una Cys.
SECp43
Aquest component de la maquinària és un tRNA associat a selenocisteïna, implicat en la expressió de les selenoproteïnes.

Es composa de 9 exons al Danio rerio. Al Xiphophorus hellerii hem trobat una dos scaffolds als quals hi havia hits amb e-values bons. A l’analitzar el primer (scaffold CM010387.1, posició 24604231-24605702 (+)), vam obtenir un alineament amb una puntuació molt bona (1000), però només hi havia 2 exons i la identitat era del 16%. Llavors vam analitzar l’altre scaffold (CM010380.1, posició 20120767-20126275 (+)) i, tot i que la puntuació de l’aliniament era menor (979), la identitat era del 65% i sí que vam trobar 9 exons. Vam descartar, per tant, el primer scaffold. La longitud de la proteïna predita és de 322 aminoàcids i tan sols acaba 6 aminoàcids abans que la query. Ambdues comencen per metionina. El SECISearch3 va predir un element SECIS que vam descartar per trobar-se a la cadena reversa (igual que a la proteïna de Danio rerio, no esperàvem trobar cap element SECIS, ja que no es tracta de una selenoproteïna).
MSRB2
Aquesta proteïna és una homòloga en cisteïna present en tots els organismes excepte en alguns paràsits i hipertermòfils. El gen de la MSRB2 està situat a la posició 11012583-11013908 (-) i consta de 4 exons.

La proteïna per la qual codifica té una longitud de 180 aminoàcids, un 34% d’aminoàcids canviats i un score de 999. Tot i que la nostra predicció no s’alinea fins després d’un petit gap, la query comença en metionina. Pensem que aquest gap al seu inici pot ser degut a la pèrdua d’un exó o a un error en la predicció.Pel que fa als elements SECIS, se n’han predit dos de grau B amb el SECISearch3 però no es troben a la regió 3’ UTR i queden descartats. Concloem que aquesta proteïna està present al genoma del X. hellerii.
MSRB3
Aquesta proteïna és també una homòloga en cisteïna. Hem trobat hits en dos scaffolds: CM010395.1 i QPIH01000032.1, de longitud 186 i 188 aminoàcids i situats a les posicions 11012562-11012684 i 9266455-9279316 respectivament.
CM010395.1

QPIH01000032.1

Ambdós contenen 1 exó i, tot i que l’scaffold CM010395.1 tenia un E-value menor, hem comprovat l’scaffold QPIH01000032.1 perquè mostrava més hits significatius i, tot i que ens ha donat un score menor, observem un nombre molt menor de gaps i a més tant la query com la nostra predicció comencen per metionina.
Pel que fa al percentatge d’aminoàcids canviats, és molt menor en l’scaffold QPIH01000032.1 (36,7% vs 87,6%), fet que ens fa pensar que no només hi ha millor homologia entre les seqüències sinó que probablement la predicció s’ajusta més a l’scaffold QPIH01000032.1.Pel que fa als elements SECIS, al igual que amb la proteïna homòloga en cisteïna MSRB2, al fer l’anàlisi amb el SECISearch3 ens ha predit dos elements SECIS de grau B però no se situen a la regió 3’ UTR. Per tant, concloem que aquesta proteïna està present al genoma del X. hellerii.
SELENOU2
El gen que codifica per a la proteïna SELENOU2 de 225 aminoàcids del peix zebra consta de 6 exons i no té cap SECIS.
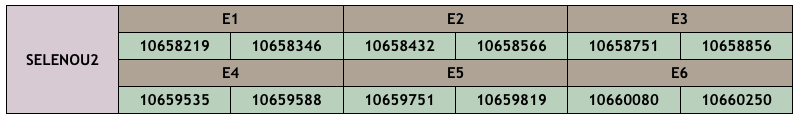
Per una banda, a X. hellerii s’ha trobat una proteïna homòloga a l’scaffold CM010386.1, posició 10658219-10660250 (-), amb una puntuació d’aliniament de 996, i prové de 6 exons. La proteïna conté 223 aminoàcids, i d’aquest són diferents el 43% en comparació amb el genoma de referència. No comença per metionina degut a què es poden observar dos gaps inicials què després es comencen a alinear amb una V. A més, té dos aminoàcids menys que la query. No va ser possible predir la selenoproteïna, i el SECIS que s’ha predit no és vàlid.
SELENOU3
Per últim, el gen que donarà lloc a la proteïna SELENOU3 conté 6 exons també i cap seqüència SECIS.
La proteïna homòloga que hem trobat a l’espècie en estudi està a l’scaffold CM010394.1, a la posició 17126170-17131955 (-), amb una puntuació d’aliniament de 1000, i conté 6 exons. La llargada d’aquesta proteïna és de 179 aminoàcids, i el 26% d’aquests es troben modificats en comparació amb el genoma de referència. Tot i que a l’inici no trobem metionina, ambdues seqüències comencen per V, i tenen la mateixa llargada. No ha estat possible predir la selenoproteïna, dels dos SECIS que s’han predit, només un es troba a la regió 3’ UTR (a la posició 25654-25732) però com que es troba molt allunyat, no el considerem vàlid i per tant, és un homòleg en cisteïna.