Discussió
Dins les selenoproteïnes analitzades hem diferenciat dos grups: les selenoproteïnes trobades a Astyanax mexicanus i les selenoproteïnes no trobades a Astyanax mexicanus.
Les selenoproteïnes que es van trobar al genoma analitzat van ser: Sel15; SelenoE; la família GPx amb GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, GPx7, GPx8; la família DIO amb DIO1 i DIO3a; SelH; SelI; SelJ; SelK; SelL; SelM; la família SelenoO amb SelO1; la família SelenoP amb SelP1 i SelP2; MsrA; la família SelenoR amb MsrB1A, MsrB2i MsrB3; la família SEPHS amb SEPHS1 i SEPHS2; la família SelenoT amb SelT1 i SelT1b; la família SelenoU amb SelU2 i SelU3; la família SelenoW amb SelW(I), SelW (II) i SelW(III); i, finalment la família TXNRD amb TXNRD2 i TXNRD3.
Pel que fa a les selenoproteïnes no trobades al genoma d’Astyanax mexicanus trobem les següents: GPx5 i GPx6; DIO2 i DIO3b; SelN; SelO2; SelS; SelT2; Sel1A, SelU1B i SelU1C i TXNRD1.
De la mateixa forma que amb les selenoproteïnes, a continuació es discutiran amb detall les següents selenoproteïnes de maquinària identificades a Astyanax mexicanus: SEPHS1 i SEPHS2; eEFsec, PSTK, SBP2, SecS, SECp43, TTPA, SELENBP1 i, finalment, RPL30.
Per altra banda les proteïnes de maquinària no trobades a Astyanax mexicanus són: ELAVL1, CELF1, EIF4A3 i, finalment, G6PD.
Les selenoproteïnes i proteïnes de maquinària d’Astyanax mexicanus (Tetra mexicà) han estat predites a partir de l’anàlisi d’homologia amb les selenoproteïnes i proteïnes de maquinària de Danio rerio (peix zebra) i Homo sapiens (humà). Es va escollir Danio rerio ja que pertany a la classe Actinopterygii, al igual que la nostra espècie, i presenta una elevada proximitat filogenètica. Per altra part, l’Homo sapiens, tot i ser una espècie més llunyana a la nostra, presenta una molt bona anotació del selenoproteoma i ens ha permès localitzar aquelles selenoprotïnes i selenoproteïnes de maquinària que no es van trobar a Danio rerio. A més, també es van buscar selenoproteïnes anotades en el genoma de Takifugu rubipres (peix fugu) que no estessin presents ni a Danio rerio ni a Homo sapiens. Es va escollir aquesta espècie ja que presenta una elevada homologia amb l’Astyanax mexicanus.
Cal comentar que totes les proteïnes que s’han agafat com a base per comparar han estat extretes de la base de dades SelenoDB 2.0. Es va observar que moltes d’elles no començaven amb metionina, fet que pot ser degut a l’anotació automàtica de la base de dades. Això no es va tenir en compte a l’hora de buscar les selenoproteïnes i proteïnes de maquinària del nostre genoma ja que pot ser que no s’hagi anotat el petit fragment que conté la metionina o que hi hagi algunes excepcions de proteïnes que no comencin amb metionina. Podem concloure, que les selenoproteïnes i proteïnes de maquinària predites a partir de proteïnes que no comencen per metionina no són la proteïna sencera però tot i així s’accepten com a vàlides a l’hora de comparar amb el nostre genoma. Per aquest motiu també ens trobem amb que la proteïna predita no comença amb metionina.
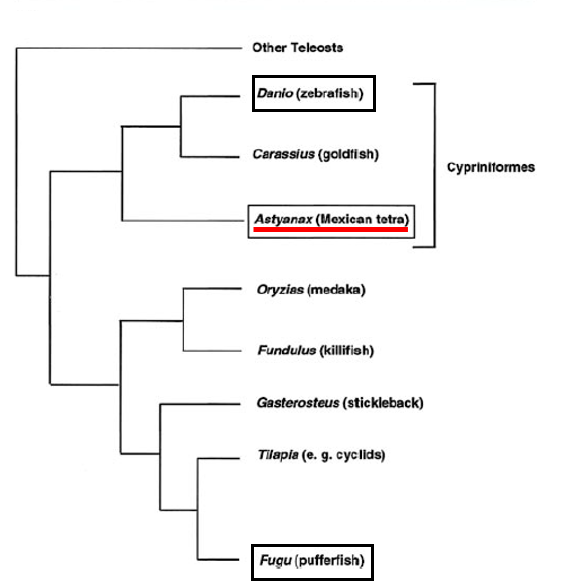
Figura 4.Arbre filogenètic dels peixos ossis
Com hem dit anteriorment, Astyanax mexicanus pertany al grup de peixos ossis. Aquest llinatge s’ha observat que posseeix un selenoproteoma major al dels mamífers. Aquest fet és degut a l’aparició de noves selenoproteïnes (SelenoE, SelenoJ i SelenoL), i a l’aparició de duplicacions (GPx1b, GPx3b, GPx4b, etc.). Per altra banda, també s’ha observat que aquest llinatge no hauria de presentar algunes selenoproteïnes com GPx5 i GPx6, que si que van aparèixer en mamífers placentaris. Tanmateix, durant l’evolució dels mamífers hi ha una tendència al reemplaçament de Sec per Cys en les selenoproteïnes.
A continuació s’analitzaran els resultats obtinguts comparant-los amb la bibliografia cercada sobre la conservació i evolució de selenoproteïnes en els peixos ossis.
Selenoproteïnes
Sel15
Aquesta proteïna de 15 kDa, que conté un domini thioredoxin-like, està situada al reticle endoplasmàtic regulant el correcte plegament de les glicoproteïnes. La quantitat de proteïna que trobem està directament relacionada amb els nivells de seleni present a l’organisme.
Aquesta proteïna es troba conservada en tots els vertebrats i també s’ha predit a Astyanax mexicanus però aquesta predicció no comença amb metionina. Cal tenir en compte que s’ha predit a partir de la Sel15 de Danio rerio anotada en el SelenoDB 2.0 que no té anotada aquesta metionina. Per aquest motiu pot ser que la proteïna predita estigui incompleta.
Aquesta proteïna es troba a l’scaffold APWO02000136.1 i conté 5 exons. Es va comprovar l’alineament amb t-coffee, donant un bon resultat. Aquesta proteïna presenta una selenocisteina (Sec) a l’exó 3, i presenta un element SECIS al extrem 3’ UTR. Tot i aquests bon resultats, Seblastian no va ser capaç de predir un alineament per una selenoproteïna en Astyanax mexicanus.
SelenoE
La SelenoE, també anomenada Fep15 (Fish Selenoprotein 15), és una proteïna exclusiva dels peixos. Es localitza al reticle endoplasmàtic i, tot i que la seva funció exacte es desconeix, es creu que podria estar involucrada en funcions úniques d’aquests organismes. Sembla haver-se originat per duplicació de SelM i només es troba com a selenoproteïna.
Durant l’anàlisi de la proteïna es va obtenir un únic scaffold, el APWO02000144.1, el qual presenta una elevada homologia amb la SelenoE de Danio rerio, la qual va ser comprovada a través de l’alineament amb t-coffee. A partir d’aquesta predicció i mitjançant el Seblastian es va poder observar que SelenoE conté 4 exons, presenta una selenocisteïna (Sec) en l’exó 2 i conté un element SECIS en l’extrem 3’ UTR.
Família GPx
La família de les glutatió peroxidases (GPx) és la família de selenoproteïnes més gran dels vertebrats i està implicada en múltiples processos fisiològics. Està formada per un conjunt d’enzims la funció més important dels quals és la protecció de l’organisme enfront el dany oxidatiu.
En mamífers, la família GPx està formada per 8 proteïnes homòlogues, de les quals cinc són selenoproteïnes (GPx1-4 i GPx6) i les altres han evolucionat a partir d’aquestes, amb la possibilitat que durant aquesta divergència hagin perdut la selenoproteïna. En el genoma d’Astyanax mexicanus s’han pogut predir correctament les proteïnes GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, GPx7 i GPx8 a partir del genoma de Danio rerio.
Tenint en compte que aquesta família de proteïnes és molt gran, ens hem trobat amb el problema de que després de realitzar cada blast obteníem molts scaffolds amb valors de e-value bons. Per tant, per tal d’assignar un scaffold a cada proteïna ens hem basat, per una part, en la llargada de l’scaffold, comprovant que aquesta fos més o menys la mateixa llargada de la query extreta del SelenoDB. Per altra part, ja que el resultat de l’alineament ens mostrava que un mateix scaffold (amb mateixes posicions genòmiques) podia correspondre a diferents proteïnes de la mateix família, va ser necessari fer un arbre filogenètic de la família. A continuació trobem l’arbre filogenètic de la família GPx que ens mostra la proximitat filogenètica corresponent entre les proteïnes d’Astyanax mexicanus i de Danio rerio.
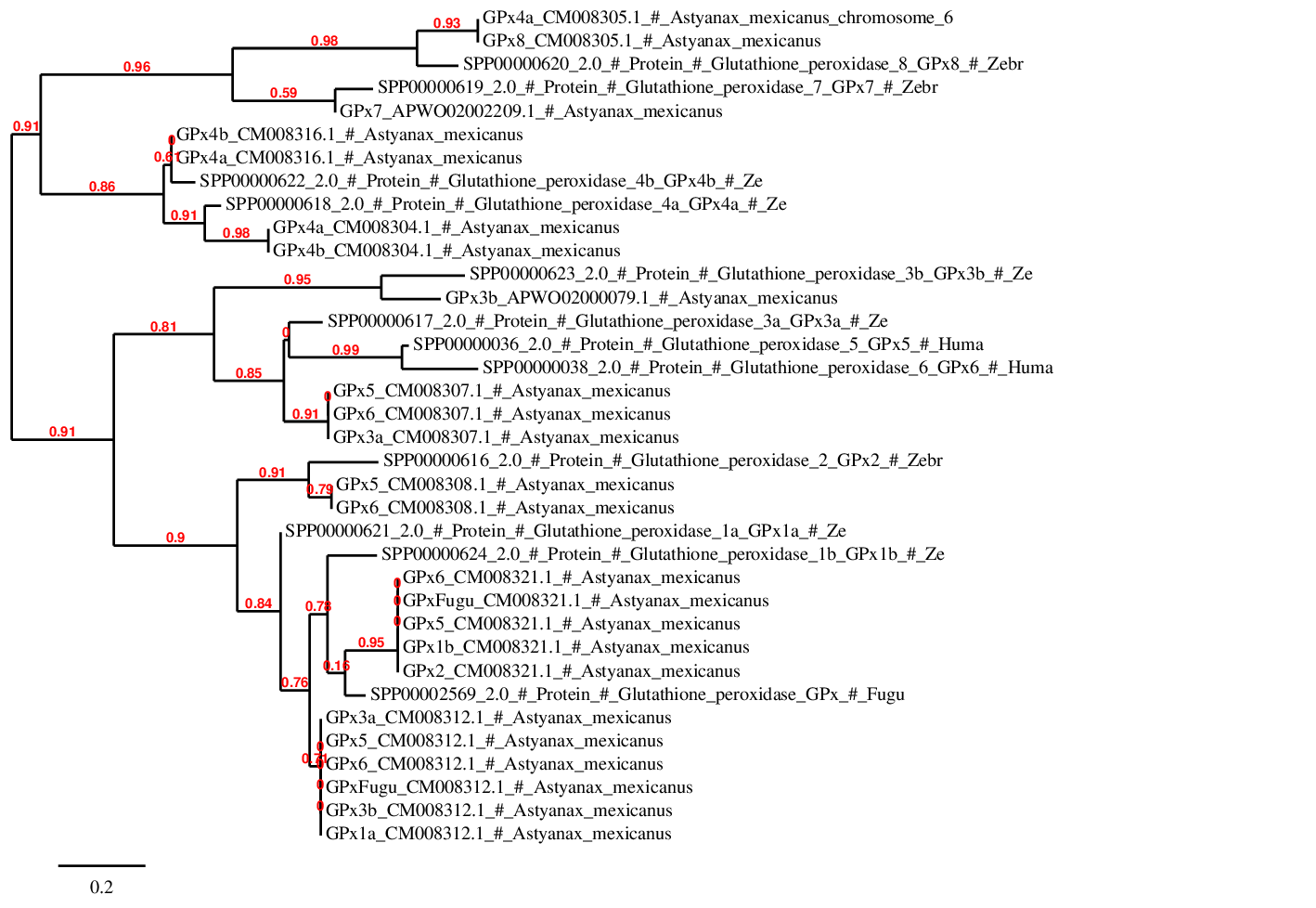
Figura 5.Arbre filogenètic de la família GPx
A partir de l’arbre es va estudiar a quina de les protïnes de Danio rerio estava més a prop filogenèticament cadascun dels scaffolds obtinguts en el nostre organisme fet que ens va permetre determinar a quina proteïna corresponia cada scaffold.
GPx1a
La proteïna GPx1a es localitza en l’scaffold CM008312.1. El gen que codifica per a la proteïna conté 2 exons i presenta una selenocisteïna (Sec) en l’exó 1. A través del t-coffee es va poder observar com presenta una elevada homologia amb la GPx1a de Danio rerio ja que l’alineament resultant fou molt bo. Un cop realitzada la predicció i a través del Seblastian es va poder localitzar un element SECIS 3’ UTR.
GPx1b
La proteïna GPx1b es localitza en l’scaffold CM008321.1. Es va comprovar l’alineament a través del t-coffee, donant un bon resultat. El gen que codifica per a la proteïna conté 2 exons, presenta una selenocisteïna (Sec) a l’exó 1 i un element SECIS en l’extrem 3’ UTR.
GPx2
En quan a la proteïna GPx2, aquesta es localitza en l’scaffold CM008308.1. Al igual que en la GPx1b, el gen que codifica per a la GPx2 conté 2 exons i presenta una selenocisteïna (Sec) a l’exó 1. Un cop comprovada la predicció observant l’alineament del t-coffee i mitjançant el Seblastian es va poder observar que conté un element SECIS en l’extrem 3’ UTR.
GPx3a
La proteïna GPx3a es localitza en l’scaffold CM008307.1. Presenta una elevada homologia amb la GPx3a de Danio rerio i l’alineament amb t-coffee mostra un bon resultat. El gen que codifica per a la proteïna conté 6 exons, presenta una selenocisteïna (Sec) a l’exó 3 un element SECIS en l’extrem 3’ UTR.
GPx3b
En quan a la proteïna GPx3b, aquesta es localitza en l’scaffold APWO02000079.1. El gen que codifica per a la proteïna conté 5 exons, presenta una selenocisteïna (Sec) a l’exó 1 i un element SECIS en l’extrem 3’ UTR. La predicció i l’alineament amb el t-coffee mostren bons resultats i elevada homologia amb Danio rerio.
GPx4a
La proteïna GPx4a es troba en l’scaffold CM008304.1, el qual mostra un bon alineament analitzat amb el t-coffee. El gen que codifica per a la proteïna conté 3 exons, una selenocisteïna (Sec) en l’exó 1 i un element SECIS en l’extrem 3’ UTR.
GPx4b
La selenoproteïna GPx4b es localitza en l’scaffold CM008316.1. El gen que codifica per a la proteïna conté 5 exons, conté una selenocisteïna (Sec) en l’exó 2 i presenta un element SECIS en l’extrem 3’ UTR. A més, presenta una elevada homologia amb la proteïna GPx4b de Danio rerio ja que els resultats del t-coffee són molt bons. Cal tenir en compte que tant la GPx4a com la GPx4b s’han predit a partir de les proteïnes anotades en el SelenoDB 2.0, les quals no comencen amb metionina. Per aquest motiu pot ser que les proteïnes predites estiguin incompletes, ja que tampoc comencen amb metionina.
GPx7 i GPx8
En quan a les proteïnes GPx7 i GPx8 ambdues són proteïnes homòlogues amb cisteïna (Cys) i no contenen elements SECIS. La primera es troba en l’scaffold APWO02002209.1 i la segona en l’scaffold CM008305.1 i mostren bons resultats en els alineaments amb el t-coffee. El gen que codifica per a la GPx7 conté 3 exons, al igual que el gen que codifica per a la proteïna GPx8. Cal tenir en compte que aquestes proteïnes s’han predit a partir de les proteïnes de Danio rerio anotades en el SelenoDB, les quals no comencen amb metionina. Per aquest motiu podria ser que les proteïnes predites no estiguin completes.
Podem observar que s’han trobat elements SECIS en totes les proteïnes de la família GPx excepte en la GPx7 i en la GPx8. Això té sentit si tenim en compte que ambdues no presenten cap selenocisteïna (Sec) en la seva seqüència proteica i per tant no es va poder trobar cap element SECIS en l’extrem 3’ UTR. Se sap que la cisteïna present en ambdues proteïnes va evolucionar a partir de la GPx4, fet que va succeir abans de la separació de peixos i mamífers.
Família DIO
La família iodotironina deionidases (DIO) està formada per enzims que catalitzen la pèrdua d'àtoms de iode de les hormones tiroidees de manera que regulen tant l'activació com la innactivació d'aquestes hormones. En Danio rerio, igual que en els mamífers, hi ha tres proteïnes DIO (DIO1, DIO2 i DIO3) que mostren homologia intra-familiar. En tots els peixos ossis s’ha observat que DIO3 està duplicada, donant com a resultats les proteïnes DIO3a i DIO3b.
Durant la cerca les proteïnes DIO en el genoma d’Astyanax mexicanus ens vam trobar amb una sèrie de problemes. El blast obtingut per a les quatre proteïnes donava com a resultat tres únics scaffolds, els mateixos per a les quatre proteïnes (DIO1, DIO2, DIO3a i DIO3b) de Danio rerio. A més de ser el mateix scaffold, les posicions del genoma també coincidien en els quatre casos. Al tenir tots tres valors de e-value bons vam procedir a filtrar els resultatsfixant-los en la llargada dels scaffolds. D’aquesta forma vam poder eliminar-ne un, el CM008308.1. A continuació, per tal d’identificar a quines proteïnes corresponien els altres dos scaffolds (CM008305.1 i CM008302.1) es va realitzar l’arbre filogenètic de la família de proteïnes DIO.

Figura 6.Arbre filogenètic de la família DIO
Mitjançant l’anàlisi d’aquest vam observar que l’scaffold CM008305.1 era el més pròxim a la proteïna
DIO1
Tal i com hem comentat anteriorment, la proteïna DIO1 es localitza en l’scaffold CM008305.1 del genoma d'Astyanax mexicanus. El gen que codifica per a aquesta selenoproteïna conté 4 exons, presenta una selenocisteïna (Sec) en l’exó 2 i un element SECIS en l’extrem 3’ UTR. Aquesta proteïna ha estat predita a partir de la proteïna DIO1 de Danio rerio anotada en el SelenoDB, la qual no comença amb metionina. És per això que la proteïna predita en el genoma del nostre organisme podria no estar completa.
DIO3a
Per altra part, la proteïna DIO3a es troba en l’scaffold CM008302.1 i presenta uns bons resultats en l’alineament amb t-coffee. El gen que codifica per a aquesta proteïna té un sol exó, el qual conté una selenocisteïna (Sec) i un element SECIS en l’extrem 3’ UTR. Aquesta proteïna ha estat predita a partir de les proteïnes DIO de Danio rerio anotades en el SelenoDB, les quals no comencen amb metionina. És per això que les proteïna DIO3a predita en el genoma del nostre organisme podria no estar completa.
Família SelenoH
La SelH es una proteïna que es troba al nucli actuant com a factor de transcripció. Protegeix les neurones contra el dany causat per UVB inhibint l’apoptosi pormoguent l’activitat mitocondrial.
Durant l’alineament amb Danio rerio es va obtenir un únic scaffold: CM008323.1. Aquest hit va donar un bon alineament amb t-coffee, la nostra predicció conté 3 exons i en el segon exó hi trobem una selenocisteïna. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure que l’Astyanax mexicanus té la selenoproteïna SelH.
Família SelenoI
La SelI es una proteïna Involucrada en el manteniment de les membranes vesiculars, regulació del metabolisme lipídic i plegament de proteïnes.
Durant l’alineament amb Danio rerio es van obtenir diversos scaffolds, però finalment ens vam quedar amb l’scaffold CM008314.1, ja que presentava un bon e-value i una llargada semblant a la proteïna amb la que estàvem comparant. Aquest hit va donar un bon alineament amb t-coffee. La nostra predicció de SelI conté 10 exons, en el desè exó hi trobem una selenocisteïna. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure que l’Astyanax mexicanus té la selenoproteïna SelI.
Família SelenoJ
La selenoproteïna SelJ té una distribució molt concreta ja que no es troba en altres vertebrats, ni com a selenoporteïna ni com a un homòleg amb cisteïna. Té la particularitat que no té una funció enzimàtica com la majoria de les selenoproteïnes sinó més aviat de caire estructural, segons estudis computacionals.
Durant l’alineament amb Danio rerio es va obtenir un únic scaffold: APWO02000036.1. Aquest hit va donar un bon alineament amb t-coffee, la nostra predicció conté 6 exons i presenta dues selenocisteïnes a l’exó 3 i una l’exó 6. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure que l’Astyanax mexicanus té la selenoproteïna SelJ.
Família SelenoK
La SelK està involucrada amb el manteniment de l’homeostasi del reticle endoplasmàtic. També es sap que està involucrada en processos de mobilització de calci durant la inflamació durant la resposta immunitària. També s’ha identificat al cor on sembla que pot actuar com a antioxidant.
Durant l’alineament amb Danio rerio es va obtenir un únic scaffold: CM008305.1. Aquest hit va donar un bon alineament amb t-coffee, la nostra predicció conté 5 exons i presenta una selenocisteïna a l’exó 4. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. Encara que Seblastian no trobés una selenoproteïna per la nostra predicció, podem concloure que l’Astyanax mexicanus té la selenoproteïna SelK degut al bon alineament amb la proteïna del genoma de referència i a la presència de l’element SECIS a l’extrem 3’UTR.
Família SelenoL
La proteïna SelL es troba a la superfamília de les thioredoxines però amb la paculiaritat que els peixos són els únics vertebrats que la presenten. La caracterísitca comuna entre aquestes és la thioredoxina, on tenim UxxU (sent U una selenocisteïna i les x aminoàcids qualsevols).
Durant l’alineament amb Danio rerio es va obtenir un únic scaffold: CM008302.1. Aquest hit va donar un bon alineament amb t-coffee, la nostra predicció conté 9 exons i presenta dues selenocisteïnes a l’exó 6 i una al l’exó 9. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure que l’Astyanax mexicanus té la selenoproteïna SelL.
Família SelenoM
La selenoproteïna SelM, es va predir a partir de la seqüència de SelM en Danio rerio. Es va intentar predir aquesta proteïna amb el nostre programa, però es presentava un error en el Fastasubseq que no vam ser capaces de solucionar. Tot i així, al córrer la nostra proteïna en el programa si que obteníem el blast, que identificava un sol hit: APWO02000095.1. Es va intentar fer el mateix procediment manualment, i tampoc es va aconseguir.
Com a alternativa, per veure si l’Astyanax mexicanus tenia aquesta selenoproteïna, es va predir a partir de blastejar les seqüències amb el Blast Protein Browser. Seguidament, a partir de la proteïna predita pel Blast Protein Browser d’Astyanax mexicanus es va realitzar l’alineament amb t-coffee Simple MSA. En aquest s’hi va entrar la seqüència de la proteïna SelM de Danio rerio i la proteïna SelM, predita pel Blast, d’Astyanax mexicanus. Es va obtenir un bon alineament amb t-coffee de la proteïna predita. Aquesta proteïna ha conservat la selenocisteïna, que ja trobàvem present en la SelM de Danio rerio. Per tant, hem hipotetitzat que Astyanax mexicanus si que té la selenoproteïna SelM.
Família SelenoO
Es troba a la mitocòndria i té una funció desconeguda. Aquesta proteïna és la selenoproteïna més llarga d’un gran nombre d’organismes des de mamífers fins a llevats. La seva expressió es veu lleugerament afectada davant la manca de seleni el que suggereix que té prioritat pel que fa a la distribució de seleni a l’organisme.
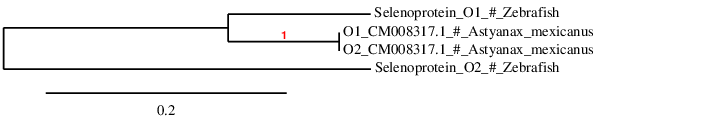
Figura 7.Arbre filogenètic de la família SelenoO
La figura mostra l’arbre filogenètic de la família de selenoproteïnes SelenoO. Per realitzar-lo, es van utilitzar les proteïnes predites a partir del l’scaffold del blast trobat al analitzar les proteïnes SelO1 i SelO2 (CM008317.1 - per ambdues proteïnes), així com les proteïnes SelO1 i SelO2 de Danio rerio. A continuació, expliquem les conclusions que n’hem extret.
En primer lloc, SelO1 i SelO2 de Danio rerio son proteïnes paràlogues, tenen un origen comú.
En segon lloc, la selenoproteïna predita a partir de l’scaffold CM008317.1, està més aprop filogenèticament a la proteïna SelO1 de Danio rerio. Per tant, hem arribat a la conclusió, que el hit CM008317.1 es correspon a la SelO1 d'Astyanax mexicanus.
Pel que fa a la selenoproteïna SelO2 de Danio rerio els hits que el blast donava no els hem posat a l’arbre ja que no tenien un alineament correcte.
SelO1
Durant l’alineament amb Danio rerio es van obtenir diversos scaffolds amb un bon e-value i una llargada similar a la de la proteïna amb la que estàvem comparant: CM008317.1, CM008306.1 i APWO02001314.1. Finalment ens vam quedar amb l’scaffold CM008317.1 per les raons que s’han comentat en l’anàlisi de l’arbre filogenétic.
Aquest hit va donar un bon alineament amb t-coffee, la nostra predicció conté 9 exons i presenta una selenocisteïna a l’exó 9. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian, encara que en troba la selenoproteïna en una altra espècie (Ictalurus punctatus). Aquest fet, pot ser degut a que la proteïna SelO1 en ambdues espècies assembli molt, i per tant, que el Seblastian te la trobi a l’altre espècie, encara que també hi sigui a Astyanax mexicanus. Podem concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna SelO1 i que aquesta es troba a l’scaffold CM008317.1.
Família SelenoP
Té relació amb el transport extracel·lular del seleni o amb la seva acció antioxidant. Majoritàriament s’expressa al fetge, que la secreta en plasma, tot i que la trobem present en molts teixits. Per altra banda, sembla que és l’encarregada de subministrar seleni al cervell i als testis. En aquestes selenoproteïnes és força normal trobar més d’una selenoproteïna.
Com podem veure al SelenoDB, hi ha dues isoformes d’aquesta proteïna en Danio rerio. Hem predit les dues isoformes en Astyanax mexicanus (anomenades SelP1 i SelP2).

Figura 8.Arbre filogenètic de la família SelenoP
SelP1
Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008316.1 i CM008305.1. El hit CM008316.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 5 exons i presenta una selenocisteïna a l’exó 1, a l’exó 4 i a l’exó 5. El hit CM008305.1 es va descartar ja que tot i presentar un bon e-value, la seva llagada no representava més d’un 80% de la proteïna que voliem predir. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian, encara que troba la selenoproteïna en una altra espècie (Ictalurus punctatus). Aquest fet, pot ser degut a que la proteïna SelP1 en ambdues espècies assembli molt, i per tant, que el Seblastian te la trobi a l’altre espècie, encara que també hi sigui a Astyanax mexicanus. Podem concloure, per tant, que l’Astyanax mexicanus té la isoforma de la selenoproteïna SelP1.
SelP2
Durant l’alineament amb Danio rerio es van obtenir un scaffold: CM008305.1. El hit CM008305.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 6 exons i presenta 13 selenocisteïnes a l’exó 6. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure, per tant, que l’Astyanax mexicanus té la isoforma de la selenoproteïna SelP2.
MsrA
Les Metionina sulfoxi reductases (Msr) són enzims dependents de tiol que catalitzen la conversió de sulfoxi-metionina (Met(O)) a metionina (Met). La majoria de les Msr són proteïnes homòlogues en cisteïna on la selenocisteïna (Sec) ha estat substituïda per una cisteïna (Cys) en la seqüència proteica.
En l’anàlisi de la subfamília MsrA, es va observar que Danio rerio presenta dues proteïnes MsrA (MrsA 1 i MsrA 2). Al blastejar aquestes dues seqüències proteiques contra el genoma del nostre organisme, es van obtenir en ambdues seqüències un mateix scaffold (CM008308.1.1) amb % d’identitat i e-values molt semblants. A partir d’aquests resultats es va hipotetitzar la existència d’una única proteïna MsrA en Atyanax mexicanus, a diferència de la duplicació present en Danio rerio.
La proteïna d’aquesta família que ha estat identificada en el nostre organisme és la MsrA 1, la qual redueix les S sulfoximetionineses. Aquesta s’ha localitzat, com s’ha comentat anteriorment, en l’scaffold CM008308.1. i el seu alineament ha estat comprovat mitjançant el t-coffee, donant bons resultats. El gen que codifica per a la proteïna conté 7 exons i no presenta cap element SECIS a l’extrem 3’ UTR, ja que en aquest cas es tracta d’un proteïna homòloga amb cisteïna (Cys). Això concorda amb la bibliografia consultada que diu que en els vertebrats la selenocisteïna d’aquesta proteïna ha estat subsituïda per una cisteïna. També cal comentar que la proteïna MsrA 1 de Danio rerio extreta del SelenoDB no comença amb metionina, al igual que la MsrA 1 que s’ha predit en el genoma del nostre organisme, fet que fa pensar que la proteïna no està sencera.
SelenoR (MSRB)
La família del Msr que oxida els residus de proteïnes. D’aquí prové el seu nom metionina sulfòxid reductasa. Està formada per diferents proteïnes algunes de les quals tenen selenocisteïna i altres homólegs amb cisteïna. Redueix la forma R de la sulfoximetionina a metionina
MsrB1A
Aquesta és una selenoproteïna ja que trobem una selenocisteïna. Durant l’alineament amb Danio rerio es van obtenir un sol scaffold: APWO02000083.1. El hit APWO02000083.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 4 exons i presenta una selenocisteïna a l’exó 3. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna MsrB1A.
MsrB2
Aquesta és una altra proteïna homòloga amb cisteïna. Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008302.1 i CM008306.1.
El hit CM008306.1 es va descartar, ja que tot i tenir un bon e-value, la seva llargada no representava més del 80% de la proteïna que s’estava intentant predir. Amés, aquest hit, en les mateixes posicions va ser triat per la proteïna MsrB3.
El hit CM008302.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 5 exons i no presenta cap selenocisteïna, presenta cisteïna. Aquesta proteïna, com era d’esperar, no conté cap element SECIS a l’extrem 3’UTR. Podem concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna MsrB2.
MsrB3
Aquesta és una altra proteïna homòloga amb cisteïna. Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008302.1 i CM008306.1.
El hit CM008302.1 es va descartar, ja que tot i tenir un bon e-value, la seva llargada no representava més del 80% de la proteïna que s’estava intentant predir. Amés, aquest hit, en les mateixes posicions va ser triat per la proteïna MsrB2.
El hit CM008306.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 6 exons i no presenta cap selenocisteïna, presenta cisteïna. Aquesta proteïna, com era d’esperar, no conté cap element SECIS a l’extrem 3’UTR. Podem concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna MsrB3.
Família SEPHS
La selenofosfat sintasa (SEPHS o SPS) és la única proteïna de maquinària de biosíntesi de Sec que alhora pot ser també una selenoproteïna, de manera que té dos gens paràlegs: SEPHS1 i SEPHS2. Un estudi recent mostra que els gens de SEPHS1 es van originar a partir d’una sèrie de duplicacions del gen SEPHS2.

Figura 9.Arbre filogenètic de la família SEPHS
Durant la cerca de les proteïnes de la família SEPHS en el genoma del nostre organisme es van obtenir dos scaffolds (CM008314.1 i CM008317.1) amb e-values i llargada bons per a cada una de les dues proteïnes SEPHS. Per tal d’assignar un scaffold a cada proteïna es va realitzar un arbre filogenètic. Després d’analitzar els resultats d’aquest i basant-nos en la proximitat filogenètica, es va assignar l’scaffold CM008314.1 a la proteïna SEPHS2 i el CM008317.1 a la proteïna SEPHS1.
SEPHS1
La proteïna SEPHS1 es situa en l’scaffold CM008317.1 i l’alineament amb t-coffee va mostrar una elevada homologia respecte la proteïna SEPHS1 de Danio rerio. El gen que codifica per a aquesta proteïna conté 8 exons i conté un element SECIS de grau B en l’extrem 3’ UTR. Tot i així, aquesta proteïna no conté cap selenocisteïna, ja que es tracta d’un homòleg de cisteïna (Cys). Aquesta fet fa pensar que segurament al llarg de l’evolució la Sec s’ha reemplaçat per una Cys però l’element SECIS s’ha mantingut. En aquest cas, tant la proteïna de Danio rerio anotada en el SelenoDB com la que hem predit en el genoma del nostre organisme comencen amb metionina.
SEPHS2
La proteïna SEPHS2 es localitza en l’scaffold CM008314.1 i l’alineament amb t-coffee va mostrar bons resultats. El gen que codifica per a aquesta proteïna conté 8 exons, presenta una selenocisteïna (Sec) en l’exó 1 i es va trobar un element SECIS en l’extrem 3’ UTR. Cal tenir en compte que aquesta proteïna s’ha predit a partir de la proteïna de Danio rerio anotada en el SelenoDB 2.0, la qual no comença amb metionina. Per aquest motiu pot ser que la proteïna predita estigui incompleta, ja que la SEPHS2 d'Astyanax mexicanus tampoc comença amb metionina.
Família SelenoT
Són selenoproteïnes thiredoxina reductasa-like amb activitat oxioreductasa. Protegeixen les neurones dopaminèrgiques davant l’estrès oxidatiu i la mort cel·lular.

Figura 10.Arbre filogenètic de la família SelenoT
La figura mostra l’arbre filogenètic de la família de selenoproteïnes SelenoT. Per realitzar-lo, es van utilitzar les proteïnes predites a partir dels diferents scaffolds del blast trobats al analitzar les proteïnes SelT1, SelT1b i SelT2 (CM008315.1 i CM008317.1 per les tres proteïnes), així com les proteïnes SelT1, SelT1b i SelT2 de Danio rerio. A continuació, expliquem les conclusions que n’hem extret.
En primer lloc, SelT1, SelT1b i SelT2 de Danio rerio tenen un origen comú. Tot i tenir les tres un origen comú, cal destacar que SelT1 i SelT1b són més properes filogenèticament entre elles que amb SelT2. És a dir, primer van divergir SelT2 del llinatge de SelT1 i SelT1b i després van divergir SelT1 i SelT1b.
En segon lloc, la selenoproteïna predita a partir de l’scaffold CM008315.1, està més aprop filogenèticament a la proteïna SelT1 de Danio rerio. Per tant, hem arribat a la conclusió, que el hit CM008315.1 correspon a la SelT1 d'Astyanax mexicanus.
En tercer lloc, la selenoproteïna predita a partir de l’scaffold CM008317.1, està més aprop filogenèticament a la proteïna SelT1b de Danio rerio. Per tant, hem arribat a la conclusió, que el hit CM008317.1 correspon a la SelT1b de l’Astyanax mexicanus.
Per últim, cap de les proteïnes predites (cap dels hits), sembla ser proper filogenèticament a SelT2.
SelT1
Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008315.1 i CM008317.1. El hit CM008315.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 5 exons i presenta una selenocisteïna a l’exó 2. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian a la nostra espècie podent concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna SelT1.
SelT1b
Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008315.1 i CM008317.1. El hit CM008317.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 5 exons i presenta una selenocisteïna a l’exó 2. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian a la nostra espècie podent concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna SelT1b.
Família SelenoU
Funció desconeguda. S'hipotetetitza que pot estar regulant milers de processos biològics gràcies a la seva funció redox.

Figura 11.Arbre filogenètic de la família SelenoU
La figura mostra l’arbre filogenètic de la família de selenoproteïnes SelenoU. Per realitzar-lo, es van utilitzar les proteïnes predites a partir dels diferents hits del blast trobats al analitzar les proteïnes SelU1A, SelU2 i SelU3 a partir de les proteïnes de Danio rerio, i les proteïnes SelU1B i SelU1C de Takifugu rubripes, així com les proteïnes SelU1A, SelU2 i SelU3 de Danio rerio i les proteïnes SelU1B i SelU1C de Takifugu rubripes. A continuació, expliquem les conclusions que n’hem extret.
En primer lloc, pel que fa a les selenoproteïnes predites a partir de Takifugu rubripes: el hit CM008324.1 és filogenèticament molt proper a SelenoU1B de Takifugu rubripes, i per tant, s’ha assignat aquesta predicció a aquesta proteïna per a fer l’anàlisis. Finalment, aquest hit no va donar resultat concloents i es va descartar. Per altra banda, no hi ha cap hit filogenèticament propera a SelU1C de Takifugu rubipes, i per tant, hem hipotetitzat que Astyanax mexicanus no té aquesta proteïna.
En segon lloc, pel que fa a les selenoproteïnes predites a partir de Danio rerio: Respecte SelU1A, veiem que no hi ha cap hit que sigui filogenèticament semblant a aquesta proteïna. Per altra banda, la selenoproteïna predita a partir de l’scaffold CM008305.1, està més aprop filogenèticament a la proteïna SelenoU2 de Danio rerio. Per tant, hem arribat a la conclusió, que aquest hit correspon a la SelU2 d'Astyanax mexicanus. Per últim, pel que fa a la proteïna SelU3 de Danio rerio és filogenèticament més propera al hit CM008321.1 de la nostra espècie predit a partir de Danio rerio. Per tant, hem hipotetitzat que aquest hit es correspon a la proteïna SelU3 d’Astyanax mexicanus.
SelU2
Durant l’alineament amb Danio rerio es va obtenir un sol scaffold: CM008305.1. El hit CM008305.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 6 exons. La selenoproteïna predita a partir d’aquest hit no presenta selenocisteïna, presenta cisteïna (cys) igual que la selenoproteïna SelU2 en Danio rerio. La selenoproteïna predita no té, com era d’esperar, cap element SECIS. Degut als bons resultats obtinguts amb l’anàlisi de la selenoproteïna predita a partir del hit CM008305.1, hem conclòs que l’Astyanax mexicanus té en el seu genoma la selenoproteïna SelU2.
SelU3
Durant l’alineament amb Danio rerio es va obtenir un sol scaffold:CM008321.1. El hit CM008321.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 6 exons. La selenoproteïna predita a partir d’aquest hit no presenta selenocisteïna, presenta cisteïna (cys) igual que la selenoproteïna SelU3 en Danio rerio. La selenoproteïna predita no té, com era d’esperar, cap element SECIS. Degut als bons resultats obtinguts amb l’anàlisi de la selenoproteïna predita a partir del hit CM008321.1, hem conclòs que l’Astyanax mexicanus té en el seu genoma la selenoproteïna SelU3.
Família SelenoW
S’expressa a múltiples teixits tot i que no es coneix la seva funció. Trobem diferents homòlegs als organismes vertebrats. Destaquem en primer lloc W2. Aquesta selenoproteïna apareix en els peixos ossis i granotes pel que sembla que era una selenoproteïna ancestral que s’ha perdut en alguns organismes. Danio rerio té dues còpies de SelW2: SelW2a i SelW2b, que és una duplicació de la primera. Els altres peixos ossis també tenen una duplicació de amb una proteïna similar a les proteïnes SelW2a i SelW2b i anomenada SelW2c, que és una duplicació del SelW2. També s’ha vist que en tots els peixos ossis, a excepció de Danio rerio, hi ha hagut una pèrdua de la selenoproteïna SelW1. La característica comuna d’aquestes selenoproteïnes és que conserven un motiu Cys-x-x-Sec.
SelW (I)
Durant l’alineament amb Danio rerio es van obtenir un scaffold: APWO02000046.1. El hit APWO02000046.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 4 exons i presenta una selenocisteïna a l’exó 2. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure i hipotetitzar, per tant, que l’Astyanax mexicanus té la selenoproteïna SelW (I).
SelW (II)
Durant l’alineament amb Danio rerio es van obtenir un scaffold: APWO02000046.1. El hit APWO02000046.1 va donar un molt bon alineament amb t-coffee, la nostra predicció conté 4 exons i presenta una selenocisteïna a l’exó 2. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure, per tant, que l’Astyanax mexicanus té la selenoproteïna SelW(II).
SelW (III)
Durant l’alineament amb Danio rerio es van obtenir un scaffold: CM008302.1. El hit CM008302.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 5 exons i presenta una selenocisteïna a l’exó 2. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian podent concloure i hipotetitzar, per tant, que l’Astyanax mexicanus té la selenoproteïna SelW (III).
Família TXNRD
Aquestes proteïnes són oxireductases del nucleòtid piridina. Redueixen tioredoxines entre d’altres substrats alhora que tenen un paper important pel que fa al metabolisme del seleni i a la protecció davant l’estrès oxidatiu. Aquest enzim actua com a homodímer que utilitza FAD com a cofactor.

Figura 12.Arbre filogenètic de la família TXNRD 1
La figura mostra l’arbre filogenètic de la família de selenoproteïnes TXNRD. Per realitzar-lo, es van utilitzar les proteïnes predites a partir dels diferents scaffolds del blast trobats al analitzar les proteïnes TXNRD1, TXNRD2 i TXNRD3 (APWO02000039.1 i CM008312.1), així com les proteïnes TXNRD1 d’Homo sapiens, i TXNRD2 i TXNRD3 de Danio rerio obtingudes de SelenoDB. A continuació, expliquem les conclusions que n’hem extret.
En primer lloc, TXNRD1 d’Homo sapiens i TXNRD3 de Danio rerio tenen un origen comú més proper entre elles que amb TXNRD2 de Danio rerio. Tot i això les tres proteïnes tenen un origen comú.
En segon lloc, la selenoproteïna predita a partir de l’scaffold APWO02000039.1, està més aprop filogenèticament a la proteïna TXNRD2 de Danio rerio. Per tant, hem arribat a la conclusió, que aquest hit correspon a la TXNRD2 d'Astyanax mexicanus i no pas a la TXNRD1 d’Homo sapiens.
En tercer lloc, la selenoproteïna predita a partir de l’scaffold CM008312.1, està més aprop filogenèticament a la proteïna TXNRD3 de Danio rerio que no pas la selenoproteïna predita a partir de l’scaffold APWO02002185.1. Per tant, hem arribat a la conclusió, que el hit CM008312.1 correspon a la TXNRD3 d'Astyanax mexicanus.
TXNRD2
Durant l’alineament amb Danio rerio es van obtenir tres scaffolds diferents: APWO02000039.1, CM008312.1 i APWO02002185.1. Es van predir les proteïnes dels hits APWO02000039.1 i CM008312.1, però un cop realitzat l’arbre filogenétic amb les prediccions, vàrem observar que el millor hit per a aquesta proteïna es corresponia al scaffold APWO02000039.1. El hit APWO02002185.1, no es va analitzar, ja que la llargada de la selenoproteïna que predeia era inferior al 80% de la proteïna que s’estava cercant. El hit APWO02000039.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 16 exons i presenta una selenocisteïna a l’exó 3 i una altra a l’exó 16. En canvi, en el genoma de Danio rerio, només presenta una selenocisteïna. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. En aquest cas no tenim una predicció de selenoproteïna amb Seblastian, però tot i això, degut al bon alineament predit per t-coffee i la presència de un element SECIS a l’extrem 3’UTR podem hipotetitzar que l’Astyanax mexicanus té la selenoproteïna TXNRD2 i que aquesta es troba en l’scaffold APWO02000039.1.
TXNRD3
Durant l’alineament amb Danio rerio es van obtenir cinc scaffolds diferents: CM008312.1, APWO02002185.1, APWO02000039.1, CM008304.1 i CM008323.1. Es van predir les proteïnes dels hits CM008312.1 i APWO02002185.1, però un cop realitzat l’arbre filogenètic amb les prediccions i els t-coffee, vàrem observar que el millor hit per a aquesta proteïna es corresponia al scaffold CM008312.1 ja que tenia una distància filogenètica menor amb aquesta i presentava un millor alineament amb la query de Danio rerio .

Figura 13.Arbre filogenètic de la família TXNRD 3
Els hits APWO02000039.1, CM008304.1 i CM008323.1, no es van analitzar, ja que la llargada de la selenoproteïna que predeien era inferior al 80% de la proteïna que s’estava cercant.
El hit CM008312.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 16 exons i presenta una selenocisteïna a l’exó 16. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. A més a més, en aquest cas també tenim una predicció Seblastian que confirma que es tracta de la TXNDR3, encara que troba la selenoproteïna en una altra espècie (Clarias batrachus). Aquest fet, pot ser degut a que la proteïna TXNRD3 en ambdues espècies assembli molt, i per tant, que el Seblastian te la trobi a l’altra espècie, encara que també hi sigui a Astyanax mexicanus. Degut a tot això, podem hipotetitzar que l’Astyanax mexicanus té la selenoproteïna TXNRD3 i que aquesta es troba en l’scaffold CM008312.1.
Selenoproteïnes no trobades
GPx5 i GPx6
En l’espècie de Danio rerio no trobem GPx5 i GPx6, ja que van aparèixer en l’origen dels mamífers placentaris a partir d’una duplicació en tàndem de GPx3. És per això que per a saber si el nostre organisme presentava GPx5 i GPx6 es va agafar com a genoma de referència el d’Homo sapiens. Durant l’alineament de les proteïnes d’Homo sapiens amb el genoma de la nostra espècie no es va obtenir cap hit bo, és a dir, que tingués uns e-values i una llargada bones. D’aquesta manera, es va poder comprovar que l’Astyanax mexicanus, al igual que Danio rerio, no presenta les proteïnes GPx5 i GPx6.
DIO2 i DIO3b
Tot i saber que la família de proteïnes DIO es troba força conservada al llarg de l’evolució, ens hem trobat amb que el nostre organisme no conté dues de les quatre proteïnes DIO que tenen la majoria de peixos, tals com Danio rerio. Aquestes dues proteïnes són DIO2 i DIO3b.
Això ho hem pogut observar ja que en blastejar les quatre proteïnes de Danio rerio amb el genoma del nostre organisme es van obtenir només dos scaffolds bons, els quals eren els mateixos per a les quatre proteïnes i compartien també posicions genòmiques. Després de realitzar un anàlisis més profund de la proximitat filogenètica d’aquests scaffolds amb les proteïnes de Danio rerio, es van assignar els dos scaffolds a DIO1 i a DIO3a, ja que presentaven millors resultats filogenètics. Aquest fet fa pensar que Astyanax mexicanus hauria pogut patir alguna mutació en les proteïnes DIO1 i DIO3a que els ha permès adquirir la funció que realitzaven DIO2 i DIO3b, les quals ha perdut com a conseqüència d’aquest fet.
Cal comentar que també es va comparar el genoma del nostre organisme amb les proteïnes DIO d’Homo sapiens, ja que vam pensar que pot ser el fet de no trobar DIO2 i DIO3b era degut a la mala anotació d’aquestes proteïnes de Danio rerio en el SelenoDB. Tot i així, no vam obtenir bons resultats d’aquesta cerca, ja que aquests eren els mateixos que s’havien obtingut al comparar amb les proteïnes de Danio rerio.
Família SelenoN
La SelN codifica per una glicoproteïna que es troba al reicle endoplasmàtic i té un paper clau tan pel que fa en relació a l’estrès oxidatiu com l’homeostasi del calci relacionat amb reaccions redox. La inhibició d’aquest gen en Danio rerio també ens mostra la seva importància en el desenvolupament muscular. En humans també causa diferents patologies.
Durant l’alineament amb Danio rerio es va obtenir un únic scaffold: CM008308.1. Respecte l’alineament amb t-coffee, cal dir que hi ha una part de la proteïna amb un bon alineament, però una altra part que no es troba tan ben alineada. Aquest fet pot ser degut a que la nostra espècie hagi perdut la proteïna i només estigui conservada una part d’aquesta, un sol domini, probablement no funcional. La nostra selenoproteïna predita conté 14 exons i presenta una selenocisteïna a l’exó 11. Aquesta proteïna conté també un element SECIS a l’extrem 3’UTR. Hem conclòs que l’Astyanax mexicanus no té la selenoproteïna SelN, o només ha conservat un domini d’aquesta, sent probablement no funcional.
SelO2
Com hem comentat en la família de SelenoO, per a la proteïna SelO2 es van trobar tres hits vàlids en el blast (scaffolds: CM008317.1, CM008306.1 i APWO02001314.1). Un cop analitzats els tres hits i realitzat l’arbre filogenètic, no es va trobar cap dels tres hits que fos filogenèticament semblant a SelO2. El hit més semblant filogenèticament a SelO2 es corresponia a l’scaffold CM008317.1, que com hem comentat, es trobava més aprop filogenéticament de SelO1 i per això l’hem assignat a SelO1. Per altra banda, analitzant els dos altres scaffolds, hem vist que no presentaven un bon alineament amb t-coffee. Es van tornar a blastejar les seqüències amb el Blast Protein Browser ja que al ser una base de dades per Internet, vam pensar que estaria més actualitzada, i vam trobar que no es trobaven hits pressents en la nostra espècie. Seguidament també van realitzar un altre cop el t-coffee Simple MSA per veure si s’obtenia un millor alineament amb els hits obtinguts, però l’alineament obtingut tampoc era gaire bo per ningun dels dos scaffolds (CM008306.1 i APWO02001314.1). Hem hipotetitzat que això pot ser degut a que SelenoDB no tingui ben anotada la proteïna SelO2 de Danio rerio o també podria ser degut a que Astyanax mexicanus hagi perdut aquesta proteïna.Família SelenoS
Els vertebrats tenen aquesta selenoproteïna conservada. La funció d’aquesta proteïna en peixos no es coneix.
Durant l’alineament amb Danio rerio es van obtenir un sol scaffold: CM008304.1. El hit CM008304.1 no va obtenir un bon alineament amb t-coffee, la nostra predicció conté 6 exons i presenta una selenocisteïna a l’exó 6. Aquesta proteïna predita conté també un element SECIS a l’extrem 3’UTR. Com no va haver-hi un bon alineament amb t-coffee amb l’únic hit que ens predia el blast, hem conclòs que l’Astyanax mexicanus no té la selenoproteïna SelS.
SelT2
Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008315.1 i CM008317.1. Com ja s’ha comentat abans, aquests scaffolds (en mateixes posicions), també es van trobar per a les proteïnes SelT i SelT1b, i un cop fet el arbre filogenètic, van ser assignats a aquestes dues proteïnes. Com ja hem dit en l’anàlisi de l’arbre filogenètic, no es va trobar cap scaffold filogenèticament proper a la proteïna SelT2 de Danio rerio en la nostra espècie. Per tant, hipotetitzem que l’Astyanax mexicanus ha perdut la selenoproteïna SelT2.
SelU1A, SelU1B i SelU1C
SelU1A
Durant l’alineament amb Danio rerio es van obtenir dos scaffolds: CM008324.1 i CM008305.1. Com hem explicat amb l’arbre filogenètic, un cop analitzats ambdós hits, vam veure que el hit CM008324.1 era més proper filogenèticament a SelU1B. El hit CM008305.1 va ser analitzat amb el nostre programa. Pel que fa a l’alineament obtingut amb t-coffee cal dir que hi ha una part de la proteïna amb un bon alineament (la meitat), però una altra part que no es troba tan ben alineada. Aquest fet pot ser degut a que la nostra espècie hagi perdut la proteïna i només estigui conservada una part d’aquesta, un sol domini, probablement no funcional. Per aquest motiu i per no haver trobat ningun hit filogenèticament semblant a SelU1A en la nostra espècie hem conclòs que l’Astyanax mexicanus podria no tenir la selenoproteïna SelU1A, o només ha conservat un domini d’aquesta, sent probablement no funcional.
SelU1B
La proteïna SelU1B va ser predita al genoma d’Astyanax mexicanus a partir de la proteïna SelU1B de Takifugu rubripes, ja que aquesta no es troba present ni en Danio rerio ni en Homo sapiens. Durant l’alineament amb Takifugu rubripes es va obtenir el hit CM008324.1 i es va analitzar. Pel que fa a l’alineament obtingut amb t-coffee cal dir que hi ha una part de la proteïna amb un bon alineament (la meitat), però una altra part que no es troba tan ben alineada. Aquest fet pot ser degut a que la nostra espècie hagi perdut la proteïna i només estigui conservada una part d’aquesta, un sol domini, probablement no funcional. Per aquest motiu, hem conclòs que l’Astyanax mexicanus podria no tenir la selenoproteïna SelU1B, o només ha conservat un domini d’aquesta, sent probablement no funcional.
SelU1C
La proteïna SelU1C va ser predita al genoma d’Astyanax mexicanus a partir de la proteïna SelU1C de Takifugu rubripes, ja que aquesta no es troba present ni en Danio rerio ni en Homo sapiens. Durant l’alineament amb Takifugu rubripes no es va obtenir cap hit bo, és a dir, que presentés uns e-values i una llargada bons. Per aquesta raó, hem hipotetitzat que la selenoproteïna SelU1C no es troba en el genoma d’Astyanax mexicanus.
TXNRD1
Durant l’alineament amb Homo sapiens es van obtenir quatre scaffolds diferents: CM008312.1, APWO02002185.1, APWO02000039.1 i CM008323.1. D’entre aquests es van seleccionar aquells que presentaven un bon e-value i representessin com a mínim un 80% de la llargada de la proteïna que es volia predir. Un cop aplicat aquest criteri, es va triar únicament l’scaffold CM008312.1 i es va analitzar. La predicció a partir de l’scaffold CM008312.1 va presentar un bon alineament amb t-coffee. Tot i això, tal i com hem dit abans, aquest scaffold amb les mateixes posicions també ha estat predit per a la selenoproteïna TXNRD3, i filogenèticament era més proper a aquesta que no a TXNRD1. La nostra hipòtesi és que Astyanax mexicanus podria no presentar la selenoproteïna TXNRD1 per dues raons: En primer lloc, perquè Danio rerio no té aquesta proteïna i és una espécie molt propera a Astyanax mexicanus, i per tant, és possible que Astyanax mexicanus tampoc la tingui en el seu genoma. En segon lloc, perquè no hem obtingut bons hits en el Blast per a la predicció d’aquesta selenoproteïna a partir d’humans en el genoma d’Astyanax mexicanus excepte l’scaffold CM008312.1 que ja hem assignat a TXNRD3 (en les mateixes posicions).
Maquinària proteica
Família SEPHS
La selenofosfat sintasa (SEPHS o SPS) és la única proteïna de maquinària de biosíntesi de Sec que alhora pot ser també una selenoproteïna, de manera que té dos gens paràlegs: SEPHS1 i SEPHS2. Un estudi recent mostra que els gens de SEPHS1 es van originar a partir d’una sèrie de duplicacions del gen SEPHS2.

Figura 9.Arbre filogenètic de la família SEPHS
Durant la cerca de les proteïnes de la família SEPHS en el genoma del nostre organisme es van obtenir dos scaffolds (CM008314.1 i CM008317.1) amb e-values i llargades bones per a cada una de les dues proteïnes SEPHS. Per tal d’assignar un scaffold a cada proteïna es va realitzar un arbre filogenètic. Després d’analitzar els resultats d’aquest i basant-nos en la proximitat filogenètica, es va assignar l’scaffold CM008314.1 a la proteïna SEPHS2 i el CM008317.1 a la proteïna SEPHS1.
SEPHS1
La proteïna SEPHS1 es situa en l’scaffold CM008317.1 i l’alineament amb t-coffee va mostrar una elevada homologia respecte la proteïna SEPHS1 de Danio rerio. El gen que codifica per a aquesta proteïna conté 8 exons i conté un element SECIS de grau B en l’extrem 3’ UTR. Tot i així, aquesta proteïna no conté cap selenocisteïna, ja que es tracta d’un homòleg de cisteïna (Cys). Aquesta fet fa pensar que segurament al llarg de l’evolució la Sec s’ha reemplaçat per una Cys però l’element SECIS s’ha mantingut. En aquest cas, tant la proteïna de Danio rerio anotada en el SelenoDB com la que hem predit en el genoma del nostre organisme comencen amb metionina.
SEPHS2
La proteïna SEPHS2 es localitza en l’scaffold CM008314.1 i l’alineament amb t-coffee va mostrar bons resultats. El gen que codifica per a aquesta proteïna conté 8 exons, presenta una selenocisteïna (Sec) en l’exó 1 i es va trobar un element SECIS en l’extrem 3’ UTR. Cal tenir en compte que aquesta proteïna s’ha predit a partir de la proteïna de Danio rerio anotada en el SelenoDB 2.0, la qual no comença amb metionina. Per aquest motiu pot ser que la proteïna predita estigui incompleta, ja que la SEPHS2 d’Astyanax mexicanus tampoc comença amb metionina.
Família eEFsec
El factor d’elongació eucariòtic (eEFsec) és una GTPasa que uneix Sec-tRNASec amb gran afinitat i especificitat. Té un paper molt important durant la descodificació mitjançant el lliurament de Sec-tRNASec al lloc de la traducció en resposta a un codó in-frame UGA.
La proteïna eEFsec es localitza en l’scaffold CM008312.1 i és un homòleg amb cisteïna (Cys). El gen que codifica per a la proteïna conté 8 exons i no presenta cap selenocisteïna sinó que hi ha hagut una pèrdua de l’àtom de seleni i ha esdevingut una cisteïna. Tot i així, els resultats del Seblastian van mostrar un element SECIS de grau B en l’extrem 3’ UTR. Per això pensem que el nostre organisme pot haver perdut la selenocisteïna però ha mantingut l’element SECIS.
Família PSTK
La fosfoseril-tRNA quinasa (PSTK) és una proteïna de maquinària conservada en eucariotes i arqueobacteris. Catalitza la reacció de seril-tRNA[Ser]Sec a la forma fosforilada fent servir una molècula d’ATP. La seva acció és essencial per la codificació específica UGA de les selenocisteïnes.
Es tracta d’una proteïna homòloga amb cisteïna (Cys) que es localitza en l’scaffold APW002000119.1. El seu gen conté 4 exons i no conté cap element SECIS. Cal tenir en compte que aquesta proteïna s’ha predit a partir de la proteïna PSTK de Danio rerio anotada al SelenoDB, la qual no comença amb metionina. Per aquest motiu pot ser que la nostra proteïna predita no estigui completa.
Família SBP2
Aquesta proteïna es localitza en l’scaffold APWO02000027.1 del genoma del nostre organisme, conté 9 exons i l’alineament amb t-coffee donà bons resultats. La seva predicció es va fer a partir del genoma de Danio rerio i es va observar que conté una selenocisteïna (Sec) en l’exó 8. Tot i això, no es va poder identificar cap element SECIS ni a través del Seblastian ni del SECISearch3. En el cas de la SBP2 de Danio rerio la proteïna conté una cisteïna, a diferència de la SBP2 del nostre organisme que conté una selenocisteïna. A més, cal tenir en compte que aquesta proteïna s’ha predit a partir de la proteïna de Danio rerio anotada al SelenoDB, la qual no comença amb metionina. Per aquest motiu, podem observar que la nostra proteïna predita tampoc comença amb metionina.
Família SecS
En els eucariotes i els arqueobacteris, la selenocisteïna sintasa (SecS) catalitza la reacció terminal de la selenocisteïna, en concret converteix O-fosfo-L-seril-tRNA[Ser]Sec a selenocisteil-tRNA[Ser]Sec gràcies a l’ús del selenofosfat com a donador de seleni.
Aquesta proteïna es localitza en l’scaffold APWOO2000275.1 i es va predir a partir del genoma de Danio rerio. El gen que codifica per a SecS conté 11 exons i no presenta cap selenocisteïna ni cap elements SECIS, ja que es tracta d’un homòleg de cisteïna (Cys).
Família SECp43
Durant l’alineament amb Danio rerio es van obtenir quatre hits al blast: CM008309.1, CM008322.1, APWO02000037.1 i CM008308.1. Els hits APWO02000037.1 i CM008309.1 van ser descartats i no es van analitzar ja que, tot i presentar un bon e-value, no presentaven una llargada corresponent al 80% de la proteïna que es volia predir. Pel que fa als scaffolds CM008308.1 i CM008322.1, van ser analitzats. L’anàlisi amb el hit CM008322.1 no va donar resultats concloents. El hit CM008308.1 va donar un bon alineament amb t-coffee, la nostra predicció conté 6 exons i no presenta selenocisteïna, presenta cisteïna. Per això, podem hipotetitzar, que l’Astyanax mexicanus podria tenir la proteïna de maquinària SECp43 i que aquesta es trobaria a l'scaffold CM008308.1.
Família TTPA
La proteïna TTPA es troba en l’scaffold APWO0200136.1 i es va predir a partir de l’alineament amb la proteïna TTPA de Homo sapiens. El seu gen conté 5 exons i no presenta cap selenocisteïna ni cap elements SECIS ja que es tracta d’un homòleg de cisteïna (Cys).
Família SELENBP1
La proteïna SELENBP1 es localitza en l’scaffold CM008309.1 i es va predir a partir del genoma de Homo sapiens. El gen que codifica per a aquesta proteïna conté 15 exons i conté una selenocisteïna (Sec) en l’exó 4. Tot i així, no es va predir cap element SECIS ni a través del Seblastian ni a través del SECISearch3. A més, s’ha observat que en el cas de la SELENBP1 d’Homo sapiens, la proteïna és un homòleg de cisteïna, a diferència de la SELENBP1 del nostre organisme que sí que conté una selenocisteïna.
Família RPL30
Aquesta proteïna es localitza en l’scaffold CM008322.1 i es va predir a partir del genoma d'Homo sapiens, ja que no es troba en Danio rerio. El gen que codifica per a la proteïna RPL30 conté 4 exons i no conté cap selenocisteïna ni cap elements SECIS, ja que es traca d’un homòleg de cisteïna (Cys).
Maquinària proteica no trobada
Família ELAVL1
La proteïna ELAVL1 va ser predita al genoma d’Astyanax mexicanus a partir de la proteïna ELAVL1 d’Homo sapiens. Durant l’alineament amb Homo sapiens es van obtenir molts hits de blast. Es va fer una tria d’aquells hits que presentessin un bon e-value i que la seva llargada es correspongués com a mínim al 80% de la proteïna que estàvem intentant predir. Després de fer aquesta selecció, només ens vam quedar amb dos hits, que estaven predits als scaffolds APWO02000150.1 i CM008305.1, respectivament. Es van analitzar aquests hits, i es va obtenir per ambdós casos molt mals alineaments amb t-coffee. La nostra hipòtesi és que Astyanax mexicanus no posseeix la proteïna de maquinaria ELAVL1 degut a tres raons raons:
En primer lloc, perquè Danio rerio no té aquesta proteïna i és una espècie molt propera a Astyanax mexicanus, i per tant, és possible que Astyanax mexicanus tampoc la tingui en el seu genoma.
En segon lloc, perquè no hem obtingut bons hits en el Blast per a la predicció d’aquesta selenoproteïna a partir d’humans en el genoma d’Astyanax mexicanus.
En tercer lloc, perquè com ja hem comentat, només hi havien dos hits que podien ser correctes (scaffolds APWO02000150.1 i CM008305.1), aquests no presentaven un cop analitzats un bon alineament amb t-coffee.
Família CELF1
La proteïna CELF1 va ser predita al genoma d’Astyanax mexicanus a partir de la proteïna CELF1 d’Homo sapiens. Durant l’alineament amb Homo sapiens es van obtenir molts hits de blast. Es va fer una tria d’aquells hits que presentessin un bon e-value i que la seva llargada es correspongués almenys al 80% de la proteïna que estàvem intentant predir. Després de fer aquesta selecció, només ens vam quedar amb un hit, que estava predit a l’scaffold APWO02000034.1. Es va analitzar aquest hit, i es va obtenir un molt mal alineament amb t-coffee. La nostra hipòtesi és que Astyanax mexicanus no posseeix la proteïna de maquinaria CELF1 degut a tres raons:
En primer lloc, perquè Danio rerio no té aquesta proteïna i és una espècie molt propera a Astyanax mexicanus, i per tant, és possible que Astyanax mexicanus tampoc la tingui en el seu genoma.
En segon lloc, perquè no hem obtingut bons hits en el Blast per a la predicció de CELF1 a partir d’humans en el genoma d’Astyanax mexicanus.
En tercer lloc, perquè com ja hem comentat, l’únic hit que podia ser correcte (scaffold APWO02001939), no presentava un cop analitzat, un bon alineament amb t-coffee.
Família EIF4A3
La proteïna EIF4A3 va ser predita al genoma d’Astyanax mexicanus a partir de la proteïna EIF4A3 d’Homo sapiens. Durant l’alineament amb Homo sapiens es van obtenir molts hits del blast. Es va fer una tria d’aquells hits que presentessin un bon e-value i que la seva llargada es correspongués almenys al 80% de la proteïna que estàvem intentant predir. Després de fer aquesta selecció, només ens vam quedar amb un hit, que estava predit a l’scaffold APWO02001939.1. Es va analitzar aquest hit, i respecte l’alineament amb t-coffee, cal dir que hi ha una part de la proteïna amb un bon alineament, però una altra part que no es troba tan ben alineada. Aquest fet pot ser degut a que la nostra espècie hagi perdut la proteïna i només estigui conservada una part de la proteïna, un sol domini, probablement no funcional. La nostra hipòtesi és que Astyanax mexicanus no posseeix la proteïna de maquinaria EIF4A3 degut a tres raons:En primer lloc, perquè Danio rerio no té aquesta proteïna i és una espècie molt propera a Astyanax mexicanus, i per tant, és possible que aquest tampoc la tingui en el seu genoma.
En segon lloc, perquè no hem obtingut bons hits en el Blast per a la predicció d’aquesta proteïna a partir d’humans en el genoma d’Astyanax mexicanus.
En tercer lloc, perquè com ja hem comentat, l’únic hit que podia ser correcte (scaffold APWO02000034.1), no presentava un cop analitzat, un bon alineament amb t-coffee.
G6PD
La proteïna G6PD va ser predita al genoma d’Astyanax mexicanus a partir de la proteïna G6PD d’Homo sapiens. Durant l’alineament amb aquest es van obtenir molts hits de blast. Es va fer una tria d’aquells hits que presentessin un bon e-value i que la seva llargada es correspongués almenys al 80% de la proteïna que estàvem intentant predir. Un cop aplicat aquest criteri, no es va obtenir cap hit que complís aquestes condicions. Per aquesta raó i pel fet que tampoc es troba a espècies filogenèticament semblants, tals com Danio rerio, hem hipotetitzat que la proteïna de maquinaria G6PD no es troba en el genoma d’Astyanax mexicanus.