 |
|||||||
| Resumen | Introducción | Materiales y Métodos | Resultados | Discusión | Conclusiones | Referencias | Agradecimientos |
|
|
IntroducciónEl selenio es un nutriente esencial para los microorganismos, animales y otros organismos eucariotas. A pesar de ello, presenta una gran toxicidad en su forma libre. En consecuencia, tanto su exceso como su déficit causan patología. La deficiencia de selenio deriva en la enfermedad de Keshan, que produce necrosis del miocardio y debilitación del corazón. Actualmente hay estudios que muestran que podría haber una asociación entre el consumo de selenio en la dieta y la prevención del cáncer de próstata, por lo que se le da aún mayor importancia a este nutriente. El exceso será tóxico debido a que sobrepasará las capacidades del organismo para quelar el selenio y, como hemos comentado anteriormente, en su forma libre éste es muy tóxico. Además, encontraremos el selenio en forma de selenocisteína, un aminoácido que se representa como Sec y U.1 SelenocisteínaLa selenocisteína no forma parte de los 20 aminoácidos principales, sino que se considera el aminoácido 21. Estructuralmente es igual a una cisteína pero en lugar de un átomo de azufre encontramos un átomo de selenio. Dadas las similitudes entre ambos aminoácidos podemos encontrar homólogos de cisteína en proteínas que en algún momento de la evolución tenían una selenocisteína en su secuencia, encontraremos así selenoproteínas y homólogos de cisteína.2 3 SelenoproteínasComo hemos comentado anteriormente, las selenoproteínas tienen como característica principal la presencia de selenocisteína en su secuencia. Son enzimas redox y hay estudios que sugieren que pueden tener efecto antioxidante, antitumoral y capacidad inmunológica referida a la capacidad de quelación del selenio.4 Las selenoproteínas están presentes en tres dominios: bacterias, arqueobacterias y la mayoría de eucariotas, pero no se encuentran en plantas. Además, encontramos gran varierad en cuanto a las especias que poseen estas proteínas. Por ejemplo, los ortólogos de una selenoproteína pueden ser homólogos de cisteína, algunas especies ni siquiera presentan dichas proteínas e incluso cambia el número de selenoproteínas que podemos encontrar en distintas especies.5 Síntesis de selenoproteínasGeneralmente su síntesis es igual a la de otras proteínas, la diferencia radica en que incluyen un aminoácido adicional. Explicaremos este aminoácido y su incorporación a la proteína en la traducción. Para poder sintetizar selenocisteínas sólo se puede usar selenio en forma de selenofosfato, porqué es la única forma en que se puede incorporar. Lo podemos obtener del selenio de la dieta gracias a la selenofosfato sintetasa (SPS2). La estructura encargada de introducir selenocisteínas a la secuencia de aminoácidos es la selenocisteinil-tRNA y su síntesis es la siguiente: La enzima seril-tRNA sintetasa une serina al tRNA que será el precursor de la selenocisteína. Entonces, la fosfoseril-tRNA quinasa (Pstk) fosforila la serina del tRNA y finalmente la selenocisteína sintetasa (SLA/LP) incorpora el selenofosfato al aminoácido. Eventualmente, esto lleva a la formación de selenocisteinil-tRNA.6 Una vez sintetizado el selenocisteinil-tRNA hay que incorporarlo a la proteína durante la traducción. El codón asignado a la selenocisteína es el UGA, que normalmente se traduce como un codón de parada. Así pues, la maquinaria de traducción debería parar la síntesis en el momento en que detecta dicho codón, pero en las secuencias que codifican para selenoproteína, la maquinaria no interpreta los codones UGA como STOP sino que introduce una selenocisteína y sigue con la traducción hasta el siguiente codón STOP.7 8 En eucariotas, la estructura responsable de este cambio en la interpretación del codón UGA es el SECIS (Selenocysteine Insertion Sequence) que es un loop de RNA situado en el extremo 3'-UTR. La maquinaria de traducción introduce selenocisteína en susodicho codón cuando hay un elemento SECIS gracias a dos proteínas: SBP2 (SECIS Binding Protein 2) y eEFsec. La proteína SBP2 se une a la estructura tridimensional del elemento SECIS y cuando SBP2 está unida a este, eEFsec se une a la maquinaria de traducción y recluta un selenocisteinil-tRNA. La incorporación de Sec y la función de eEFsec como factor de elongación permite que la traducción siga hasta el siguiente codón STOP.6 7 9 Gavialis gangeticusGavialis gangeticus también conocido como gavial o gharial, es un cocodriliano nativo de la parte norte del subcontinente de la índia. La población global actual se estima en menos de 235 individuos y está considerada actualmente como una especie en peligro critico de extinción. Es uno de los cocodrilos más grandes del mundo, puede llegar a medir 6.25m y, con 110 dientes puntiagudos e interdigitados y su estrecho hocico, está muy adaptado a la caza de peces.
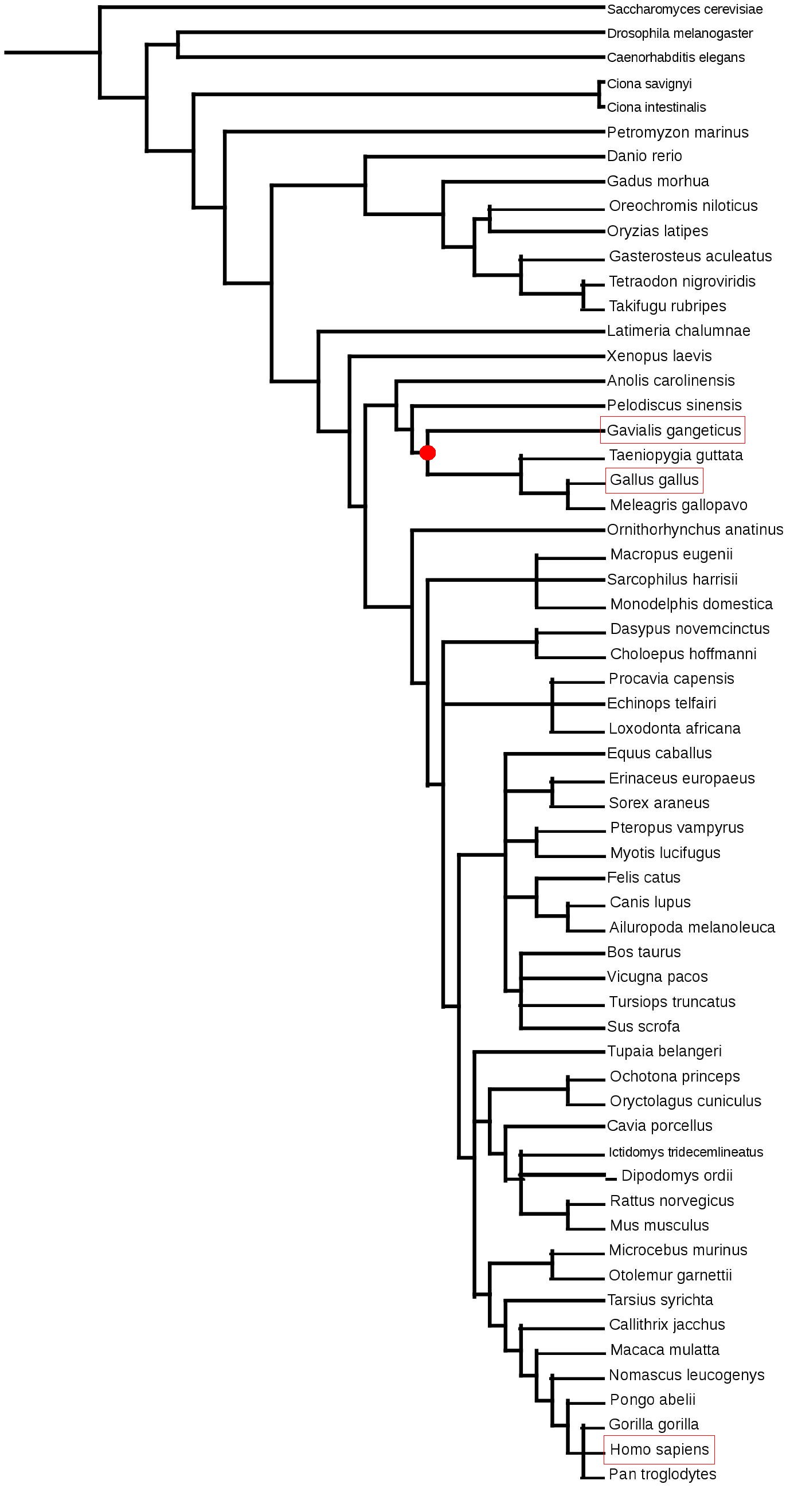
Los gaviales ocupaban antes la mayoría de los ríos del subcontinente indio, pero actualmente solo habitan en un 2% de todo su hábitat original.10 Dominio: Eukarya Reino: Animalia Filum: Chordata Clase: Reptilia Orden: Crocodilia Familia: Gavialidae Género: Gavialis Especie: G. gangeticus Elección del genoma de referenciaLos genomas filogenéticamente más próximos para los cuales tenemos notación de selenoproteínas disponible son las aves. Concretamente, hemos escogido el genoma del pollo (Gallus gallus) porqué, al ser un animal modelo de investigación, estará probablemente más estudiado.11 Además, también hemos usado como referencia el genoma humano porqué es el que está mejor estudiado en la actualidad. 
|