Selenoproteïnes
El requadre vermell de les proteïnes indica que no són selenoproteïnes en P.coquereli degut a la falta de l'element SECIS o l'absència de U.
Els colors verd/vermell indiquen si s'han trobat resultats o no.
| Proteïna | Sec humà | BLAST | Exonerate | Prot (aa) | Scaffold | Localització gen (*) | T-coffee | SECIS | |||||
| SeciSearch3 | Seblastian | Imatge | |||||||||||
| FAMÍLIA DI | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DI1 | 14083582-14096555 | ||||||||||||
| DI2 | 19911742-19928515 | ||||||||||||
| DI3 | 2947064-2947897 | ||||||||||||
| FAMÍLIA GPx | |||||||||||||
| GPx1 | 12270918-1271776 | ||||||||||||
| GPx2 | 5826257-5829181 | ||||||||||||
| GPx3 | 2141584-2148087 | ||||||||||||
| GPx4 | 3517665-3493844 | ||||||||||||
| GPx6 | 701683-702139 | ||||||||||||
| FAMÍLIA Sel | |||||||||||||
| Sel15 | 11748610-11795147 | ||||||||||||
| SelH | 2413222-2413815 | ||||||||||||
| Sell.A | 2230387-2263516 | ||||||||||||
| Sell.B | 3834908-3836070 | ||||||||||||
| SelK | 16342077-16347813 | ||||||||||||
| SelM | 792350-794803 | ||||||||||||
| SelN | 3264076-3276444 | ||||||||||||
| SelO | 3674731-3690021 | ||||||||||||
| SelP | - | ||||||||||||
| SelR | 801583-804872 | ||||||||||||
| SelS | 1053328-1060392 | ||||||||||||
| SelT | 9201195-9241725 | ||||||||||||
| SelV | - | ||||||||||||
| SelW.A | 4442672-4443168 | ||||||||||||
| SelW.B | 2800959-2801202 | ||||||||||||
| SelW.C | 14083430-1408544 | ||||||||||||
| SPS2.A | 50700666-5089015 | ||||||||||||
| SPS2.B | 2090301-2091694 | ||||||||||||
| SPS2.C | 5751951-5753241 | ||||||||||||
| SPS2.D | 10096654-10098102 | ||||||||||||
| FAMÍLIA TR | |||||||||||||
(*) La localització del gen s'ha obtingut a partir de la la coordenada incical del primer exó i de la coordenada final de l'últim exó.
Maquinària
A la maquinària no vam poder executar l’EXONERATE ni el TCOFFEE i, per tant, no vam poder realizar l’alineament. Creiem que això pot ser degut a que la seqüència proteica tenia una longitud major a la del scaffold (aquest mostra regions no seqüenciades de manera correcta).
DI1
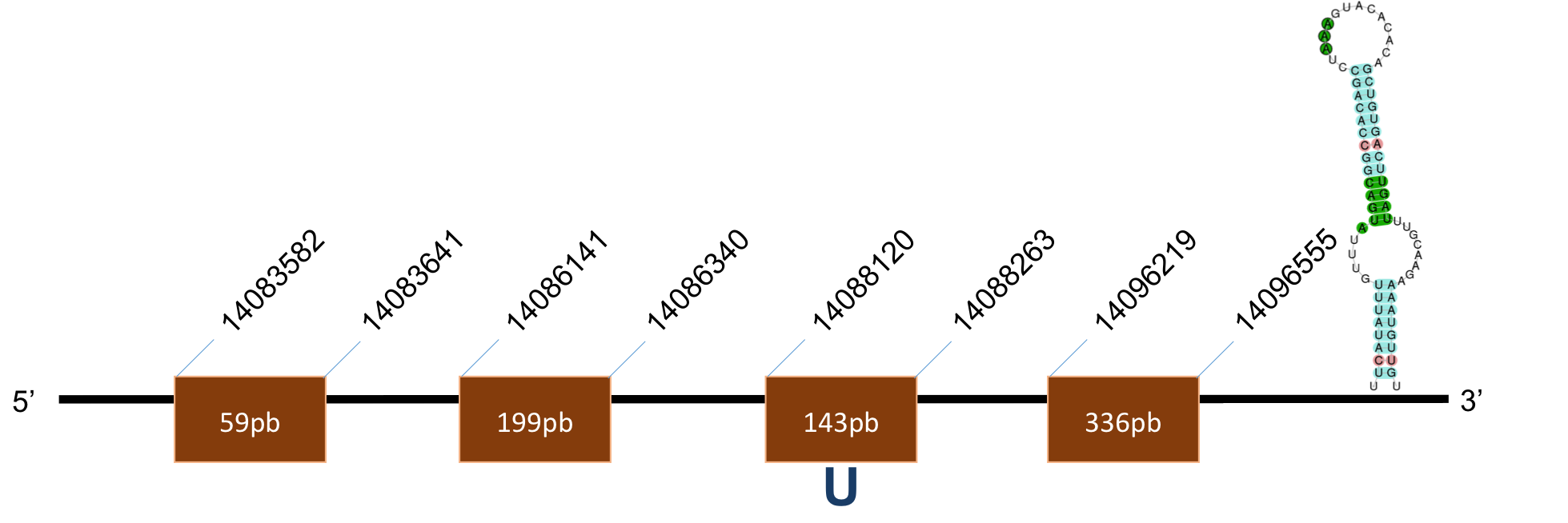
La proteïna DI1 està localitzada al scaffold KQ017406,1 del genoma de Prophitecus coquereli. Trobem un hit clar (identitat> 75%, E-value < 10-20) a l'output del BLAST.
L’alineament presenta un score de 1000, sent aquesta la puntuació màxima donada pel software utilitzat (TCOFFEE), per a una proteïna de 4 exons, amb una selenocisteïna conservada a la posició 375 de l’exó 1. L'element SECIS és de grau A i, es posiciona a les coordenades 23997-22067 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 1295 pb.
DI2
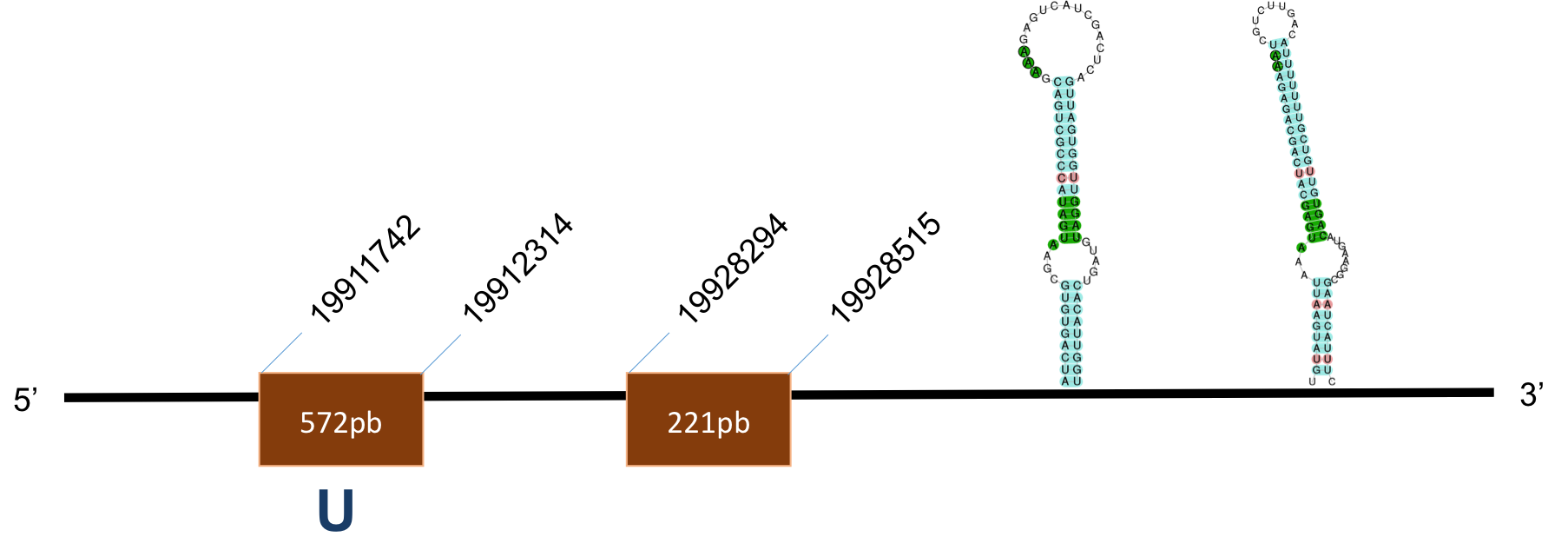
La proteïna DI2 està localitzada al scaffold KQ024846,1 del genoma Prophitecus coquereli. Trobem dos hits (identitat > 84%, E-value < 10-39 i 10-113, tractant-se d’un exó petit i un exó gran, respectivament) l'output del BLAST.
L’alineament presenta un score de 999, per a una proteïna de 2 exons, amb una selenocisteïna que coincideix amb la d'Homo sapiens i una selenocisteïna a prop de 3’UTR a la query, no es troba conservada a P.coquereli. Trobem dos element SECIS, un de grau A que es posiciona a les coordenades 23997-22067 de la target. I un altre de grau A que es posiciona a les coordenades 59210-59287.
DI3
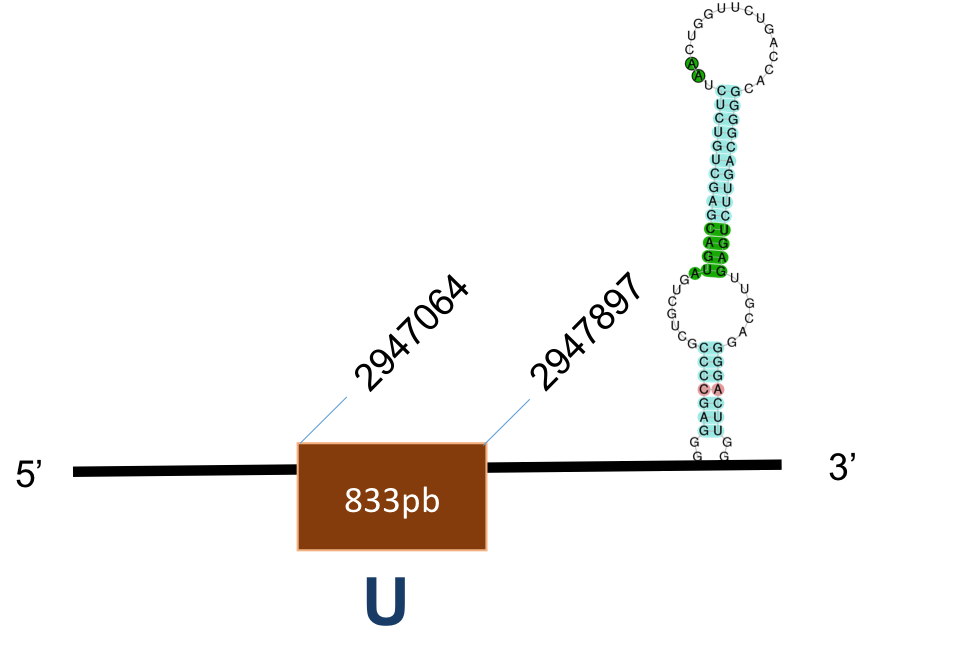
La proteïna DI3 està localitzada al scaffold KQ022136,1 del genoma de P.coquereli. Trobem un hit (identitat 97% i un E-value 10-176). Hem observat que aquesta proteïna no conté introns.
L’alineament presenta un score de 1000, per a una proteïna amb 1 exó. Trobem un element SECIS de grau A a les posicions 24351-24427. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 974 pb.
GPx1
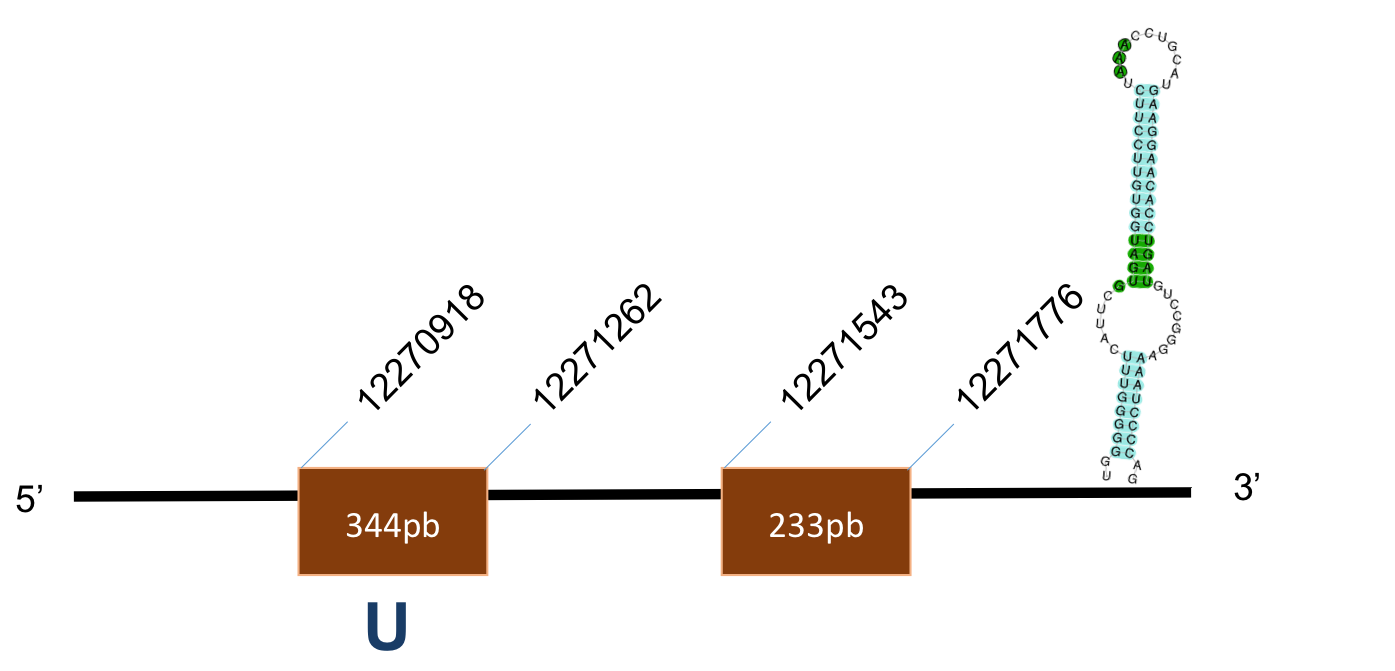
La proteïna GPx1 està localitzada al scaffold KQ012994,1 del genoma de P.coquereli. Trobem un hit òptim (identitat > 92% i un E-value < 10-36) a l'output del BLAST.
L’alineament presenta un score de 993, per a una proteïna amb dos exons de la mateixa mida, aproximadament, d’uns 200-300 aminoàcids. Es tracta d’una proteïna petita.
L’element SECIS és de grau A i es posiciona a les coordenades 24882-24956. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 493 pb.
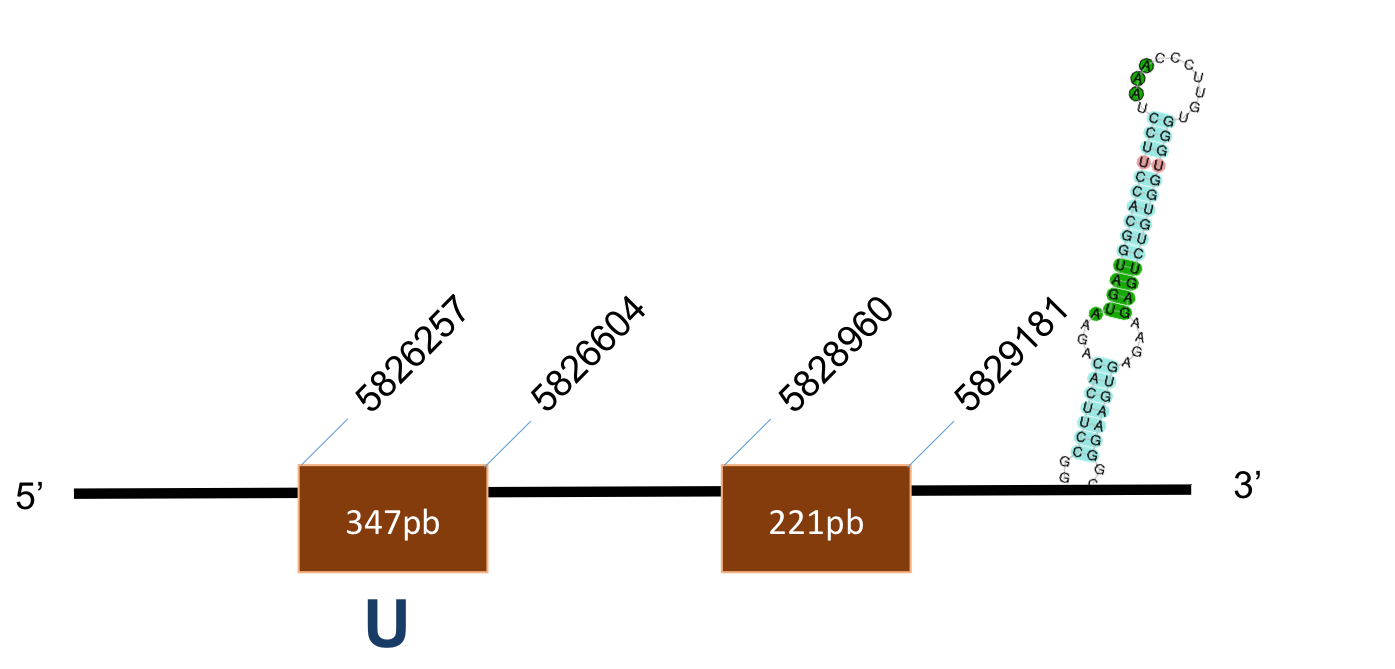
GPx2
La proteïna GPx2 es troba localitzada al scaffold KQ024846,1 del genoma de P.coquereli. Trobem un hit clar (identitat > 96% i E-value < 10-40) a l'output del BLAST.
L’alineament presenta un score de 1000, sent aquesta la puntuació màxima donada pel software utilitzat (TCOFFEE), per a una proteïna de 2 exons, amb una selenocisteïna. L’element SECIS és de grau A i, es posiciona a les coordenades 24718-24782 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 667 pb.
GPx3
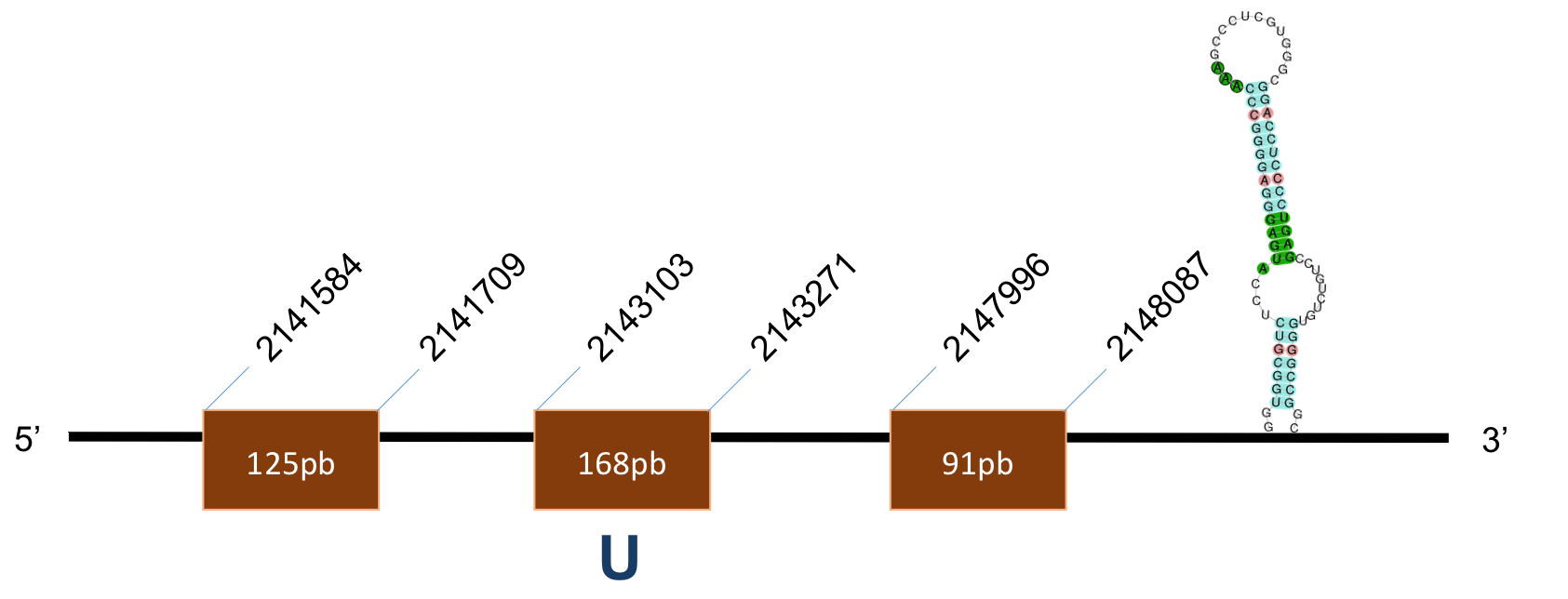
La proteïna GPx3 es troba localitzada al scaffold KQ025735,1 del genoma de P.coquereli. Trobem un hit òptim (identitat > 73% i E-value < 10-4) a l'output del BLAST.
L’alineament presenta un score de 998, per a una proteïna de 3 exons. L’element SECIS és de grau A i, es posiciona a les coordenades 24320-24393 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 1053 pb.
GPx4
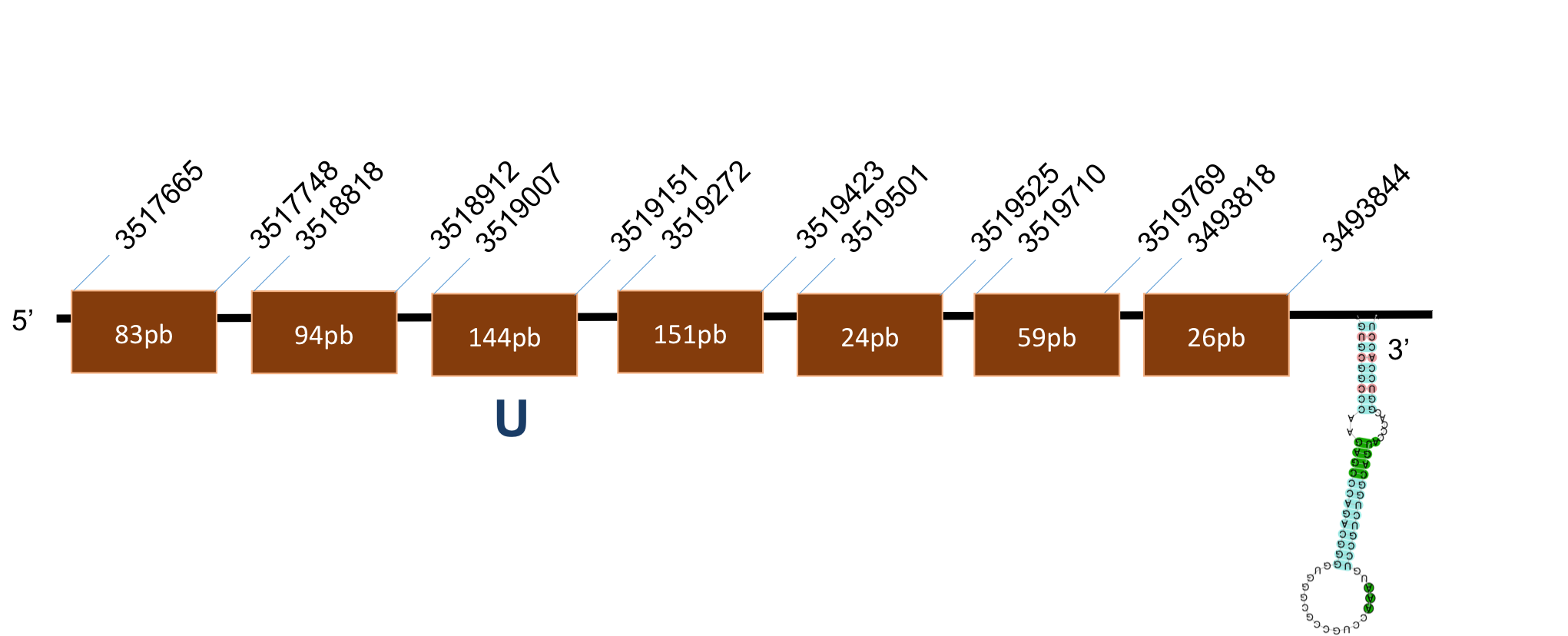
La proteïna GPx4 es troba localitzada al scaffold KQ021870,1 del genoma de P.coquereli. Trobem un hit destacat (identitat > 90% i E-value < 10-56) a l'output del BLAST.
L’alineament presenta un score de 1000, per a una proteïna de 7 exons. L’element SECIS és de grau A i es posiciona a les coordenades 26113-26184 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 426 pb.
GPx6
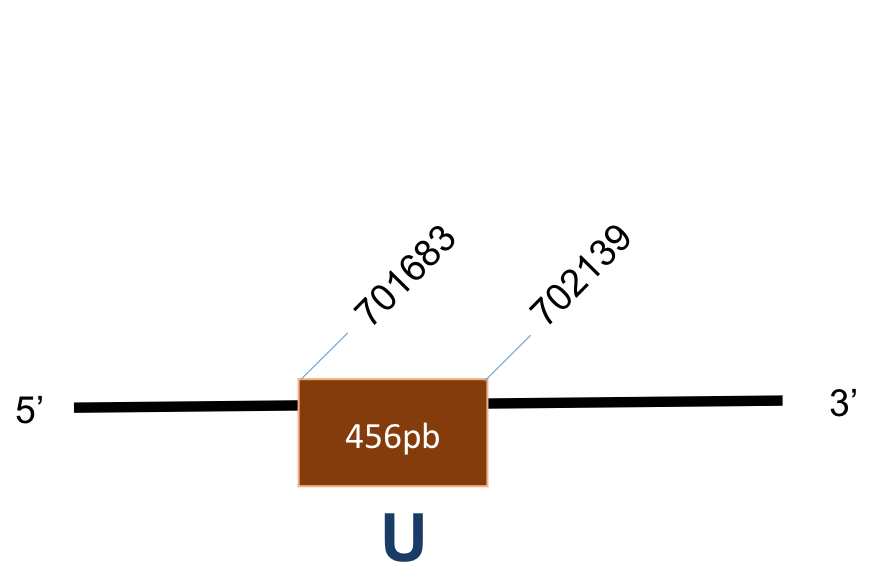
La proteïna GPx6 es troba localitzada al scaffold KQ021614,1 del genoma de P.coquereli. Trobem un hit òptim (identitat entre 45% - 84%,- i E-value < 10-55) a l'output del BLAST.
L’alineament presenta un score de 981, per a una proteïna d’un exó. No es troba element SECIS.
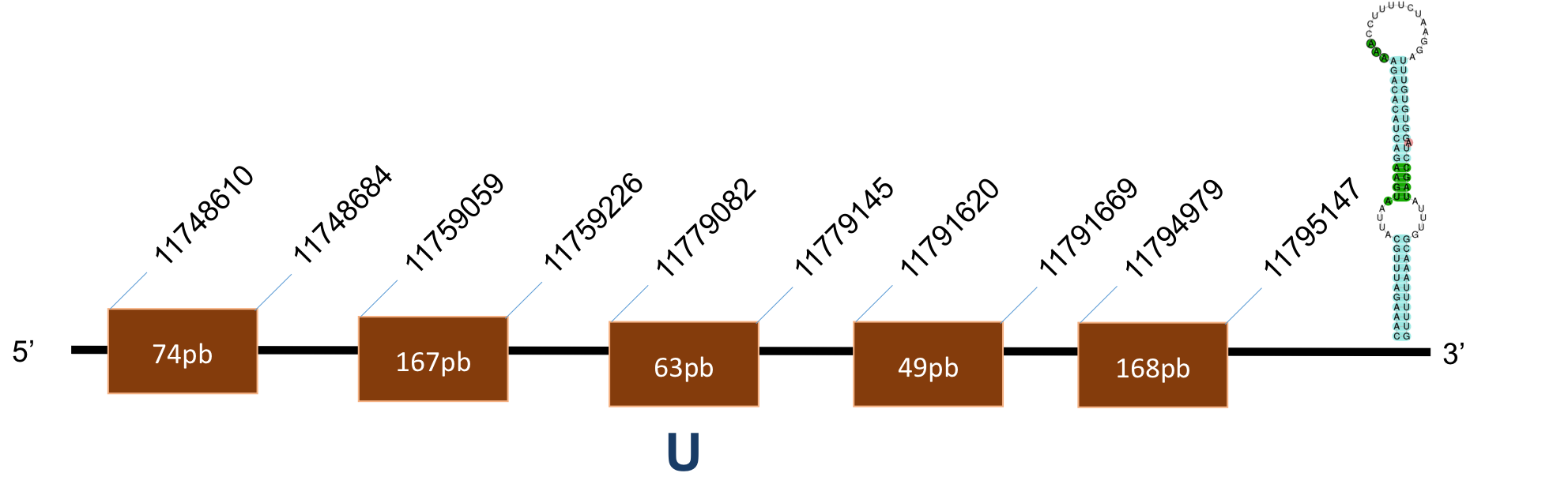
Sel 15
La proteïna Sel15 es troba localitzada al scaffold KQ027066,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant de 90% i E-value < 10-4) a l'output del BLAST. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli.
L’alineament presenta un score de 1000, per a una proteïna de 5 exons. L’element SECIS és de grau A i, es posiciona a les coordenades 72066-72144 de la target.
Sel H
La proteïna SelH es troba localitzada al scaffold KQ021537,1 del genoma de P.coquereli. Trobem dos hits; el hit òptim té una identitat al voltant de 90% i E-value < 10-11) a l'output del BLAST.
L’alineament presenta un score de 1000, per a una proteïna de 3 exons. L’element SECIS és de grau A i, es posiciona a les coordenades 24146-24213 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 1020 pb.
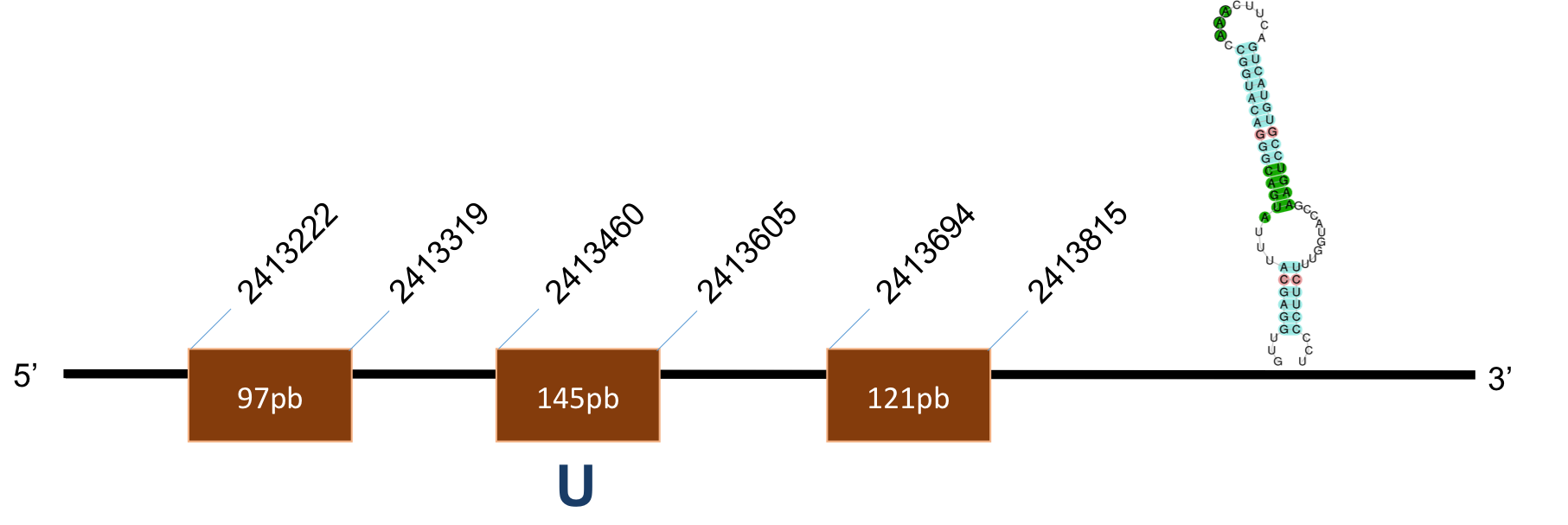
Sel I
Hem dividit els resultats d’aquesta proteïna segons dos hits considerats òptims i que es volien avaluar individualment. Els hem anomenat SelI.A i SelI.B.
SelI A
La proteïna SelI.A es troba localitzada al scaffold KQ023624,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant de 80% i E-value al voltant de 10
L’alineament presenta un score de 1000, per a una proteïna de 10 exons. L’element SECIS és de grau A i es posiciona a les coordenades 59394-59471 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 1294 pb.
SelI B
La proteïna SelI.B es troba localitzada al scaffold KQ013437,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant de 80% i E-value al voltant de 10-13) a l'output del BLAST. Identitat dividida en dues regions: en la regió N-terminal amb una longitud de 300 aminoàcids aproximadament amb una identitat de 85% i un E-value de 10-171, ja que el hit és molt llarg. I la regió C-terminal amb una longitud de 90 aminoàcids aproximadament amb una identitat de 49% i un E-value de 10-17.
El genoma de P.coquereli presenta un residu I al lloc de la Sec en H.sapiens.
L’alineament presenta un score de 978, per a una proteïna de 2 exons. La query conté una U, que no coincideix amb cap de les U’s del hit. L’element SECIS és de grau B i es posiciona a les coordenades 7782-7858 de la target.
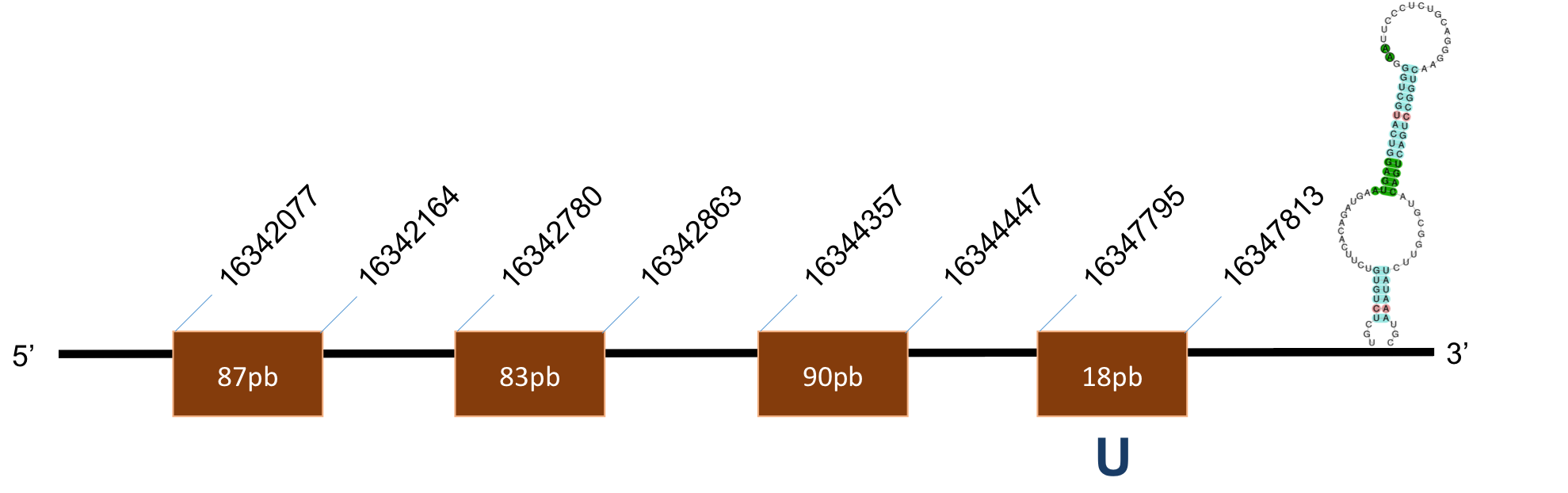
Sel K
La proteïna SelK es troba localitzada al scaffold KQ012994,1 del genoma de P.coquereli. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli.
Com s'ha comprovat a la bibliografia, es tracta d’una molècula petita de 94 aminoàcids. S’han trobat dos hits, dels quals un té un E-value molt petit i l’altre té una identitat al voltant d’un 70% i un E-value inferior a 10-7.
L’alineament presenta un score de 1000, per a una proteïna de 4 exons. L’element SECIS és de grau A i es posiciona a les coordenades 23423-23510 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 784 pb.
Sel M
La proteïna SelM es troba localitzada al scaffold KQ025591,1 del genoma de P.coquereli. Trobem un hit destacat (identitat de 67% i E-value < 10-34) a l'output del BLAST. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli.
L’alineament presenta un score de 1000, per a una proteïna de 5 exons. Conté dos elements SECIS, un és de grau B i, es posiciona a les coordenades 16366-16442 de la target i l’altre és de grau A i es posiciona a les coordenades 24885-24956.
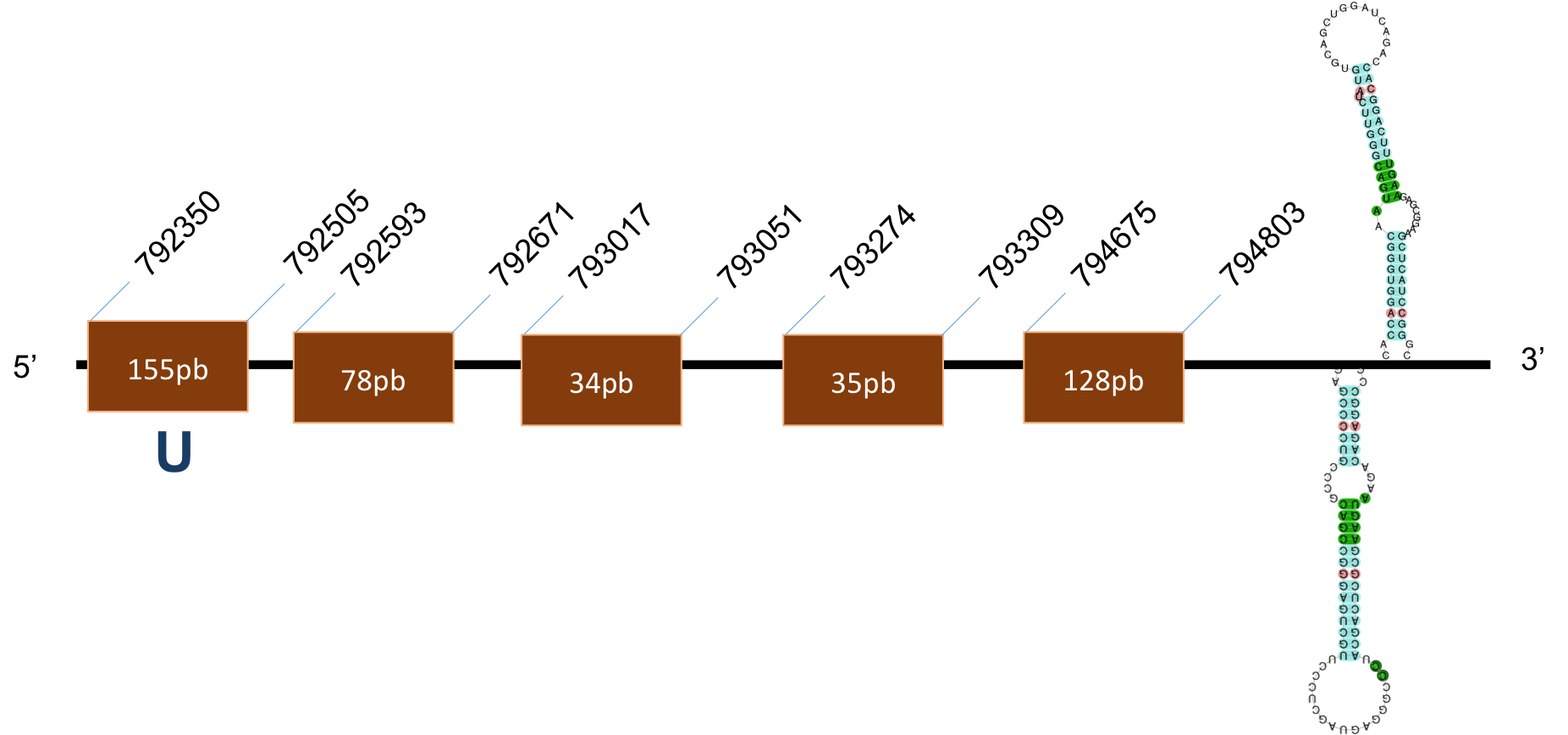
Sel N
La proteïna SelN es troba localitzada al scaffold KQ021947,1 del genoma de P.coquereli. Trobem un hit destacat (identitat > 80% i E-value < 10-16) a l'output del BLAST. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli.
L’alineament presenta un score de 1000, per a una proteïna d'11 exons. Conté dos elements SECIS, un és de grau A i, es posiciona a les coordenades 23735-23802 de la target i l’altre és de grau B i es posiciona a les coordenades 26658-26742. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 2886 pb.
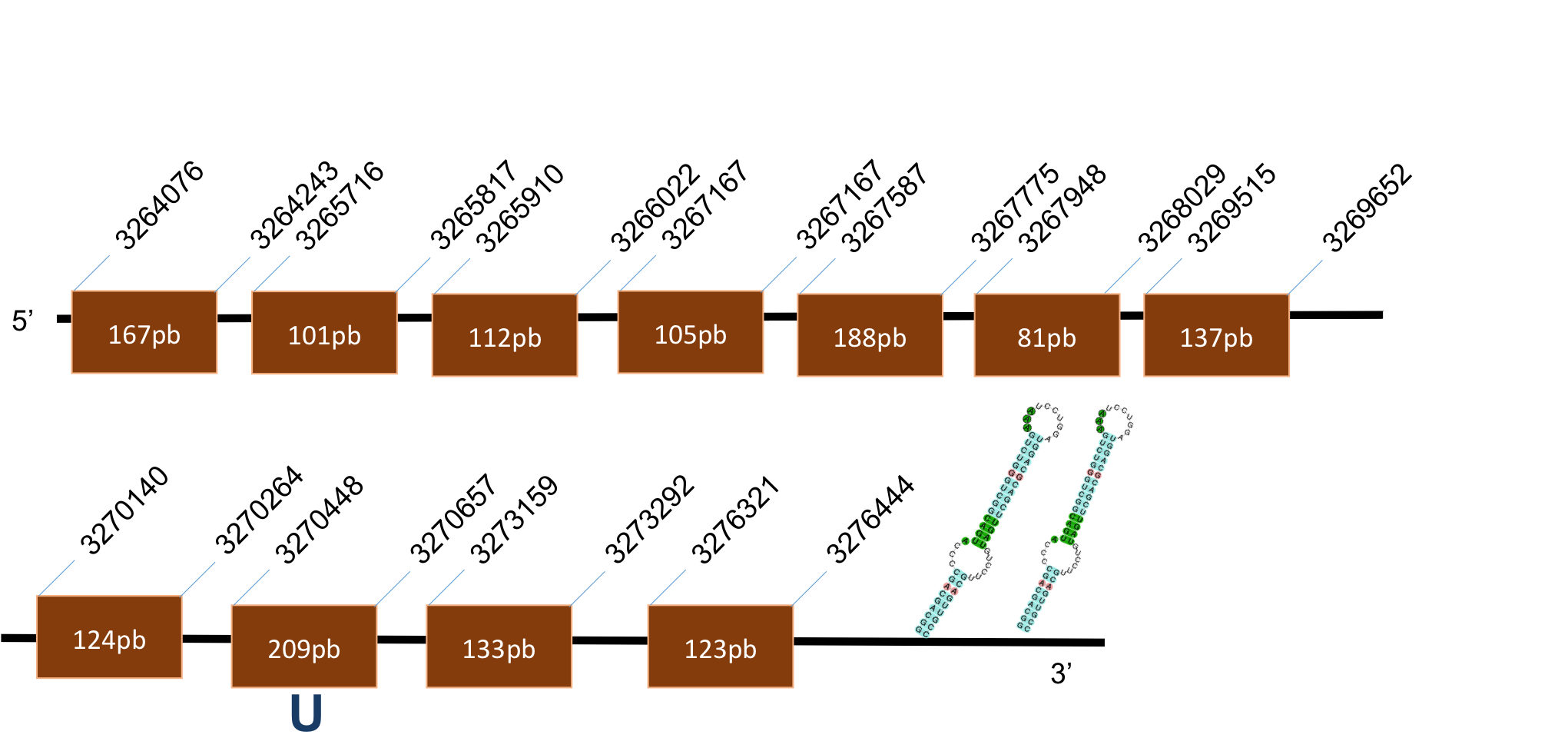
Sel O
La proteïna SelO es troba localitzada al scaffold KQ022558,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant d’un 74% i E-value < 10-17) a l'output del BLAST. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli.
L’alineament presenta un score de 989, per a una proteïna de 9 exons. L’element SECIS és de grau A i es troba a les posicions 24837-24913. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 92 pb.
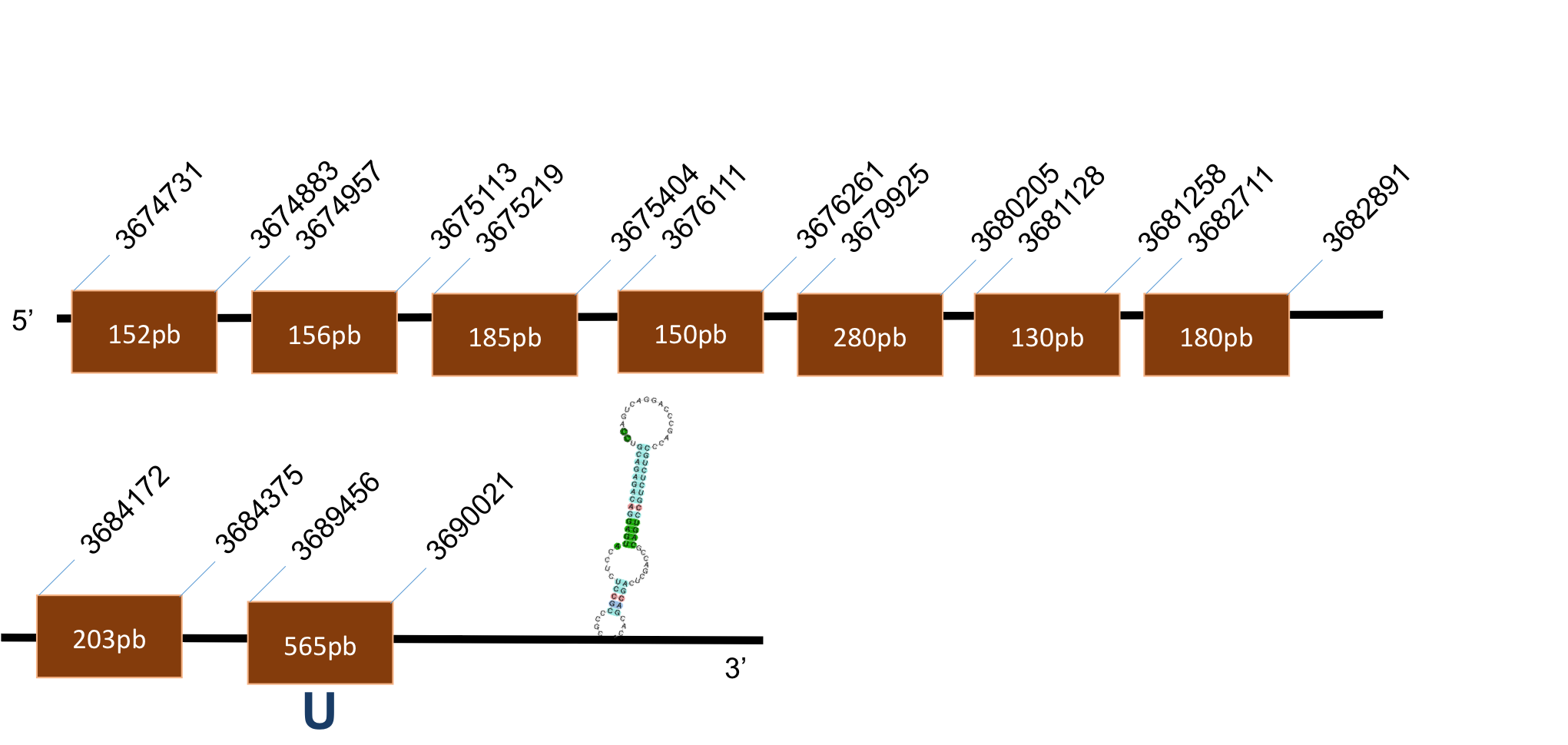
Sel P
La proteïna SelP es troba localitzada al scaffold KQ029394,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant del 80% i E-value < 10-17) a l'output del BLAST.
L’alineament ha sortit malament degut a un error a l’EXONERATE que no hem sabut diagnosticar. És per això que ens dóna un alineament buit. Conté dos elements SECIS, un és de grau A i es posiciona a les coordenades 24226-24291 de la target i l’altre és i es troba a la posició 24665-24737. Els elements SECIS han estat predits amb el programa Search3 i, per tant no hem obtingut cap alineament.
Sel R
La proteïna SelR es troba localitzada al scaffold KQ022291,1 del genoma de P.coquereli. Trobem un hit destacat (identitat > 80% i E-value < 10-16) a l'output del BLAST. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli.
L’alineament presenta un score de 1000, per a una proteïna de 4 exons. L’element SECIS és de grau A i es posiciona a les coordenades 23456-23528 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 507 pb.
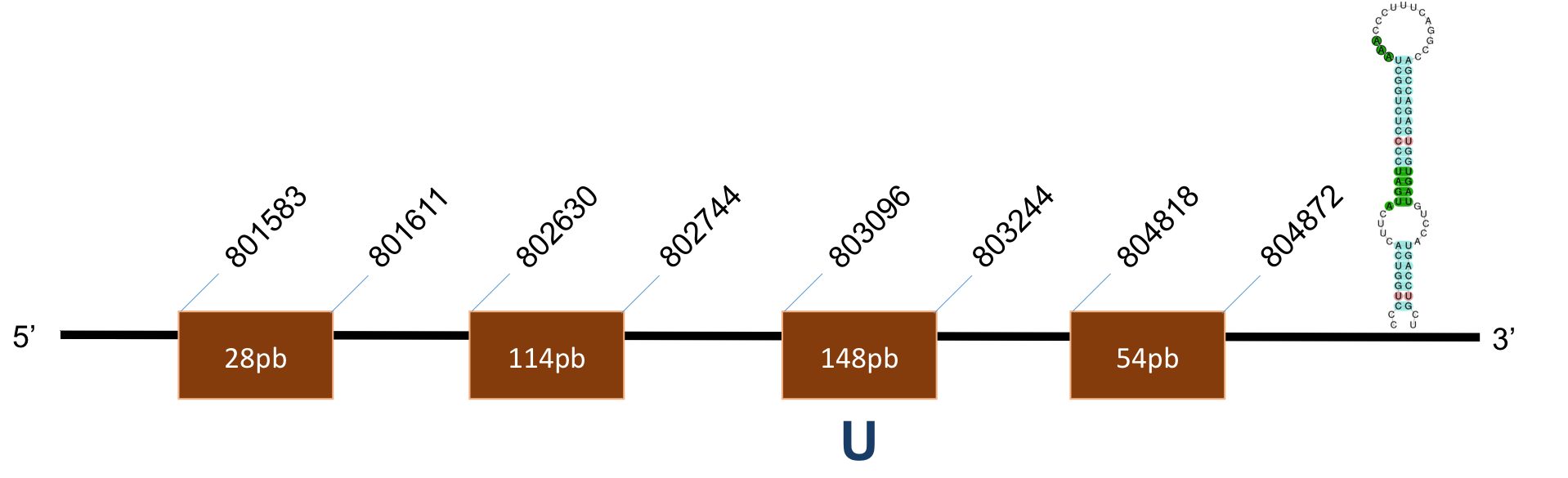
Sel S
La proteína SelS es troba localitzada al scaffold KQ021336,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant d’un 90% i E-value < 10-8) a l'output del BLAST. No presenta selenocisteïna sinó un gap en P.coquereli.
L’alineament presenta un score de 992, per a una proteïna de 6 exons. Conté dos elements SECIS, un és de grau A i es posiciona a les coordenades 24539-24618 de la target i l’altre és de grau B i es posiciona a 2991-3080.
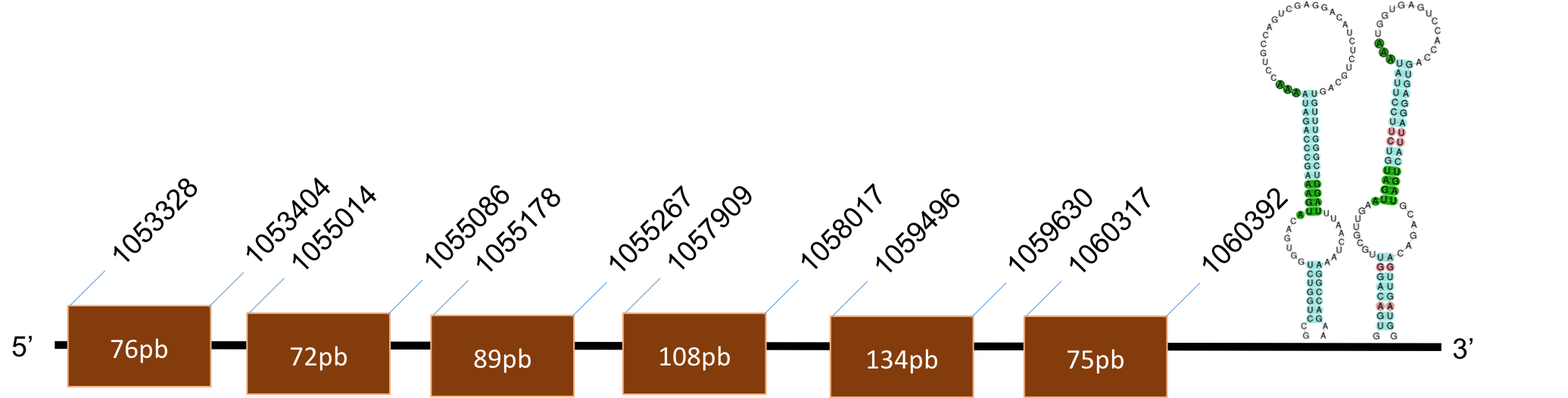
Sel T
La proteïna SelT es troba localitzada al scaffold KQ021402,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant del 90% i E-value < 10-10) a l'output del BLAST. La posició de la selenocisteïna coincideix entre H.sapiens i P.coquereli i, a més, trobem una altra Sec a la proteïna de P.coquereli.
L’alineament presenta un score de 993, per a una proteïna de 5 exons. L’element SECIS és de grau A i es posiciona a les coordenades 24339-24413 de la target.
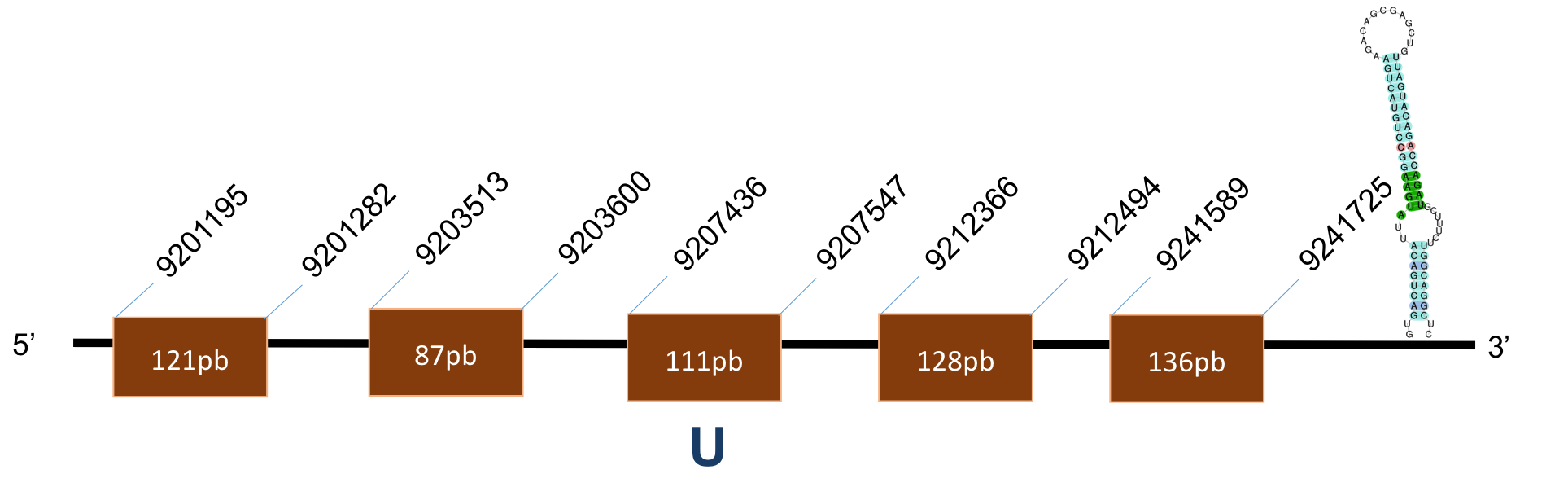
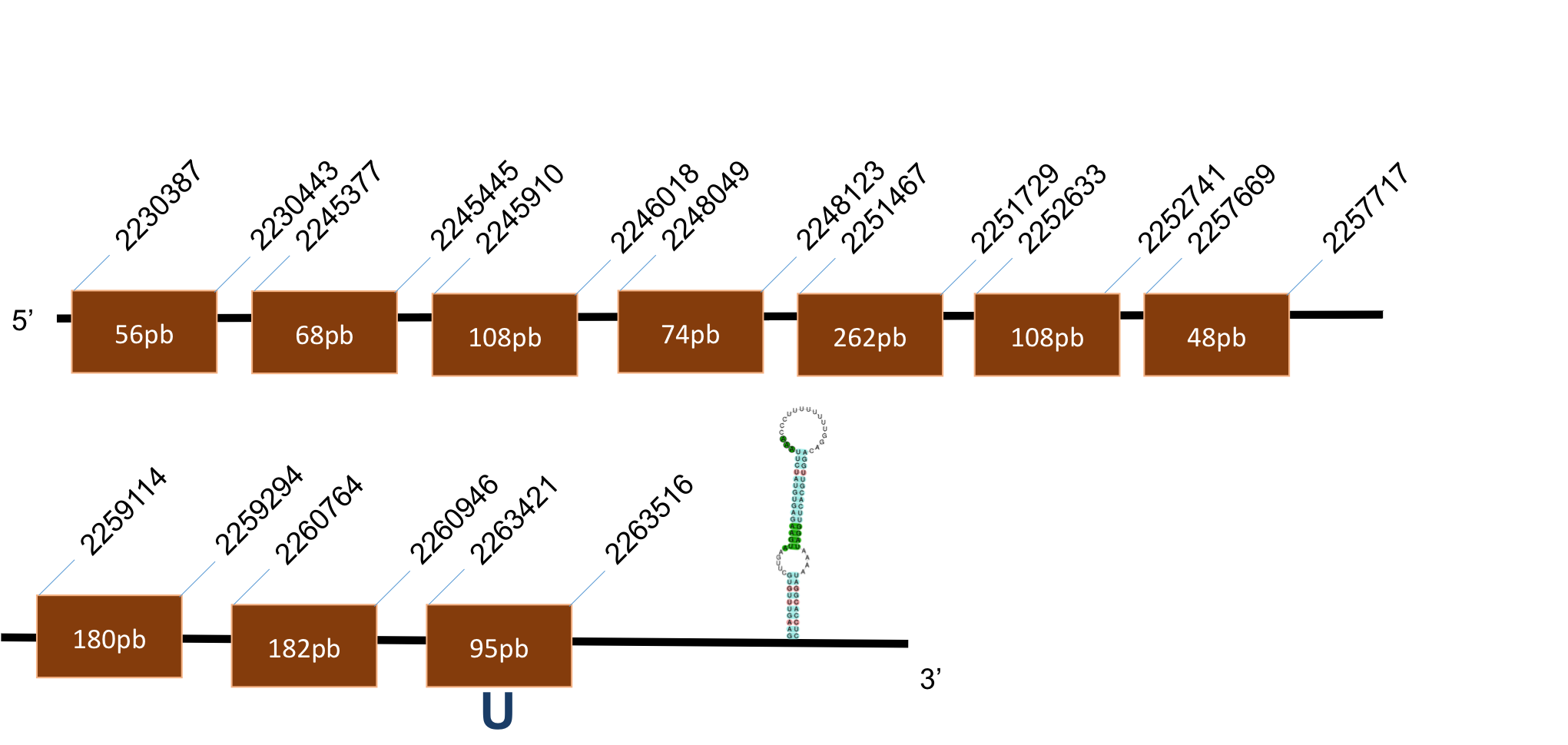
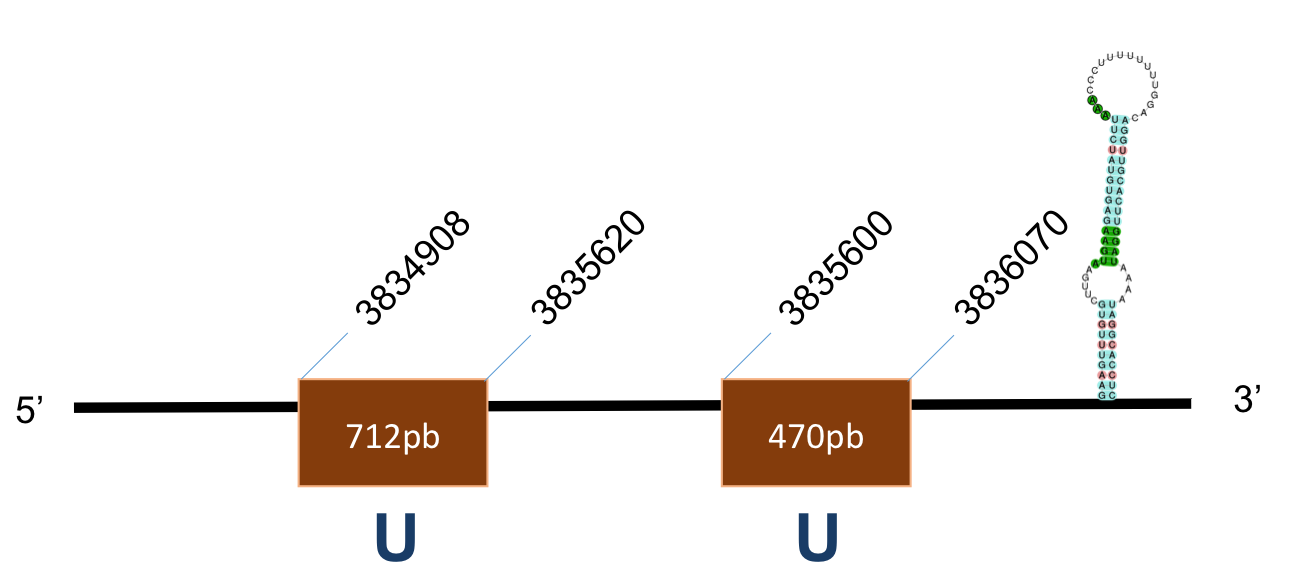
Sel V
La proteïna SelV es troba localitzada al scaffold KQ025180,1 del genoma de P.coquereli. Trobem un hit (identitat al voltant del 50% i E-value < 10-5) a l'output del BLAST. El genoma d’H.sapiens presenta dues Sec que no es troben al P.coquereli, a més, a la proteïna de P.coquereli trobem Secs que no es troben al H.sapiens.
L’alineament presenta un score de 1000, per a una proteïna de “11 exons”, però no ho són exactament ja que es troben superposats. L’element SECIS és de grau A i es posiciona a les coordenades 23136-23216 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 2002 pb.
Sel W
Trobem 3 hits, és una proteïna de 87 aminoàcids. A continuació presentem els tres hits de la SelW’s:
SelW A
La proteïna SelW A es troba localitzada al scaffold KQ023958,1 del genoma de P.coquereli. Trobem un hit destacat (identitat 96% i E-value 10-8) a l'output del BLAST. A la posició de la Sec d'H.sapiens (a prop de l’extrem 5’) trobem un gap a P.coquereli. No hi ha selenocisteïna a la target.
L’alineament presenta un score de 999, per a una proteïna de 3 exons. L’element SECIS és de grau A i es posiciona a les coordenades 27772-27852 de la target. No hi ha selenocisteïna a la target.
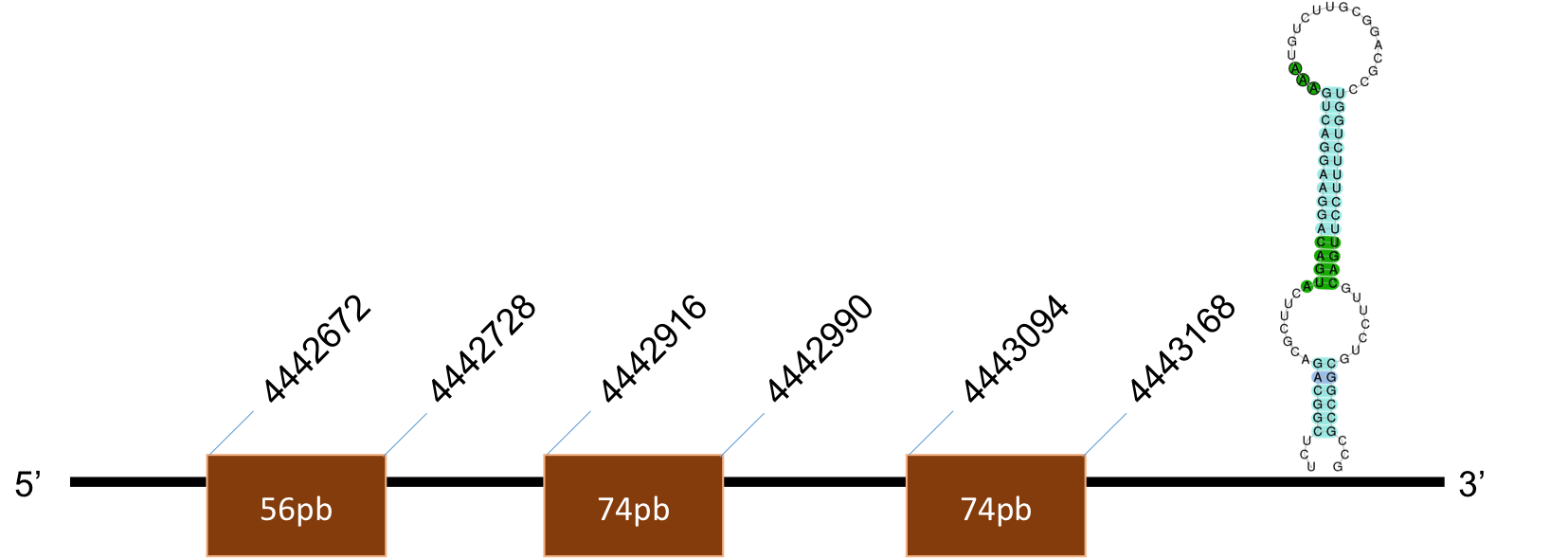
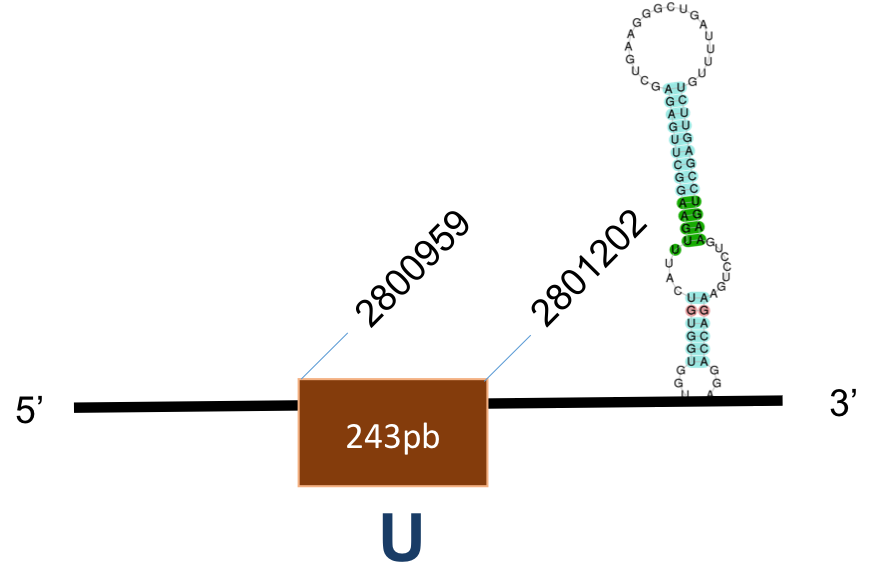
SelW B
La proteïna SelW B es troba localitzada al scaffold KQ028730,1 del genoma de P.coquereli. Trobem un hit (identitat 43% i E-value 10-6) a l'output del BLAST. A la posició de la Sec d'H.sapiens (a prop de l’extrem 5’) trobem un residu de Q a P.coquereli.
L’alineament presenta un score de 967, per a una proteïna de 1 exó. L’element SECIS és de grau B i es posiciona a les coordenades 48825-48897 de la target. Trobem una selenocisteïna en una posició diferent a la de la query.
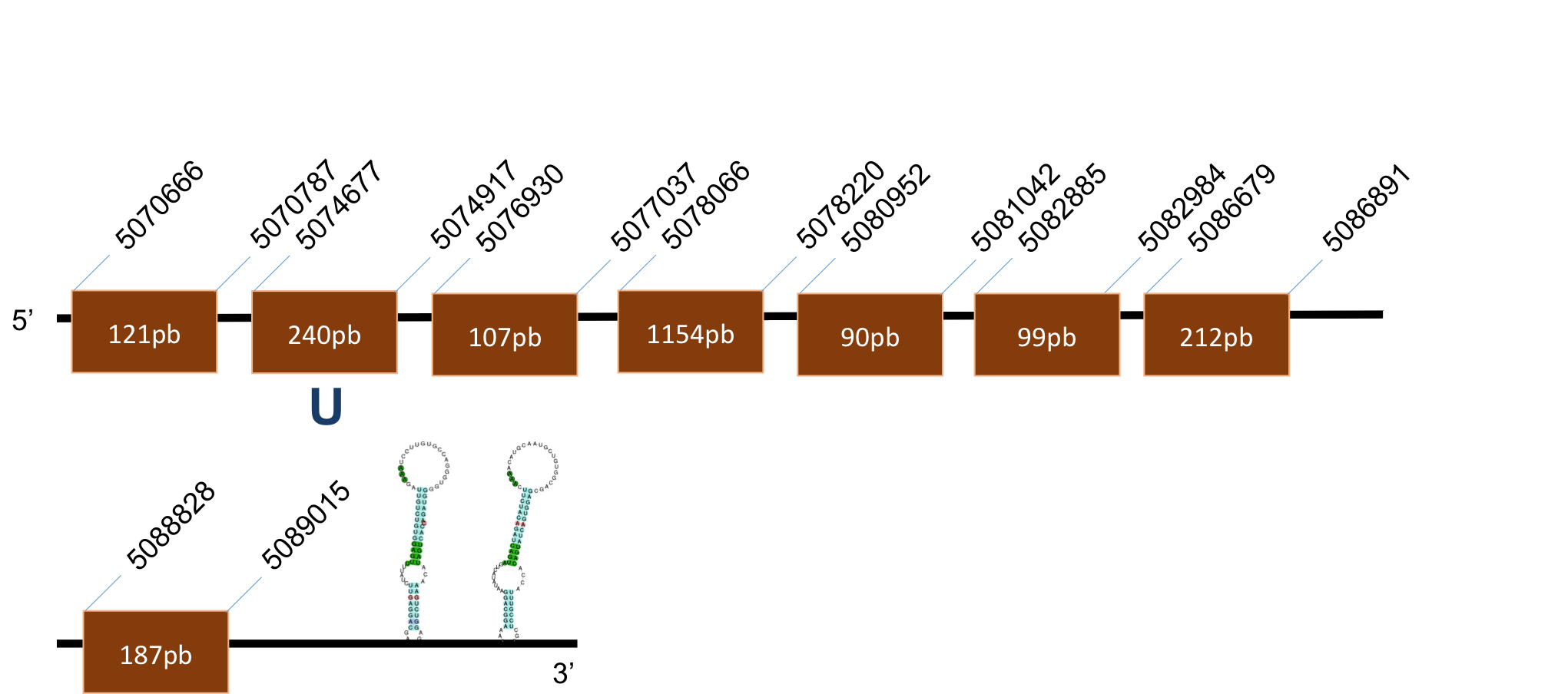
SelW C
La proteïna SelW C es troba localitzada al scaffold KQ025180,1 del genoma deP.coquereli. Trobem un hit destacat (identitat 84% i E-value 0,21) a l'output del BLAST.
L’alineament presenta un score de 988, per a una proteïna de 2 exons. L’element SECIS és de grau A i es posiciona a les coordenades 22966-23046 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 2002 pb.
SPS2
Trobem més d’un hit òptim per a aquesta proteïna i, per tant, hem volgut analitzar-les independentment.
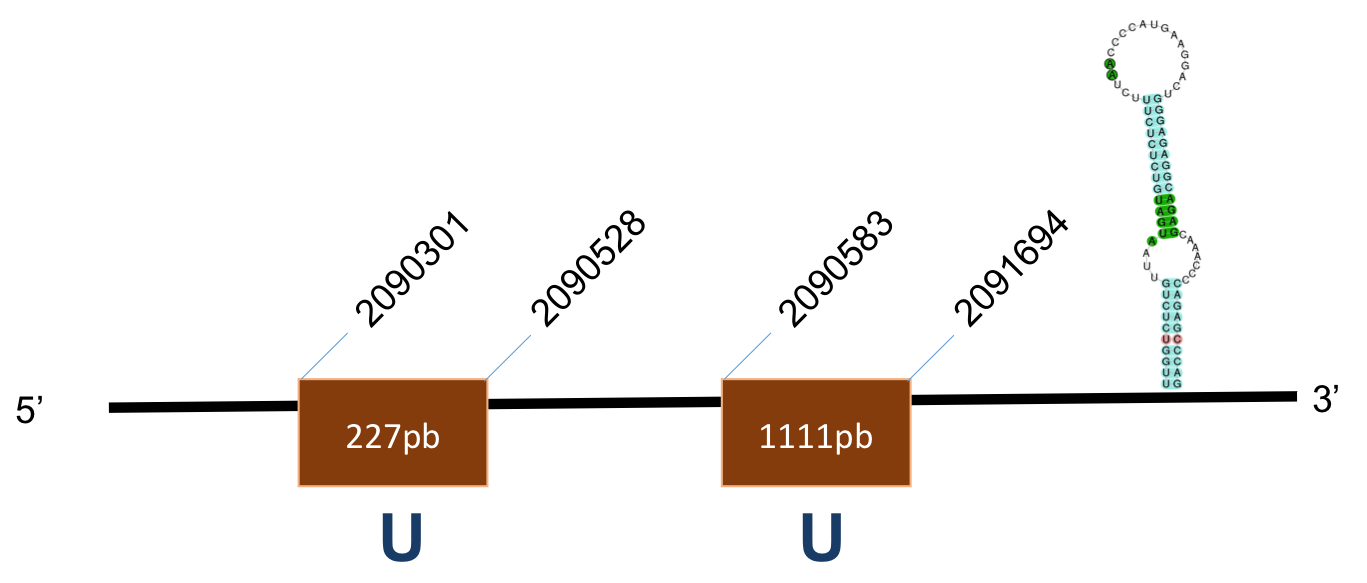
SPS2 A
La proteïna SPS2A es troba localitzada al scaffold KQ026955,1 del genoma de P.coquereli. Trobem un hit (identitat al voltant del 70% i E-value < 10-7) a l'output del BLAST. La posició de la Sec a la query i a la target no coincideix.
L’alineament presenta un score de 998, per a una proteïna de 8 exons. Conté dos elements SECIS, un és de grau B i es posiciona a les coordenades 8962-9047 de la target i l’altre és de grau B i es posiciona a 50432-50509.
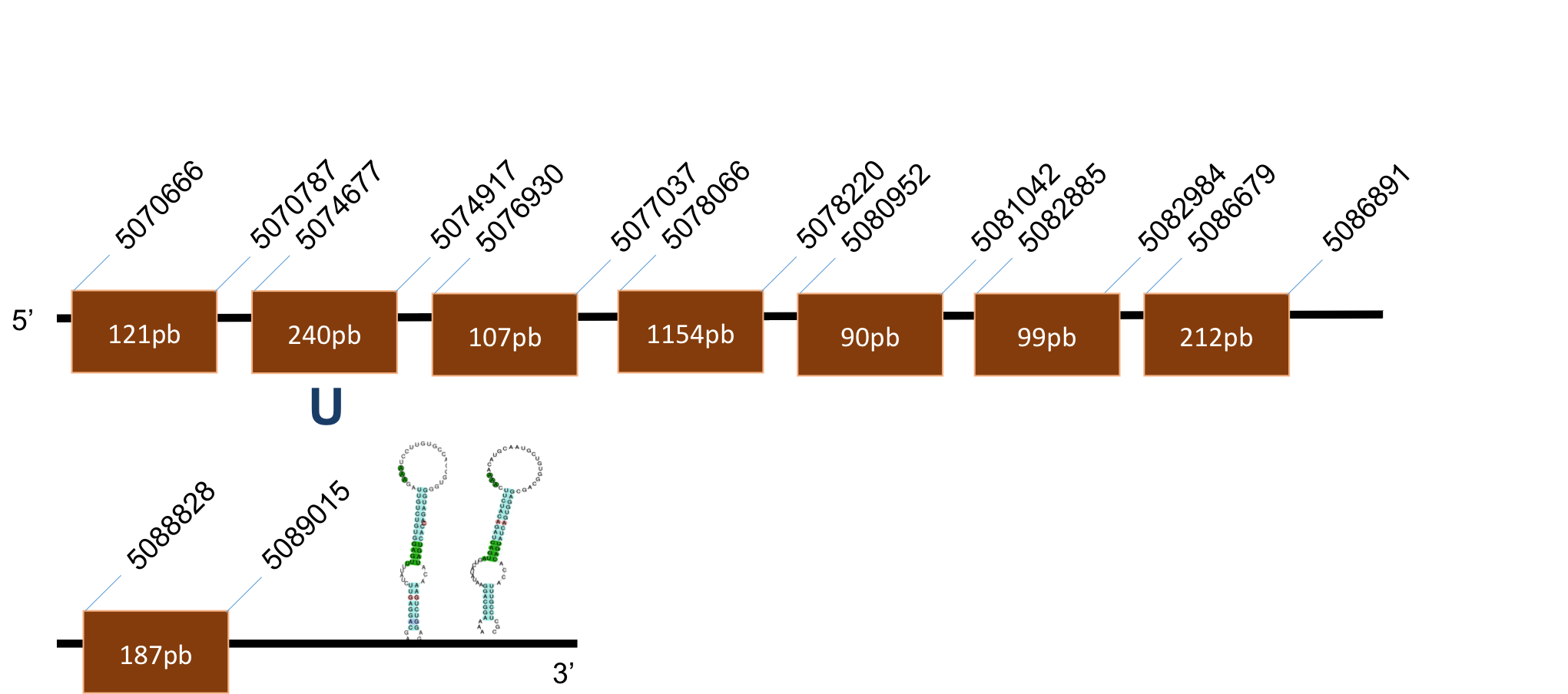
SPS2 B
La proteïna SPS2B es troba localitzada al scaffold KQ012996,1 del genoma de P.coquereli. Trobem un hit (identitat al voltant del 75% i E-value 10-4) a l'output del BLAST. Trobem una Sec que coincideix a la query i a la target i a més una Sec que només es troba a la proteïna de P.coquereli.
L’alineament presenta un score de 990, per a una proteïna de 2 exons. L’element SECIS és de grau A i es posiciona a les coordenades 26790-26865 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 1703 pb.
SPS2 C
La proteïna SPS2C es troba localitzada al scaffold KQ021625,1 del genoma de P.coquereli. Trobem un hit destacat (identitat al voltant de 60% i E-values de 10-30 i 10-116) a l'output del BLAST. Hi ha una Sec que coincideix, en canvi, hi ha una altra on tant la query com la target la tenen però no coincideixen en la posició.
L’alineament presenta un score de 961, per a una proteïna de 2 exons. L’element SECIS és de grau B i es posiciona a les coordenades 26779-26855 de la target.
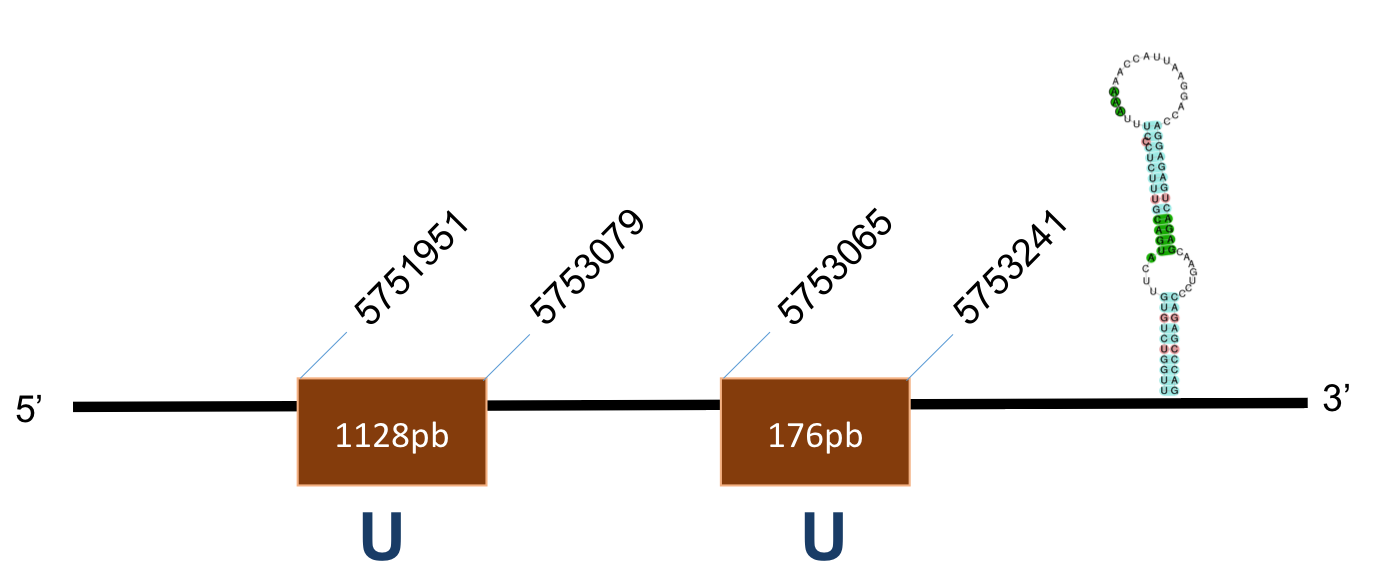
SPS2 D
La proteïna SPS2D es troba localitzada al scaffold KQ026623,1 del genoma de P.coquereli. Trobem un hit (identitat 84% i E-value 0.0) a l'output del BLAST.
L’alineament presenta un score de 1000, per a una proteïna d'1 exó. L’element SECIS és de grau A i es posiciona a les coordenades 24457-24534 de la target. La distància entre la selenocisteïna –UGA i l’element SECIS obtinguts per Seblastian és de 1734 pb.
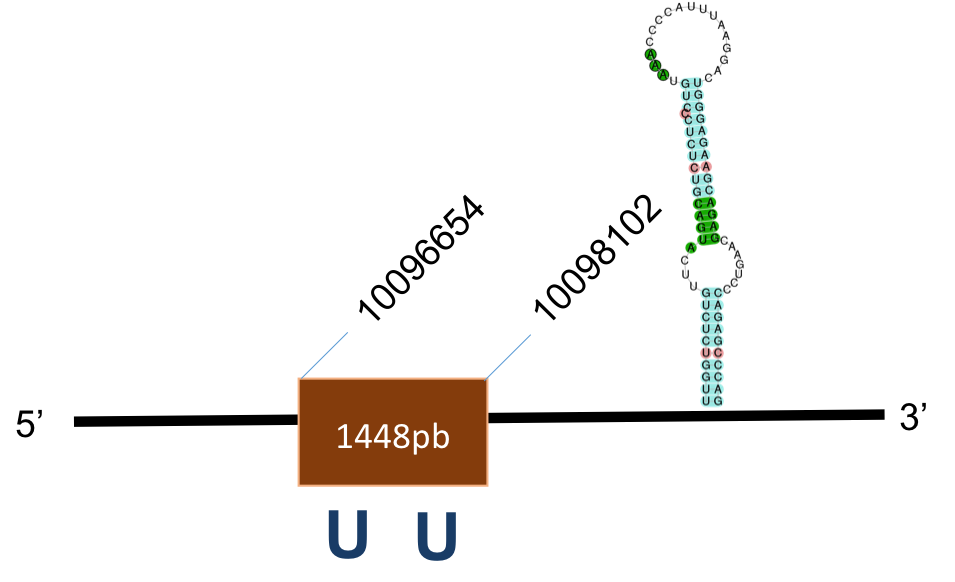
TR's
A la família de les TR’s no vam poder executar l’EXONERATE ni el TCOFFEE i, per tant, no vam poder realizar l’alineament. Creiem que això pot ser degut a que la seqüència proteica tenia una longitud major a la del scaffold (aquest mostra regions no seqüenciades de manera correcta).
