DISCUSIÓN
Los criterios que seguimos para analizar las distintas proteínas son éstos;
En general, siempre que hemos podido, hemos intentado usar como query problema las proteínas de Pteropus vampyrus, ya que es una especie de megaquiróptero próxima a Eidolon helvum que tiene las selenoproteínas anotadas e identificadas. Cuando eso no ha sido posible (ya sea porque o bien las selenoproteínas a estudiar en Pteropus vampyrus no estaban bien anotadas o porque no se encontraban presentes), hemos usado las proteínas de Homo sapiens como modelo para la obtención de querys problema.
Para seleccionar qué scaffold de todos los obtenidos en el blast era más probable que codificase para una determinada proteína, tuvimos en cuenta diferentes aspectos. Para empezar, sólo consideramos como válidos aquellos scaffolds que fueran significativos (que tuvieran un e value menor a 10-4). Además, seleccionamos aquellos que presentaran una gran homología con la query problema en el programa t coffee (elevado porcentaje de identidad y similitud de secuencias), y que cubrieran una gran parte de la query problema. Por último, elegimos los scaffolds que contuvieran la selenocisteína (U), en caso de que ésta estuviera en la query problema inicial (ya fuese de Homo sapiens o de Pteropus vampyrus). Todas estas condiciones nos permitieron, en la mayoría de los casos, quedarnos con un único scaffold.
Respecto a los elementos SECIS, comentar que solo contamos como válidos aquellos predichos en el mismo strand que el scaffold en cuestión y a una distancia de entre 0 y 6000 nucleótidos del último nucleótido predicho por el exonerate/genewise.
Selenoproteínas en Eidolon helvum
Iodotironina deiodinasas 1, 2 i 3
DI1:
Para estudiar si la proteína DI1 se encuentra presente o no en Eidolon helvum, usamos como query problema la DI1 de Pteropus vampyrus, ya que ésta se encuentra bien anotada.
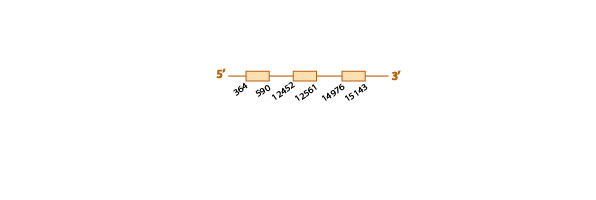
Al hacer el blast, obtuvimos distintos scaffolds significativos (con un e value menor a 10-4). Seleccionamos KE815153.1, ya que era el que tenía un valor de e value menor (2e-63) y además cubría completamente la query problema desde el primero hasta el último aminoácido, cubriendo en consecuencia también la selenocisteína (indicándonos ya una prueba de la conservación de esta proteína en Eidolon helvum).
En el resultado del tcoffee se observaba mucha homología de secuencias, y en el análisis exonerate advertimos la presencia de un codón UGA (codón stop que en las selenoproteínas codifica una selenocisteína) en nuestro scaffold alineado con un codón Unk (desconocido) en la query problema, indicando la presencia de una selenocisteína en el scaffold seleccionado. Todos estos resultados, se observan en esta tabla .
Finalmente, con SECISearch3 predijimos un elemento SECIS de grado A en el strand correspondiente (el reverso), y a una distancia de 1000 nucleótidos a 3' del último nucleótido codificante predicho en el scaffold seleccionado. Además Seblastian predijo homología entre nuestro scaffold y la DI1 de Ovis aries.
Con todos los resultados obtenidos (elevada homología del scaffold con la query problema, conservación de la seleocisteína, predicción de un elemento SECIS y homología de nuestro scaffold con la DI1 de otra especie), podemos concluir que DI1 se encuentra conservada en el genoma de Eidolon helvum.
Ésta es al predicción de la estructura de esta proteína obtenida según nuestros resultados:
DI2:
Para estudiar si DI2 se encuentra o no conservada en Eidolon helvum, usamos como query problema la DI2 de Pteropus vampyrus, que estaba bien anotada.
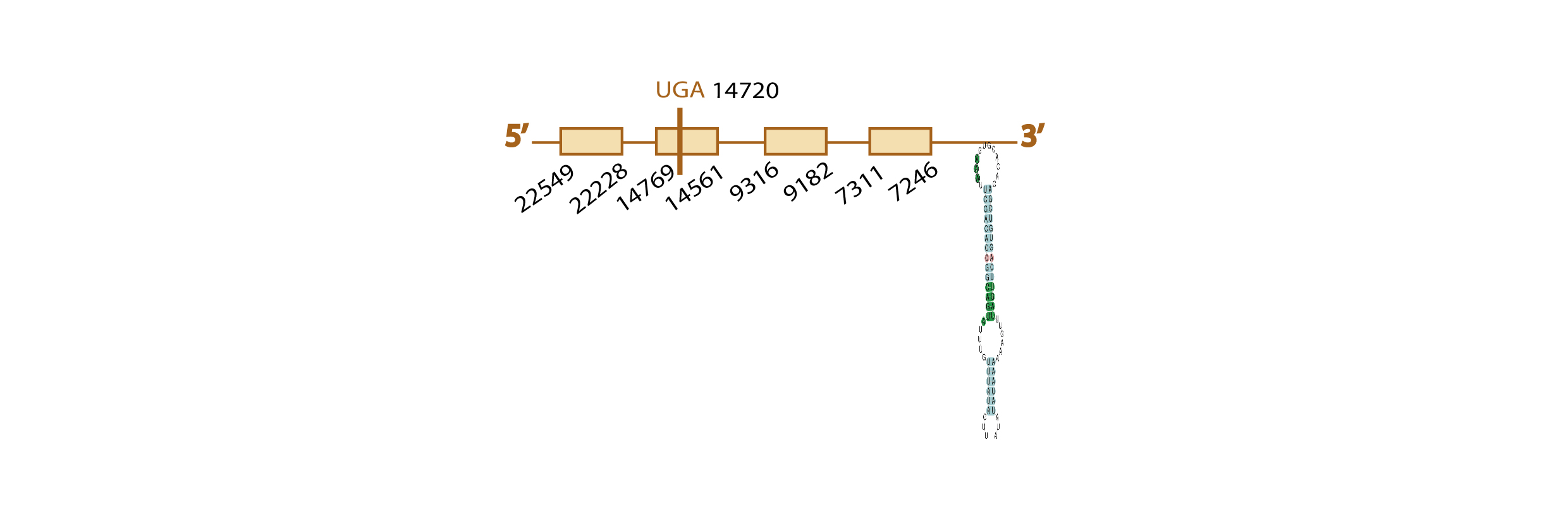
Al hacer el blast, obtuvimos distintos scaffolds significativos, pero seleccionamos KE817286.1, ya que era el hit que cubría casi completamente toda la query problema (excepto los 70 aminoácidos iniciales de los 264 aminoácidos totales) con una elevada homología de secuencias observada en el tcoffee, contenía la selenocisteína y además poseía un valor de e value muy bajo (e-122).
Los resultados predichos con exonerate y genewise fueron bastante similares (ver esta tabla), y además en el análisis exonerate, no sólo se observaba mucha homología de secuencias como ya se había visto en tcoffee, sino que también se presenciaba una selenocisteína (codón Unk de la query alineado con un codón UGA del scaffold seleccionado).
No fue posible predecir la presencia o ausencia de elementos SECIS con SECISearch3, ni la posible homología de nuestro scaffold con la DI2 de alguna otra especie con Seblastian, ya que el gen que codifica para la DI2 se encontraba casi al final del scaffold seleccionado, y por lo tanto el scaffold no englobaba la región donde podríamos encontrar el elemento SECIS.
Con todos los resultados obtenidos comentados, aunque no podamos saber si hay o no hay elementos SECIS, ni podamos predecir homología con la DI2 de otra especie con Seblastian, es muy probable que la selenoproteína DI2 se encuentre conservada en el genoma de Eidolon helvum.
Ésta es la predicción de la estructura de esta proteína en Eidolon helvum en base a los resultados obtenidos:
DI3:
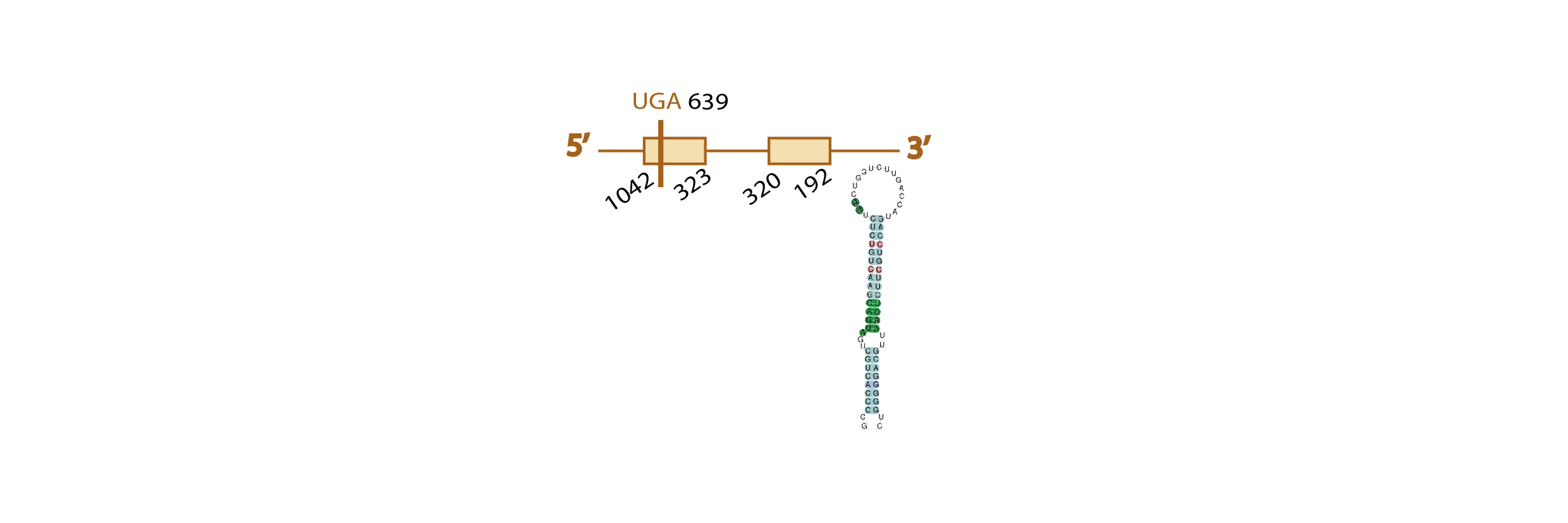
Para saber si esta proteína se encuentra o no conservada en Eidolon helvum, usamos como query problema la secuencia de DI3 de Homo sapiens, ya que la de Pteropus vampyrus no está bien anotada.
Al hacer el blast, obtuvimos distintos scaffolds significativos, pero escogimos AWHC01168030.1, ya que no sólo era el que tenía un valor de e value menor (e-150), sino que también cubría una mayor proporción de la query problema (desde el aminoácido 39 hasta último de la query, el 278, conteniendo la selenocisteína) con elevada homología (observada en el programa tcoffee).
Las predicciones del genewise y del exonerate de este scaffold fueron bastante similares, y en el análisis exonerate se observó la presencia de una posible selenocisteína (codón UGA del genoma de Eidolon helvum alineado con codón Unk de la query problema) (ver esta tabla).
Finalmente, con SECISearch3 predijimos un elemento SECIS de grado A en el strand correspondiente (el forward) y a una distancia adecuada, y el programa Seblastian predijo homología de nuestro scaffold con la DI3 de Macaca mulatta.
Con los resultados observados y comentados (elevada homología del scaffold seleccionado con la query problema, conservación de la selenocisteína y predicción de un elemento SECIS y de homología con la DI3 de otra especie), podemos concluir que la proteína DI3 se encuentra conservada en Eidolon helvum, y ésta es la predicción de su estructura en base a nuestros resultados:
Al analizar las distintas GPX, vimos que éstas eran muy similares las unas a las otras, e incluso, al hacer el comando blast, había muchos scaffolds predichos que daban hits significativos para varias GPX. Sabiendo que al tratarse de proteínas distintas no podían encontrarse codificadas en la misma región del genoma (es decir, en el mismo scaffold), para seleccionar qué scaffold era más probable que codificase para una determinada GPX seguimos los criterios mencionados en la introducción.
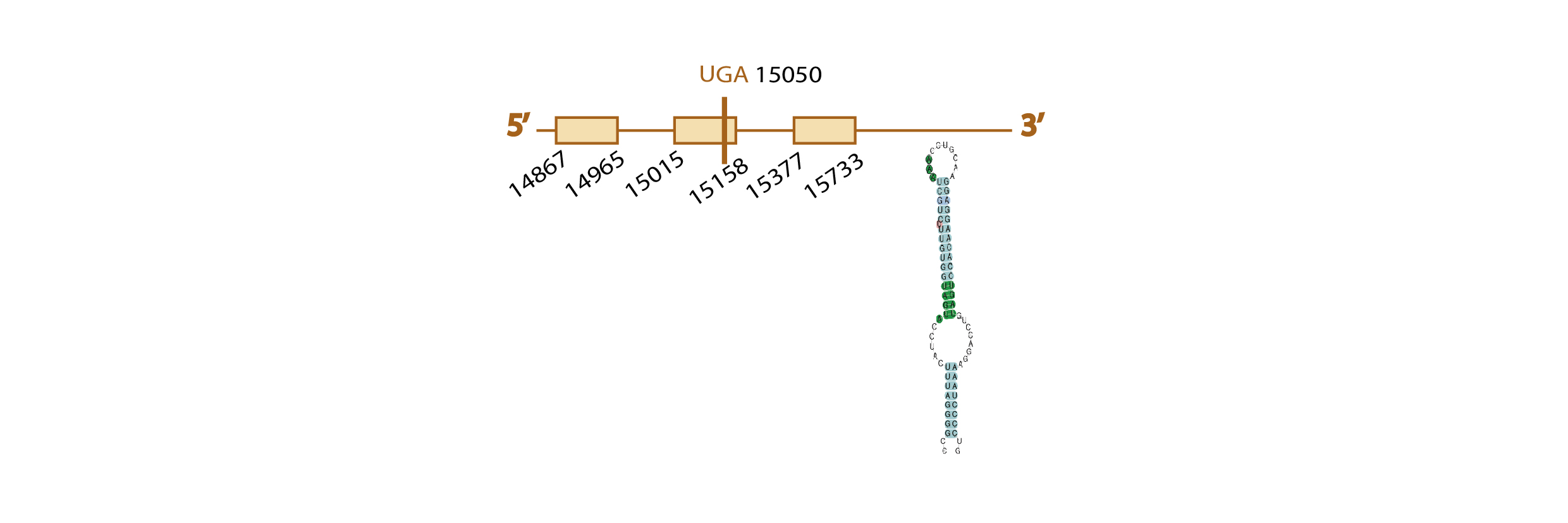
GPX1:
Para estudiar si la GPX1 se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína GPX1 de Homo sapiens, ya que la GPX1 de Pteropus vampyrus no se encontraba bien anotada (su secuencia no empezaba por el aminoácido metionina). En la query problema de Homo sapiens, observamos una U, indicando la posición de la selenocisteína.
Al hacer el comando blast, obtuvimos distintos hits (scaffolds) significativos. Descartamos aquellos que no cubrían la selenocisteína de la query problema, los de baja identidad y aquellos que cubrían sólo parcialmente la query problema de Homo sapiens, quedando como posibles scaffolds para GPX1 los siguientes: KE813470.1, KE751547.1, KE814389.1. Finalmente seleccionamos el scaffold KE751547.1 (e value 3e-68) ya que era el que presentaba una mayor homología con la query problema al hacer el análisis con tcoffee.
Al analizar este scaffold con el comando exonerate y con el genewise, observamos una predicción muy similar con los dos programas (observar la tabla) y también vimos que el scaffold contenía 3 exones (ver esquema de abajo).
Además, en el exonerate observamos en nuestro scaffold un codón stop, el codón UGA (que es el que codifica para la selenocisteína), alineado con un codón "Unknown" (éste programa no distingue el aminoácido U de la query problema, y lo marca como desconocido) en nuestra query problema, lo que es indicativo de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, predecimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (el forward) y a una distancia del último nucleótido del último exon predicho adecuada. Además, el programa Seblastian, encontró homología del scaffold seleccionado con la GPX1 de Canis lupus familiaris.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína, la predicción del elemento SECIS y la homología del scaffold seleccionado con la GPX1 de otra especie), son indicativos de la conservación de la selenoproteína GPX1 en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
GPX2:
Para estudiar si la GPX2 se encuentra conservada o no en Eidolon helvum, usamos como query problema la GPX2 de Pteropus vampyrus, ya que ésta se encontraba bien anotada (de hecho, es la única GPX de esta especie de Megaquirópteros que se encuentra bien anotada y que por lo tanto hemos podido usar en nuestro estudio).
Al hacer el comando blast, obtuvimos distintos hits significativos. Seleccionamos el scaffold (hit) KE814389.1, ya que presentaba el e value más significativo (2e-73) y cubría con mayor homología (elevada identidad, 97%) en el tcoffee una mayor proporción de la query problema (de hecho este scaffold presenta dos fragmentos que cubren completamente la query problema, desde el primero hasta el último aminoácido, el 190). Lógicamente, al cubrir toda la query, este scaffold también cubría la posible selenocisteína observada en la query problema. También predijimos que este scaffold conteía dos exones (mirar esquema de abajo).
En el análisis tcoffee observamos una elevada (casi perfecta) homología entre secuencias que cubre toda la longitud de la query problema. En el exonerate encontramos que el codón desconocido (Unk, indicativo de un codón stop) se alineaba en el scaffold seleccionado con un codón UGA, el que codifica para la selenocisteína, confirmando por lo tanto la presencia de una selenocisteína en este scaffold.
Finalmente, a través del programa SECISearch3, predijimos un elemento SECIS en el strand correspondiente (el forward) de grado A, que se localizaba en el genoma en una posición razonable, a unos 200 nucleótidos del último nucleótido del último exón de nuestro scaffold. El programa Seblastian, identificó homología de nuestro scaffold con la GPX2 de jabalí (Sus scrofa).
Con todos los resultados comentados anteriormente, concluimos que la selenoproteína GPX2 se encuentra conservada en Eidolon helvum.
Aquí mostramos la estructura predicha de esta selenoproteína según los resultados obtenidos:
GPX3:
Para estudiar si la GPX3 se encuentra o no conservada en Eidolon helvum, usamos como query problema la GPX3 de Homo sapiens, ya que la de Pteropus vampyrus se encontraba mal anotada.
En el blast obtuvimos distintos hits significativos, y seleccionamos tres que presentaban un valor de e value significativo y que además cubrían una mayor proporción de la query problema (incluyendo la selenocisteína) que el resto: KE810295.1, KE751547.1 i KE814389.1. Finalmente, seleccionamos el scaffold KE810295.1 (e value 4e-33) ya que cubría una mayor proporción de la query problema (197 aminoácidos de los 226 totales) con una mayor identidad (más del 70% en los cuatro fragmentos en los que se subdivide), es decir, era el scaffold que presentaba un mejor resultado (mayor homología con la query problema) en el tcoffee. Predijimos que este gen contenía 5 exones, descritos en el esquema que presentamos más abajo.
Los resultados obtenidos con el exonerate y el genewise fueron bastante similares (ver tabla), además, en el análisis exonerate observamos el alineamiento de un codón stop (UGA) del scaffold seleccionado con un codón desconocido en la query problema, que como ya hemos visto es indicativo de que en esa posición del scaffold es donde se encuentra la selenocisteína de esta proteína.
No pudimos predecir ningún elemento SECIS real con el programa SECISearch3. El único elemento SECIS predicho en este programa era de bajo grado (grado C) y se encontraba en el strand equivocado (forward en vez de reverse), por lo que no podemos afirmar que exista un elemento SECIS en esta proteína en Eidolon helvum. Seblastian tampoco predijo ninguna homología del scaffold seleccionado con alguna GPX3 de otra especie.
Con los distintos resultados obtenidos, aunque no hayamos predicho ningún elemento SECIS ni ningún resultado con el Seblastian, la elevada homología observada entre la query problema y el scaffold seleccionado y la conservación de la selenocisteína, nos indican la conservación de ésta selenoproteína en Eidolon helvum. Podría ser que el scaffold seleccionado no englobase la región donde se encuentra el posible elemento SECIS y por este motivo no lo hayamos encontrado.
Con los resultados obtenidos, elaboramos el esquema de la predicción de la estructura de GPX3 en el genoma de Eidolon helvum:
GPX4:
Para estudiar si la GPX4 se encontraba o no conservada en Eidolon helvum, usamos como query problema la GPX4 de Homo sapiens, ya que la de Pteropus vampyrus se encontraba mal anotada.
En el blast obtuvimos distintos hits significativos. De entre todos aquellos que presentaban un valor de e value menor y que además cubrían una mayor parte de la query problema, seleccionamos el scaffold AWHC01282061.1 (e value 4e-31), ya que éste era el scaffold que presentaba una mayor homología con la query problema en el análisis con tcoffee. También era el scaffold que cubría una mayor proporción de la query problema (160 de 197 aminoácidos totales) con elevada identidad y además cubría la región de la query donde se encuentra la selenocisteína.
Los resultados que obtuvimos con el exonerate y el genewise eran bastante similares (ver tabla), y además en la preedicción hecha con el exonerate se observaba el alineamiento del codón stop UGA del scaffold seleccionado con el codón desconocido (Unk) en la query problema, indicativo de la presencia de la selenocisteína en este scaffold.
Finalmente, con el SECISearch3 predijimos un elemento SECIS de grado A en el strand correspondiente (el reverse), a una distancia adecuada, y el Seblastian predijo homología de nuestro scaffold con la GPX4 de Cyprinus carpio.
Con todos los resultados obtenidos (elevada homología e identidad del scaffold con la query problema, conservación de la selenocisteína y predicción de un elemento SECIS y de homología con la GPX4 de otra especie), podemos concluir que la selenoproteína GPX4 se encuentra conservada en Eidolon helvum.
Aquí mostramos un esquema de la predicción de la estructura de esta proteína en el genoma de Eidolon helvum:
GPX5:
Para estudiar si la GPX5 se encuentra o no conservada en Eidolon helvum, usamos la query de la GPX5 de Homo sapiens como query problema, ya que la de Pteropus vampyrus se encontraba mal anotada. No observamos ninguna selenocisteína en la query problema, ya que la GPX5 humana es un homólogo de cisteína.
Al hacer el blast obtuvimos muchos hits (scaffolds) con un valor de e value significativo. Seleccionamos inicialmente aquellos scaffolds con un valor de e value menor que cubrían una mayor parte de la query problema, quedándonos con KE755429.1, KE751547.1, KE813470.1, KE814389.1 y KE810295.1. Para seleccionar de entre estos posibles uno sólo, analizamos los distintos scaffolds con el tcoffee. El scaffold KE810295.1 lo descartamos, ya que ya lo habíamos seleccionado para la predicción de la GPX3, porque presentaba una homología mucho mayor para la GPX3 que para la GPX5 (además los análisis subsiguientes que hicimos con este scaffold y la query de GPX3 humana nos confirmaron que KE810295.1 contiene la región codificante de GPX3, tal como vimos anteriormente).
Finalmente seleccionames el scaffold KE755429.1 (e value 1e-33), ya que cubría una gran parte de la query problema (146 aminoácidos de los 221 totales), y era el scaffold que presentaba una mayor homología e identidad con dicha query en el análisis con tcoffee.
La homología y las predicciones realizadas con el exonerate y el genewise con este scaffold nos dieron resultados muy similares (ver tabla). Con el tcoffee observamos que la homología entre este scaffold y la query problema era muy elevada, aunque la parte inicial de la query no estaba alineada con ninguna región del scaffold, ya que éste delimita una región de nuestro genoma que no se alinea con nuestra query hasta su aminoácido 72 (cosa que ya vimos en el blast). En el programa genewise tampoco detectamos ninguna posible selenocisteína en el scaffold, por lo que deducimos que GPX5 en Eidolon helvum es también un homólogo de cisteína.
No predijimos ningún elemento SECIS con SECISearch3. El análisis con Seblastian tampoco predijo ninguna selenoproteína.
Con todos los resultados obtenidos, deducimos que GPX5, debido a la elevada homología del scaffold seleccionado con la query problema, se encuentra conservada en nuestra especie. La ausencia de predicción de ningún elemento SECIS se debe a la ausencia de selenocisteína en nuestro scaffold. Hipotetizamos que GPX5 inicialmente era una selenoproteína, que a lo largo de la evolución ha perdió la selenocisteína, pasando a ser una cisteína.
Basándonos en los resultados obtenidos, ésta es la predicción que hacemos de la posible estructura de GPX5 en Eidolon helvum:
GPX6:
Para saber si la GPX6 se encuentra o no conservada en Eidolon helvum, usamos como query problema la GPX6 de Homo sapiens, no porque la GPX6 de Pteropus vampyrus estuviera mal anotada como en otros casos, sino porque la GPX6 no se encuentra en este megaquiróptero.
Al hacer el análisis blast, obtuvimos distintos hits significativos. Sin embargo, todos los hits que obtuvimos pertenecían a scaffolds que no sólo presentaban una baja homología con la query problema en el tcoffee, sino que además eran scaffolds que pertenecían a otras GPXs.
Con estos resultados, y considerando que la GPX6 tampoco se encuentra presente en la especie próxima a Eidolon helvum que usamos como modelo (Pteropus vampyrus), hipotetizamos que la GPX6 no se encuentra presente en el genoma de Eidolon helvum, y que es probable que la pérdida de GPX6 tuviera lugar en un ancestro común de ambos megaquirópteros.
GPX7:
Para saber si esta proteína se encuentra o no conservada en Eidolon helvum, usamos como query problema la GPX7 de Homo sapiens, ya que la de Pteropus vampyrus se encontraba mal anotada. La GPX7 en Homo sapiens no continene ninguna selenocisteína, ya que es un homólogo de cisteína.
Al hacer el blast, obtuvimos distintos hits significativos. Procedimos a analizar con tcoffee los distintos scaffolds significativos, y nos dimos cuenta que había dos scaffolds de elevada identidad y homología con la query problema que eran contiguos. El scaffold KE813456.1 (e value 6e-51) era el que cubría con elevada homología (identidad >90%) la mayor parte de la query problema (cubría 139 aminoácidos de los 187 totales, desde el aminoácido 47 hasta el 186 de la query problema). Los primeros 48 aminoácidos estaban cubiertos en parte por el scaffold KE781696.1.
Para procedir con los siguientes análisis (exonerate, genewise, SECISearch3, Seblatian...), usamos el scaffold KE813456.1, ya que era el que cubría la mayor parte de la query problema, y para hacer estos análisis no se pueden seleccionar dos scaffolds a la vez.
Los resultados predichos con el genewise y con el exonerate fueron bastante similares (ver tabla).
Con el genewise, no observamos ninguna selenocisteína en nuestro scaffold, asumimos entonces que GPX7 en E. helvum es un homólogo de cisteína.
Finalmente, no predijimos ningún elemento SECIS con SECISearch3, ni ninguna selenoproteína homóloga a nuestro scaffold con Seblastian.
Con los resultados obtenidos, aunque no hayamos encontrado ningún elemento SECIS (la cual cosa es lógica, ya que GPX7 no presenta ninguna selenocisteína), debido a la elevada homología que presenta el scaffold seleccionado con la query problema, deducimos que la GPX7 se encuentra consevada en nuestra especie. Hipotetizamos que la GPX7 inicialmente era una selenoproteína, que a lo largo de la evolución ha perdido la selenocisteína, pasando a ser una cisteína.
Teniendo en cuenta los resutados obtenidos, la estructura de GPX7 es ésta:
GPX8:
Para estudiar si la GPX8 se encuentra o no conservada en Eidolon helvum, usamos la query de Homo sapiens como query problema, ya que la de Pteropus vampyrus se encontraba mal anotada. No observamos ninguna selenocisteína en la query problema, ya que GPX8 humana es un homólogo de cisteína.
Una vez hecho el blast, observamos 4 posibles scaffolds, todos ellos seleccionados por el programa por tener un e-value menor a 10-4.
Entre estos 4 elegimos el de menor e-value, que a su vez cubría toda nuestra query problema y tenía un muy buen alineamiento en el tcoffe con un score de 100. Éste es el scaffold: KE808138.1.
Tanto el genewise como el exonerate nos predijeron la misma proteína, con 3 exones.
Ningún elemento SECIS fue predicho, ya que la proteína no contenía ninguna selenocisteína, y tampoco predijimos ningún resultado con el Seblastian.
La alta homología de nuestra query problema con el genoma de Eidolon helvum confirma que esta proteína se encuentra conservada en esta especie.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
En el genoma de Homo sapiens encontramos 3 proteínas dentro de esta familia: TR1,TR2 y TR3. Para estudiar si las encontramos o no en Eidolon helvum utilizaremos las querys de Homo sapiens, ya que las de Pteropus vampyrus se encuentran mal anotadas.
TR1:
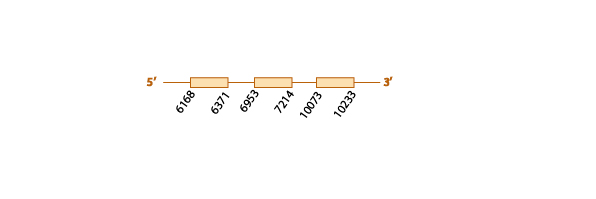
Para estudiar si esta proteína se encuentra conservada en Eidolon helvum realizamos un blast y obtuvimos 4 scaffolds posibles. De estos, solo encontramos la selenocisteína en uno de ellos, pero este scaffold pertenecía a la proteína TR2 (porque presentaba mayor homología en el tcoffee que TR1). Escogimos entonces el scaffold KE766852.1 con un valor de e-value de 1e-37 y con una elevada identidad (96%). En el tcoffe obtuvimos un score de 99 aunque el alineamiento obtenido no nos cubría la parte inicial ni la final de la proteína, donde se encontraba la selenocisteína. Por tanto, no sabemos si ésta se encuentra conservada o no en Eidolon helvum.
Tanto el genewise como el exonerate predijeron la misma proteína con 11 exones.
Ningún elemento SECIS fue predicho. Podríamos hipotetizar que la proteína se encuentra conservada, pero al no haberse conservado la selenocisteína es posible que la TR1 en Eidolon helvum no sea funcional.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
TR2:
Para estudiar si esta proteína se encuentra conservada en Eidolon helvum realizamos un blast y obtuvimos 6 scaffolds posibles. De estos 6 scaffolds, seleccionamos KE791502.1 , con un valor de e-value de 2e-14, porque contenía la parte final de la proteína con la selenocisteína.
En el tcoffe obtuvimos un score de 99 y, lógicamente, el alineamiento, que era de elevada homología, solo se realizaba con la parte final de la proteína.
En el exonerate observamos un codón desconocido ("Unk") de la query alineado con un codón UGA indicando la conservación de la selenocisteina.
Tanto el genewise como el exonerate predijeron la misma proteína (ver tabla), de 3 exones.
Con SECISearch3 predijimos un elemento SECIS de grado A en una posición adecuada. Seblastian predijo homología de secuencias de nuestro scaffold con la TR2 de Homo sapiens, dando robustez a nuestra hipótesis de conservacion de esta selenoproteína en Eidolon helvum.
Con todos los resultados obtenidos (elevada homología de secuencias, conservación de la selenocisteína, y predicción de un elemento SECIS y de homología con TR2 de otra especie), podemos hipotetizar que la TR2 se encuentra conservada en Eidolon helvum. Sin embargo, cabe destacar que el scaffold seleccionado solo cubría una región muy pequeña de la query problema. Podría ser que la fracción no cubierta de la query problema se encontrase en otro scaffold.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
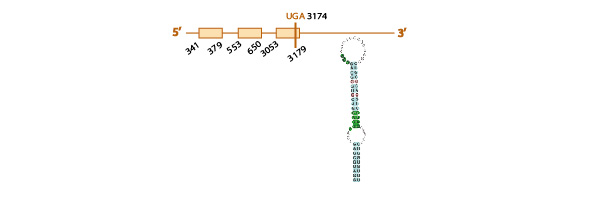
TR3:
Para estudiar si esta proteína se encuentra conservada en Eidolon helvum realizamos un blast y obtuvimos 4 scaffolds posibles. Escogimos el scaffold KE790577.1, porque era el que presentaba un mejor valor de e-value (4e-33), cubría una mayor parte de la proteína y contenía la selenocisteína.
El tcoffe obtenido tenía un score de 99 y presentaba una elevada homología. En el exonerate observamos que la selenocisteína de la query (codón "Unk") se alineaba con un codón UGA en nuestro scaffold, indicando conservación de la selenocisteína.
Tanto el genewise como el exonerate predijeron una proteína de 10 exones.
Predijimos un elemento SECIS en el strand correcto (el reverse) y a una distancia adecuada del último nucleótido codificante, y el programa Seblastian predijo homología de nuestro scaffold con TR3 de Homo sapiens, confiriendo robustez a nuestra hipótesis de coservación de esta selenoproteína en Eidolon helvum.
Con todos los datos obtenidos (elevada homología con la query, conservación de la selenocisteína, y predicción de un elemento SECIS y de homología con una TR3 de otra especie), concluimos que TR3 se encuentra conservada en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
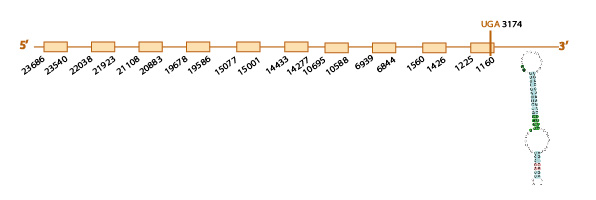
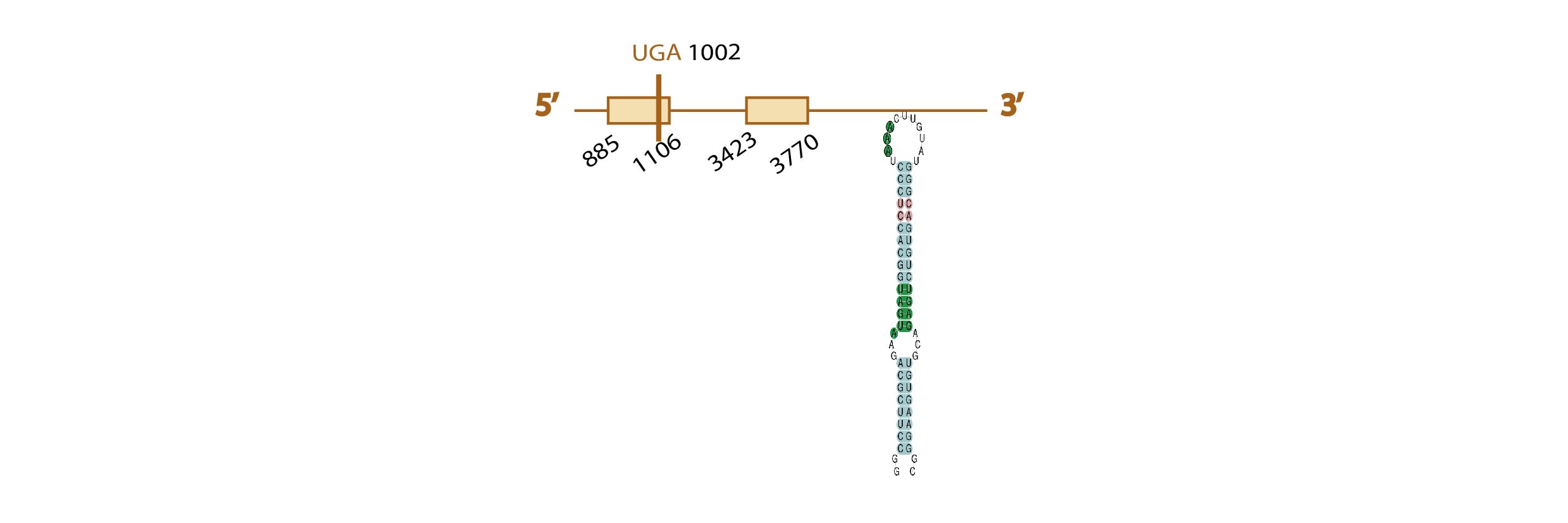
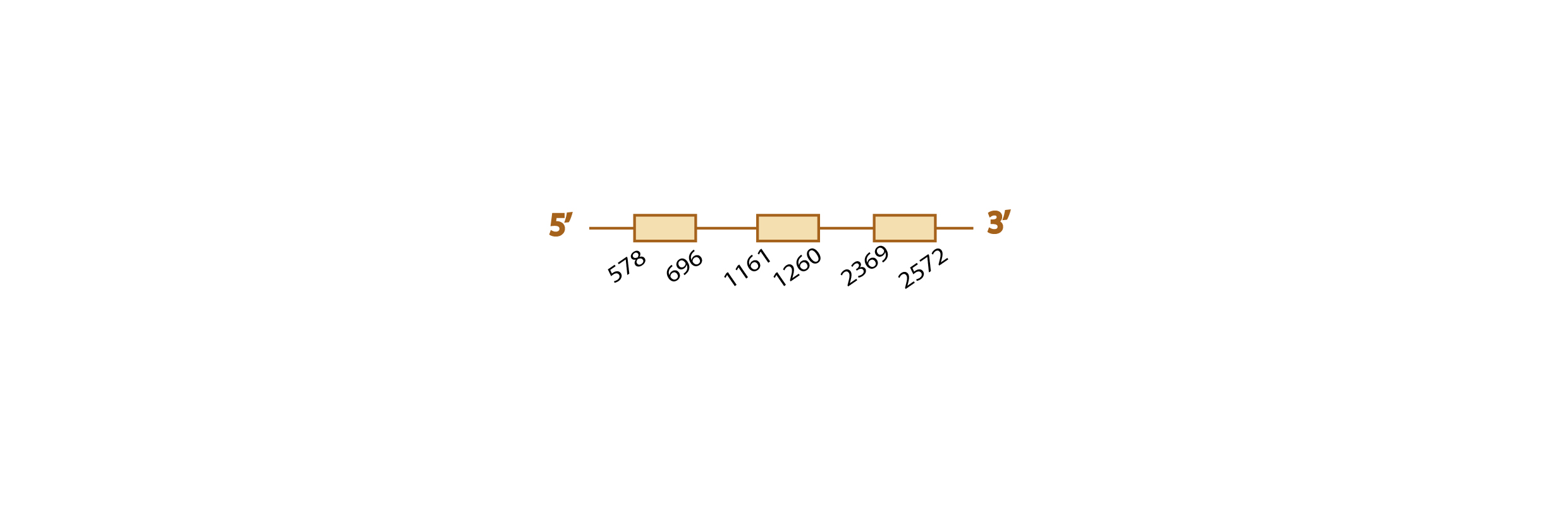
Para estudiar si la proteína Sel15 se encuentra presente o no en Eidolon helvum, usamos como query problema la Sel15 de Pteropus vampyrus, ya que ésta se encuentra bien anotada.
Al hacer el blast, obtuvimos distintos scaffolds significativos (con un e value menor a 10-4). Seleccionamos KE778117.1 (e value 5e-19) ya que era el scaffold que cubría la query problema en mayor proporción, incluyendo la posible selenocisteína.
En el resultado del tcoffee se observaba mucha homología de secuencias, aunque la parte inicial de la query no se encontraba presente en el scaffold. En el an´lisis exonerate, advertimos la presencia de un codón UGA (codón stop que en las selenoproteínas codifica una selenocisteína) en nuestro scaffold alineado con un codón "Unk" (desconocido) en la query problema, indicando la presencia de una selenocisteína en el scaffold seleccionado. Todos estos resultados, se observan en esta tabla.
Finalmente, con SECISearch3 predijimos un elemento SECIS de grado A en el strand correspondiente (el forward) a una distancia adecuada, y con Seblastian predijimos homología entre nuestro scaffold y la Sel15 de Oryctolagus cuniculus.
Con todos los resultados obtenidos (elevada homología del scaffold con la query problema, conservación de la selenocisteína, predicción de un elemento SECIS y homología de nuestro scaffold con la Sel15 de otra especie), podemos concluir que Sel15 se encuentra conservada en el genoma de Eidolon helvum. Ésta es al predicción de la estructura de esta proteína obtenida según nuestros resultados:
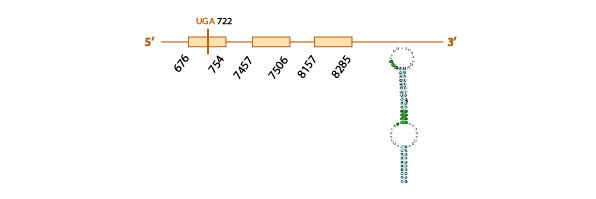
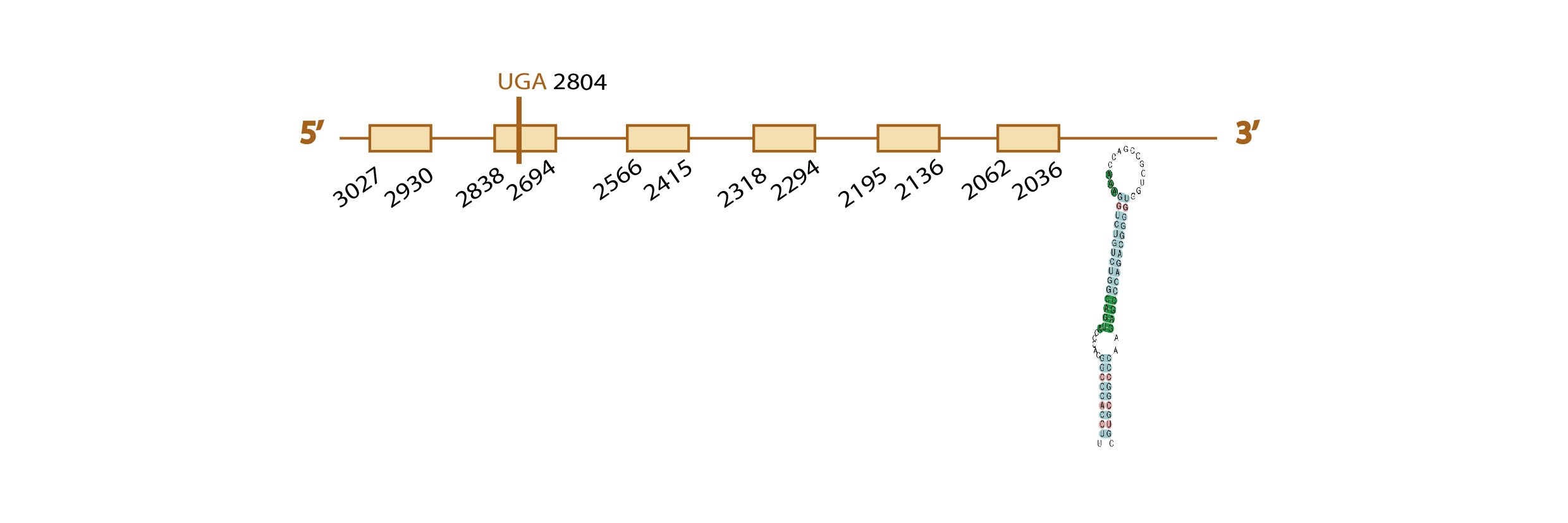
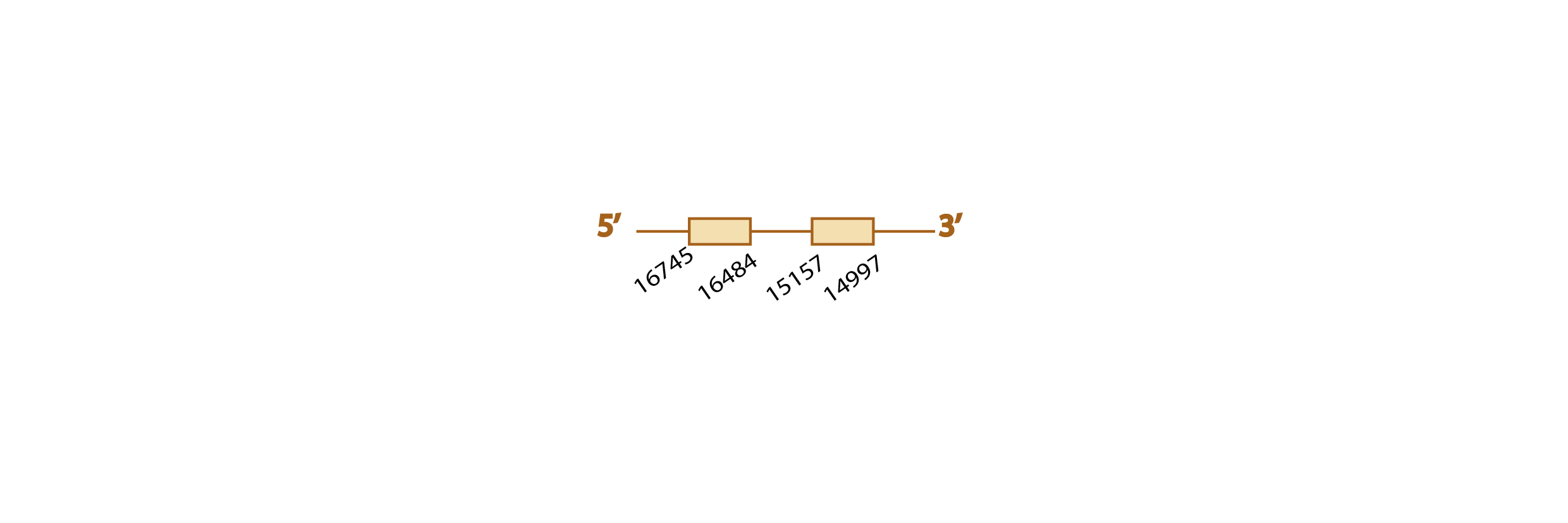
Para estudiar si la proteína SelH se encuentra presente o no en Eidolon helvum, usamos como query problema la SelH de Pteropus vampyrus, ya que ésta se encuentra bien anotada.
Al hacer el blast, obtuvimos distintos scaffolds significativos (con un e value menor a 10-4). Seleccionamos KE809834.1 (3e-37) ya que era el scaffold que cubría la query problema en mayor proporción, incluyendo la posible selenocisteína.
En el resultado del tcoffee se observaba mucha homología de secuencias, aunque la parte inicial de la query no se encontraba presente en el scaffold. En el análisis exonerate, advertimos la presencia de un codón UGA (codón stop que en las selenoproteínas codifica para una selenocisteína) en nuestro scaffold alineado con un codón "Unk" (desconocido) en la query problema, indicando la presencia de una selenocisteína en el scaffold seleccionado. Todos estos resultados, se observan en esta tabla.
No fue posible predecir la presencia de elementos SECIS con SECISearch3, ni la posible homología de nuestro scaffold con la SelH de alguna otra especie con Seblastian, ya que el gen que codifica para SelH se encontraba casi al final del scaffold seleccionado, y por lo tanto el scaffold no englobaba la región donde podríamos encontrar el elemento SECIS.
Con todos los resultados obtenidos, aunque no podamos saber si hay o no hay elementos SECIS, ni podamos predecir homología con la SelH de otra especie con Seblastian, es muy probable que la selenoproteína SelH se encuentre conservada en el genoma de Eidolon helvum.
Ésta es al predicción de la estructura de esta proteína obtenida según nuestros resultados:
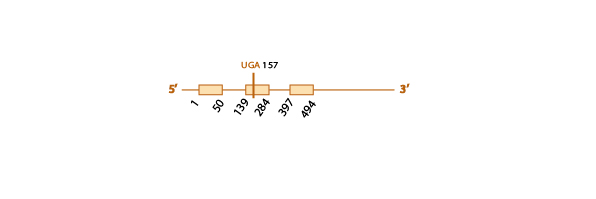
Para estudiar si la SelI se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelI de Homo sapiens, ya que a pesar de que la de Pteropus vampyrus estaba bien anotada, los resultados que obtuvimos con esta query en el programa tcoffee eran de muy mala calidad.Sin embargo, usando la query de Homo sapiens pudimos estudiar la homología de secuencias de esta proteína con la de Eidolon helvum.
En la query problema de Homo sapiens observamos una U, indicando la posición de la selenocisteína.
Al hacer el blastall, obtuvimos diferentes hits (scaffolds) significativos. Descartamos aquellos que no cubrían la selenocisteína de la query problema, los de baja identidad y aquellos que cubrían parcialmente la query problema. Así, nos quedamos con el scaffold AWHC01202482.1, que presentaba una e value de 2e-38, contenía la selenocisteína y cubría casi toda la query (solo le faltaba un fragmento de la query que va del aminoácido 1 al 18 de un total de 397 aminoácidos). Además, era el que presentaba mayor homología con la query problema al hacer el análisis con el t-coffee.
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene 9 exones (ver esquema más abajo).
Además, en el exonerate observamos en nuestro scaffold un codón stop UGA (que codifica para la selenocisteína), alineado con un codón "Unknown" en nuestra query problema, cosa que es indicativa de la presencia de una selenocisteína en nuestro scaffold. Recordar que el programa no distingue el aminoácido U y que por eso lo marca como desconocido.
Finalmente, con el programa SECISearch3, predijimos un elemento SECIS. Sin embargo, éste se encontraba en el strand contrario a nuestro scaffold por lo que no podemos considerarlo válido.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema y la conservación de la selenocisteína), nos indican que es muy probable que esta selenoproteína se encuentre conservada en Eidolon helvum, aunque la ausencia de elemento SECIS nos impide assegurarlo. Sin embargo, podría ser que sí que hubiese un elemento SECIS en esta proteína pero que no lo podamos detectar porque no esté incluído en el scaffold seleccionado.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
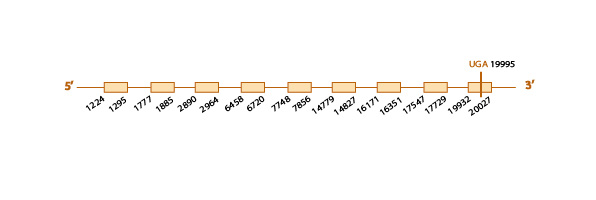
Para estudiar si la SelK se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelK de Homo sapiens, por la misma razón que ya vimos en SelI. En la query problema de Homo sapiens observamos una U, indicando la posición de la selenocisteína.
Al hacer el blastall, obtuvimos 7 hits (scaffolds) significativos. Para empezar, descartamos 4 de ellos pues no contenían la selenocisteína. De entre los 3 restantes elegimos el scaffold KE785780.1, ya que cubría toda la query a excepción del primer aminoácido, tenía un valor de e value muy bajo (8e-13), y conservaba la selenocisteína (encontramos el codón UGA, codificante de la selenocsiteína, alineado con un codón "Unknown" en el exonerate, indicando la presencia de una selenocisteína en el scaffold). Además, era el scaffold que presentaba mayor homología con la query problema cuando miramos el t-coffee, con un score de 99.
Al analizar este scaffold con el exonereate y con el genewise, observamos que hay una pequeña diferencia entre los inicios predichos por ambos programas, ya que el exonerate sitúa el inicio en 10742 mientras que el genewise lo sitúa en 13713 (ver tabla). Además, vimos que el scaffold contiene 4 exones (ver esquema más abajo).
Finalmente, con el programa SECISearch3, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (el forward) y a una distancia adecuada. A su vez, con el Seblastian vimos que el scaffold elegido para la SelK presentaba gran homología con la SelK de Chinchilla lanigera, confiriendo robusteza a nuestra hipótesis de que la SelK está conservada.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína, la predicción del elemento SECIS y la predicción de homología con la SelK de otra especie con Seblastian), son indicativos de la conservación de la SelK en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
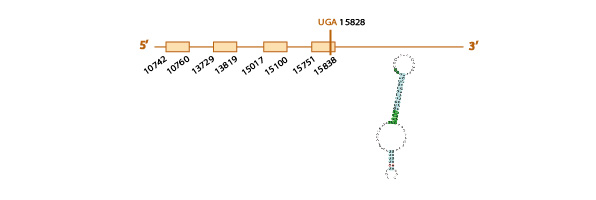
Para estudiar si la SelM se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelM de Pteropus vampyrus ya que estaba bien anotada. En la query problema de Pteropus vampyrus encontramos una U, indicando la posición de la selenocisteína.
Al hacer el blastall, obtuvimos un único hit (scaffold) significativo KE819481.1, que presentaba una e value de 2e-25, y cubría con una gran homología la query problema en el t-coffee, con un score de 98.
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene 5 exones (ver esquema más abajo).
Además, en el exonerate observamos en nuestro scaffold un codón stop UGA (que codifica para la selenocisteína), alineado con un codón "Unknown" en nuestra query problema, cosa que es indicativa de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, predijimos 2 elementos SECIS. Sin embargo, descartamos el segundo ya que se encontraba demasiado lejos (a más de 6000 nucleótidos del último nucleótido predicho por el exonerate/genewise). El elemento SECIS con el que nos quedamos es de grado A y se sitúa en el mismo strand de nuestro scaffold (el reverse) y a una distancia adecuada. A su vez, con el Seblastian vimos que el scaffold elegido para la SelM presentaba gran homología con la SelM precursora de Equus caballus, cosa que nos da robusteza a nuestra hipótesis de que la SelM está conservada.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína y la predicción del elemento SECIS y de una selenoproteína homóloga de otra especie), son indicativos de la conservación de la SelM en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
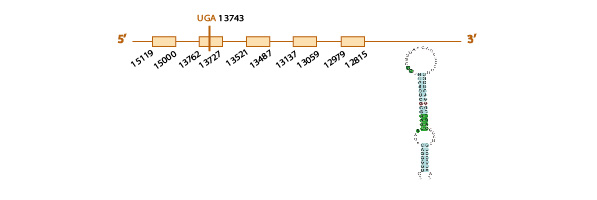
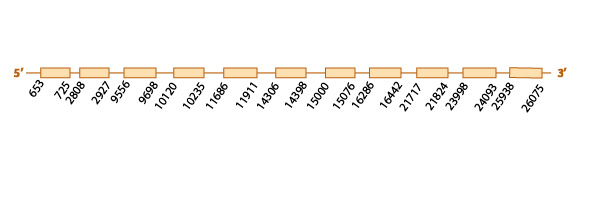
Para estudiar si la SelN se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelN de Homo sapiens, ya que la SelN de Pteropus vampyrus no se encontraba bien anotada. En la query problema de Homo sapiens encontramos 2 selenocisteínas. Sin embargo, con el blastall obtuvimos un único scaffold (KE799186.1) y éste sólo contenía una de las dos posibles selenocisteínas (la segunda) de la query. Aun así, la homología observada con la query problema es muy elevada en el t-coffee.
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene 11 exones (ver esquema más abajo).
Además, en el exonerate observamos en nuestro scaffold un codón stop UGA (que codifica para la selenocisteína), alineado con un codón "Unknown" en nuestra query problema, cosa que es indicativa de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, predijimos 4 elementos SECIS. Sin embargo, tuvimos que descartar 2 de ellos porque se encontraban en el strand contrario a nuestro scaffold, y un tercero porque se encontraba dentro de la región codificante. Así, nos quedamos con un elemento SECIS de grado A en el mismo strand de nuestro scaffold (el reverse) y a una distancia del último nucleótido del último exón predicho adecuada.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína y la predicción del elemento SECIS), son indicativos de la conservación de la SelN en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:

Para estudiar si la SelO se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelO de Homo sapiens, ya que la SelO de Pteropus vampyrus no se encontraba bien anotada (su secuencia no empezaba por el aminoácido metionina). En la query problema de Homo sapiens encontramos una U, indicando la posición de la selenocisteína.
Al hacer el blastall, obtuvimos 3 hits (scaffolds) significativos, pero elegimos KE752061.1 porque era el único que contenía la selenocisteína. Además presentaba un valor de e value bajo (3e-30), era el que cubría una mayor parte de la query y el que presenta mayor homología con la query problema la hacer el análisis con t-coffee.
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene 9 exones (ver esquema más abajo).
Además, en el exonerate observamos en nuestro scaffold un codón stop UGA (que codifica para la selenocisteína), alineado con un codón "Unknown" en nuestra query problema, cosa que es indicativa de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (el forward) y a una distancia del último nucleótido del último exón predicho adecuada. En cambio, al contrario de lo esperado el programa Seblastian no nos ha predicho ninguna selenoproteína.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína y la predicción del elemento SECIS), son indicativos de la conservación de la SelO en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
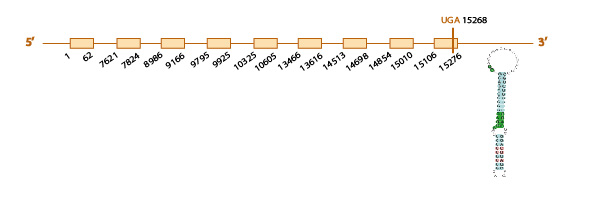
Para estudiar si la SelP se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelP de Pteropus vampyrus ya que estaba bien anotada. En la query problema de Pteropus vampyrus encontramos 10 Us, indicando la posición de 10 selenocisteínas.
Al hacer el blastall, obtuvimos un único hit (scaffold) significativo KE762614.1, que cubría varias de las posibles selenocisteínas. Además, este scaffold presentaba un e value de 3e-92, y una gran homología con la query problema cuando miramos el t-coffee, con un score de 97.
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene 4 exones (ver esquema más abajo).
Además, en el exonerate observamos en nuestro scaffold 9 codones stop UGA (que codifican para la selenocisteína), alineados con un codón "Unknown" en nuestra query problema, cosa que es indicativa de la presencia de 9 selenocisteínas en nuestro scaffold.
Finalmente, con el programa SECISearch3, predijimos 2 elementos SECIS, ambos posibles. Los dos son de grado A y están situados en el mismo strand de nuestro scaffold (el forward) a una distancia del último nucleótido del último exón predicho adecuada. Esto coincide con lo que sabemos ya que SelP es la única selenoproteína que contiene 2 elementos SECIS [6].
A su vez, con el Seblastian vimos que el scaffold elegido para la SelP presenta gran homología con la SelP precursora de Equus caballus, cosa que nos da robusteza a nuestra hipótesis de que la SelP está conservada.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de las selenocisteínas y la predicción de los 2 elementos SECIS), son indicativos de la conservación de la SelP en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
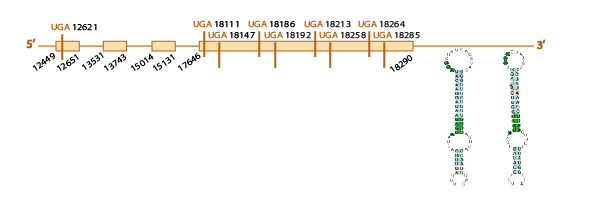
Familia de Selenoproteínas R (SelR)
La familia de las Methionine R sufoxide reductase o SelR está formada por tres proteínas denominadas SelR1, SelR2 y SelR3. Para determinar si esta familia se encuentra conservada en Eidolon helvum hemos utilizado como query problema la SelR1 de Pteropus vampyrus y la SelR2 y la SelR3 de Homo sapiens, pues la primera no estaba corrrectamente anotada y la segunda no estaba presente en el genoma de Pteropus vampyrus.
Sel R1:
Al realizar el blastall obtuvimos varios resultados significativos, de entre estos descartamos aquellos que no cubrían la selenocisteína de la query problema, los de baja identidad y aquellos que cubrían sólo parcialmente la query problema. Finalmente seleccionamos el scaffold KE760349.1 (e-value 4e-22) ya que presentaba mayor homología al realizar el tcoffee. El tcoffee (score 99) tenía una homología muy buena, casi perfecta con nuestra query, presentando la selenocisteína en el lugar correcto.
Al analizar este scaffold con exonerate y genewise, observamos una predicción muy similar con los dos programas (observar la tabla) y también vimos que el scaffold contiene 3 exones (ver esquema de abajo). Además, en el exonerate observamos en nuestro scaffold un codón stop, el codón UGA (que codifica para la selenocisteína), alineado con un codón "Unknown" de la query problema, la cual cosa como ya hemos visto es indicativa de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (forward) y a una distancia del último nucleótido del último exon predicho adecuada (<6000 nucleótidos), y el programa Seblastian predijo homología de nuestro scaffold con la SelR1 de Pan troglodytes.
Los resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína y la predicción de un elemento SECIS y de homología de nuestro scaffold con la SelR1 de otra especie), son indicativos de la conservación de la selenoproteína SelR1 en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
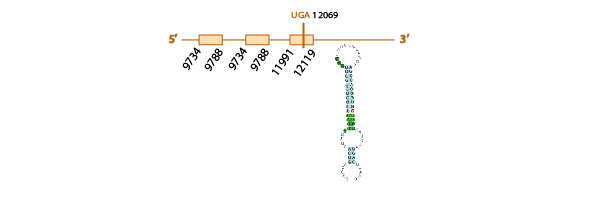
Sel R2:
Al realizar el blastall obtuvimos varios resultados significativos, de entre éstos descartamos aquellos que no cubrían la selenocisteína de la query problema, los de baja identidad y aquellos que cubrían sólo parcialmente la query problema. Finalmente seleccionamos el scaffold KE755266.1 (e-value 5e-22), ya que presentaba mayor homología con la query al realizar el tcoffee. El tcoffee (score 99) tenía una homología muy buena y casi perfecta con nuestra query a excepción del inicio de la proteína, que no se encuentra conservada en este scaffold.
Al analizar este scaffold con el exonerate y el genewise, observamos una predicción muy similar con los dos programas (observar la tabla) y también vimos que el scaffold contiene 4 exones (ver esquema de abajo). Ni nuestra query inicial (human) ni la proteína anotada en P.vampyrus, presentaba ninguna selenocisteína. De la misma forma, ésta tampoco estaba presente en nuestro scaffold de E. helvum. SelR2, por lo tanto, se trata de un homólogo de cisteína. Podría ser que la pérdida de la selenocisteína sucediera en un ancestro común de estas tres especies, o bien que por un proceso de convergencia evolutiva, las tres especies independientemente presentasen alguna mutación responsable de la pérdida de la selenocisteína.
Finalmente, con el programa SECISearch3 no predijimos ningún elemento SECIS en nuestro scaffold.Tampoco obtuvimos ningún resultado con Seblastian.
Con los distintos resultados obtenidos, aunque SelR2 no se encuentre completa en nuestro scaffold, con la elevada homología presentada entre la query problema y nuestro scaffold, podemos hipotetizar que SelR2 se encuentra conservada en Eidolon helvum. Podría ser que la parte de la proteína que no está presente en nuestro scaffold estuviera presente en algun scaffold contiguo al nuestro.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
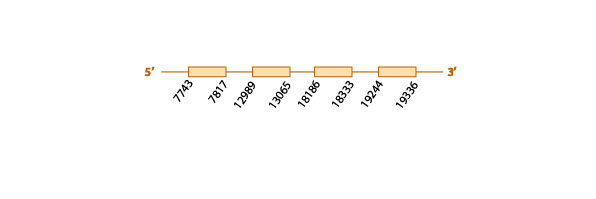
Sel R3:
Al realizar el blastall obtuvimos varios resultados significativos, de entre éstos seleccionamos el scaffold KE754241.1 (e-value 3e-17) que presentaba la mayor homología en comparación con los demás al realizar el tcoffee. Sin embargo, comentar que ningún scaffold significativo se alineaba completamente con la query problema.
Al analizar este scaffold con exonerate y genewise, observamos una predicción muy similar con los dos programas (observar la tabla) y también vimos que el scaffold contiene 2 exones (ver esquema más abajo). Ni nuestra query problema (human) ni la proteína anotada en P.vampyrus presentaban ninguna selenocisteína (son homólogos de cisteína), por lo que del mismo modo ésta tampoco estaba presente en nuestro scaffold de E. helvum.
Finalmente, con el programa SECISearch3 no predijimos ningún elemento SECIS en nuestro scaffold. Tampoco obtuvimos ningún resultado con Seblastian.
Con los distintos resultados obtenidos no podemos determinar que la proteína se encuentre conservada en nuestra especie, solo podemos hipotetizar que si está conservada su secuencia nucleotídica se encuentra fragmentada en distintos scaffolds. Teniendo en cuenta que esta proteína no está presente en Pteropus vampyrus, es muy probable que SelR3 se haya perdido en algún ancestro común entre P. vampyrus y Eidolon helvum.
Para estudiar si la SelS se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SelS de Homo sapiens, ya que la SelS de Pteropus vampyrus no se encontraba bien anotada (su secuencia no empezaba por el aminoácido metionina).
En la query problema de Homo sapiens encontramos una U, indicando la presencia de la selenocisteína. Sin embargo, con el blastall obtuvimos 3 scaffolds significativos que no contenían (ninguno de los tres) la selenocisteína. Elegimos el scaffold KE756635.1 que era el que presentaba un e value menor (1e-12) aunque la homología de este scaffold con la query problema era muy baja (de hecho, ningún scaffold significativo daba una buena homología con la query problema).
Además, tampoco observamos ningún codón stop UGA (que codifica para la selenocisteína) alineado con un codón "Unknown" en nuestra query problema (la cual cosa ya preveíamos al saber que este scaffold no cubría la selenocisteína presente en la query humana).
Finalmente, con el programa SECISearch3, al contrario de lo que esperábamos, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (el forward) y a una distancia del último nucleótido del último exón predicho adecuada.
Podría ser que la proteína de Eidolon helvum sí contenga una selenocisteína, pero que no la detectáramos por no estar incluída en nuestro scaffold. El programa Seblastian no nos predijo ninguna selenoproteína.
Con los resultados obtenidos, podemos deducir que la SelS no está conservada en Eidolon helvum.
SelT es una proteína que aunque se encuentra en P. vampyrus no está correctamente anotada, por lo que hemos utilizado la correspondiente query de Homo sapiens.
Al realizar el blastall obtuvimos varios resultados significativos, de entre éstos descartamos aquellos que no cubrían la selenocisteína de la query problema y los de baja identidad. Comentar que no había ningún scaffold que cubriera la mayoría de la query problema.
Finalmente seleccionamos el scaffold AWHC01032017.1 (e-value 1e-18) porque en el programa t-coffee (score 100) presentaba una homología muy buena con la región que cubría la selenocisteína. Hay que destacar que la anterior región de la query problema se encontraba conservada en otro scaffold, el KE819888.1.
Al analizar el scaffold elegido con exonerate y con genewise, observamos una predicción muy similar con los dos programas (ver la tabla) y también vimos que el scaffold contiene 2 exones (ver esquema de abajo). Además, en el exonerate observamos en nuestro scaffold un codón stop, el codón UGA (que codifica para la selenocisteína), alineado con un codón "Unknown", la cual cosa es indicativa de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, no encontramos ningún elemento SECIS en el scaffold seleccionado. Esto tiene sentido ya que en este scaffold no se encuentra la región final de la proteína.
Con los resultados obtenidos, a pesar de que no hemos podido predecir ninún elemento SECIS, la elevada homología observada entre la query problema y el scaffold seleccionado y la conservación de la selenocisteína, son indicativas de la conservación de esta proteína en Eidolon helvum. Podemos hipotetizar que esta proteína se encuentra en dos scaffolds distintos, AWHC01032017.1 y KE819888.1.
Aquí mostramos un esquema predictivo de esta possible selenoproteína con toda la información obtenida:
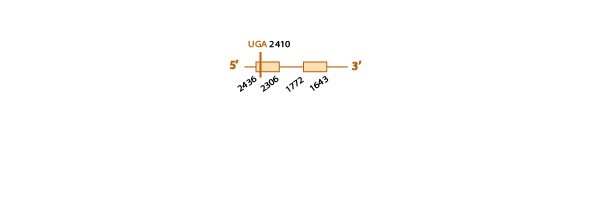
Ésta es una familia de selenoproteínas compuesta por tres miembros: SelU1, SelU2 y SelU3. Ni SelU1 ni SelU2 estaban bien anotadas en Pteropus vampyrus y por tanto usamos las correspondientes querys de Homo sapiens. En cambio sí que pudimos utilizar la query de SelU3 de Pteropus vampyrus ya que estaba bien anotada.
SelU1
Al realizar el blastall obtuvimos varios resultados significativos, de entre éstos descartamos aquellos que no cubrían la selenocisteína de la query problema, los de baja identidad y aquellos que cubrían sólo parcialmente la query problema.
Finalmente seleccionamos el scaffold KE816858.1 (e-value 1e-24), ya que era el que presentaba mayor homología al realizar el tcoffee.
El análisis tcoffee (score 99) presentaba una homología muy buena, casi perfecta con nuestra query.
Al analizar este scaffold con exonerate y con genewise, observamos una predicción muy similar con los dos programas (ver la tabla) y también vimos que el scaffold contiene 5 exones (ver esquema más abajo). Ni nuestra query inicial (Homo sapiens), ni la proteína anotada en P.vampyrus presentaban ninguna selenocisteína. Tampoco predijimos ninguna selenocisteína en el scaffold seleccionado de E. helvum, la cual cosa nos indica que SelU1 es un homólogo de cisteína.
Finalmente, con el programa SECISearch3 no encontramos ningún elemento SECIS, tal como esperaríamos al saber que esta proteína no contiene ninguna selenocisteína. Tampoco obtuvimos ningún resultado con el análisis de Seblastian.
Los distintos resultados obtenidos de elevada homología de este scaffold con la query, son indicativos de la conservación de SelU1 en Eidolon helvum.
Aquí mostramos un esquema predictivo de SelU1 con toda la información obtenida:
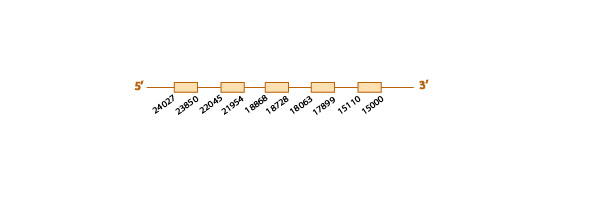
SelU2
Observamos que la query problema no contenía ninguna selenocisteína.
Al realizar el blastall obtuvimos varios resultados significativos, y de entre éstos descartamos aquellos de baja identidad.
Finalmente seleccionamos el scaffold KE819056.1 (e-value 2e-18), ya que presentaba mayor homología al realizar el tcoffee, con un score de 99. Sin embargo, la región inicial no la encontramos en nuestro scaffold.
Al analizar este scaffold con el comando exonerate y con el genewise, observamos una predicción muy similar con los dos programas (ver la tabla) y también vimos que el scaffold contiene 6 exones (ver esquema más abajo), y que no contiene ninguna selenocisteína. Por lo tanto, consideramos que SelU2 es un homólogo de cisteína en E. helvum.
Finalmente, con el programa SECISearch3 no predijimos ningún elemento SECIS (tal como esperaríamos), ni tampoco ninguna selenoproteína homóloga con el programa Seblastian.
Los distintos resultados obtenidos de elevada homología de este scaffold con la query, son indicativos de la conservación de SelU2 en Eidolon helvum.
Aquí mostramos un esquema predictivo de SelU2 con toda la información obtenida:
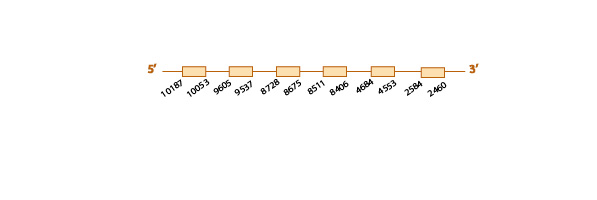
SelU3
Al realizar el blastall obtuvimos varios resultados significativos, de entre éstos descartamos los de baja identidad y aquellos que cubrían sólo parcialmente la query problema.
Finalmente seleccionamos el scaffold KE769133.1 (e-value 3e-36), ya que presentaba mayor homología respecto los demás en el tcoffee, con un score de 100 (casi perfecta).
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con los dos programas (ver la tabla) y también vimos que el scaffold contiene 7 exones (ver esquema más abajo).
Ni nuestra query inicial de P.vampyrus, ni la proteína anotada en humanos presentaban ninguna selenocisteína. Por tanto, tampoco la encontramos en el scaffold seleccionado de E. helvum, con lo que dedujimos que SelU3 es un homólogo de cisteína en nuestra especie.
Finalmente, con el programa SECISearch3 no encontramos ningún elemento SECIS, tal como esperaríamos al saber que esta proteína no contiene ninguna selenocisteína. Tampoco predijimos ninguna selenoproteína homóloga con Seblastian.
Los resultados obtenidos de elevada homología del scaffold seleccionado con la query problema son indicativos de la conservación de esta proteína en Eidolon helvum.
Aquí mostramos un esquema predictivo de esta proteína con toda la información obtenida:
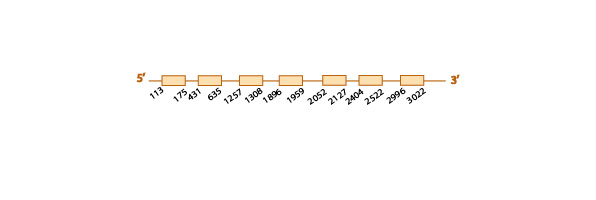
Familia de selenoproteínas W (SelW)
Es una familia de selenoproteínas compuesta por dos miembros SelW1 y SelW2. Ni SelW1 ni SelW2 se encuentran bien anotadas en Pteropus vampyrus y por tanto hemos utilizado las correspondientes proteínas de Homo sapiens como query problema.
SelW1
Al realizar el blastall obtuvimos un único scaffold KE761826.1 significativo, con una e-value de 5e-09. Además, presentaba una homología casi perfecta al realizar el tcoffee, con un score de 99. Sin embargo, la región inicial de la proteína no se encontraba presente en el scaffold, y es precisamente aquí donde se encuentra la posible selenocisteína. .
Al analizar este scaffold con exonerate y con genewise, observamos una predicción muy similar con los dos programas (ver la tabla) y también vimos que el scaffold contiene 3 exones (ver esquema más abajo).
Finalmente, con el programa SECISearch3, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (forward) y a una distancia adecuada.
Los distintos resultados obtenidos no nos permiten confirmar la correcta conservación de la selenoproteína SelW1 en Eidolon helvum, habría que determinar si la región inical, la que contiene la selenocisteína, se encuentra en otro scaffold.
Hipotetizamos que la gran homología observada entre el scaffold seleccionado y la query problema, junto con la predicción de un elemento SECIS, indican conservación de SelW1 en Eidolon helvum, aunque la ausencia de selenocisteína en este scaffold nos impiden saber si esta proteína es funcional o no en E. helvum.
Aquí mostramos un esquema predictivo de esta selenoproteína con toda la información obtenida:
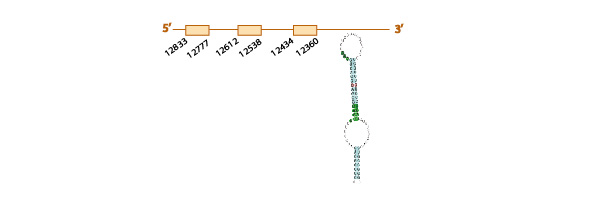
SelW2
Los resultados tanto del blast como del exonerate, genewise y tcoffee son totalmente idénticos al de la proteína anterior, por lo que podemos determinar que en nuestra especie solo hay una proteína de esta familia (SelW1). Esto podría deberse a la presencia de una duplicación de SelW1 en el genoma humano que no se ha producido en Eidolon helvum. En la especie P. vampyrus no hay ninguna de las dos proteínas anotadas, lo que refuerza nuestra hipótesis.
SelV
Para determinar si esta proteína se encuentra o no conservada en Eidolon helvum, usamos como query problema la secuencia de la SelV de Homo sapiens, ya que la SelV no está presente en Pteropus vampyrus.
Al realizar el blastall obtuvimos solo un resultado significativo, el del scaffold KE799521.1 (e-value 4e-08), y el tcoffee (score 73) apenas presentaba ningún match, y el alineamiento tampoco cubría la selenocisteína presente en la query problema.
Al analizar este scaffold con exonerate y con genewise, observamos una predicción muy similar con los dos programas (observar la tabla) y también vimos que el scaffold contiene 7 exones.
Con el programa SECISearch3, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (forward) y a una distancia del último nucleótido del último exon predicho adecuada (<6000 nucleótidos).
Aunque la predicción de un elemento SECIS normalmente es indicativa de la conservación de la proteína en cuestión, en este caso la baja homología del único scaffold significativo con la query problema descarta la conservación de esta proteína en Eidolon helvum. El hecho de que esta proteína tampoco esté conservada en Pteropus vampyrus respalda nuestra hipótesis; es probable que esta selenoproteína se perdiera en algún ancestro común de ambas especies de megaquirópteros.
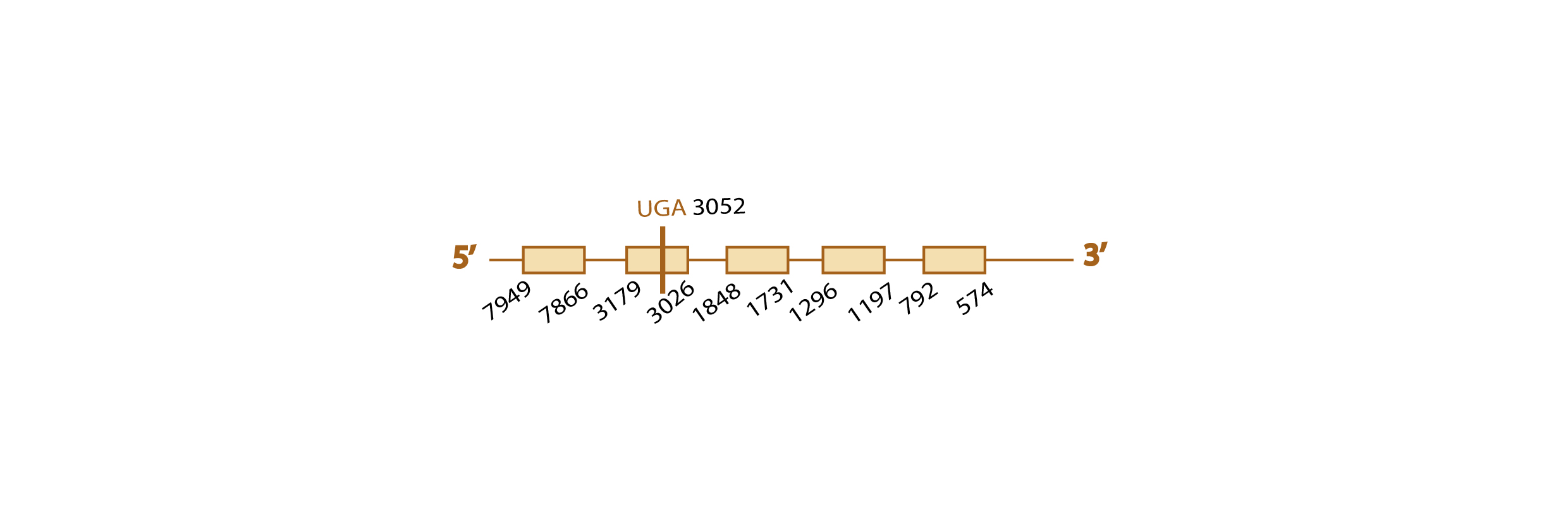
MsrA
Para saber si esta proteína se encontraba o no conservada en Eidolon helvum, usamos como query problema la proteína MsrA de Pteropus vampyrus, que estaba bien anotada. Observamos que ya en la query problema no había ninguna selenocisteína. MsrA es un homólogo de cisteína.
Al hacer el análisis blast, obtuvimos distintos scaffolds significativos. Vimos que los 4 scaffolds más significativos obtenidos (AWHC01279060.1, KE757833.1, KE794280.1 i KE749494.1) eran contiguos y cubrían la query problema con elevada homología (vista en el tcoffee) desde el aminoácido 43 hasta el último aminoácido de la query problema (el 180). Dedujimos que el hecho de que el gen para esta proteína se encontrase fragmentado en distintos scaffolds se debería probablemente a algun problema en el ensamblaje del genoma de Eidolon helvum.
Debido a la dicha fragmentación del gen en los 4 scaffolds descritos, no pudimos continuar con los análisis, y por lo tanto no podemos afirmar que esta proteína se encuentre conservada en Eidolon helvum con certeza, aunque la elevada homología observada en todos los scaffolds para la query problema nos indican que es probable que así sea.
Aquí describimos la identidad observada de los 4 scaffolds diferentes para la query problema y la región de la query que cubren:
- AWHC01279060.1: cubre 42 aminoácidos de la query (68-110aa) con una identitat del 95% y un valor de e value de 3e-20
- KE757833.1: cubre 36 aminoácidos de la query (144-180aa) con una identidad del 94% y un valor de e value de 3e-15
- KE794280.1: cubre 35 aminoácidos de la query problema (109-144aa) con una identidad del 94% y un valor de e value de 6e-14
- KE749494.1: cubre 26 aminoácidos de la query problema (43-69aa) con una identidad del 86% y un valor de e value de 2e-06
Proteínas de maquinaria
Para estudiar si esta proteína se encuentra o no conservada en Eidolon helvum, usamos como query problema la secuencia de SBP2 de Homo sapiens, ya que la de Pteropus vampyrus se encontraba mal anotada.
Al hacer el blast, obstuvimos dos únicos scaffolds significativos. Escogimos KE806098.1 (reverse strand), ya que tenía un e value muy bajo (2e-46), y era el hit que cubría una mayor proporción de la query problema (el otro scaffold cubría menos de 100 aminoácidos de una query con 854 aminoácidos totales).
En el análisis con tcoffee se observó una gran homología de secuencias entre la query problema humana y el scaffold seleccionado. Además, el scaffold cubría la query problema casi en su totalidad, exceptuando los primeros 57 aminoácidos.
La predicción hecha con el genewise y con el exonerate dio resultados similares. Todos estos resultados se encuentran en esta tabla.
Lógicamente, al tratarse de una proteína de maquinaria que no contiene ninguna selenocisteína, no predijimos ningun elemento SECIS con SECISearch3. Tampoco obtuvimos ninguna predicción con Seblastian.
Con los resultados obtenidos, podemos hipotetizar que SBP2 se encuentra conservada en Eidolon helvum. Ésta es la predicción de la estructura de SBP2 en el genoma de Eidolon helvum en base a nuestros resultados:
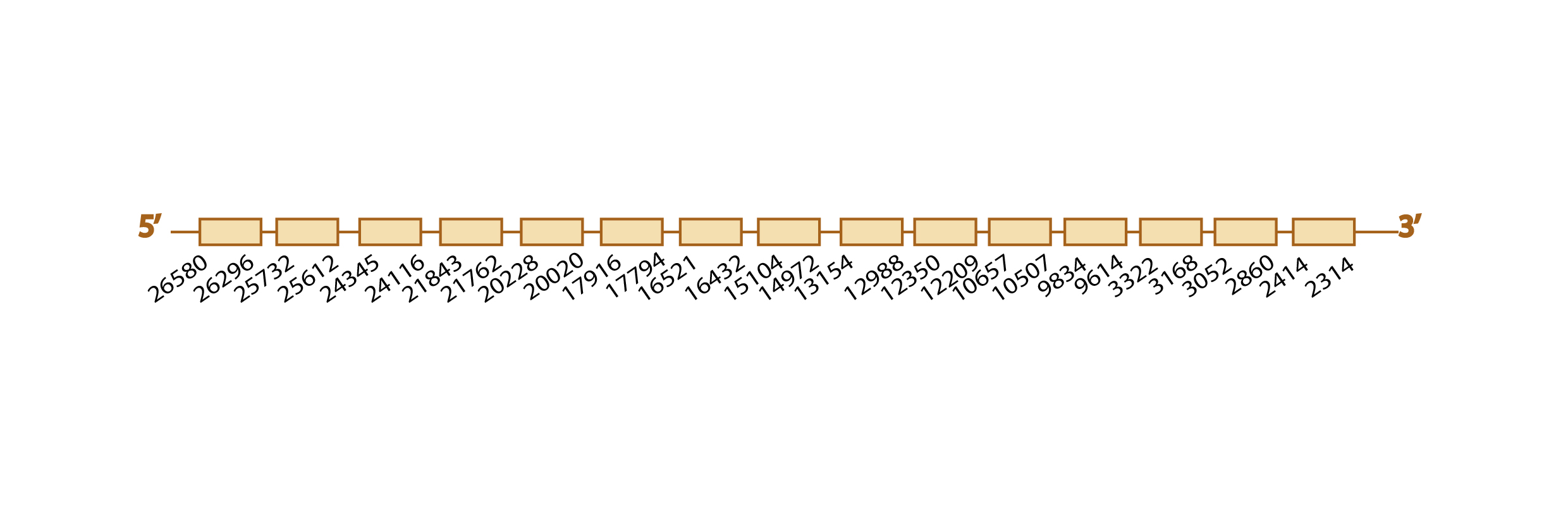
Para estudiar si la SPS1 se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SPS1 de Pteropus vampyrus, ya que estaba bien anotada. Lógicamente, al tratarse de una proteína de maquinaria de síntesis de selenoproteínas, en esta query problema no encontramos ninguna selenocisteína (U).
Al hacer el blastall, obtuvimos cuatro hits (scaffolds) significativos. Descartamos uno de ellos porque ya lo habíamos utilizado para la Selenophosphate Synthetase 2 y nos había dado unos resultados muy buenos. Descartamos también aquellos que presentaban baja identidad o que cubrían parcialmente la query problema. Por tanto, nos quedamos con el scaffold AWHC01215926.1, ya que era el único que cubría toda la query y era el que tenía un e value menor (e-134).
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene 3 exones (ver esquema de abajo).
Finalmente, con el programa SECISearch3 o Seblastian tampoco predijimos ningún elemento SECIS, tal como esperarábamos.
Los distintos resultados obtenidos (sobre todo la elevada homología observada entre ambas secuencias) son indicativos de la conservación de SPS1 en Eidolon helvum.
Aquí mostramos un esquema predictivo con toda la información obtenida:
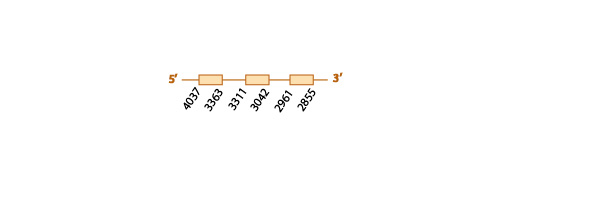
Para estudiar si la SPS2 se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SPS2 de Pteropus vampyrus, ya que se encontraba bien anotada. En esta query problema observamos una U, indicando la presencia de una selenocisteína. Esto se debe a que a pesar de formar parte de la maquinaria de síntesis, esta proteína puede considerarse también una selenoproteína.
Al hacer el bastall, obtuvimos 4 hits (scaffolds) significativos. Descartamos uno de ellos porque no contenía la selenocisteína. Finalmente, nos quedamos con el scaffold KE805803.1, ya que era el que presentaba la mayor homología con la query problema al hacer el análisis con el t coffee. Además, tenía un e value muy bajo (0'000...) y era el scaffold que cubría una mayor proporción de la query problema.
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla) y también vimos que el scaffold contiene un solo exón (ver esquema más abajo).
Además, en el exonerate observamos en nuestro scaffold un codón stop UGA (que codifica para la selenocisteína), alineado con un codón "Unknown" en nuestra query problema, cosa que es indicativa de la presencia de una selenocisteína en nuestro scaffold.
Finalmente, con el programa SECISearch3, predijimos un elemento SECIS de grado A en el mismo strand de nuestro scaffold (el reverse) y a una distancia del último nucleótido del último exón predicho adecuada.
A su vez, con el Seblastian vimos que el scaffold elegido para la SPS2 presentaba gran homología con la selenoproteína de Canis lupus familiaris, cosa que nos da robusteza a nuestra hipótesis de que la SPS2 está conservada.
Los distintos resultados obtenidos (la elevada homología de este scaffold con la query problema, la conservación de la selenocisteína y la predicción del elemento SECIS y de homología con otra selenoproteína), son indicativos de la conservación de la SPS2 en Eidolon helvum.
Aquí mostramos un esquema predictivo con toda la información obtenida:
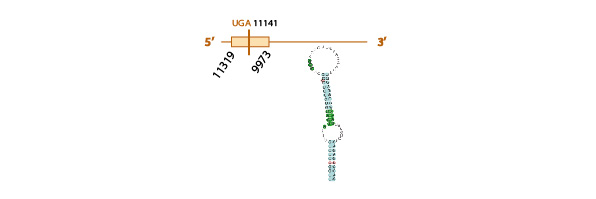
Para estudiar si la SecS se encuentra o no conservada en Eidolon helvum, usamos como query problema la proteína SecS de Homo sapiens, ya que a pesar de que la de Pteropus vampyrus estaba bien anotada, los resultados que obtuvimos con esta query en el programa tcoffee eran de muy mala calidad. Sin embargo, usando la query de Homo sapiens pudimos estudiar la homología de secuencias de esta proteína con la de Eidolon helvum. En esta query problema de Homo sapiens no encontramos ninguna U, ya que se trata de una proteína de maquinaria de síntesis de selenoproteínas y no de una selenoproteína per sé.
Al hacer el blastall, obtuvimos 3 hits (scaffolds) significativos. No encontramos grandes diferencias en cuanto a la homología ni el score del t coffee entre los diferentes scaffolds por lo que al final elegimos el KE796598.1, ya que era el scaffold que cubría más query problema. Sin embargo, cabe destacar que sólo cubre media query (del aminoácido 270 al 501).
Al analizar este scaffold con el exonerate y con el genewise, observamos una predicción muy similar con ambos programas (ver tabla), y también vimos que el scaffold contiene 5 exones (ver esquema más abajo).
Finalmente, con el programa SECISearch3 o Seblastian tampoco predijimos ningún elemento SECIS.
La elevada homología observada entre la query problema y el scaffold seleccionado son indicativos de la conservación de esta proteína en Eidolon helvum, aunque este scaffold no cubra la totalidad de la query problema.
Aquí mostramos un esquema predictivo de la proteína con toda la información obtenida:
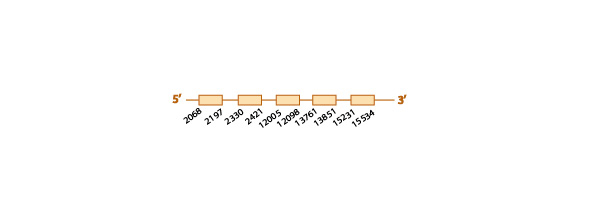
Con el objetivo de estudiar si esta proteína se encuentra conservada en Eidolon helvum utilizamos como query problema la proteína de Homo sapiens, ya que la de Pteropus vampyrus se encontraba mal anotada. Esta query problema no presentaba ninguna selenocisteína (U).
Después de hacer el blast obtuvimos 7 scaffolds. Entre todos ellos nos quedamos con el que tenía un mejor e-value (4e-21), no solo por su bajo valor sino también porque cubría toda la query problema con una identidad del 96%. Este scaffold es el KE815164.1.
Al analizar este scaffold con el programa exonerate y el genewise observamos discrepancias. Mientras que los dos programas predecían el inicio de la proteína en el mismo punto, el genewise predecía el final aproximadamente 10.000 nucleótidos antes que el exonerate. Esto alteraba la predicción de la estructura de la proteína; 9 exones contenía ésta según exonerate, y 6 exones según genewise.
La alta homología observada en este scaffold con la query problema es indicativa de su conservación en Eidolon helvum, pese a que no predijimos ningún elemento SECIS, tal como esperábamos, porque esta proteína es de maquinaria y no es própiamente una selenoproteína.
Aquí mostramos un esquema predictivo de esta proteína con toda la información obtenida de la predicción hecha por exonerate:
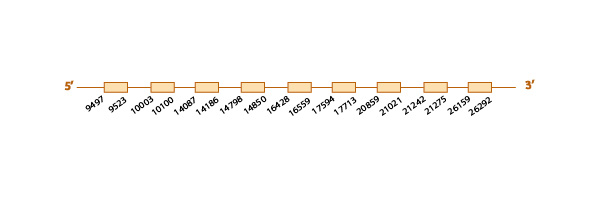
Para estudiar si encontramos esta proteína conservada en Eidolon helvum, hemos utilizado como query problema la de Homo sapiens, ya la de Pteropus vampyrus se encontraba mal anotada.
Con el blast obtuvimos dos scaffolds. Elegimos el que presentaba una mayor identidad (91%) y major score en el tcoffe (98) con la query problema, a pesar de que el otro scaffold tenía un e-value menor.
Tanto el genewise como el exonerate predijeron la misma proteína, conteniendo ésta 6 exones.
No se ha encontrado ninguna posible selenocisteína ni se ha predicho ningún elemento SECIS. Esto concuerda con nuestra idea inicial, ya que se trata de una proteína de maquinaria.
La elevada homología de este scaffold con la query problema, nos hace pensar que esta proteína se encuentra conservada en el genoma de Eidolon helvum.
Aquí mostramos un esquema predictivo de la proteína con toda la información obtenida:
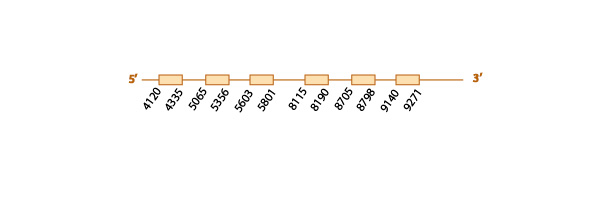
Se trata de una proteína que forma parte de la maquinaria de las selenoproteínas y que está bien anotada en Pteropus vampyrus, por tanto hemos elegido la eEFsec de esta especie como query problema.
Al realizar el blastall obtuvimos varios resultados significativos, de entre éstos seleccionamos el scaffold KE772629.1 (e-value 2e-36) ya que presentaba la mayor homología en el análisis de tcoffee en comparación con los demás. Sin embargo, este scaffold cubría solamente el final de la query problema y no presentaba total homología. Hay que destacar que había otro scaffold que cubría la parte inicial de la proteína.
Al analizar el scaffold seleccionado con el exonerate y con el genewise, observamos una predicción muy similar con los dos programas (ver esta tabla) y también vimos que el scaffold contiene 3 exones (ver esquema de abajo).
Finalmente, tal como esperaríamos tal tratarse de una proteína de maquinaria, con el programa SECISearch3 no predijimos ningún elemento SECIS.
Con los distintos resultados obtenidos, podemos confirmar la conservación de esta proteína en el genoma de Eidolon helvum. Ésta proteína se encuentra fragmentada en dos scaffolds distintos, por este motivo la homología presentada entre el scaffold seleccionado y la query problema solo es total en un fragmento de la query inicial. Aquí mostramos un esquema predictivo de la proteína con toda la información obtenida: