Introducción a las selenoproteínas
Importancia del selenio
En 1817 el químico sueco J. J. Berzelius descubrió el selenio que, desde 1957, se considera un micronutriente fisiológicamente esencial para gran variedad de especies, entre las que se incluye la humana. El déficit de selenio en humanos es muy infrecuente, pero alteraciones en la homeostasis de este elemento pueden condicionar la respuesta inmune o estar implicadas en procesos neurodegenerativos, enfermedad cardiovascular o cáncer. Por otro lado, se ha observado que el exceso de selenio causa toxicidad (selenosis).
Los beneficios del selenio se obtienen de su incorporación al aminoácido selenocisteína, que a su vez forma parte de un conjunto de proteínas conocidas como selenoproteínas. Este hecho evita la toxicidad de este elemento y permite su participación en procesos biológicos tan relevantes como el correcto funcionamiento del sistema inmunitario y la función tiroidea, la protección antitumoral, la homeostasis REDOX y el efecto antioxidante que protege del daño celular causado por las especies reactivas de oxígeno (ROS).
¿Qué son las selenoproteínas?
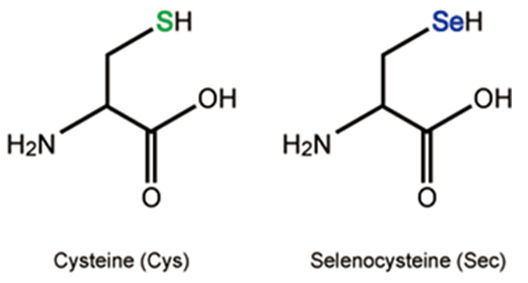
Las selenoproteínas son un conjunto de proteínas que se caracterizan por incorporar en su secuencia el aminoácido selenocisteína, un análogo de la cisteína en el que un átomo de selenio sustituye al átomo de azufre. El selenio y el azufre pertenecen al mismo grupo en la tabla periódica, lo que les confiere propiedades similares. Sin embargo, el pKa de la selenocisteína es menor que el de la cisteína (5,2 vs 8,3), lo cual proporciona una mayor reactividad y mejor eficiencia catalítica a las selenoproteínas. Además de selenoproteínas, existen en ciertas especies homólogos en cisteína, que son selenoproteínas que han sustituido el aminoácido selenocisteína por una cisteína. Es relevante destacar que no se ha encontrado en la naturaleza el proceso de mutagénesis inversa, es decir, de cisteína a selenocisteína, de manera que parece poco probable la aparición de nuevas selenoproteínas.
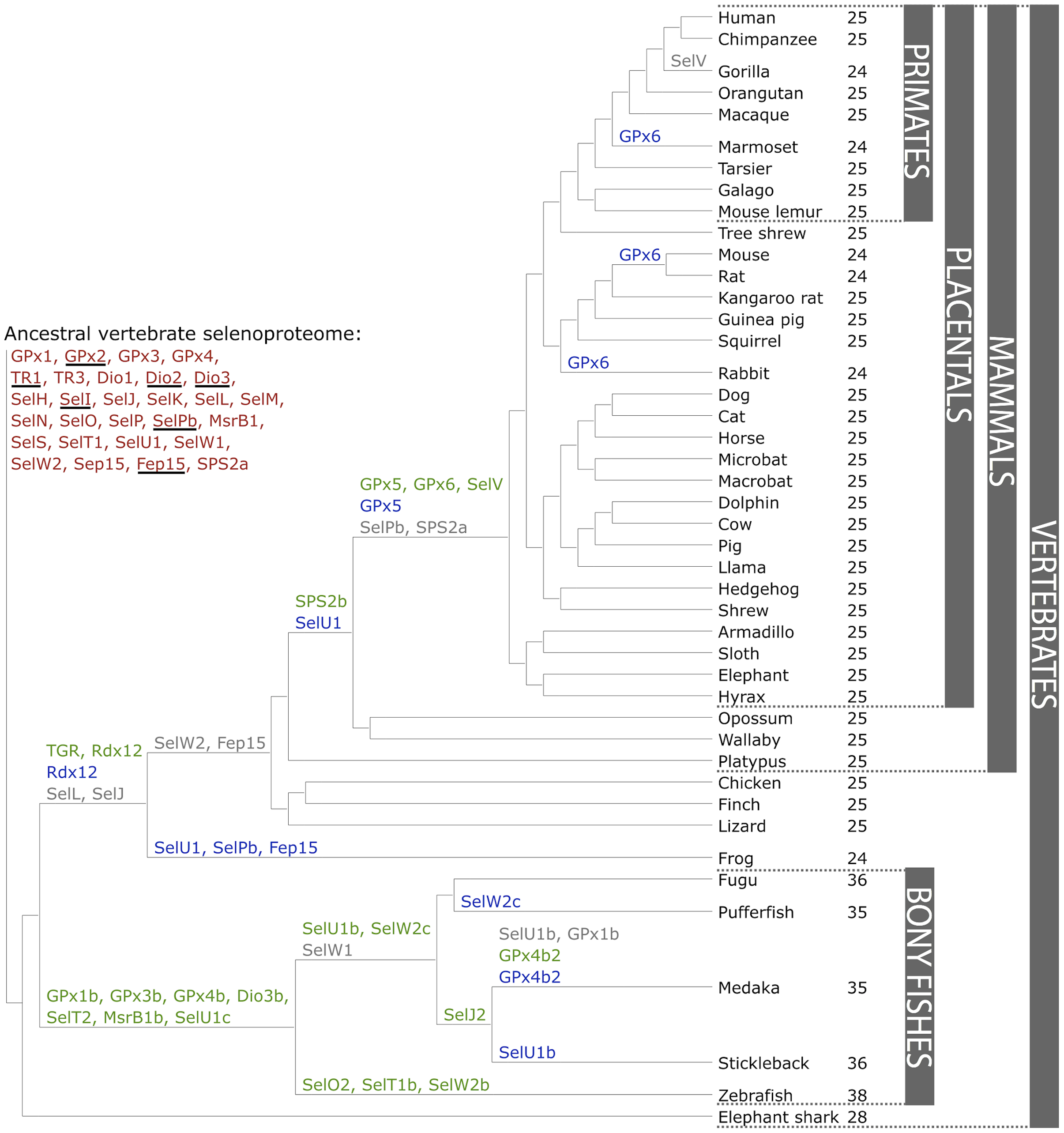
La existencia de selenoproteínas es muy común entre organismos eucariotas, aunque hay bastante diversidad entre especies. Cada selenoproteína tiene una historia evolutiva diferente, puesto que pueden estar presentes en una cierta especie o grupo taxonómico, ausentes en otro grupo filogenéticamente cercano y presentes de nuevo en un grupo filogenéticamente alejado. Se han descrito 25 selenoproteínas en humanos, 24 de las cuales también existen en roedores. También se conocen 3 selenoproteínas en Drosophila melanogaster y 1 en Caenorhabditis elegans, pero no se ha descrito ninguna en especies vegetales.
Las selenoproteínas humanas son: 5 glutatión peroxidasas (GPX1, GPX2, GPX3, GPX4 y GPX6), 3 yodotironina deyodinasas (DI1, DI2, DI3), Sel15, 1 selenofosfato sintasa (SPS2), SelH, SelK, SelI, SelM, SelN, SelP, SelO, SelR1, SelS, SelT, SelV, SelW1, y 3 tioredoxina reductasas (TR1, TR2 y TR3). La mayoría contienen una única selenocisteína, a excepción de SelN, SelP y algunas isoformas de DI.
Biosíntesis de las selenoproteínas
La incorporación de una selenocisteína (U) en una selenoproteína requiere una maquinaria de traducción específica que permita decodificar el codón UGA del mRNA, ya que este triplete se interpreta habitualmente como un codón STOP. Este hecho dificulta considerablemente la predicción y anotación de selenoproteínas en un genoma recién secuenciado.
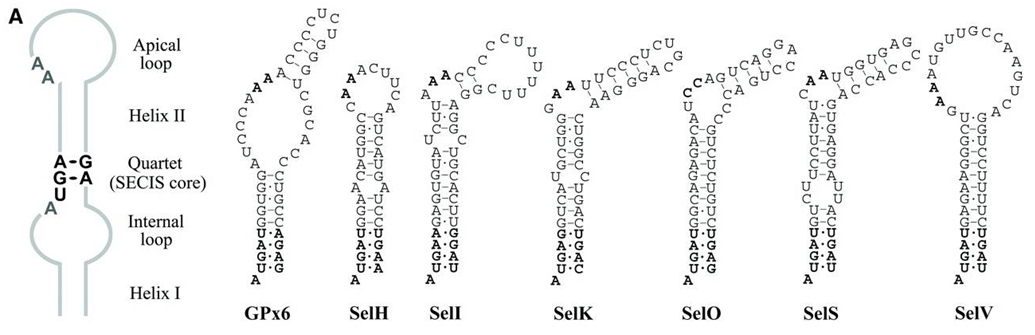
Un elemento clave para diferenciar un codón STOP de un codón codificante para selenocisteína es la existencia de un elemento SECIS (SElenoCysteine Insertion Sequence), situado en la región 3’ UTR de ciertos genes de eucariotas y archeas. Se trata de elementos con una estructura trimensional conservada en forma de loop, pero sin conservación de la secuencia nucleotídica, ya que solo se conserva un cuarteto de nucleótidos que puede variar entre selenoproteínas. Se denominan SECIS canónicos aquellos que conservan el cuarteto AUGA, y el resto se conocen como no canónicos. Existen softwares informáticos que predicen su existencia a partir de un patrón estructural.
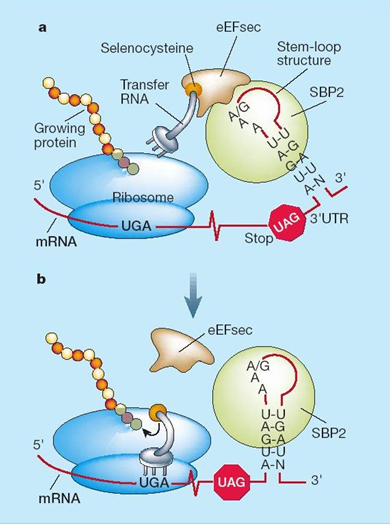
El elemento SECIS recluta la maquinaria de traducción específica e impide la terminación de la proteína cuando aparece el codón UGA en la secuencia del mRNA. Primeramente, recluta a SBP2, una proteína que se une al elemento SECIS y al factor de elongación eEFsec, que es específico para la traducción de selenoproteínas. A continuación, eEFsec aproxima el tRNA específico de selenocisteína (tRNAsec) y su entrada al lugar A del ribosoma. Por último, el tRNAsec interacciona con el codón UGA del mRNA, lo cual permite la incorporación de este aminoácido. El mRNA de selenoproteína continúa su traducción mientras la maquinaria no encuentre un codón STOP.
Por otro lado, la síntesis del aminoácido selenocisteína requiere dos selenofosfato sintasas (SPS1 y SPS2), que son las dos enzimas encargadas que transformar el selenio procedente de la dieta en monoselenofosfato. Concretamente, SPS1 se encarga de reciclar el selenio de otras selenocisteínas, mientras que SPS2 sintetiza el monoselenofosfato. De esta manera, el monoselenofosfato puede incoporarse al seril-tRNAser por acción de la fosfoseril-tRNA quinasa (PSTK), que se encarga de la fosforilación del seril-tRNAser para que sea posible la transferencia del monoselenofosfato.
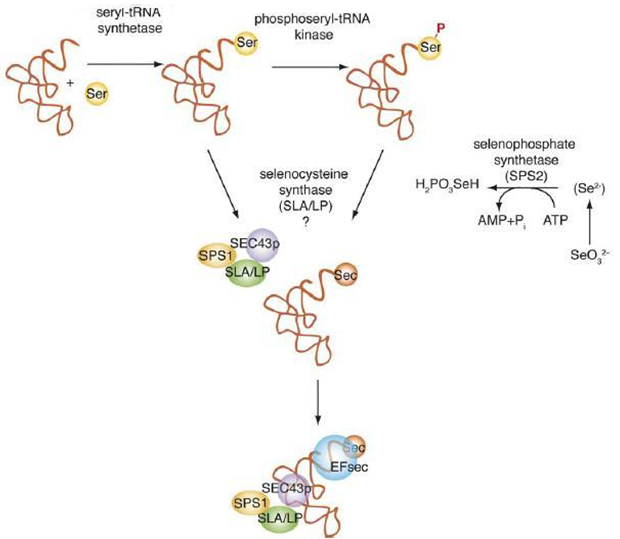
También cabe destacar el papel de otras dos proteínas: por un lado, SLA/LP es una selenocisteína sintasa que participa en el paso de fosfoseril-tRNAser a tRNAsec mediante la defosforilación de la serina anteriormente fosforilada por PTSK; por otro lado, Secp43 participa en la metilación del tRNAsec y la estabilización del complejo SECIS-SBP2/eEFsec/tRNAsec.
Filogenia de selenoproteínas en Alligator mississippiensis
La especie más cercana a Alligator mississippiensis en este árbol filogenético es, probablemente, el lagarto. Si se consideran las posiciones del lagarto y el humano en esta filogenia de selenoproteínas, cabe esperar que SelV y GPX6 no formen parte del genoma del aligátor, ya que se originan mucho después de la separación de Alligator mississippiensis en el árbol filogenético. Se sabe que algunas selenoproteínas ancestrales como Fep15 y SelW2 se perdieron antes de la separación de los reptiles, por lo que, teniendo en cuenta que tampoco están presentes en el selenoproteoma humano, se asume que no se encuentran en el genoma de Alligator mississippiensis.