Selenoproteïnes
Les selenoproteïnes són proteïnes que incorporen l’aminoàcid selenocisteïna en qualsevol posició.
La Selenocisteïna (Sec o U) es considera el 21è aminoàcid, s’assembla a la Cisteïna però enlloc d’un sofre presenta un àtom de seleni (Se), això fa que les propietats químiques d’aquests dos aminoàcids siguin similars. El Seleni es troba en els organismes vius en forma de selenoproteïnes. Aquest és un nutrient essencial per a molts microorganismes i també pels eucariotes. La seva manca pot donar lloc a diferents malalties (per exemple: malaltia de Keshan, problemes musculars...) d'altra banda, l’excés de seleni és tòxic. S’han fet diversos estudis respecte l’efecte del suplement de seleni sobre diferents malalties (càncer de pròstata...), però encara no hi ha resultats concloents.
Degut a que la majoria de selenoproteïnes són enzims que participen en reaccions redox, es creu que poden tenir capacitat de protecció antioxidant.
Les selenoproteïnes apareixen d’una manera molt amplia en l’evolució. Les trobem tant en Archea, bacteris i eucariotes. El nombre varia d’una espècie a un altre, els peixos tenen prop de trenta selenoproteïnes, mentre que el cuc C. elegans en té només una. Es coneixen 25 en humans i 3 en Drosophila melanogaster però no s’ha trobat cap en plantes.
A vegades trobem Cys en un gen ortòleg d’una selenoproteïna en el lloc de la selenocisteïna. Per exemple: en C. elegans només s’ha trobat una selenoproteïna. Però si busquem els gens ortòlegs de selenoproteïnes en altres organismes, en el seu genoma veiem que no trobem selenocisteïna sinó cisteïna.
Biosíntesi de les selenoproteïnes
El codó que codifica per a la selenocisteïna és l’UGA, el qual normalment és un codó STOP de terminació de la traducció.
El que fa que aquest codó no sigui reconegut com a Stop és una estructura 3D al 3’UTR anomenada SECIS ( SElenoCystein Insertion Sequence), que produeix una recodificació del codó UGA com a codó per la selenocisteïna.
SECIS recluta a la proteïna SBP2 (Secis binding protein) i aquesta recluta al factor d'elongació específic de selenoproteïnes (eEFsec), el qual apropa el tRNA de Sec al codó UGA del mRNA de les selenoproteïnes. El tRNA incorpora l’U a la cadena de aminoàcids sense que el ribosoma interpreti el UGA com a codó Stop. El tRNA continuarà fins que el ribosoma reconeixi un nou codó de terminació.
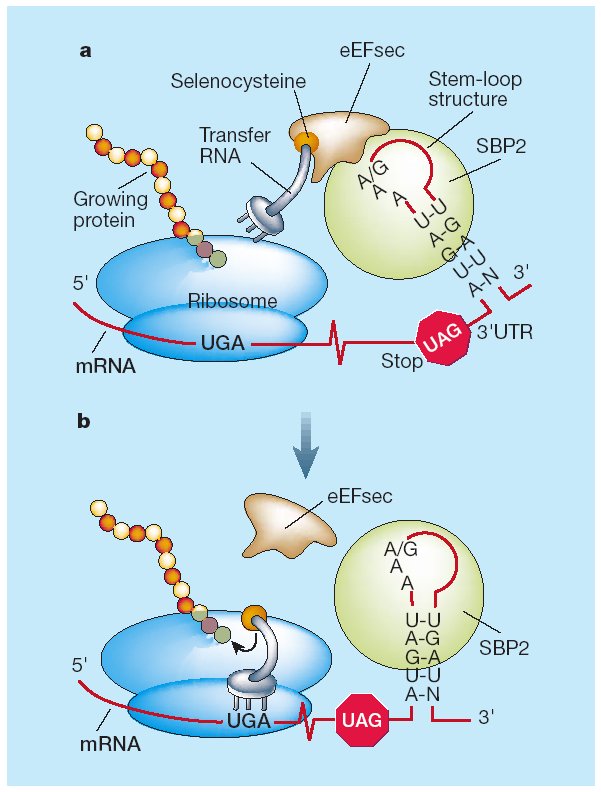
El tRNA Sec es sintetitza a partir de tRNA Ser (serina). En aquest procés hi participa: les selenofosfat sintetases SPS1 i SPS2, la selenocisteïna sintasa SLA/LP, la Phosphoseryl tRNA Kinase (PSTK) i la Selenocysteine associated protein Sec43p.
Identificació de les selenoproteïnes
Les selenoproteïnes són difícils de trobar en els genomes ja que per error un codó UGA es considera d’Stop sent en realitat, una selenocisteïna. Per poder identificar-les a nivell de gen hem de buscar un codó UGA i l’element SECIS a l’extrem 3’UTR.
Els elements SECIS mesuren unes 100-150 bases. Són molt diversos en quant a seqüència i no hi ha un patró regular. Entre tots ells només tenen conservats un quartet de nucleòtids, dos A’s propers a aquest i l’estructura 3D.
Una estratègia per trobar a aquests SECIS pot ser buscar les seqüències que es puguin plegar i formar aquesta estructura 3D coneguda (loop-helix-loop). PatScan és un dels diversos programes que poden fer aquesta cerca. Se li dóna el patró estructural i un conjunt se seqüències i el programa mostra quina/es d’aquestes pot adoptar aquesta estructura.
Història natural de les selenoproteïnes
Els selenoproteomes coneguts, ens ofereixen una visió fins a cert punt desconcertant de l’evolució d’aquestes proteïnes. Cada una d’elles sembla tenir la seva pròpia història evolutiva: poden ser presents en una determinada espècie o grup taxonòmic, absents en un altre grup filogenèticament proper, i en canvi tornar a ser presents (com a selenoproteïnes o homòlegs amb Cys) en un grup taxonòmic filogenèticament allunyat.
Cada nou genoma analitzat aporta noves sorpreses i fa que calgui repensar fets que es donaven gairebé per establerts. Per exemple, fins fa poc es pensava que les selenoproteïnes eren essencials per la vida animal, donat que tots els animals coneguts tenien selenoproteïnes i C. elegans manté tota la maquinària molecular necessària només per sintetitzar una selenoproteïna. Tanmateix, després de l’obtenció de la seqüència del genoma de dotze espècies de Drosophila, es va descobrir, mitjançant anàlisis computacionals, que D. willistoni no té selenoproteïnes i ha perdut la capacitat de codificar-ne. L’anàlisi d’un nombre de genomes d’insectes seqüenciats el darrer any ha permès comprovar que les selenoproteïnes s’han extingit en moltes altres espècies d’insectes tot i que n’hi ha d’altres que encara les mantenen.
Les selenoproteïnes en els genomes de protists
Entre els grups taxonòmics en el quals la distribució de selenoproteïnes és més diversa i, en conseqüència, més desconeguda, es troben els protists. Òbviament, això es deu en part, a la gran diversitat taxonòmica que caracteritza aquests organismes i a la gran distància filogenètica que els separa.
Les selenoproteïnes que hem estudiat en protists són: la família DI i Sel3.
La Iodotironina deiodinasa 1 (D1) és un enzim pertanyent a la família deiodinasa, que es troba bàsicament al fetge. Esdevé crucial en l'activació de les hormones tiroidees, ja que converteix la prohormona tiroxina (T4) en tri-iodetironina activa (T3); aquesta producció de T3 es creu que contribueix a la major part de T3 circulant en mamífers. La DI1 també degrada ambdues hormones per desiodació. La D1 no s’ha trobat ni en amfibis ni en peixos pulmonats.
La selenoproteïna Iodotironina deiodinasa 2 (DI2) és un dels membres de la família d'enzims que controlen el nivell d'activitat de les hormones tiroidees als diferents teixits. La DI2, igual que la DI 1, catalitza la desiodació de l'hormona tiroxina T4 per a l'obtenció de T3. La DI2 s'expressa únicament al cervell, tiroides i teixit adipós. S'ha observat que els nivells de T3 al cervell fetal estan controlats per l'activitat de DI2 i DI3. Així doncs, aquesta família de selenoproteïnes és molt important durant el desenvolupament embrionari del sistema nerviós.
La Iodotironina deiodinasa III (D3) catalitza la desiodació de T4 i T3 als metabòlits inactius rT3 i T2, respectivament. Aquest enzim està altament expressat en l’úter de femelles embarassades, placenta i teixits fetals i neonatals, suggerint que juga un paper essencial en la regulació de la inactivació de l’hormona tiroidea durant el desenvolupament embrionari. Tant D3 com D2 es creu que estan present en tots els organismes amb la possible excepció dels rèptils escamats.
Sel3 és una selenoproteïna d'aproximadament 351 aminoàcids que es troba en espècies del gènere Plasmodium. Va ser caracteritzada en Plasmodium falciparum, on està codificada en un sol exó. Sel3 s'expressa en diferents moments del cicle de vida de Plasmodium, amb el nivells d'expressió més alts en trofozoïts i schistozonts. Aquest fet suggereix que les selenoproteïnes poden tenir diferents funcions segons l'etapa del cicle vital en que es trobin. Sel3 té un residu de Selenocisteïna (Sec) a la part N-terminal, dins l'ORF.
Tornar a dalt
Protists
Els tres Dominis reconeguts en la natura viva són Eucariota, Arquea i Eubacteria (els dos últims també anomenats Arqueabacteria i Bacteria, respectivament). En els últims anys, els eucariotes s’han classificat en 4 regnes: animals, plantes, fongs i protists. Mentre que els tres primers regnes són grups monofilètics ben definits, el Regne Protist és parafilètic: conté organismes que es troben més relacionats amb els membres dels altres regnes que amb altres espècies de protists.

El Regne Protist comprèn tots aquells organismes que no pertanyen a cap altre regne, per això és el més divers dels regnes. Conté més de 200.000 espècies conegudes i encara en queden moltes per descobrir, a mode d’exemple hi pertanyen algues, floridures, paràsits (Plasmodium falciparum), protozous, etc. Entre ells no hi ha gaire en comú a part de ser organismes eucariotes unicel·lulars que existeixen com a cèl·lules independents o bé com a colònies, les quals no es s’especialitzen en teixits. Una altra característica comuna entre els organismes protists és que cap es troba adaptat plenament a la vida en presència d’aire, de manera que els que no són totalment aquàtics, habiten en ambients terrestres humits o a l’interior d’altres organismes.
La classificació dels protists ha estat modificada diversos cops en els últims 20 anys. Gràcies a les noves tècniques de comparació de seqüències entre espècies, s’ha eliminat el problema de l’escassetat o ambigüitat dels caràcters morfològics dels protists a l’hora d’establir la seva classificació. Per tant, ara és quan es comença a definir grups monofilètics de protists.
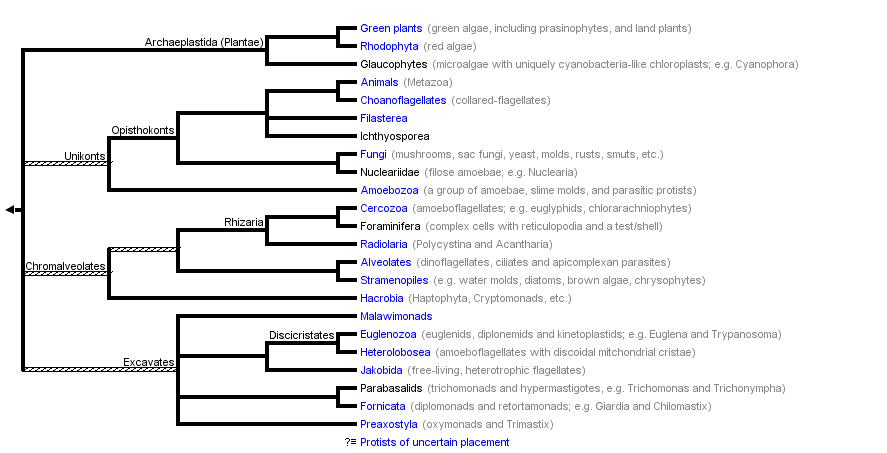
El nostre estudi consisteix en cercar els selenoproteomes dels protists dels quals s’ha seqüenciat el genoma aquest mateix any 2011. Els trobem marcats en blau a la imatge següent i descrits breument més avall.
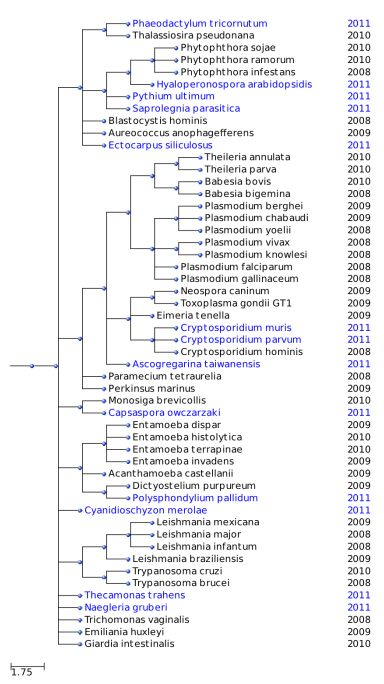
Feu click sobre la imatge per veure-la en gran.
Ascogregarina taiwanensis: Paràsit protozou que desenvolupa trofozoïts en les cèl·lules que revesteixen l’intestí mitjà de les larves de Aedes Albopictus (mosquit tigre).
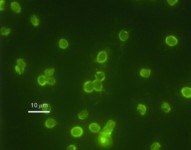
Blastocystis hominis: Protozou que causa quadres diarreics. Es transmet entre animals i humans per la ingestió de quists presents en aigües o aliments contaminants amb matèria fecal. Presenta una gran diversitat morfològica. Generalment són organismes de forma esfèrico-ovalada, incolors, hialins i refringents. La mida varia entre 5- 40 μm de diàmetre, amb una massa central granular rodejada per refringència amb un o dos núclis.
Cyanidioschyzon merolae: Alga vermella unicel·lular que viu en ambients molt àcids. Aquesta alga té un únic cloroplast i una única mitocòndria, però careix de vacúol i paret cel·lular. El seu genoma va ser el primer genoma complet d’alga que es va seqüenciar el 2004. És una de les algues vermelles més primitives.
Cryptosporidium muris i C. parvum: Protists paràsits al que se li associa una malaltia anomenada criptosporidiosis diarreica en éssers humans. El paràsit es transmet en el medi ambient mitjançant quists resistents que, un cop ingerits es desenquisten en l’intestí prim i donen lloc a la infecció dels teixits epitelials intestinals. .

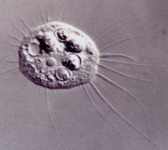
Capsaspora owczarzaki: Paràsit eucariota del cargol Biomphalaria glabrata, que actua com a hoste intermediari de Schistosoma, un platelmint causant de la esquistosomiasis, una malaltia mortal que afecta a més de 200milions de persones al mon. El coneixement de la seqüència del genoma de Capsaspora proporciona una visió de la coevolució hoste-paràsit. També podria ser utilitzat per desenvolupar estratègies en el control d’aquest paràsit humà devastador.
Ectocarpus siliculosus: Alga marró filamentosa sense branques, que es situa sobre plantes o altres substrats formant barbes, les quals poden créixer fins a mig metre. El seu genoma ha estat el primer de macroalga marró en ser seqüenciat, amb l’esperança que serveixi de model d’aquest tipus d’organismes.
Hyaloperonospora arabidopsidis: Espècie de protista pertanyent a la familia Peronosporaceae. és un paràsit obligat de la planta Arabidopsis thaliana , amb una molt alta especialització per a aquesta espècie hoste.

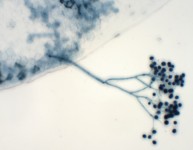
Naegleria gruberi: Ameba flagelada no patògena que destaca per la seva capacitat de canviar de la forma ameboide a la flagelada. En condicions normals, N.gruberi creix i es divideix com una amoeba, però en condicions de falta de nutrients N.gruberi esdevé un flagelat, tot desenvolupant un esquelet de microtúbuls. No obstant, aquest estadi flagelat és transitori. El genoma de N.gruberi és clau a l'hora d’estudiar processos evolutius d’ameba.
Polysphondylium pallidum: Llim cel·lular que té forma d'arbre ramificat a diferència d'altres llims que tenen una morfologia no ramificada. Principalment viu en el terra dels boscos o en els fems. La seva distribució és força àmplia i es pot trobar a Àfrica, a América, al Canadà i al Japó. La seva mitosis és un procés molt estudiat.
Phaeodactylum tricornutum: És una de les dues diatomees en tenir el seu genoma seqüenciat, l’altra és Thalassiosira pseudonana. A diferència d’altres diatomees, P.tricornutum existeix en diferents morfologies (fusiforme, radiat i oval), i els canvis en la forma de la cèl·lula poden ser estimulats per les condicions ambientals. Aquesta característica pot ser utilitzada per a la investigació en les bases moleculars de la morfogènesi i el seu control. A més, P. tricornutum genera frustules silicades, fet que proporciona l’oportunitat d’estudiar el procés de nanofabricació basada en silici en diatomees.
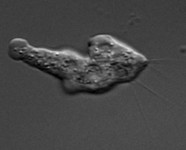

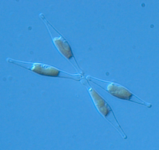
Pythium ultimum: Es troba en el sòl i té una amplia distribució terriorial així com també un rang d’hostes molt extens. Juntament amb els seus parents propers Phytophthora, són patògens de plantes d’importància econòmica per a l’agricultura mundial. P.ultimum ocasiona el podriment de les arrels de les plantes, provocant la seva mort.
Saprolegnia parasitica: Són floridures d’aigua que actuen de paràsits sobre una àmplia varietat de peixos (com ara el salmó), amfibis i crustacis, rellevants en aqüicultura i en ecosistemes marins. Causen la malaltia anomenada saprolegniosi , la qual és considerada una molèstia per a les empreses salmoneres. La seva importàcia és tal que s’ha seqüenciat tot el seu genoma, inclòs el ADN mitocondrial.
Thecamonas trahens: Protist eucariota unicel·lular, biflagelat i extès territorialment, tot i que es troba principalment en ambients marins, també en aigua dolça i el sòl. Va ser triat per a la seqüenciació del genoma. Creix vigorosament a altes densitats, sense deixar grans quantitats d’aliment bactèria que pugui interferir en l’experimentació bioquímica.