TblastN
El TblastN obtingut de llençar diferents GPx de diferents organismes contra el genoma de P. marinus ha donat el següent resultat:

Clica la imatge per veure-la gran
Hem seleccionat el contig gi|126302502|gb|AAXJ01000003.1| per seguir amb l'estudi de GPx ja que és el que té un millor e-value i a més alinea amb totes les GPxs i organismes, exceptuant la GPx5 de C. Elegans. Per veure l'alineament prem aquí.
Predicció de GPx en P. marinus
Per predir les GPx en P. marinus s’han seleccionat les GPx que tenen un millor e-value, que són la GPx1 humana, la murina, la de ximpanzé i la de C. elegans, la GPx2 humana i murina; la GPx3 de Saccharomyces cerevisiae, i la GPx4 de Drosophila melanogaster.
La predicció exònica l'hem realitzat amb el programa Exonerate, i no ha donat cap resultat.
La predicció peptídica i del cDNA realitzada amb el programa Genewise, ha donat les següents prediccions per cada GPx analitzada:
GPx1 Homo sapiens

Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx1 Mus musculus

Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx1 Pan troglodytes

Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx1 Caenorhabditis elegans

Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx2 Homo sapiens
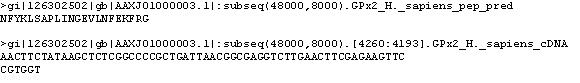
Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx2 Mus musculus
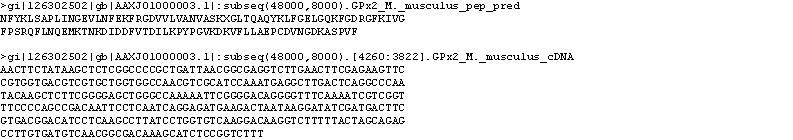
Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx2 Saccharomyces cerevisiae
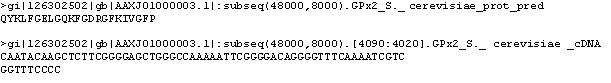
Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx3 Saccharomyces cerevisiae
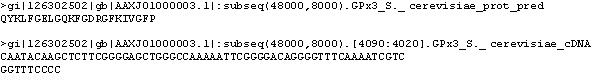
Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
GPx4 Drosophila melanogaster

Clica la imatge per veure-la gran
Per veure el resultat complet del programa Genewise prem aquí
Alineament del GPx predit
T-coffee L'alineament amb el programa T-coffee de les proteïnes predites amb les proteïnes de partida, dónen els següents resultats:
GPx1 Homo sapiens
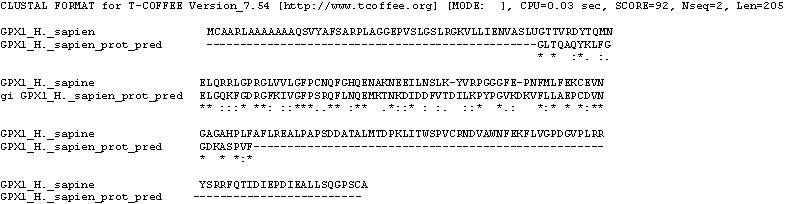
Clica la imatge per veure-la gran
GPx1 Mus musculus

Clica la imatge per veure-la gran
GPx1 Pan troglodytes

Clica la imatge per veure-la gran
GPx1 Caenorhabditis elegans

Clica la imatge per veure-la gran
GPx2 Homo sapiens

Clica la imatge per veure-la gran
GPx2 Mus musculus
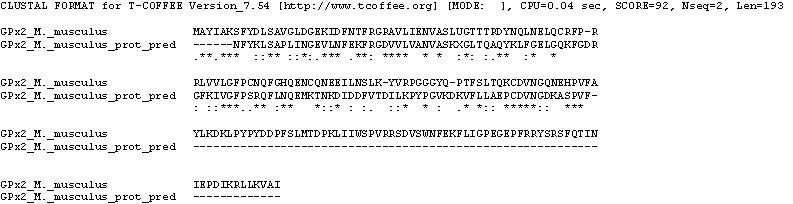
Clica la imatge per veure-la gran
GPx2 Saccharomyces cerevisiae

Clica la imatge per veure-la gran
GPx3 Saccharomyces cerevisiae

Clica la imatge per veure-la gran
GPx4 Drosophila melanogaster

Clica la imatge per veure-la gran
Blastcl3 nr El resultat del blastcl3 per les GPxs predites dóna els següents resultats:
GPx1 Homo sapiens
GPx1 Mus musculus
GPx1 Pan troglodytes
GPx1 Caenorhabditis elegans
GPx2 Homo sapiens
GPx2 Mus musculus
GPx2 Saccharomyces cerevisiae
GPx3 Saccharomyces cerevisiae
GPx4 Drosophila melanogaster
Cerca d'elements SECIS
Al analitzar la seqüència dels 1000 nucleotids posteriors a l'alineament del contig gi|126302502|gb|AAXJ01000003.1|, amb el que totes les GPx s’alineen, exceptuant la GPx5 de Caenorhabditis elegans, amb el programa SECISearch, s'obté el següent element SECIS tipus 1, amb un score de 23.93. Per veure la seqüència clica aquí
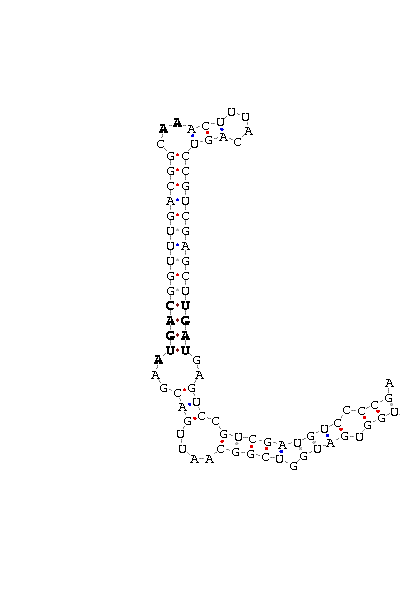
Clica la imatge per veure-la gran
Arbre filogenètic
Utilitzant l'anàlisi filogenètic de la web MABL, s'ha construit el següent arbre filogenètic:

Clica la imatge per veure-la gran

