 |
ACANTHAMOEBA CASTELLANII |
 |
TR (Thioredoxin reductase)
TR
Función: enzima implicada en reaciones de óxido-reducción
Resultados
TR1 (Homo sapiens)
Tblastn
En el Contig15518_Contig14235_Contig10107_Contig18526 se alinea la U:
Genewise
Predicción de la proteína homóloga a TR1 de Homo sapiens en el Contig15518_Contig14235_Contig10107_Contig18526
PEDHTYDYDLIVIGGGSGGLAAAKEAGRLGKKVALLDFVVPTPTGTTWGLGGTCVNVGCIPKKLMHQAALLGESLKDA
QHYGWAVPDNVNHDWEKMVNAVQDHIGSLNWGYRVALREKNVNYLNAYGVFVDSHTLECTDRAKKVTRVTARRF
LVATGGRPKYPDIPGDREFGITSDDFFSLPTPPGKTLVVGASYVALECAGFVRGLGYDTTVMVRSILLRGFDQQLANMIG
QYMECHGIKFVRSAVPTKVEKLESGKLRVTFQQDGVEGVEEYDTVMWAIGREAETKKIGLDKAGVQVDRIGKIHTVME
RTNVPHIYAIGDIIDEPSQRSLELTPVAIKAGILLVRRLYAGSTQPMDYINVPTTVFTPIEYGAIGYSEEDAIAQFGEDNLEIY
HSYFKPLEWTIAERDDNVCYAKLICDKRDSERVVGFHVLGPNAGEITQGFGTAMKAGATKSTFDATVGIHPTTAEEFTT
LEVTKRSGVEAQKKGC
Menú TR
TR2 (Homo sapiens)
Tblastn
En el Contig15518_Contig14235_Contig10107_Contig18526 se alinea la U:
Genewise
Predicción de la proteína homóloga a TR2 de Homo sapiens en el Contig15518_Contig14235_Contig10107_Contig18526DYDLIVIGGGSGGLAAAKEAGRLGKKVALLDFVVPTPTGTTWGLGGTCVNVGCIPKKLMHQAALLGESLKDAQHYGWA
VPDNVNHDWEKMVNAVQDHIGSLNWGYRVALREKNVNYLNAYGVFVDSHTLECTDRAKKVTRVTARRFLVATGG
RPKYPDIPGDREFGITSDDFFSLPTPPGKTLVVGASYVALECAGFVRGLGYDTTVMVRSILLRGFDQQLANMIGQYMECH
GIKFVRSAVPTKVEKLESGKLRVTFQQDGVEGVEEYDTVMWAIGREAETKKIGLDKAGVQVDRIGKIHTVMERTNVPH
IYAIGDIIDEPSQRSLELTPVAIKAGILLVRRLYAGSTQPMDYINVPTTVFTPIEYGAIGYSEEDAIAQFGEDNLEIYHSYFKP
LEWTIAERDDNVCYAKLICDKRDSERVVGFHVLGPNAGEITQGFGTAMKAGATKSTFDATVGIHPTTAEEFTTLEVTKR
SGVEAQKKGC
Menú TR
TR3 (Homo sapiens)
Tblastn
En el Contig15518_Contig14235_Contig10107_Contig18526 se alinea la U:
Genewise
Predicción de la proteína homóloga a TR3 de Homo sapiens en el Contig15518_Contig14235_Contig10107_Contig18526EDHTYDYDLIVIGGGSGGLAAAKEAGRLGKKVALLDFVVPTPTGTTWGLGGTCVNVGCIPKKLMHQAALLGESLKDAQ
HYGWAVPDNVNHDWEKMVNAVQDHIGSLNWGYRVALREKNVNYLNAYGVFVDSHTLECTDRAKKVTRVTARRFL
VATGGRPKYPDIPGDREFGITSDDFFSLPTPPGKTLVVGASYVALECAGFVRGLGYDTTVMVRSILLRGFDQQLANMIGQ
YMECHGIKFVRSAVPTKVEKLESGKLRVTFQQDGVEGVYDTVMWAIGREAETKKIGLDKAGVQVDRIGKIHTVMERTN
VPHIYAIGDIIVDEPSQRSLELTPVAIKAGILLVRRLYAGSTQPMDYINVPTTVFTPIEYGAIGYSEEDAIAQFGEDNLEIYHS
YFKPLEWTIAERDDNVCYAKLICDKRDSERVVGFHVLGPNAGEITQGFGTAMKAGATKSTFDATVGIHPTTAEEFTTLEV
TKRSGVEAQKKGC
Menú TR
TR1 (Drosophila melanogaster)
Tblastn
Genewise
Predicción de la proteína homóloga a TR1 de Drosophila melanogaster en el Contig15518_Contig14235_Contig10107_Contig18526YDYDLIVIGGGSGGLAAAKEAGRLGKKVALLDFVVPTPTGTTWGLGGTCVNVGCIPKKLMHQAALLGESLKDAQHYGW
AVPDNVNHDWEKMVNAVQDHIGSLNWGYRVALREKNVNYLNAYGVFVDSHTLECTDRAKKVTRVTARRFLVATG
GRPKYPDIPGDREFGITSDDFFSLPTPPGKTLVVGASYVALECAGFVRGLGYDTTVMVRSILLRGFDQQLANMIGQYMEC
HGIKFVRSAVPTKVEKLESGKLRVTFQQDGVEGVEEYDTVMWAIGREAETKKIGLDKAGVQVDRIGKIHTVMERTNVP
HIYAIGDIIVDEPSQRSLELTPVAIKAGILLVRRLYAGSTQPMDYINVPTTVFTPIEYGAIGYSEEDAIAQFGEDNLEIYHSYF
KPLEWTIAERDDNVCYAKLICDKRDSERVVGFHVLGPNAGEITQGFGTAMKAGATKSTFDATVGIHPTTAEEFTTLEVTK
RSGVEAQKKGC
Menú TR
TR2 (Drosophila melanogaster)
Tblastn
Genewise
Predicción de la proteína homóloga a TR2 de Drosophila melanogaster en el Contig15518_Contig14235_Contig10107_Contig18526TYDYDLIVIGGGSGGLAAAKEAGRLGKKVALLDFVVPTPTGTTWGLGGTCVNVGCIPKKLMHQAALLGESLKDAQHYG
WAVPDNVNHDWEKMVNAVQDHIGSLNWGYRVALREKNVNYLNAYGVFVDSHTLECTDRAKKVTRVTARRFLVAT
GGRPKYPDIPGDREFGITSDDFFSLPTPPGKTLVVGASYVALECAGFVRGLGYDTTVMVRSILLRGFDQQLANMIGQYME
CHGIKFVRSAVPTKVEKLESGKLRVTFQQDGVEGVEEYDTVMWAIGREAETKKIGLDKAGVQVDRIGKIHTVMERTNV
PHIYAIGDIIVDEPSQRSLELTPVAIKAGILLVRRLYAGSTQPMDYINVPTTVFTPIEYGAIGYSEEDAIAQFGEDNLEIYHSY
FKPLEWTIAERDDNVCYAKLICDKRDSERVVGFHVLGPNAGEITQGFGTAMKAGATKSTFDATVGIHPTTAEEFTTLEVT
KRSGVEAQKKGC
Menú TR
Protein blast
Solo se hacen los protein BLAST del péptido más largo, puesto que los hits en exactamente la misma región ·en el mismo Contig· no son más que una sola y única proteína (más información en el apartado de Discusión)
TR1 contra Homo sapiens
TR1 contra todos los organismos
Discusión
En las tres proteínas TR presentes en Homo sapiens los resultados son similares, pues el tBLASTn alinea la selenocisteína de la proteína de Homo sapiens en el hit con e-value menor, es decir, más significativo.
En GeneWise obtenemos unas secuencias proteicas muy parecidas a las humanas (resultado), todas ellas en realidad reflejando una sola proteína de Acanthamoeba castellanii. A pesar de la alta calidad del alineamiento, la predicción de la proteína a partir de la secuencia genómica se detiene al llegar al codón TGA que corresponde a la selenocisteína, y por tanto no obtenemos la proteína entera. Para resolverlo se cambió el codón stop por otro que codifica para una cisteína (TGA ' TGC) y se volvió a realizar el t_coffee.
En realidad, como se ha dicho, lo que observamos es que Acanthamoeba castellaniii presenta una única selenoproteína TR que presenta homología con las tres TRs de Homo sapiens, ya que la secuencia proteica de TR1, TR2 y TR3 no son muy variables entre ellas. Una hipótesis razonable que encajaría con este escenario sería que la proteína ancestral de las TR se triplicó en Homo sapiens dando lugar a los tres miembros de la familia; en Acanthamoeba castellaniii, por el contrario, en base a lo que se ha podido observar, no se dio ningún suceso similar.
Al realizar el protein BLAST se observan correspondencias significativas con las tres clases de "thioredoxin reductase", independientemente de cual sea la secuencia proteica obtenida de Acanthamoeba castellanii que se utilice.
Para corroborar estos resultados realizamos un t_coffee que nos muestra la homología entre las tres TRs de Homo sapiens y la potencial de Acanthamoeba castellanii.
Una vez comprobado que realmente hay una proteína homóloga en el genoma, se buscó su elemento SECIS downstream. Se encontró uno con un COVE score significativo de 28'27:
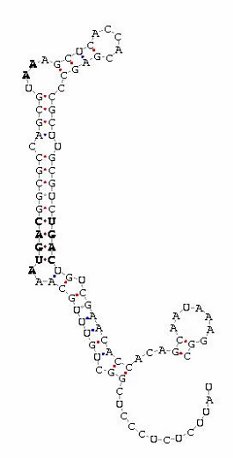
Para finalizar, se buscó, mediante un t_coffee de elementos SECIS, cual de las TR de las distintas familias existentes (TR1, TR2 y TR3) tenía una mayor homología con la TR presente en el genoma de Acanthamoeba castellanii.
Este múltiple alineamiento nos sirvió para realizar un árbol filogenético con ClustalW, donde vemos que la TR de Acanthamoeba castellaniii se encuentra más próxima a TR1:
A pesar de todo, lo más probable es que, si la hipótesis sobre la triplicación de TR en Homo sapiens posterior a la especiación respecto a Acanthamoeba castellanii - y la conservación de una única copia en el genoma de esta última - es cierta, el árbol realizado no proporcione ninguna información consistente.
En definitiva, podemos concluir que hay una TR en el genoma de Acanthamoeba castellanii.