| Introducció | Objectius | Metodologia | Programes | Resultats | Conclusions | Referències | Autores | Agraïments |
RESULTATS
L'anàlisi exhaustiu dels resultats que aquí es detallen es troba en l'apartat de Conclusions. |
||||||||||||||||||||||||||||||
Anàlisi de la complexitat algorísmica i del cost computacional de l'algorisme simple correspondència exacta versus l'algorisme de Knuth-Morris-Pratt Per tal de determinar el guany computacional de l'algorisme de Knuth-Morris-Pratt respecte l'algorisme simple de correspondència exacta, s'ha implementat dos programes per a cadascun d'ells, la funció dels quals és generar tots les possibles patrons d'un mRNA concret i fer la comprovació de si eren únics o no en aquest mateix mRNA, mitjançant l'algorisme corresponent.
|
||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||
Reproducció dels resultats del mRNA de ratolí a partir de l'algorisme de Knuth-Morris-Pratt Per corroborar els resultats obtinguts pel Dr. Robert Castelo, del gen de la EPO de Mus musculus, hem implementat el nostre programa de cerca de subseqüències úniques (explicat detalladament a Programes) basat en l'algorisme de Knuth-Morris-Pratt, però únicament en el cromosoma 5 de ratolí per estalviar temps, ja que el gen de la EPO es troba en aquest.
Els resultats obtinguts són que efectivament les següents subseqüències són úniques:
|
||||||||||||||||||||||||||||||
CGACAGTCGAGTTC
AGTGGTCTACGTAG
GGGTCTACGCCAAC
|
||||||||||||||||||||||||||||||
A més a més, el programa també informa del desplaçament de les mateixes en el cromosoma, un fet important a l'hora d'usar aquests resultats per a la seva aplicació en relació amb el dopatge genètic, sobretot en aquells casos en què les subseqüències trobades pertanyin a dos exons diferents separats per algun intró. El desplaçament trobat per a cadascuna d'aquestes subseqüències és el següent: |
||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||
Com que el nostre programa té una estratègia automàtica de moure la llargada (per trobar les subseqüències úniques mínimes) i sols ho comprovem en el cromosoma 5, aconseguim trobar també subseqüències inferiors a 14 nucleòtids per a la primera i la tercera subseqüències, que si féssim la comprovació per tot el genoma de ratolí, ja no serien úniques.
Per a la primera subseqüència el programa ens troba una subseqüència de 12 nucleòtids: CGACAGTCGAGT, amb un desplaçament de 136.403.348. I per a la tercera subseqüència trobem una subseqüència d'11 nucleòtids: GGGTCTACGCC, amb un desplaçament de 136.401.415. Si es compara el desplaçament d'aquestes subseqüències més curtes amb el desplaçament de les subseqüències de les que provenien, no concorda la posició inicial. Això és perquè totes les subseqüències s'han trobat a l'strand negatiu. Tenint en compte aquest fet el desplaçament ja concorda.
Si es volen consultar els outputs d'aquesta cerca, clicar sobre cada un d'ells:
|
||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||
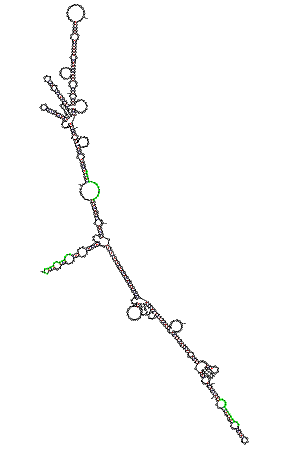
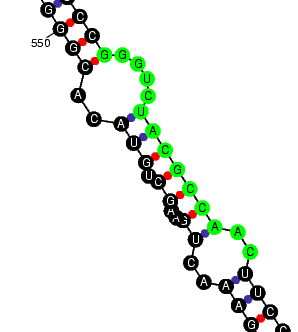
Predicció de l'estructura secundària del mRNA de la EPO Per determinar si les subseqüències úniques trobades es localitzaven en un regió exposada de l'estructura secundària del mRNA de l'EPO de ratolí hem utilitzat el programa mFOLD. Aquest programa fa prediccions de l'estructura secundària d'una seqüència de mRNA, així com també d'una de DNA. Els resultats obtinguts són els següents:
|
||||||||||||||||||||||||||||||
A continuació es poden observar amb més detall les tres subseqüències úniques trobades al gen de la EPO de ratolí. |
||||||||||||||||||||||||||||||