| Introducción | Objetivos | Metodología | Programas | Resultados | Conclusiones | Referencias | Autoras | Agradecimientos |
RESULTADOS
El análisis exhaustivo de los resultados que aquí se detallan se encuentran en el apartado de Conclusiones. |
||||||||||||||||||||||||||||||
Análisis de la complejidad algorítmica y del coste computacional del algoritmo simple de correspondencia exacta versus el algoritmo de Knuth-Morris-Pratt Para determinar la ganancia computacional del algoritmo de Knuth-Morris-Pratt respecto el algoritmo simple de correspondencia exacta, se han implementado dos programas para cadauno de ellos, la función de los cuales es generar todos los patrones posibles de un mRNA concreto y hacer la comprobación de que fuesen únicos o no en este mismo mRNA, mendiante el algoritmo correspondiente.
|
||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||
Reproducción de los resultados del mRNA de ratón a partir del algoritmo de Knuth-Morris-Pratt Para corroborar los resultados obtenidos por el Dr. Robert Castelo, del gen de la EPO de Mus musculus, hemos implementado nuestro programa de búsqueda de subsecuencias únicas (explicado detalladamente en Programas) basado en el algoritmo de Knuth-Morris-Pratt, pero únicamente en el cromosoma 5 de ratón para ahorrar teimpo, ya que el gen de la EPO se encuentra en éste.
Los resultados obtenido son que efectivamente las siguientes subsecuencias son únicas:
|
||||||||||||||||||||||||||||||
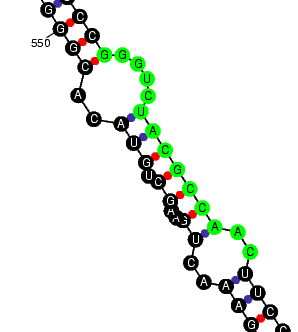
CGACAGTCGAGTTC
AGTGGTCTACGTAG
GGGTCTACGCCAAC
|
||||||||||||||||||||||||||||||
Además, el programa también informa del desplazamiento de las mismas en el cromosoma, un hecho importante a la hora de utilizar estos resultados para su aplicación en relación al dopaje genético, sobretodo en aquellos casos en que las subsecuencias halladas pertenecen a dos exones diferentes separados por algún intrón. El desplazamiento hallado para cada una de estas subsecuencias es el siguiente: |
||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||
Como nuestro programa tiene un estrategia automática de mover la longitud (para encontrar las subsecuencias únicas mínimas) y sólo lo comprobamos en el cromosoma 5, consiguiendo encontrar también subsecuenicas inferiores a 14 nucleótidos para la primera y tercera subsecuencias, que si hiciéramos la comprobación por todo el genoma de ratón, ya no serían únicas. |
||||||||||||||||||||||||||||||
Para la primera subsecuencia el programa encuentra una subsecuencia de 12 nucleótidos: CGACAGTCGAGT, con un desplazamiento de 136.403.348. Y para la tercera subsecuencia encontramos una subsecuencia de 11 nucleótidos: GGGTCTACGCC, con un desplazamiento de 136.401.415. Si se compara el desplazamiento de estas subsecuencias más cortas con el desplaamiento de las subsecuencias de las que provenían, no concuerda la posición inicial. Ésto es así porque todas las subsecuencias se han encontrado en el strand negativo. Teniendo en cuenta este hecho el desplazamiento ya concuerda.
Si se quieren consultar los outputs de esta búsqueda, clicar sobre cada uno de ellos:
|
||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||
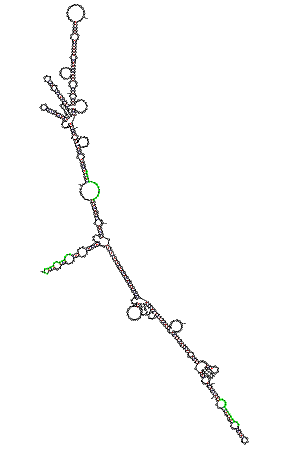
Predicción de la estructura secundaria del mRNA de la EPO Para determinar si las subsecuencias únicas encontradas se localizan en una región expuesta de la estructura secundaria del mRNA de la EPO de ratón hemos utilizado el programa mFOLD. Este programa hace predicciones de la estructura secundaria de una secuencia de mRNA, así como también de una de DNA. Los resultados obtenidos son los siguientes:
|
||||||||||||||||||||||||||||||
A continuación se pueden observar con más detalle las tres subsecuencias únicas encontradas en el gen de la EPO de ratón. |
||||||||||||||||||||||||||||||