Obtenció de la seqüència

Fitxer apocluster.fa

Fitxer apocluster.fa
Llargada de la seqüència = 500.000 pb.
Freqüència dels diferents nucleòtids:
Els resultats són cinc fitxers (per apo1 i per apo2 cinc més):
| apo1.seq.stderr | Progress report |
| apo1.seq.out | Annotation of masked sequence |
| apo1.seq.cat | Crossmatch information |
| apo1.seq.tbl | Summary of the repeat content |
| apo1.seq.masked | Masked Sequence |
| apo2.seq.stderr | Progress report |
| apo2.seq.out | Annotation of masked sequence |
| apo2.seq.cat | Crossmatch information |
| apo2.seq.tbl | Summary of the repeat content |
| apo2.seq.masked | Masked Sequence |
Per treure informació sobre les regions repetitives haurem de treballar amb el fitxer repeat.seq.tbl, el qual ens diu que:
Els fitxer obtinguts sobre l'emmascarament de la seqüència ens indiquen que aproximadament cada subseqüència (apo1 i apo2) conté un 40% d'elements repetitius. Aquest es troben repartits per tota la seqüència, fet que ens indica que aquesta no forma part ni d'un telòmer ni d'un centròmer.
En el següent gràfic podem observar com es troben distribuïts els diferents elements repetitius al llarg de la nostra seqüència. A més a més, també podem observar com els elements més abundants, tant per apo1 com per apo2,són els Sine/Alu i els Line/L1.
Gràfic: apo1.seq.out.ps
Si ens fixem amb els resultats obtinguts tant en el gràfic per la subseqüència apo1 com per la apo2,podem observar com les prediccions obtingudes pels tres programes varien una mica l'una de l'altre, les diferències les trobem principalment en el nombre de gens i exons. Pel que fa a apo1,podem observar com el fgenesh prediu 8 gens, el Genscan 7 i el Geneid 9. Pel que fa a apo2, fgenesh prediu 5 gens, Genscan 5 i Geneid 4. Aquesta diferència ve determinada pel fet que en algun programa es prediu un únic gen, mentre en els altres aquest es troba dividit en dos.
Fitxers obtinguts després de canviar el format del fitxer d'elements repetitius a una sola línia.
Agrupament elements repetitius i predicció de gens:
Validació amb ESTs humans:
En els següents gràfics podem observar el resultat del megablast, que ens mostra el tipus i la distribució de tots els ESTs d'humans que trobem en la seqüència apo1 i apo2.
Spliced ESTs:
Spliced ESTs i predicció de gens:
En aquests gràfics podem observar els spliced ESTs i les prediccions de gens realitzades pels tres programes agrupades, d'aquesta manera podrem determinar quines estructures gèniques estan més suportades pels spliced ESTs. El fet de trobar el mateix EST alineat més d'una vegada en la seqüència, és un fort indicador de que aquella regió és una zona d'splicing i que per tant és molt probable l'existència d'un gen. Tenint en compte aquest fet i observant les gràfiques obtingudes, hem determinar aquells gens que tenen més suport i dels quals analitzarem les proteïnes predites.
Mirant el gràfic hem considerat que els gens que estaven millor suportats per ESTs eren, per apo1 el gen 4 i 5 de Geneid i per apo2 el gen 4 de Geneid i el 2 de Genscan.
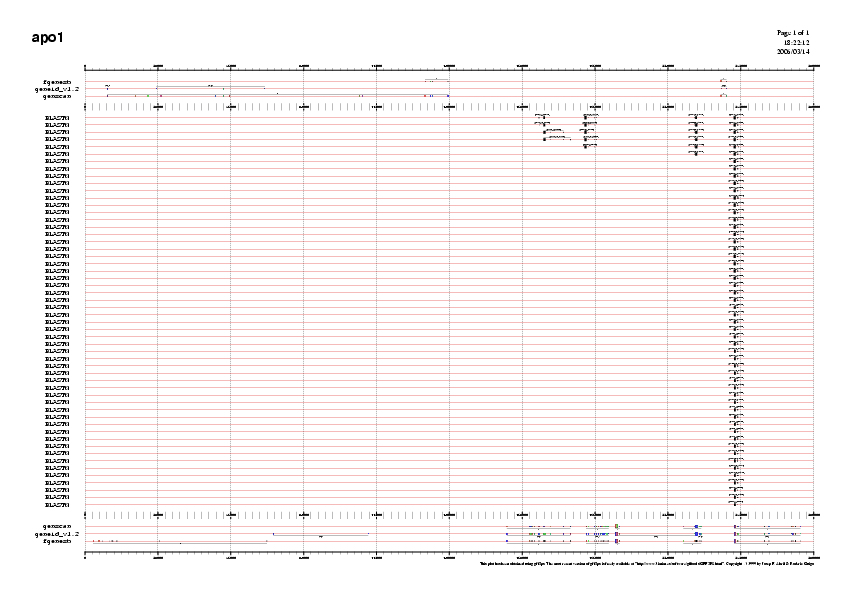
Resultat per apo2: apo2.pspliced+predictions+noframe.ps
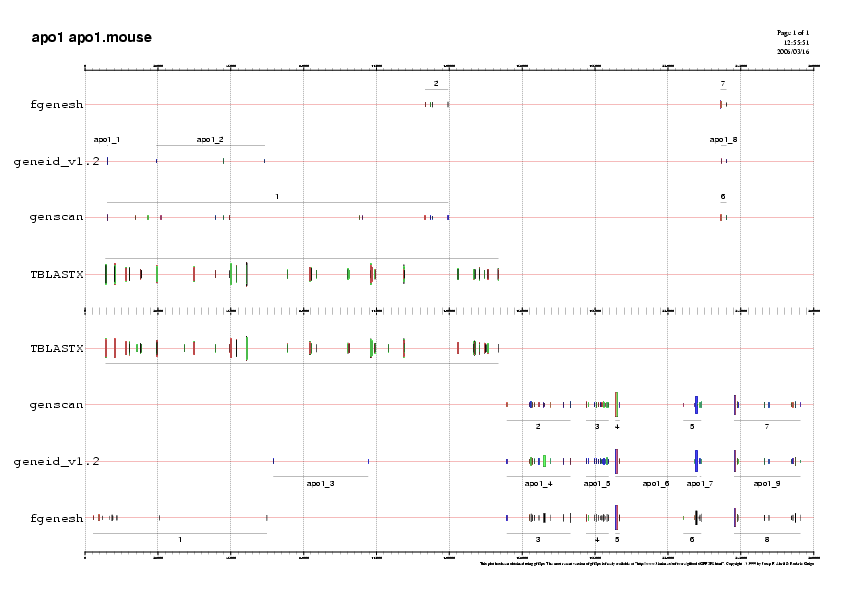
Validació amb TBLASTX:
Seqüències que utilitzem per fer el TBLastX (les utilitzades com a base de dades han estat obtingudes gràcies al Blat):
En els dos gràfics resultants podem observar el resultat del tblastx agrupat amb les prediccions de gens. Si observem els gràfics podem veure com no hi ha validació de gens. Per tant no afegirem cap més gen per l'anàlisi de proteïnes.

Resultats BlastP apo1:

Llargada de la query= 401
El millor aliniament ha sigut per:
Zinc-finger protein ZPR1 (Zinc finger protein 259)
Length= 459 Score = 776 bits (2004), Expect = 0.0 Identities = 401/401 (100%)
Resultats BlastP apo2:
Llargada de la query= 1391
El millor aliniament ha sigut per:
Apolipoprotein A-I precursor (Apo-AI) (ApoA-I) [Contains: Apolipoprotein A-I(1-242)]
Length= 267 Score = 473 bits (1217), Expect = 2e-133 Identities = 253/253 (100%)
Serine/threonine-protein kinase SNF1-like kinase 2 (Qin-induced kinase)
Length=926 Score = 385 bits (988), Expect = 8e-107 Identities = 200/360 (55%), Positives = 251/360 (69%)
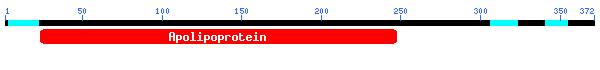
Llargada de la query= 372
El millor aliniament ha sigut per:
Apolipoprotein A-IV precursor (Apo-AIV) (ApoA-IV)
Length= 396 Score = 395 bits (1016) Expect = 1e-110 Identities = 206/235 (87%)
Resultats de l'INTERPRO per apo1:
Es tracta d'una proteïna no caracteritzada.
Es tracta d'un zinc finger: zf-ZPR1 amb dos dominis amb els següents E-values: 1,4e-86 i 4,4e-93
Resultats de l'INTERPRO per apo2:
Apolipoprotein amb E-value de 5,3e-167.
Proteïna Serina/Treonina Kinasa amb E-value de 6,9e-84
Apolipoprotein amb un E-value de 2,2e-141
ClustalW (per la proteïna 4 desconeguda).
En cap dels tres alineaments realitzats hem pogut observar una clara similitud entre les tres seqüències, per tant no podem definir la funció de la proteïna.
| Inici | Discussió |