
| INICI ~ OBJECTIU ~ INTRODUCCIÓ ~ MATERIALS ~ MÈTODES ~ EXPLICACIÓ DETALLADA DEL SCRIPT ~ RESULTATS ~ CONCLUSIÓ ~ REFERÈNCIES ~ AUTORES ~ |
RESULTATS
 | RESULTATS |
1.Què són els ESTs?
Els ESTs (Expressed Sequence Tag) són petites seqüències de DNA (normalment d'uns 200 a 500 nucleòtids) que són generats a partir de la seqüenciació d'un o dels dos extrems del gen expressat.
A partir del cDNA es pot seqüenciar uns quants centenars de nucleòtids per crear dos tipus diferents d' ESTs.
Si només es seqüencia la porció del començament del cDNA creem els 5' ESTs, que normalment són una porció del trancrit que codifica per una proteïna. Aquestes regions tendeixen a estar conservades entre les diferents espècies.
Si, en canvi, seqüenciem la porció del final del cDNA produïm els 3' EST. Aquests són generats a partir de la porció 3' del transcrit i probablement cauran a una regió no-codificant, o a una regió d'UTRs (regió no traduïda), i per tant, tendiran a estar menys conservats entre les diferents espècies.
A més a més, els 3' ESTs són una font comú d'STSs ("Sequence Tagged Site", seqüències curtes de DNA fàcilment reconegudes) per la seva probabilitat de ser únics per una espècie en particular i perquè ens donen informació addicional de què es pot tractar d'un gen expressat.
La seqüenciació amb ESTS és molt ràpida i barata. Els seus resultats ens poden donar informació sobre gens coneguts, similaritats amb gens coneguts, contaminació o bé el descobriment de gens novells.
Per a més informació sobre els ESTs feu click en aquest link.

2. Validació de la predicció de gens
A partir dels 23 gens possibles que ens van sortir ( script ) per veure si realment aquests eren o no possibles gens novells, vam anar a la pagina de UCSC Genome Browser on Human July 2003 Freeze. , on a la finestra de "position" vam posar l'inici i el final de la transcripció de cadascun dels 23 cromosomes per veure si només es trobaven dins de geneid o genscan, o bé també es podien veure a Ensembl i/o RefSeq ( annex2 )
Un cop realitzada aquesta operació ens vam quedar amb 7 possibles canditats a gens novells. A aquests els hi vam fer un BLAST amb els paràmetres següents:
MEGABLAST : choose database = est_human
que els vam anomenar així: blastchr_.txt ( script )Options: select from Homo Sapiens [ORGN] Format:Plain text Word Size:16 Number of: Descriptions: 1000 and Aligments: 1000
Aligment View: Hit Table
Un cop realitzades les transformacions adients ( script ) vam arribar a les anotacions dels gens predits com possibles gens novells.
A causa que molts ESTs només soporten un sol exó seleccionarem aquells que es repeteixin més d'un cop.Ho farem mitjançant les següents comandes:
gawk '{if($1 >='2'){print $0}}' genchr2_2065.1.txt > egrepgenchr2_2065.1.txt cut -f2 egrepgenchr2_2065.1.txt > 2egrepgenchr2_2065.1.txt Amb la primera comanda seleccionem només aquelles anotacions que estan repetides 1 o més vegades.
Amb la segonda comanda fem que no ens surti el número que indica les repeticions (uniq -c) dins el fitxer.
| NÚMERO DE CROMOSOMA | IDENTIFICACIÓ | ANOTACIONS | FITXER |
| chr2 | chr2_2065.1. | 2egrepgenchr2_2065.1.txt | genchr2_2065.1.txt |
| chr5 | chr5_1143.1. | 2egrepgenchr5_1123.1.txt | genchr5_1123.1.txt |
| chr6 | chr6_1127.1. | 2egrepgenchr6_1127.1.txt | genchr6_1127.1.txt |
| chr6_928.1. | 2egrepgenchr6_928.1.txt | genchr6_928.1.txt | |
| chr7 | chr7_1259.1. | 2egrepgenchr7_1259.1.txt* | genchr7_1259.1.txt |
| chr7_1204.1. | 2egrepgenchr7_1204.1.txt | genchr7_1204.1.txt | |
| chr7_1204.1. | 2egrepgenchr7_292.1.txt | genchr7_292.1.txt |
* Al clicar el fitxer d'anotacions veiem que està en blanc ja que no hi ha cap anotació repetida.

3. Discussió
Com ja s'ha comentat els ESTS són petites seqüències de RNAm que han estat seqüenciats un cop s'han passat al cDNA per retrotranscripció. Per tant, aquests aliniaments aporten informació de possibles regions codificants. Si aquests ESTs trobats amb el programa Megablast s'aparellen en aquelles regions de les diferents seqüències genòmiques estudiades, s'obtindrà un reforçament de la hipòtesi que aquests poden ser reals i, per tant, en el nostre cas, seràn gens novells no descrits anteriorment.
GRÀFIC 1: chr2_2065
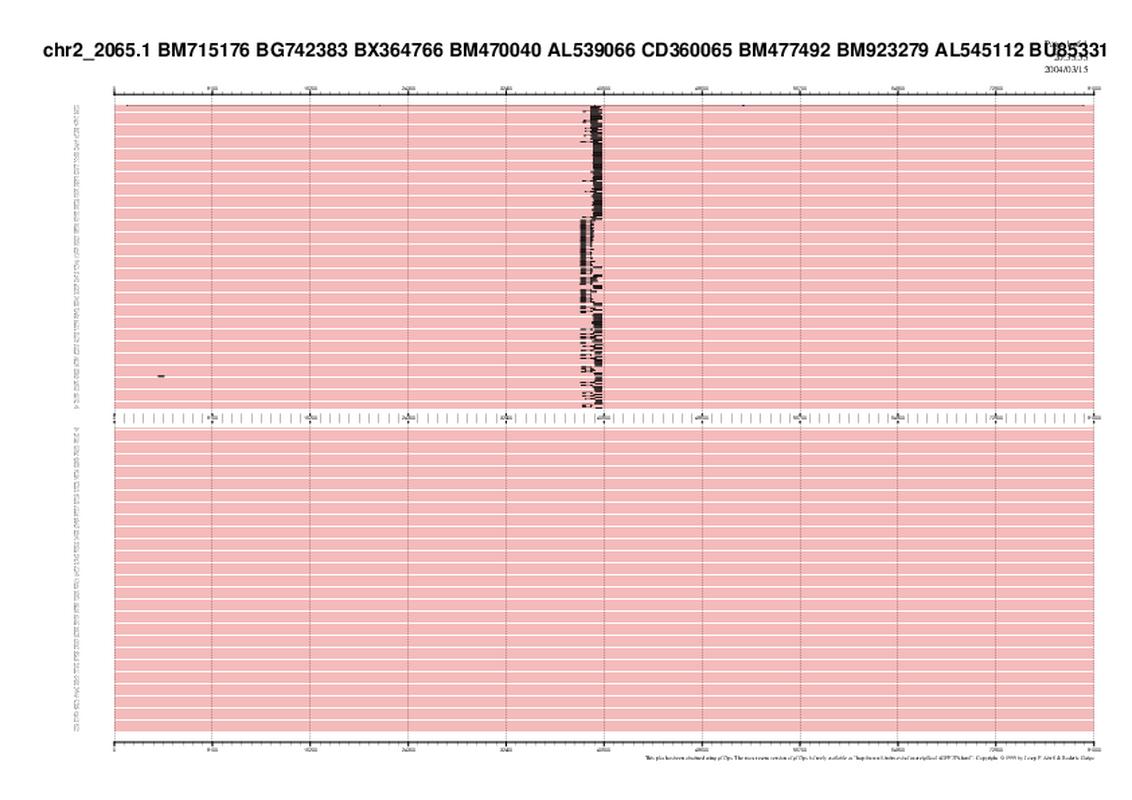 Aquesta imatge ens mostra el chr2_2065, com es pot comprovar hi havia una gran quantitat d'ESTs que s'aparellaven amb la nostra seqüència. Ara bé, n'hi ha tants que el resultat és difícil de comentar, ja que ens van sortir aproximadament uns 200 ESTs. A més, quan vam realitzar els diferents passos per tal d'obtenir l'imatge, ens van donar una mena d'errors, que no sabem si s'han vist o no reflexats en el resultat final. Simplement, comentar que vam voler realitzar aquest tipus de gen, ja que volíem saber que passava quan hi havia un gran nombre d'ESTs. Per tal de donar uns bons resultats ens fixarem però, en les imatges posteriors, ja que contenen menys ESTs.
Aquesta imatge ens mostra el chr2_2065, com es pot comprovar hi havia una gran quantitat d'ESTs que s'aparellaven amb la nostra seqüència. Ara bé, n'hi ha tants que el resultat és difícil de comentar, ja que ens van sortir aproximadament uns 200 ESTs. A més, quan vam realitzar els diferents passos per tal d'obtenir l'imatge, ens van donar una mena d'errors, que no sabem si s'han vist o no reflexats en el resultat final. Simplement, comentar que vam voler realitzar aquest tipus de gen, ja que volíem saber que passava quan hi havia un gran nombre d'ESTs. Per tal de donar uns bons resultats ens fixarem però, en les imatges posteriors, ja que contenen menys ESTs.
| GRÀFIC 2: chr5_1143 | GRÀFIC 3: chr6_1127 |
| GRÀFIC 4: chr7_292 |
GRÀFIC 5: chr7_1204
|
A continuació comentarem els gràfics chr5_1143,chr6_1127,chr7_292,chr7_1204, ja que els resultats són molt semblants en tots ells. El que s'observa és una gran quantitat d'alineaments d'ESTS en regions UTR (regions no traduïdes d'RNAm).
Una possible explicació és que com que nosaltres estem treballant amb unes anotacions de gens que han estan "predites", aquestes prediccions poden ser errònies. El que pot passar, per exemple, és que a la predicció del gen li falti alguna part codificant upstream i per tant, sembla que l'aliniï amb l'UTR.
Una altra explicació, tot i que menys probable, seria que realment algun alineament dels ESTS fos amb els UTRs dels gens.
També s'observa en altres casos, que hi ha ESTs que es solapen amb un sol exó i part de l'intró. El motiu podria ser el mateix que el que s'ha comentat anteriorment. És a dir, que la predicció no sigui bona i que l'EST ens estigui donant l'exó tal i com és realment.
GRÀFIC 6: chr6_928
Aquesta imatge ens mostra el chr6_928; com es pot comprovar és l'únic resultat gràfic en què es pot observar un possible correcte resultat. Ja que l'EST amb l'anotació BX92581 se solapa entres dos exons del gen, és a dir, creua el llindar d'un intró, tot i això,observem que el solapament no és perfecte.
Com s'ha pogut observar, ha estat molt difícil trobar més d'un gen on els ESTS s'aliniin creuant un intró perfectament. Tot i amb això, l'evidència és la que hi ha, ja que la base de dades dels ESTS no és ni molt menys complerta, i les prediccions, per una altra banda,tampoc no ho son.
Per tal de corraborar si l'anotació chr6_928.1 correspon a un gen novel farem el següent:
En primer lloc, buscarem la seqüència proteica del gen, mitjançant la comanda:
$ egrep 'chr6_928.1' /disc8/genomes/H.sapiens/golden_path_200307/database/geneidPep.txt > chr6_928.1.aa.fa
amb el que ens dona:
chr6_928.1 MGTLPARRHIPPWVKVPEDLKDPEVFQVQTRLLKAIFGECAQYWRAAPARESQPARGSHLFSPT
GPDGSRIPYIEQVSKAMLELKALESSDLTEVVVYGSYLYKLRTKWMLQSMAEWHRQRQERGMLKLAEAMNALELGPWMK*
A continuació, a través de la pàgina de NCBI fem un BLASTP, és a dir, proteina contra proteines, contra la base de dades per defecte 'nr', amb
els paràmetres per defecte.
Del resultat que ens surt, mirem la primera seqüència amb la que
s'alinea (clicant al identificador que ens porta al registre NCBI
de la proteïna amb que s'ha alineat) i així podrem distinguir que és el que hem trobat.
El resultat del BLASTP ens torna la proteïna de RefSeq XP_087901 amb les següents caracteristíques:
LOCUS XP_291161 116 aa linear PRI 19-FEB-2004
DEFINITION similar to developmental pluripotency associated 5; embryonal stem cell specific gene 1 [Homo sapiens].
ACCESSION XP_291161
VERSION XP_291161.1 GI:29740698
DBSOURCE REFSEQ: accession XM_291161.1
KEYWORDS .
SOURCE Homo sapiens (human)
MODEL REFSEQ: This record is predicted by automated computational analysis. This record is derived from an annotated genomic sequence (NT_007299) using gene prediction method: GNOMON, supported by EST evidence.
Aquesta anotació ha estat obtinguda mitjançant el mètode GNOMON de predicció de gens. Aquest es basa en la predicció de regions no cobertes pels alineaments de RefSeq, que poden incloure transcrits, pseudogens i transcrits no codificants, que són representats com "misc_RNA" features. També s'hi indica que aquesta compta amb el suport de l'evidència dels EST's.
Com es pot observar, la data de l'anotació é,s del dia 19 de febrer de 2004.
Per tant, podem dir que el gen anotat amb la ID:chr6_928.1 és una bon candidad a ser un nou gen.
Si voleu mes informacio, fer click a conclusió .
 |