Selenoproteïnes de Tupaia chinensis
Forteza A., Garcia M., Jarque M., Martinez S., Merenciano M., Roca-Umbert A.
Les selenoproteïnes
Les selenoproteïnes són proteïnes que incorporen un aminoàcid anomenat selenocisteïna en la seva estructura. La selenocisteïna és l’aminoàcid número 21 i es representa amb les abreviatures Sec i U. Les selenoproteïnes es troben distribuïdes en els tres dominis de la vida: Bacteris, Archeae i Eucariotes, tot i així en eucariotes són més comunes. El nombre de selenoproteïnes en les diferents espècies varia molt segons el taxó. Per exemple, s'han determinat 3 selenoproteïnes en Drosophila melanogaster, 25 en humans i unes 30 en peixos. Tanmateix, no tots els organismes presenten selenoproteïnes, de moment en plantes no se n'ha trobat cap. [1] [2] [3]
SELENOCISTEÏNA
L'aminoàcid selenocisteïna presenta una estructura molt semblant a la cisteïna, però incorpora un àtom de seleni en comptes d’un de sofre. En la taula periòdica, el seleni i el sofre ocupen la mateixa columna, fet que els hi confereix propietats químiques semblants, tot i que el seleni resulta ser un element més reactiu que el sofre. A vegades la seqüència ortòloga d’una selenoproteïna conté una cisteïna en comptes d’una selenocisteïna.
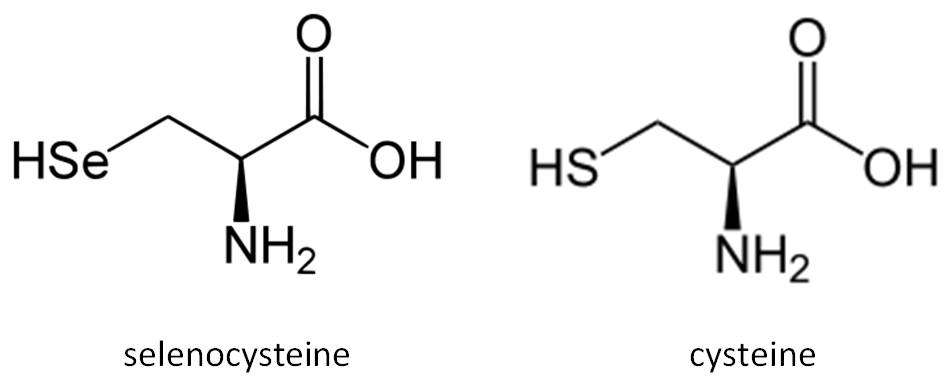
Fig.1 Estructura molecular selenocisteïna
El seleni és un micronutrient essencial pels animals, microorganismes i altres eucariotes. La seva manca pot provocar diferents alteracions, per exemple, la malatia de Keshan, el nom de la qual prové de la província xinesa de Kesh, on hi havien nivells baixos de seleni. El síndrome primari d'aquesta malaltia és la necrosi miocàrdica que porta a debilitament del cor. Contràriament, un excés de seleni pot conduir a una intoxicació greu anomenada selenosi. [1] [3]
SÍNTESI
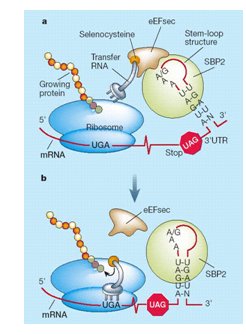 Quan es sintetitzen les selenoproteïnes, la maquinària transcripcional llegeix el codó UGA i no l’interpreta com un STOP, sinó que en aquesta posició incorpora una selenocisteïna i segueix la traducció fins que es troba un codó STOP.
En eucariotes, el mecanisme mitjançant el qual aquest codó s’interpreta de manera diferent està mediat per l’extrem 3’UTR (final del mRNA). A l'extrem dels gens que codifiquen per selenoproteïnes hi ha una estructura tridimensional anomenada SECIS (Selenocystein Insercion Sequence). L'element SECIS és una estructura secundària que té forma de loop i s'encarrega de reclutar la maquinària de traducció evitant la terminació, fent que el codó UGA sigui traduït a selenocisteïna. Per realitzar aquesta tasca, recluta una proteïna anomenada SECIS Binding Protein (SBP2) la qual uneix un factor d’elongació específic de selenoproteïnes (eEFsec). Aquest factor aproxima el tRNA de la selenocisteïna al codó UGA del mRNA de la selenoproteïna, facilitant per tant, la traducció de la selenocisteïna. [3] Fig.2 Traducció selenocisteïna
Quan es sintetitzen les selenoproteïnes, la maquinària transcripcional llegeix el codó UGA i no l’interpreta com un STOP, sinó que en aquesta posició incorpora una selenocisteïna i segueix la traducció fins que es troba un codó STOP.
En eucariotes, el mecanisme mitjançant el qual aquest codó s’interpreta de manera diferent està mediat per l’extrem 3’UTR (final del mRNA). A l'extrem dels gens que codifiquen per selenoproteïnes hi ha una estructura tridimensional anomenada SECIS (Selenocystein Insercion Sequence). L'element SECIS és una estructura secundària que té forma de loop i s'encarrega de reclutar la maquinària de traducció evitant la terminació, fent que el codó UGA sigui traduït a selenocisteïna. Per realitzar aquesta tasca, recluta una proteïna anomenada SECIS Binding Protein (SBP2) la qual uneix un factor d’elongació específic de selenoproteïnes (eEFsec). Aquest factor aproxima el tRNA de la selenocisteïna al codó UGA del mRNA de la selenoproteïna, facilitant per tant, la traducció de la selenocisteïna. [3] Fig.2 Traducció selenocisteïna
Per tant, les proteïnes que participen en la incorporació del codó Sec en les selenoproteïnes són:
- La proteïna SBP2 (SECIS Binding Protein): uneix a la SECIS i recluta un tRNA específic per la Sec, incorpora l’aminoàcid a la cadena peptídica i es continua la traducció.
- La proteïna eEFsec (Sec elongation factor): uneix SBP2 i permet que continuï l’elongació quan s’incorpora la Sec.
Per altra banda, hi ha les proteïnes necessàries per sintetitzar la selenocisteïna. Hi ha unes 10 proteïnes o factors involucrats necessaris per la síntesi d'aquesta proteïna. És important destacar que és l’únic aminoàcid que es sintetitza directament en el tRNA: inicialment aquest conté una serina i llavors s’incorpora el selenofosfat formant el selenocisteinil-ARNt. Aquestes són les proteïnes implicades:
- Seryl-tRNA sintetasa: permet la unió de la serina a tRNA.
- La selenofosfat sintetasa SPS2: catalitza la reacció de seleni a selenofosfat.
- Phosphoseryl-tRNA kinasa Pstk: permet la fosforilació de la serina.
- SEC43p i SPS1: formen part del complex que permet la incorporació del selenofosfat.
- La selenocisteïna sintasa SLA/LP o SecS: permet la incorporació del selenofosfat a l’aminoàcid.[3]
SELENOCISTEÏNA
L'aminoàcid selenocisteïna presenta una estructura molt semblant a la cisteïna, però incorpora un àtom de seleni en comptes d’un de sofre. En la taula periòdica, el seleni i el sofre ocupen la mateixa columna, fet que els hi confereix propietats químiques semblants, tot i que el seleni resulta ser un element més reactiu que el sofre. A vegades la seqüència ortòloga d’una selenoproteïna conté una cisteïna en comptes d’una selenocisteïna.

Fig.1 Estructura molecular selenocisteïna
El seleni és un micronutrient essencial pels animals, microorganismes i altres eucariotes. La seva manca pot provocar diferents alteracions, per exemple, la malatia de Keshan, el nom de la qual prové de la província xinesa de Kesh, on hi havien nivells baixos de seleni. El síndrome primari d'aquesta malaltia és la necrosi miocàrdica que porta a debilitament del cor. Contràriament, un excés de seleni pot conduir a una intoxicació greu anomenada selenosi. [1] [3]
SÍNTESI
 Quan es sintetitzen les selenoproteïnes, la maquinària transcripcional llegeix el codó UGA i no l’interpreta com un STOP, sinó que en aquesta posició incorpora una selenocisteïna i segueix la traducció fins que es troba un codó STOP.
En eucariotes, el mecanisme mitjançant el qual aquest codó s’interpreta de manera diferent està mediat per l’extrem 3’UTR (final del mRNA). A l'extrem dels gens que codifiquen per selenoproteïnes hi ha una estructura tridimensional anomenada SECIS (Selenocystein Insercion Sequence). L'element SECIS és una estructura secundària que té forma de loop i s'encarrega de reclutar la maquinària de traducció evitant la terminació, fent que el codó UGA sigui traduït a selenocisteïna. Per realitzar aquesta tasca, recluta una proteïna anomenada SECIS Binding Protein (SBP2) la qual uneix un factor d’elongació específic de selenoproteïnes (eEFsec). Aquest factor aproxima el tRNA de la selenocisteïna al codó UGA del mRNA de la selenoproteïna, facilitant per tant, la traducció de la selenocisteïna. [3] Fig.2 Traducció selenocisteïna
Quan es sintetitzen les selenoproteïnes, la maquinària transcripcional llegeix el codó UGA i no l’interpreta com un STOP, sinó que en aquesta posició incorpora una selenocisteïna i segueix la traducció fins que es troba un codó STOP.
En eucariotes, el mecanisme mitjançant el qual aquest codó s’interpreta de manera diferent està mediat per l’extrem 3’UTR (final del mRNA). A l'extrem dels gens que codifiquen per selenoproteïnes hi ha una estructura tridimensional anomenada SECIS (Selenocystein Insercion Sequence). L'element SECIS és una estructura secundària que té forma de loop i s'encarrega de reclutar la maquinària de traducció evitant la terminació, fent que el codó UGA sigui traduït a selenocisteïna. Per realitzar aquesta tasca, recluta una proteïna anomenada SECIS Binding Protein (SBP2) la qual uneix un factor d’elongació específic de selenoproteïnes (eEFsec). Aquest factor aproxima el tRNA de la selenocisteïna al codó UGA del mRNA de la selenoproteïna, facilitant per tant, la traducció de la selenocisteïna. [3] Fig.2 Traducció selenocisteïna
Per tant, les proteïnes que participen en la incorporació del codó Sec en les selenoproteïnes són:
- La proteïna SBP2 (SECIS Binding Protein): uneix a la SECIS i recluta un tRNA específic per la Sec, incorpora l’aminoàcid a la cadena peptídica i es continua la traducció.
- La proteïna eEFsec (Sec elongation factor): uneix SBP2 i permet que continuï l’elongació quan s’incorpora la Sec.
Per altra banda, hi ha les proteïnes necessàries per sintetitzar la selenocisteïna. Hi ha unes 10 proteïnes o factors involucrats necessaris per la síntesi d'aquesta proteïna. És important destacar que és l’únic aminoàcid que es sintetitza directament en el tRNA: inicialment aquest conté una serina i llavors s’incorpora el selenofosfat formant el selenocisteinil-ARNt. Aquestes són les proteïnes implicades:
- Seryl-tRNA sintetasa: permet la unió de la serina a tRNA.
- La selenofosfat sintetasa SPS2: catalitza la reacció de seleni a selenofosfat.
- Phosphoseryl-tRNA kinasa Pstk: permet la fosforilació de la serina.
- SEC43p i SPS1: formen part del complex que permet la incorporació del selenofosfat.
- La selenocisteïna sintasa SLA/LP o SecS: permet la incorporació del selenofosfat a l’aminoàcid.[3]
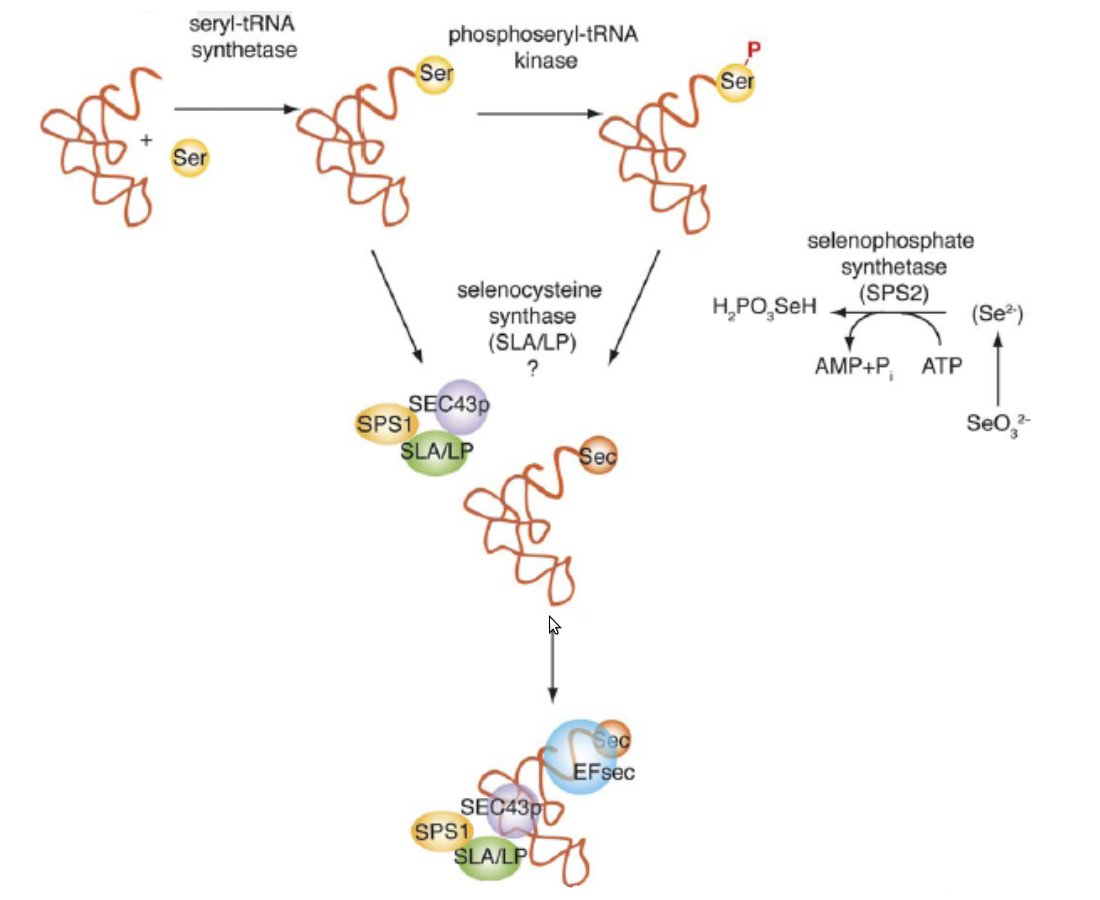
Fig.3 Síntesi selencisteïna

Anotació i predicció
La identificació computacional de selenoproteïnes és complicada degut a que el codó UGA, codificant per a la selenocisteïna, es considera sistemàticament un codó STOP. Per aquest motiu, no existeix un mètode exacte per a predir la localització de selenoproteïnes al genoma.
D'entrada, una forma per reconèixer aquesta pauta de lectura alternativa del codó UGA seria la cerca d'elements SECIS. El problema d’aquesta aproximació és que els elements SECIS tenen una conservació de seqüència molt baixa en les selenoproteïnes. D’una seqüència de 100 nucleòtids, només es conserven dues A en la posició del loop i unes 5 posicions en l’hèlix II entre les selenoproteïnes humanes. Aquest fet dificulta la identificació d’aquestes estructures. Tot i que no hi ha cap conservació a nivell de seqüència entre els SECIS, aquests tenen una estructura tridimensional loop-helix-loop característica que permet la seva identificació mitjançant programes bioinformàtics com SECIsearch3.0/Seblastian.
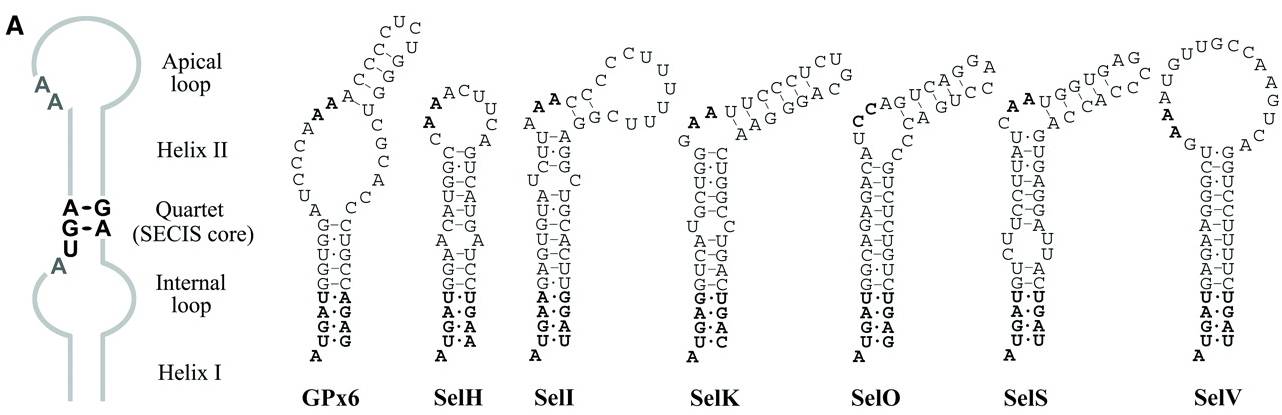
Fig.4 Estructura dels elements SECIS
Podem concloure que un bon procediment per a predir selenoproteïnes en un genoma que encara no ha estat analitzat és:
1. Buscar seqüències de selenoproteïnes ja anotades en espècies properes en el genoma nou, és a dir,
buscar proteïnes homòlogues.
2. Cercar elements SECIS.
Superposant aquesta informació obtindrem els gens candidats, que seran aquells en els quals trobem elements SECIS a l'extrem 3'UTR.

D'entrada, una forma per reconèixer aquesta pauta de lectura alternativa del codó UGA seria la cerca d'elements SECIS. El problema d’aquesta aproximació és que els elements SECIS tenen una conservació de seqüència molt baixa en les selenoproteïnes. D’una seqüència de 100 nucleòtids, només es conserven dues A en la posició del loop i unes 5 posicions en l’hèlix II entre les selenoproteïnes humanes. Aquest fet dificulta la identificació d’aquestes estructures. Tot i que no hi ha cap conservació a nivell de seqüència entre els SECIS, aquests tenen una estructura tridimensional loop-helix-loop característica que permet la seva identificació mitjançant programes bioinformàtics com SECIsearch3.0/Seblastian.

Fig.4 Estructura dels elements SECIS
Podem concloure que un bon procediment per a predir selenoproteïnes en un genoma que encara no ha estat analitzat és:
1. Buscar seqüències de selenoproteïnes ja anotades en espècies properes en el genoma nou, és a dir,
buscar proteïnes homòlogues.
2. Cercar elements SECIS.
Superposant aquesta informació obtindrem els gens candidats, que seran aquells en els quals trobem elements SECIS a l'extrem 3'UTR.

Tupaia chinensis

CLASSIFICACIÓ CIENTÍFICA
- Regne: Animal
- Filum: Cordats
- Classe: Mamífers
- Ordre: Scadentia
- Familia: Tupaiidae
- Gènere: Tupaia
- Espècie: Tupaia chinensis
Fig.6 Tupaia chinensis
MODEL ANIMAL
Tupaia chinensis (també anomenada musaranya arborícola xinesa) té una àmplia distribució en el sud i sud-est d'Àsia, així com al sud-oest de la Xina. Tupaia chinensis pertany a la família Tupaiidae, la qual té una sèrie de característiques úniques ideals per a ser utilitzada com a model animal: mida petita, relació de la massa cerebral i corporal alta, cicle reproductiu i de vida curt, un baix cost de manteniment, i el més important, està situada filogenèticament molt propera als primats. Així, Aquestes característiques fan que sigui un model animal alternatiu als primats en la investigació biomèdica. Actualment, hi ha nombrosos intents per emprar tupaies com a models per a una varietat de trastorns humans, incloent malalties infeccioses, metabòliques, neurològiques, psiquiàtriques i càncers.
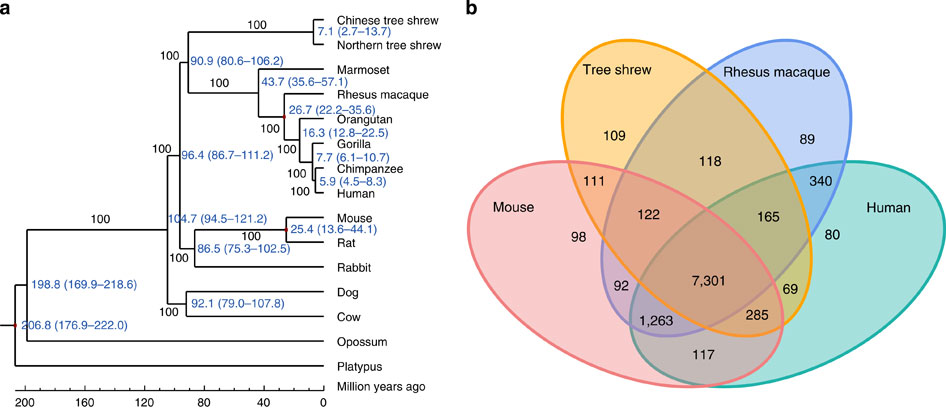
Fig.7 Relació de la Tupaia chinensis (Northem Tree Shrew) amb els mamífers més propers. a| Arbre filogenètic. b| Diagrama de Venn de la família de gens de Tupaia chinensis amb humans, macacos rhesus i el ratolí.
Tot i així, són pocs els laboratoris que les utilitzen, en part per l’accés limitat a aquest recurs animal i per la manca de reactius específics. D'altra banda, els grans obstacles per a la promoció d'aquests estudis segueixen sent, sobretot la manca d’un genoma de gran qualitat i una visió global dels perfils d'expressió gènica. [5]

Fig.7 Relació de la Tupaia chinensis (Northem Tree Shrew) amb els mamífers més propers. a| Arbre filogenètic. b| Diagrama de Venn de la família de gens de Tupaia chinensis amb humans, macacos rhesus i el ratolí.
Tot i així, són pocs els laboratoris que les utilitzen, en part per l’accés limitat a aquest recurs animal i per la manca de reactius específics. D'altra banda, els grans obstacles per a la promoció d'aquests estudis segueixen sent, sobretot la manca d’un genoma de gran qualitat i una visió global dels perfils d'expressió gènica. [5]

Filogènia de les selenoproteïnes en vertebrats
L'estudi de selenoproteïnes permet caracteritzar els genomes i pot ser útil per veure la proximitat filogenètica entre diferents organismes. El selenoproteoma és el conjunt de proteïnes que són o que estan relacionades amb les selenoproteïnes.
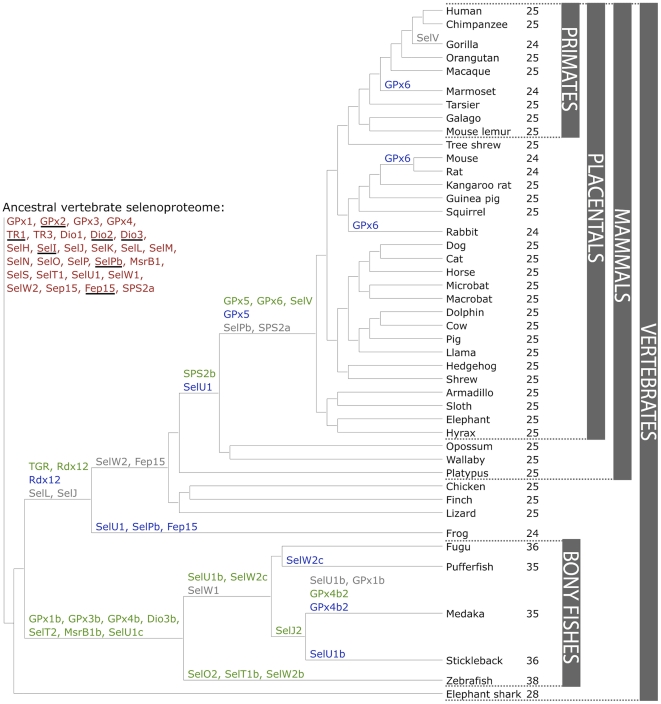
Fig.5 Evolució del selenoproteoma dels vertebrats
La imatge superior mostra que al llarg de la evolució de les selenoproteïnes en els vertebrats, han tingut lloc 20 duplicacions (verd), 9 delecions (gris) i 13 mutacions als homòlegs amb cisteïna (blau). A la dreta, es mostra el nombre de selenoproteïnes predites a cada espècie. [2]
L’objectiu del nostre treball és estudiar i analitzar el selenoproteoma de Tupaia chinensis, i ho farem buscant homologia amb el genoma del ratolí (Mus musculus) i l’humà (Homo sapiens sapiens). Donada la proximitat de T.chinensis als primats, es va escollir el selenoproteoma d'H.sapiens ja que considerem que és un dels que està millor anotat. D’altra banda, donada la controvèrsia en la classificació filogenètica de la T.chinensis en els últims anys (ja que anteriorment es classificava més propera als rosegadors que als primats) hem utilitzat també el selenoproteoma de M.musculus. Així, l'estudi contibuirà a entendre millor les relacions filogenètiques entre espècies i l'evolució i funció d'aquesta família de proteïnes.

Fig.5 Evolució del selenoproteoma dels vertebrats
La imatge superior mostra que al llarg de la evolució de les selenoproteïnes en els vertebrats, han tingut lloc 20 duplicacions (verd), 9 delecions (gris) i 13 mutacions als homòlegs amb cisteïna (blau). A la dreta, es mostra el nombre de selenoproteïnes predites a cada espècie. [2]
L’objectiu del nostre treball és estudiar i analitzar el selenoproteoma de Tupaia chinensis, i ho farem buscant homologia amb el genoma del ratolí (Mus musculus) i l’humà (Homo sapiens sapiens). Donada la proximitat de T.chinensis als primats, es va escollir el selenoproteoma d'H.sapiens ja que considerem que és un dels que està millor anotat. D’altra banda, donada la controvèrsia en la classificació filogenètica de la T.chinensis en els últims anys (ja que anteriorment es classificava més propera als rosegadors que als primats) hem utilitzat també el selenoproteoma de M.musculus. Així, l'estudi contibuirà a entendre millor les relacions filogenètiques entre espècies i l'evolució i funció d'aquesta família de proteïnes.

INTRODUCCIÓ
LINKS
QUI SOM?
Som un grup d'estudiants de quart de Biologia Humana de la Universitat Pompeu Fabra-FCSV. Hem realitzat aquest projecte per l'assignatura de Bioinformàtica. Si et vols posar en contacte amb nosaltres clica Aquí