INTRODUCCIÓN¿Qué son las selenoproteínas?Las selenoproteínas son un grupo exclusivo de proteínas que incluyen en su estructura algún residuo de selenocisteína (Sec), considerado el aminoácido número 21 del código genético. Este inusual aminoácido es un análogo de cisteína (Cys) y contiene selenio en lugar de azufre. El selenio es un micronutriente esencial en muchos organismos y su deficiencia se ha asociado a numerosas condiciones fisiopatológicas como la enfermedad cardiovascular, los desórdenes neuromusculares, el cáncer o la inflamación (Vyacheslav M et al, 2014).Los residuos de Sec y Cys ocupan posiciones homólogas, sin embargo la Sec es un aminoácido más reactivo que la cisteína y forma parte del dominio catalítico de la proteína. En las selenoproteínas de funcioón conocida estaá siempre involucrado en reacciones de oxidorreducción. Desde el punto de vista evolutivo y debido a la similitud de sus propiedades químicas, ha habido evolución específica de linaje de Sec a Cys y por ello es posible encontrar homólogos de selenoproteínas que incorporan Cys en lugar de Sec, definiéndose como genes ortólogos o parálogos (Zhang Y et al, 2006). La incorporación de Sec a la cadena polipeptídica está determinada por su codificación por el codón UGA, un codón que usualmente especifica la terminación de la traducción proteica. Se produce cuando existe una estructura tallo-bucle conservada en la región 3'-UTR del mRNA de las selenoproteínas, conocida como elemento SECIS (del inglés SElenoCysteine Insertion Sequence) (Lobanov AV at al, 2009) (Figura 1). 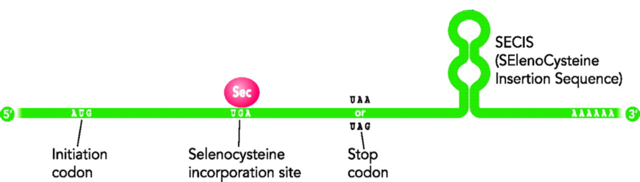 Figura 1. Representación esquemática de la estructura del mRNA de una selenoproteína. Se observa la dualidad del codón UGA codificando, por un lado, por el aminoácido Sec y, por el otro, indicando la finalización de la traducción. En la región 3'UTR destaca la presencia del elemento SECIS con una estructura de loop conservada (modificado de Moghadaszadeh B et al, 2006).Biosíntesis de selenoproteínasLas selenoproteínas se caracterizan por contener el aminoácido Sec. Este residuo está codificado por el codón UGA (normalmente un codón STOP) presente en el marco de lectura abierto del gen, que se decodifica como Sec durante la síntesis ribosomal de proteínas a partir de un mecanismo específico montado sobre el aparato traduccional canónico. La principal señal que dirige la recodificación del codón UGA es una estructura secundaria presente exclusivamente en los transcritos de las selenoproteínas, el elemento SECIS. Es decir, el elemento SECIS es el que desambigua el significado del codón UGA y activa la maquinaria de inserción de Sec.La Sec es el único aminoácido cuya síntesis ocurre en su propio tRNA (no existe la síntesis del aminoácido libre), llamado tRNA[Ser]Sec. Este tRNA se aminoacila con una serina en una reacción catalizada por la seril-tRNA sintetasa (SerS) para dar lugar a la estructura base para la biosíntesis de la Sec. A continuación habrá una fosforilación de la serina por parte de la fosfoseril-tRNA quinasa (PSTK). El selenio es a su vez forforilado por la selenofosfato sintetasa 2 (SPS2) y se añade a la serina ya fosforilada. Sobre esta estructura la Sec sintetasa (SecS) produce el residuo de Sec formando así el Sec-tRNA[Ser]Sec (Bellinger FP et al, 2009) (Figura 2). 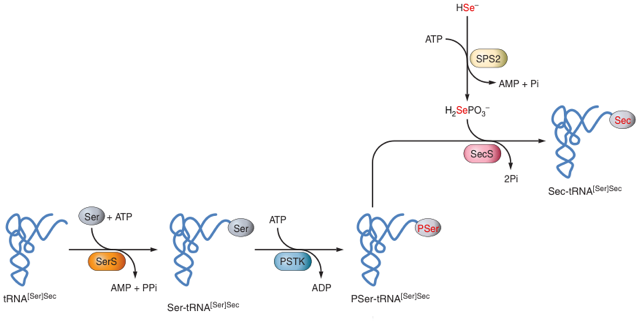 Figura 2. Mecanismo de síntesis de la Sec. La SerS aminoacila el tRNA[Ser]Sec con serina a partir de SerS dando lugar al Ser-tRNA[Ser]Sec. Éste útlimo es fosforilado por PSTK, que proporciona el sustrato a la SecS junto con el selenio fosforilado para la síntesis de Sec-tRNA[Ser]Sec (modificado de Labunskyy VM et al, 2014).Para la incorporación del Sec-tRNA[Ser]Sec al ribosoma, éste debe ser reconocido por un factor de elongación específico denominado EFSec. EFSec no interacciona directamente con el elemento SECIS, sino que lo hace a través de la proteína de unión a SECIS, conocida como SBP2. Además, otros factores como la proteína ribosomal L30 (que es un factor eucariótico de iniciación de la traducción, también llamado eIF4a3) participan en el proceso de inclusión de la Sec en respuesta a la presencia del codón UGA en el marco de lectura del gen (Bellinger FP et al, 2009) (Figura 3). 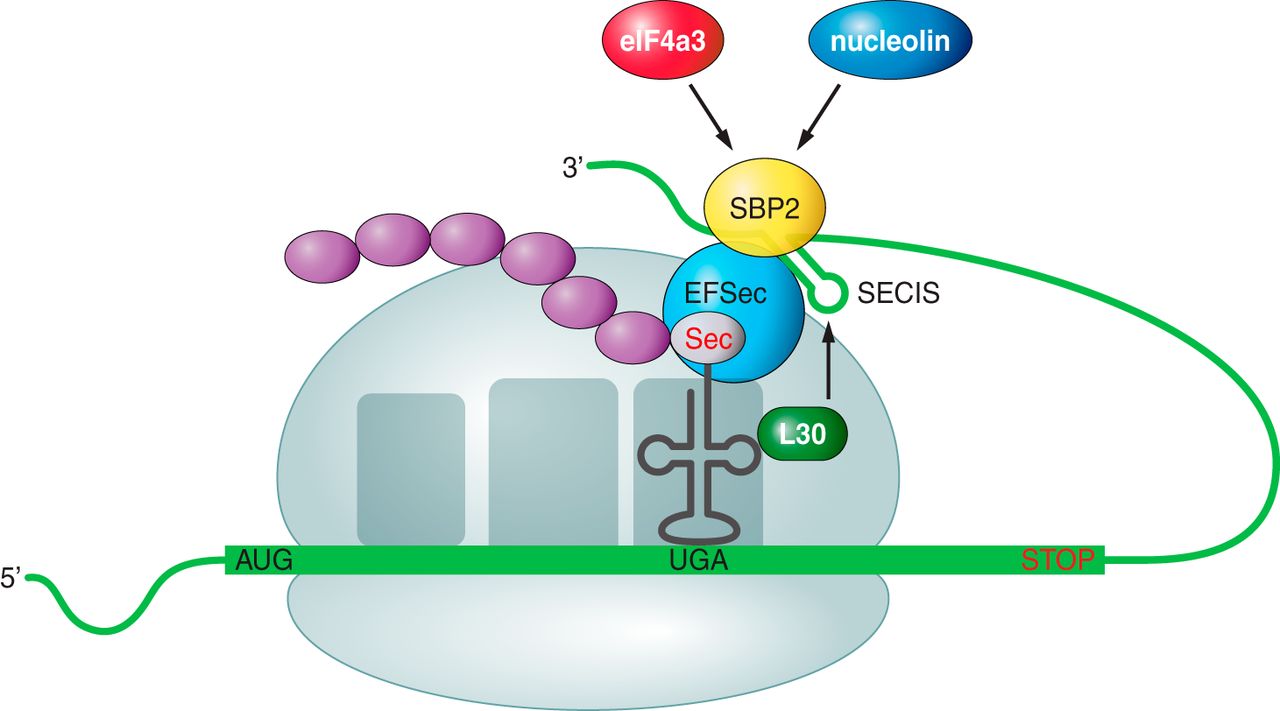 Figura 3. Mecanismo de inserción de la Sec a la cadena polipeptídica. El elmento SECIS es un intermediario del ensamblaje de los factores EFSec, SBP2 y L30 al mRNA de las selenoproteínas permitiendo la incorporación de Sec en respuesta a la presencia del codón UGA (modificado de Labunskyy VM et al, 2014).La similitud global del mecanismo de síntesis e incorporación de Sec en los tres dominios de la vida sugiere que este fenómeno apareció una sola vez en la evolución, previo a la separación de dichos dominios. La existencia de linajes que no incorporan Sec indica que en algunos casos se perdió esta capacidad ancestral y esto fundamenta la idea de que existen diferentes presiones selectivas que determinan el mantenimiento o la pérdida de la incorporación de Sec. Familias de selenoproteínasLos efectos biológicos del selenio están mayormente mediados por las selenoproteínas, que están presentes en arqueas, bacterias y eucariotas. Con algunas excepciones, el aminoácido Sec se localiza en el centro activo del enzima, donde media las reacciones de catálisis redox (Arnér ES, 2010). Por ello, los roles fisiológicos que adoptan las selenoproteínas dependen en gran medida de la presencia de Sec, de forma que mutaciones puntuales que afecten dicho aminoácido pueden fácilmente desencadenar su inactivación.La clasificación de las selenoproteínas en familias depende de la localización de Sec en la cadena de aminoácidos (Figura 4). Por un lado, un primer grupo lo constituyen aquellas familias que tienen la Sec cerca del extremo C-terminal de la proteína. Lo integran, por ejemplo, las tioredoxina reductasas (TrxRs) y las selenoproteínas S, I, O y K. Por otro lado, la posición de Sec en el extremo N-terminal forma un segundo grupo en el que destacan las glutatión peroxidasas (GPxs), las yodotironin deodinasas (DIOs), las selenoproteínas H, M, N, T, V y W y la selenofosfato sintetasa 2 (SPS2) (Lu J and Holmgren A, 2009). Aunque las vías moleculares a través de las cuales las selenoproteínas llevan a cabo sus distintos papeles biológicos sean muy diversas y en muchos casos desconocidas, la mayoría actúan como enzimas antioxidantes extremadamente eficientes formando parte de los sistemas de tiorredoxina y de glutatión (Kryukov GV et al, 2003). Estos sistemas mantienen la homeostasis redox de las células a expensas del poder reductor del NADPH y participan, entre otros procesos fisiológicos, en el metabolismo de las hormonas tiroideas, la maduración de los espermatozoides y la función muscular (Labunskyy VM et al, 2014). 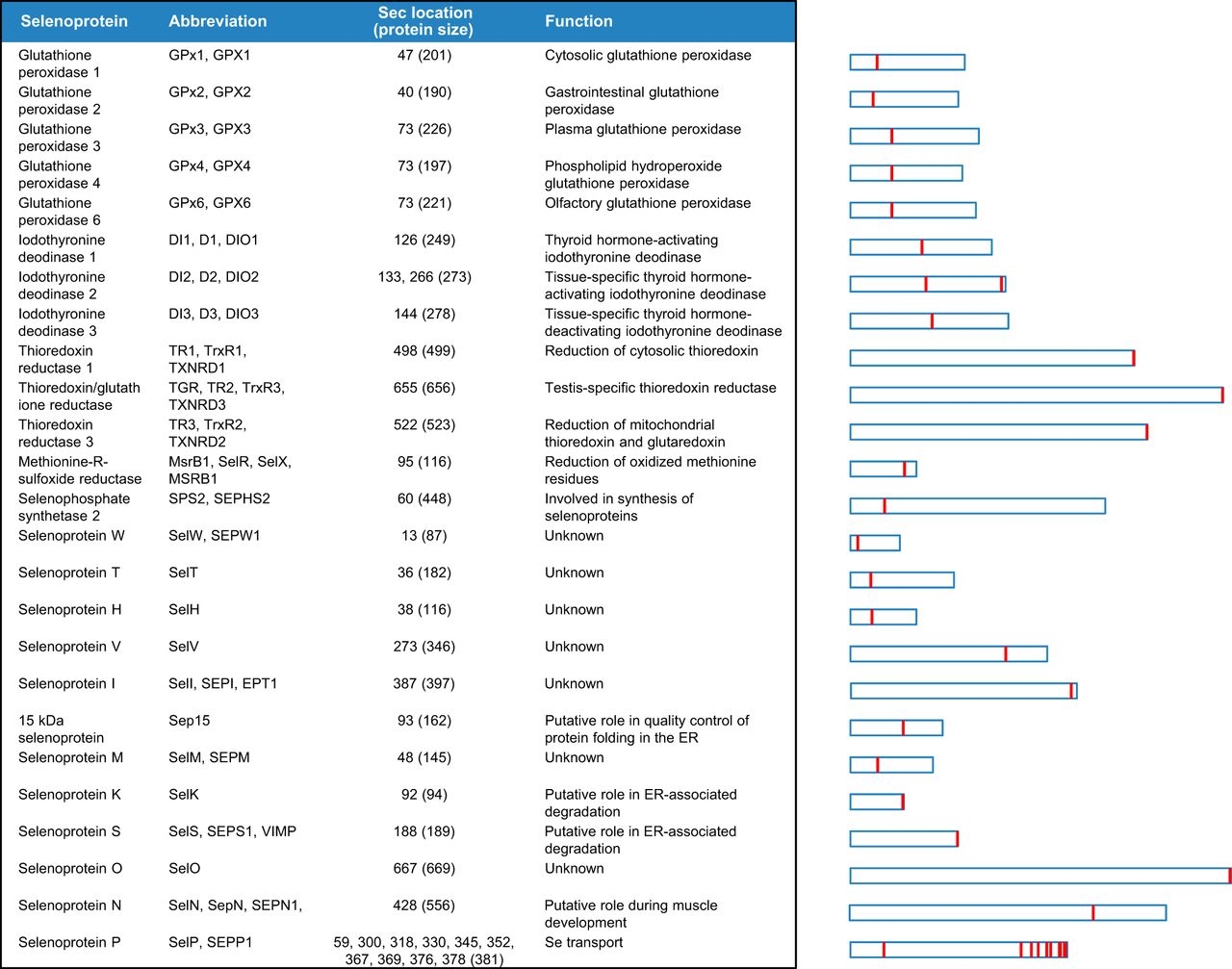 Figura 4. Clasificación y funciones de distintas familias de selenoproteínas. A la derecha se muestra un esquema de la longitud relativa de cada tipo de selenoproteína y en rojo se marca la localización de la Sec (Labunskyy VM et al, 2014).Evolución de las selenoproteínasSe llama selenoproteoma al conjunto de selenoproteínas de un organismo y su evolución está definida por las condiciones ambientales en las que viven los organismos así como la disponibilidad de selenio (Lobanov AV at al, 2009). Por ejemplo, los organismos acuáticos se caracterizan por presentar los selenoproteomas más grandes. Por el contrario, en los organismos terrestres la cantidad selenoproteínas es marcadamente más reducida. Una de las hipótesis con más peso para explicar dicha situación es que la función antioxidante propia de las selenoproteínas juega un papel clave en la protección de las células frente a las concentraciones elevadas de oxígeno (Jakupoglu C et al, 2005). Una de las implicaciones de la transición al hábitat terrestre es la limitación al acceso de varios elementos esenciales como el selenio, que es notablemente menor. Como resultado, muchos organismos terrestres como las plantas superiores, las levaduras, los hongos y ciertas especies de animales pierden familias de selenoproteínas o bien las reemplazan por homólogos que contienen cisteína (Lobanov AV et al, 2009).La mayor parte de selenoproteínas identificadas tienen un origen ancestral y principalmente se han generado a lo largo de la evolución a partir de duplicaciones génicas. La anotación de genomas, la aplicación de métodos bioinformáticos para la predicción de nuevas selenoproteínas y los modelos de reconstrucción filogenética han permitido caracterizar 45 familias de selenoproteínas en vertebrados, de las cuales 20 se generan a partir de duplicaciones de una selenoproteína ya existente y 6 forman parte del selenoproteoma ancestral (Mariotti M et al., 2012) (Figura 5). El mismo estudio concluye que el selenoproteoma de mamíferos se ha mantenido relativamente estable y se compone de un total de 28 familias, habiendo por cada especie de mamífero hasta 25 genes codificantes para selenoproteínas. Asimismo, se identifican 21 selenoproteínas comunes en todos los vertebrados mientras que el resto únicamente se encuentra en determinados linajes. Esto muestra los posibles orígenes de las selenoproteínas que incluyen la duplicación génica y la sustitución por cisteína. 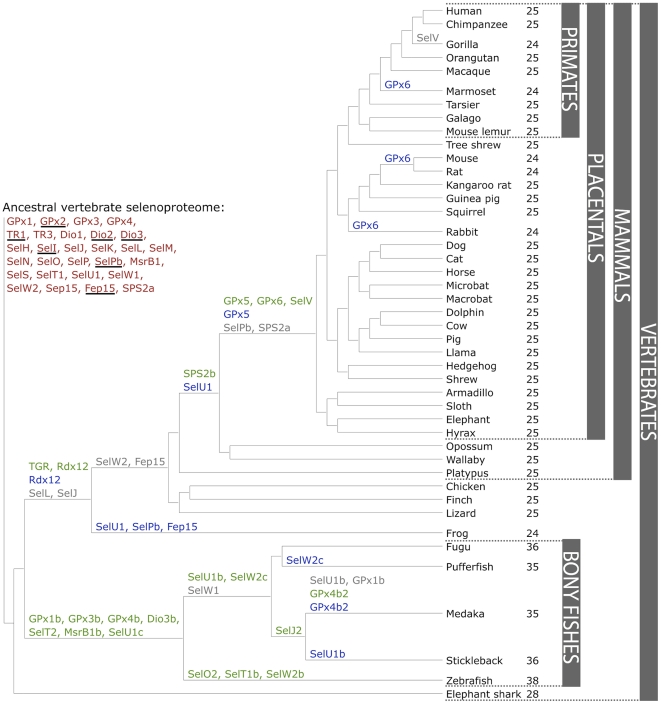 Figura 5. Evolución del selenoproteoma de vertebrados. Las selenoproteínas ancestrales se señalan en rojo y sus cambios evolutivos son reflejados mediante el árbol filogenético. Aquellas selenoproteínas subrayadas son exclusivas de vertebrados, mientras que el color verde indica la formación de una nueva selenoproteína por duplicación génica. En gris se muestran las selenoproteínas perdidas a lo largo de los linajes y en azul se señalan los casos en el que se ha producido un reemplazo por un homólogo de cisteína (Mariotti M et al, 2012).Anotación y predicciónEl papel dual del codón UGA, la dificultad en la predicción y anotación de selenoproteínas además de la gran variabilidad en la función de las distintas familias descritas sustenta la falta de conocimiento que aún se posee sobre este grupo de proteínas. Sin embargo, el reciente desarrollo de aproximaciones computacionales ha permitido la identificación y caracterización de los selenoproteomas de un gran número de especies (Castellano S et al, 2008). Uno de los métodos empleados para la predicción de genes codificantes para selenoproteínas en un genoma se basa en la comparación con secuencias génicas de selenoproteínas pertenecientes al genoma de la especie filogenéticamente más cercana cuyo selenoproteoma ya esté anotado en bases de datos. Los algoritmos usados están diseñados para localizar el codón UGA en el marco de lectura del gen de la selenoproteína del genoma de referencia. De la misma forma, la identificación de los elementos SECIS en la región 3'UTR de los genes candidatos también contribuye a una mejor definición de las predicciones obtenidas. Ambos sistemas, siempre y cuando vayan acompañados de una correcta interpretación de los resultados, permiten el reconocimiento de selenoproteínas que contienen Sec y de los correspondientes homólogos de cisteína. Además existen programas como Seblastian que combinan ambas estrategias de predicción (Mariotti M et al, 2013).En conclusión, el análisis de genomas secuenciados que permitan analizar la presencia y distribución de selenoproteínas juntamente con el estudio detallado de su filogenia, llevará a una mejor comprensión de la función e implicación de las selenoproteínas en la fisiología y la patología humana. Macaca nemestrinaEl macaco de cola de cerdo sureño (Macaca nemestrina) es una especie de primate catarrino de la familia Cercopithecidae. Habita en la península de Malaya, Borneo, Sumatra e isla Bangka. Es un macaco de mediano tamaño, omnívoro y que habita principalmente en los bosques, pero que con el avance de los humanos también se le encuentra en plantaciones y jardines. Anteriormente se consideran subespecies de este taxón a las ahora especies: Macaca leonina (macaco cola de cerdo norteño), Macaca pagensis (el macaco de Pagai) y Macaca siberu (el macaco de Siberut). Para más información clique aquí. |


