Selenoproteïnes en Eptesicus fuscus
Cebrián M.C, Franco M, Gallardo P, Gallego E.
| ABSTRACT | INTRODUCCIÓ | MATERIALS I MÈTODES | RESULTATS | DISCUSSIÓ | CONCLUSIONS | REFERÈNCIES | AGRAÏMENTS | CONTACTA'NS |
INTRODUCCIÓ
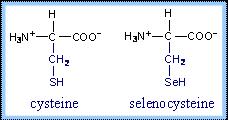
Les selenoproteïnes es caracteritzen per la presència de, com a mínim, un residu de selenocisteïna (Sec o U) a la seva seqüència. Sec es considera l'aminoàcid 21, i difereix de la cisteïna (Cys) per la identitat d'un únic àtom: Sec té seleni enlloc del sulfur propi de Cys. Una de les característiques que Sec confereix a les selenoproteïnes és la seva alta nucleofilicitat inherent, proporcionant-li una alta reactivitat amb els electròfils. D'aquesta manera, s'ha observat la seva acció en reaccions redox en la protecció front els radicals lliures, així com en la regulació de la funció tiroidea i del sistema immunitari.
Això implica, doncs, que les selenoproteïnes tindran unes propietats químiques diferents que han de resultar útils per la cèl·lula, per compensar el cost de tenir una maquinària específica per a la síntesi d'aquest aminoàcid que reconegui el codó UGA com a codificant per Sec, enlloc d'identificar-lo com a STOP.
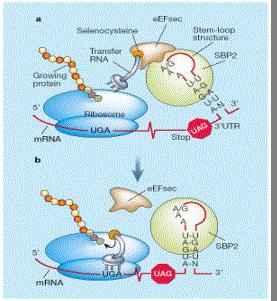
Aquesta recodificació és possible gràcies als elements SECIS (Seleno Cystein Insertion Sequence), una estructura de RNA 3D que en el cas dels eucariotes es troba situada a l’extrem 3’-UTR (extrem 3’ no traduït) dels gens de les selenoproteïnes, o bé downstream del codó UGA en el cas dels procariotes.
L’estructura dels SECIS consta d’un total de 60 nucleòtids, però només 4 d’aquests estan conservats en el core. Tot i això, l’estructura 3D dels SECIS està molt conservada. Aquesta estructura característica dels SECIS fa possible el reclutament de la proteïna SBP2 (SECIS Binding Protein 2), a la qual s’uneix eEFsec (factor d'elongació específic de selenoproteïnes). D’aquesta manera, el tRNA propi de la selenocisteïna (tRNAsec) arriba al codó UGA propi de la selenoproteïna i permet la traducció, la qual acabarà quan es trobi un nou codó de terminació.
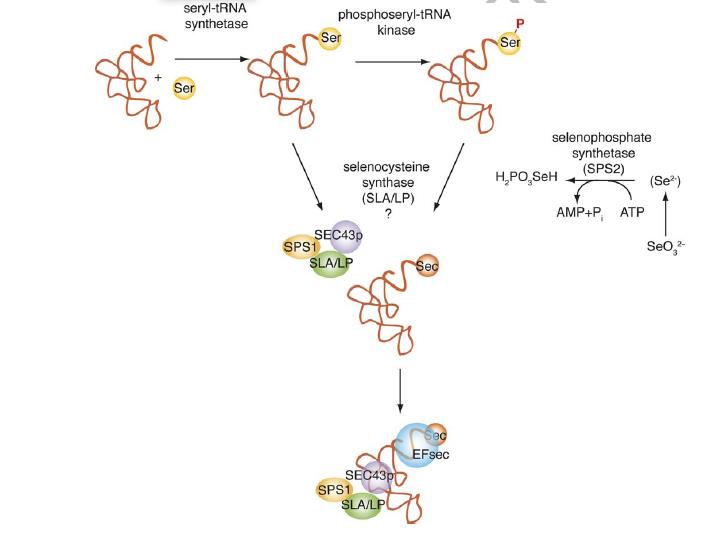
Per tant, per a poder sintetitzar les selenoproteïnes es necessita un conjunt de maquinària específica. Inicialment, s’ha de sintetitzar la selenocisteïna a partir de la serina per l’acció de Selenofosfat sintetasa 1 i 2 (SPS1 i SPS2), transformant el seleni de la dieta a monoselenofosfat, que gràcies a PSTK (Phosphoseryl tRNA kinase) es transferirà a fosfoseril-tRNAser. Posteriorment, SLA/LP (Soluble Liver Antigen/Liver-Pancreas) el transformaran en selenocistenil-tRNAsec per la desfosforilació de la serina fosforilada per PTSK prèviament. Finalment, Secp43 (Selenocystein associated protein) estabilitza el complex SECIS-SBP2-eEFsec-Selenocistenil-tRNAsec i participa en la metilació de tRNAsec.
Un cop està la selenocisteïna sintetitzada, s’ha de poder incorporar a la selenoproteïna. Aquest procés és possible gràcies a SBP2 (SECIS binding protein) que a l’unir-se al SECIS recluta el complex eEFsec-Selenocistenil-tRNAsec al ribosoma. A continuació, eEFsec (eukaryotic elongation factor, selenocysteine-tRNA-specific) proporciona el Sec-tRNASec necessari al ribosoma per a la incorporació de Sec a la proteïna que s’està sintetitzant. Per últim, tRNAsec reconeix el codó UGA, s’hi uneix i transporta la Sec fins al ribosoma.
DIFICULTATS EN LA IDENTIFICACIÓ DE SELENOPROTEÏNES
La identificació d'una nova selenoproteïna és possible per l'anàlisi de proteïnes de bases de dades d'un ancestre proper. D'aquesta manera es pot establir el patró d'intercanvis Sec/Cys. Algorismes de cerca per homologia (com el programa NCBI-BLAST), s'han usat extensament per descriure la distribució de les famílies de selenoproteïnes. Tot i això, es considera que l'ús de Sec en diferents famílies de selenoproteïnes no és necessàriament homòleg, ja que es creu que l'ús de Sec ha estat un procés independent (origen polifilètic) en les diferents famílies de selenoproteïnes.
Cal destacar que un gran problema de l'estudi de les selenoproteïnes és la falta d'anotació (més que la falta de seqüenciació), de manera que les cerques per homologia resulten en proteïnes truncades, fent que els gens de les selenoproteïnes s'hagin d'anotar en seqüències genòmiques. A més, cal destacar que l'anotació de genomes, en comparació amb la generació de seqüències, és més difícil, cara i necessita més temps. I això encara es compleix més en les selenoproteïnes, ja que el paper dual del codó UGA (STOP i codificant per selenocisteïna) confon els programes de predicció de gens, donant lloc a la malaanotació dels gens de les selenoproteïnes.
És per això que s'han desenvolupat programes específics per la identificació de selenoproteïnes (Secis/Seblastian), basats principalment en la identificació de les estructures SECIS. Però com ja hem comentat anteriorment, la característica més conservada en els elements SECIS és l’estructura 3D i no tant la seqüència nucleotídica. És per això que el resultat predit pel Seblastian és una imatge que mostra l’estructura 3D del SECIS predit. Per a millorar l’eficiència de la identificació de les selenoproteïnes s’usa conjuntament la informació dels elements SECIS i de la localització dels exons, podent reduir considerablement la identificació de selenoproteïnes errònies.
A l’actualitat estan identificades 21 famílies de selenoproteïnes en eucariotes superiors: Glutathione Peroxidases (GPx), Iodothyronine Deiodinase (DI), Selenoprotein 15 (15kDa o Sel15), Fish selenoproteIn 15 (Fep15), SelM, SelH, SelI, SelJ, SelK, SelL, SelN, SelO, SelP, SelR, SelS, SelT, SelU, SelV, SelW, Thioredoxin Reductases (TR), SelenoPhosphate Synthetase (SPS). A més, diverses d’aquestes famílies inclouen diverses selenoproteïnes.
DISTRIBUCIÓ FILOGENÈTICA DE LES SELENOPROTEÏNES
Les famílies de selenoproteïnes tenen distribucions filogenètiques molt variades. Tot i haver-se identificat en els tres dominis de la vida (arquees, bacteris i eucariotes), no s’han identificat selenoproteïnes ni en llevats ni en plantes superiors, i en canvi, sí que trobem proteïnes amb l’homòleg cisteïna (per tant, les selenoproteïnes no són essencials). Hi ha algunes famílies de selenoproteïnes que només es troben en procariotes, mentre que altres només existeixen en eucariotes. Algunes famílies de selenoproteïnes tenen una distribució molt restringida i només es troben en filogènies molt concretes, i no tenen tan sols homòlegs amb Cys en altres genomes. S'ha observat que tot i que la distribució de selenoproteïnes en eucariotes és molt dispersa, encara ho és més en els procariotes.
SELENI A LA VIDA
El seleni és l'element químic de símbol Se, de número atòmic 34 i pes atòmic 78.96. Aquest element es presenta de manera natural en el medi ambient i és alliberat tant per processos naturals com per activitats humanes. És un micronutrient essencial per a la fisiologia de diverses espècies, incloent la humana. El principal resevori de seleni en els metazous són les selenoproteïnes.
SELENOPROTEÏNES EN ELS MAMÍFERS
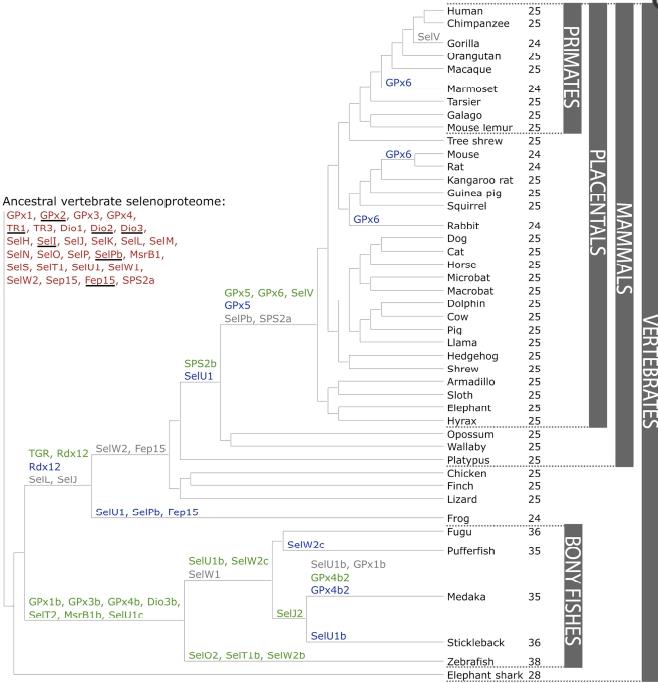
L’estudi de les selenoproteïnes i del selenoproteoma (conjunt de selenoproteïnes o bé proteïnes relacionades amb les selenoproteïnes) permet descobrir la proximitat filogenètica entre diferents organismes. Com podem veure a la imatge (Mariotti et al.), al llarg de l’evolució de les selenoproteïnes en els vertebrats hi ha hagut 20 duplicacions (representades en color verd), 9 delecions (representades en color gris) i 13 mutacions a homòlegs amb cisteïna (representades en color blau).
Eptesicus fuscus
Nom comú: Ratpenat morè, "Big brown bat".
Taxonomia
| Phylum | Chordata |
| SubPhylum | Vertebrata |
| Class | Mammalia |
| Order | Chiroptera |
| Family | Vespertilionidae |
| Genus | Eptesicus |
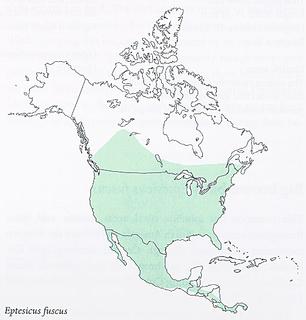
Hàbitat
Eptesicus prové del grec i significa “volador de casa”, ja que necessiten un forat petit per poder entrar a les llars on habiten en dobles parets. Per tant, els podem trobar a ciutats, pobles i àrees rurals i, menys freqüentment, a regions amb boscos molt densos. Poden viure en habitatges humans, graners o esglésies. Anteriorment es creu que habitava en buits d’arbres, coves naturals o obertures en roques.
Durant les estacions fredes, aquests ratpenats migren a coves o mines. E. fuscus normalment pengen sols durant la hibernació, tot i que es poden trobar en grups de menys de 20 individus.
És raonable especular que poblacions d'aquest ratpenat han incrementat conjuntament amb un augment de la població humana.
Distribució
Big brown bat (Eptesicus fuscus) és un dels ratpenats més comuns a Nord-Amèrica. Viu també a Alaska, Canadà central, alguns estats del EUA, Mèxic i Amèrica Central.
Descripció física
Els ratpenats són els únics mamífers amb les extremitats anteriors especialitzades pel vol. Les membranes per volar connecten el cos amb les ales, cames i cua. A més de les ales, els ratpenats també utilitzen les dues cames durant el vol. E.fuscus és un dels ratpenats més llargs, amb una envergadura de 305 mm i una longitud total d’entre 94 i 130 mm. El seu pes oscil·la entre 13 i 20 g, i rarament arriba als 30 g. Aquesta espècie presenta dimorfisme sexual en la mida, ja que les femelles són lleugerament més grans que els mascles.
El crani d’Eptesicus fuscus és relativament gran i conté 32 dents, les quals són esmolades i pesades. El nas és ample, els ulls grans i brillants i les orelles arrodonides.
La cua és menys de la meitat de la longitud del cos total i la punta es projecta lleugerament més enllà de l’uropatagi.
El color de pelatge depèn de la ubicació i les subespècies. Oscil·la entre marrons rosats i més foscos. Les parts del ratpenat nues (la cara, les orelles, les ales, la cua i la membrana) són totalment negres. Ocasionalment, s'han trobat individus amb taques blanques a les ales i en menor freqüència, albins.
Eptesicus fuscus pot viure fins 19 anys en el medi salvatge, amb una esperança de vida superior en el cas dels mascles. La majoria però, moren en el seu primer hivern.
El crani d’Eptesicus fuscus és relativament gran i conté 32 dents, les quals són esmolades i pesades. El nas és ample, els ulls grans i brillants i les orelles arrodonides.

La cua és menys de la meitat de la longitud del cos total i la punta es projecta lleugerament més enllà de l’uropatagi.
El color de pelatge depèn de la ubicació i les subespècies. Oscil·la entre marrons rosats i més foscos. Les parts del ratpenat nues (la cara, les orelles, les ales, la cua i la membrana) són totalment negres. Ocasionalment, s'han trobat individus amb taques blanques a les ales i en menor freqüència, albins.
Eptesicus fuscus pot viure fins 19 anys en el medi salvatge, amb una esperança de vida superior en el cas dels mascles. La majoria però, moren en el seu primer hivern.
Comportament i reproducció
Les femelles formen colònies de maternitat la mida de les quals pot variar de 5 a 700 individus. Els mascles de l'espècie romanen sols o en grups petits durant aquest temps. Ambdós sexes es troben de nou al final de l'estiu.
Hibernen en diverses estructures, ja sigui en entorns artificials o naturals, preferint temperatures fresques i poden tolerar condicions més extremes que altres ratpenats.
Eptesicus fuscus utilitza l'ecolocalització per evitar els obstacles i per capturar les preses volant, de manera que pot seguir els insectes entre la vegetació evitant al mateix temps els obstacles. Aquests ratpenats emeten senyals eco localitzadores a través de la boca oberta. Són capaços d'obtenir imatges acústiques mitjançant la integració d'informació del ressò en relació amb les seves emissions.
Hibernen en diverses estructures, ja sigui en entorns artificials o naturals, preferint temperatures fresques i poden tolerar condicions més extremes que altres ratpenats.
Eptesicus fuscus utilitza l'ecolocalització per evitar els obstacles i per capturar les preses volant, de manera que pot seguir els insectes entre la vegetació evitant al mateix temps els obstacles. Aquests ratpenats emeten senyals eco localitzadores a través de la boca oberta. Són capaços d'obtenir imatges acústiques mitjançant la integració d'informació del ressò en relació amb les seves emissions.
Alimentació
Eptesicus fuscus és un ratpenat insectívor, de manera que s'alimenta principalment d'escarabats fent servir el seu crani robust i les seves poderoses mandíbules per mastegar el dur exosquelet d’aquests. També menja altres insectes voladors com arnes, mosques, vespes, formigues i libèl·lules.
Abans d’entrar a la hibernació i per poder superar-la han hagut d’acumular suficients reserves (es creu suficient una quantitat aproximada d’un terç del pes abans d’hibernar).
Abans d’entrar a la hibernació i per poder superar-la han hagut d’acumular suficients reserves (es creu suficient una quantitat aproximada d’un terç del pes abans d’hibernar).
Depredació
Trien refugis aïllats per protegir-se de molts depredadors. Els joves al caure són sovint preses de serps, óssos rentadors i gats. També són capturats a vegades per mussols i falcons quan surten dels seus refugis.
Importància pels humans
Com que E. fuscus són mamífers insectívors ajuden a consumir moltes plagues d’insectes. Tot i això, hi ha preocupacions respecte als ratpenats i el virus que causa la ràbia, ja que tots els mamífers són susceptibles a aquesta malaltia. A més, transmeten la histoplasmosi.
Torna a dalt